유전자 발현
Gene expression| 시리즈의 일부 |
| 생화학 |
|---|
 |
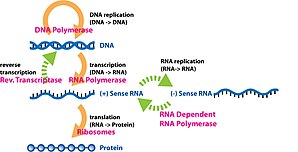
유전자 발현이란 유전자 정보가 기능성 유전자 산물의 합성에 이용되는 과정으로 단백질 또는 비코드 RNA를 최종 생산하고 궁극적으로 표현형에 영향을 준다.이 제품들은 종종 살아 남지만 전이 RNA(tRNA)과 작은 핵 RNA(소핵라이보 핵산) 같은non-protein-coding 유전자, 그 제품이 기능non-coding RNA. 유전자 발현 분자 생물학 먼저 프랜시스 크릭에 의해 1958,[1]의 더 이상 그의 1970년 article,[2]에로 인해 팽창되는 개발품의 센트럴 도그마에 집약되어 있proteins 있다. subse역전사와[3][4][5] RNA [6]복제에 대한 발견을 제안합니다.
유전자 발현 과정은 알려진 모든 생명체(유핵 생물 포함), 원핵 생물(박테리아와 고세균), 그리고 바이러스에 의해 이용됨)에 의해 평생 고분자 기계를 생성하기 위해 사용된다.
유전학에서, 유전자 발현은 유전자형이 표현형, 즉 관찰 가능한 특성을 발생시키는 가장 근본적인 단계이다.DNA에 저장된 유전자 정보는 유전자형을 나타내지만 표현형은 그 정보의 "해석"에서 비롯된다.이러한 표현형은 종종 유기체의 구조와 발달을 조절하거나 특정 대사 경로를 촉매하는 효소로 작용하는 단백질의 합성에 의해 발현된다.
유전자 발현 과정의 모든 단계는 단백질의 전사, RNA 스플라이싱, 번역 및 번역 후 변형을 포함하여 변조(조절)될 수 있다.유전자 발현 조절은 세포 내에 존재하는 특정 유전자 생성물(단백질 또는 ncRNA)의 시기, 위치 및 양을 제어하고 세포 구조와 기능에 큰 영향을 미칠 수 있다.유전자 발현 조절은 세포 분화, 발달, 형태 형성 및 모든 유기체의 다양성과 적응성의 기초이다.따라서 유전자 조절은 진화적 변화의 기질 역할을 할 수 있다.
메커니즘
문자 변환
DNA 가닥에서 RNA 복사를 생성하는 것을 전사라고 하며, RNA 중합효소에 의해 수행되며, RNA 중합효소는 뉴클레오티드 염기의 상호보완성 법칙에 따라 성장하는 RNA 가닥에 한 번에 하나의 리보뉴클레오티드를 추가합니다.이 RNA는 RNA에서 티민(T)이 우라실(U)로 대체되는 것을 제외하고 템플릿 3µ → 5µ DNA [7]가닥과 상보적이다.
원핵생물에서, 전사는 전사를 시작하기 위해 시그마 인자 단백질의 도움을 받아 프리브노 박스라고 불리는 DNA 염기서열을 결합할 필요가 있는 단일 유형의 RNA 중합효소에 의해 수행됩니다.진핵생물에서, 전사는 세 가지 유형의 RNA 중합효소에 의해 핵에서 수행되는데, 각 RNA 중합효소는 과정을 시작하기 위해 프로모터라고 불리는 특별한 DNA 배열과 일련의 DNA 결합 단백질(전사인자)을 필요로 한다.RNA 중합효소 I은 리보솜 RNA(rRNA) 유전자의 전사를 담당한다.RNA 중합효소 II(Pol II)는 모든 단백질 코드화 유전자와 일부 비코드화 RNA(예: snRNA, snoRNA 또는 긴 비코드화 RNA)를 전사한다.RNA 중합효소 III는 5S rRNA, 전달 RNA(tRNA) 유전자 및 일부 작은 비코드 RNA(예: 7SK)를 전사한다.전사는 중합효소가 터미네이터라고 불리는 염기서열을 만나면 끝납니다.
mRNA 처리
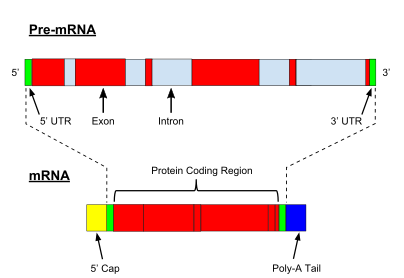
원핵생물 단백질 코드 유전자의 전사가 단백질로 번역될 준비가 된 메신저 RNA를 만드는 반면, 진핵생물 유전자의 전사는 먼저 성숙한 RNA가 되기 위해 일련의 수정을 거쳐야 하는 RNA의 1차 전사를 남긴다.성숙 과정에 관여하는 유형과 단계는 코딩 및 비코딩 사전RNA 사이에서 다르다. 즉, mRNA와 tRNA 모두에 대한 사전RNA 분자는 스플라이싱을 거치지만 관련된 단계와 기계는 다르다.[8]비부호화 RNA의 처리는 아래에 설명되어 있습니다(비부호화 RNA 성숙).
prem 처리RNA는 사전 mRNA의 5' 말단에 7-메틸구아노신(mG7)을 첨가하여 엑소뉴클라아제에 의한 분해로부터 RNA를 보호하는 효소 반응 세트인 5' 캡핑을 포함한다.그런7 다음 mG 캡은 캡 결합 복합 헤테로다이머(CBC20/CBC80)에 의해 결합되며, 이는 세포질로 mRNA를 내보내는 데 도움을 주며 RNA가 소멸되는 것을 방지합니다.
또 다른 변형은 3' 절단 및 폴리아데닐화이다.일반적으로 단백질 코드 배열과 종단자 사이에 있는 pre-mRNA에 폴리아데닐화 신호 배열(5µ-AAA-3µ)이 존재할 경우 발생한다.사전 mRNA는 먼저 분해된 후 약 200개의 아데닌(A)을 첨가하여 폴리(A) 꼬리를 형성하며, 이는 RNA를 분해로부터 보호합니다.폴리(A) 꼬리는 mRNA 수출 및 번역 재개시에 필요한 다중 폴리(A) 결합 단백질(PABP)에 의해 결합됩니다.데데닐화의 역공정에서는 CCR4-Not 3--5 ex 엑소핵산가수분해효소에 의해 폴리(A) 꼬리가 짧아지고, 이는 종종 완전한 전사 붕괴로 이어진다.
진핵생물 pre-mRNA의 매우 중요한 변형은 RNA 스플라이싱이다.대부분의 진핵생물 사전 mRNA는 엑손과 인트론이라고 불리는 번갈아 일어나는 부분으로 구성되어 있다.스플라이싱 과정에서 스플라이싱으로 알려진 RNA-단백질 촉매 복합체는 두 가지 에스테르 전이 반응을 촉매하며, 두 가지 에스테르 전이 반응은 인트론을 제거하고 라리아 구조의 형태로 방출한 다음 인접한 엑손들을 함께 스플라이싱한다.일부 인트론 또는 엑손은 성숙한 mRNA에서 제거되거나 유지될 수 있습니다.이 대체 접합이라고 불리는 것은 단일 유전자에서 유래한 일련의 다른 전사들을 만들어냅니다.이러한 전사물이 잠재적으로 다른 단백질로 번역될 수 있기 때문에, 스플라이싱은 진핵생물 유전자 발현의 복잡성과 종 프로테옴의 크기를 확장합니다.
광범위한 RNA 처리는 진핵 생물의 핵에 의해 가능해진 진화상의 이점일 수 있다.원핵생물에서는 전사와 번역이 함께 일어나는 반면, 진핵생물에서는 핵막이 두 과정을 분리하여 RNA가 처리되는 데 시간을 준다.
비부호화 RNA
대부분의 유기체에서 ncRNA(non-coding genes)는 추가 처리를 거친 전구체로서 전사된다.리보솜 RNA(rRNA)의 경우, 종종 하나 이상의 rRNA를 포함하는 사전 rRNA로 전사된다.pre-rNA는 snoRNA라고 불리는 약 150개의 서로 다른 작은 뉴클레오러스 제한 RNA 종에 의해 특정 부위에서 분해되고 수정된다(2µ-O-메틸화 및 pseudouridine 형성).SnoRNA는 단백질과 관련지어 SnoRNP를 형성한다.snoRNA 부분과 대상 RNA의 염기쌍을 형성하여 정확한 부위에 개조를 위치시키면서 단백질 부분은 촉매반응을 한다.진핵생물, 특히 RNase라고 불리는 snoRNP에서 MRP는 45S 사전 RNA를 28S, 5.8S 및 18S rRNA로 분해합니다.rRNA와 RNA 처리 인자는 [9]핵소체라고 불리는 큰 집계를 형성합니다.
transfer RNA(tRNA)의 경우, 예를 들어 RNase [10]P에 의해 5' 배열이 제거되고, tRNase Z[11] 효소에 의해 3' 말단이 제거되며, 뉴클레오티딜 전이효소에 [12]의해 비템플레이트화 3' CCA 꼬리가 첨가된다.마이크로 RNA(miRNA)의 경우 miRNA는 먼저 캡과 폴리A 꼬리를 가진 1차 전사물 또는 pri-miRNA로 전사되며, 드로샤 및 파샤 효소에 의해 세포핵에서 프리미RNA로 알려진 짧은 70-뉴클레오티드 스템루프 구조로 처리된다.수출 후 엔도핵산가수분해효소 다이커와의 상호작용에 의해 세포질 중 miRNA가 성숙하도록 처리되며, 이는 또한 아르고나이트 단백질로 이루어진 RNA 유도 사일런싱 복합체(RISC)의 형성을 개시한다.
snRNA 및 snoRNA 자체도 기능성 RNP 복합체의 일부가 되기 전에 일련의 수정을 거칩니다.이것은 핵체나 카잘체라고 불리는 특수 구획에서 이루어진다.이들의 염기는 구조적으로 snoRNA와 유사한 작은 Cajal 체특이적 RNA(scaRNA) 그룹에 의해 메틸화되거나 의사유리기화된다.
RNA수출
진핵생물에서 대부분의 성숙한 RNA는 핵에서 세포질로 내보내져야 한다.일부 RNA가 핵에서 기능하는 반면, 많은 RNA는 핵구멍을 통해 세포로 [13]운반된다.RNA의 수출은 수출로 알려진 특정 단백질과의 연관성을 필요로 한다.특정 exportin 분자는 특정 RNA 타입의 수출을 담당합니다.또한 mRNA 수송은 Exon Junction Complex(EJC)와의 올바른 연관성을 필요로 합니다.이것에 의해, 수출전에 mRNA의 올바른 처리가 완료됩니다.어떤 경우에 RNA는 시냅스와 같은 세포질의 특정 부분으로 추가로 운반된다; 그런 다음 RNA의 [14]특정 배열에 링커 단백질을 통해 결합하는 운동 단백질에 의해 견인된다.
번역.
일부 RNA(비코드 RNA)의 경우 [15]성숙한 RNA가 최종 유전자 생성물입니다.메신저 RNA(mRNA)의 경우 RNA는 하나 이상의 단백질 합성을 코드하는 정보담체이다.단일 단백질 배열(진핵생물에서 공통)을 가진 mRNA는 단일 단백질 배열(원핵생물에서 공통)을 가진 mRNA는 폴리시스트론(polycistronic)으로 알려져 있다.
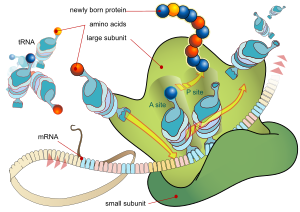
각 mRNA는 5µ 미번역 영역(5µUTR), 단백질 코드 영역 또는 개방형 판독 프레임(ORF), 3µ 미번역 영역(3µUTR)의 3가지 부분으로 구성됩니다.부호화 영역은 세쌍둥이를 형성하기 위해 유전자 코드에 의해 부호화된 단백질 합성에 대한 정보를 전달한다.코드화 영역의 뉴클레오티드의 각 트리플렛은 코돈이라고 불리며, 전달 RNA의 안티코돈 트리플렛과 상보적인 결합 부위에 대응한다.항상 동일한 안티코돈 서열을 가진 전달 RNA는 동일한 유형의 아미노산을 가지고 있다.이어서 아미노산은 부호화 영역의 세쌍의 순서에 따라 리보솜에 의해 연쇄된다.리보솜은 RNA가 메신저 RNA에 결합하는 것을 돕고 각각의 전달 RNA에서 아미노산을 추출하여 [16][17]구조 없는 단백질을 만든다.각 mRNA 분자는 많은 단백질 분자로 변환되는데,[18][19] 포유류의 경우 평균 2800개입니다.
원핵생물에서 번역은 일반적으로 전사의 지점에서 일어나며, 종종 아직 생성 과정에 있는 메신저 RNA를 사용한다.진핵생물에서 번역은 쓰여지는 단백질이 어디에 있어야 하는지에 따라 세포의 다양한 영역에서 일어날 수 있다.주요 위치는 수용성 세포질 단백질의 세포질 및 세포막으로의 수출 또는 삽입을 위한 단백질의 내소체막이다.소포체에서 발현되어야 하는 단백질은 번역 과정 도중에 인식된다.이는 리보솜에 결합하고 성장 중인(나소)[20] 아미노산 사슬에서 신호 펩타이드를 발견하면 이를 내소체로 유도하는 단백질인 신호 인식 입자에 의해 지배됩니다.
접이식
각 단백질은 일련의 mRNA에서 아미노산의 선형 사슬로 변환될 때 전개된 폴리펩타이드 또는 랜덤 코일로 존재합니다.이 폴리펩타이드에는 발달된 3차원 구조(주변 그림의 왼쪽)가 없다.그런 다음 폴리펩타이드는 랜덤 [21]코일로부터 특징적이고 기능적인 3차원 구조로 접힙니다.아미노산은 서로 상호작용하여 잘 정의된 3차원 구조, 즉 접힌 단백질(그림의 오른쪽)을 만들어 냅니다.결과적인 3차원 구조는 아미노산 배열(Anfinsen의 도그마)[22]에 의해 결정된다.
비록 기능성 단백질의 일부 부분이 [23]펼쳐져 있을 수 있지만, 정확한 3차원 구조는 기능하기 위해 필수적이다.의도된 모양으로 접히지 않으면 일반적으로 독성 프리온을 포함한 다른 특성을 가진 비활성 단백질이 생성됩니다.몇몇 신경퇴행성 질환과 다른 질병들은 잘못 접힌 [24]단백질의 축적에서 기인하는 것으로 여겨진다.면역체계가 특정 단백질 구조에 대한 항체를 생성하지 않기 때문에 많은 알레르기가 단백질의 [25]접힘에 의해 발생합니다.
샤페론이라고 불리는 효소는 새롭게 형성된 단백질이 [26]기능하는 데 필요한 3차원 구조를 얻는 것을 돕는다.마찬가지로, RNA 샤페론은 RNA가 기능적인 [27]형태를 갖추도록 도와줍니다.단백질 접힘을 돕는 것은 진핵생물에서 소포체의 주요 역할 중 하나이다.
이행 장소
진핵생물이나 원핵생물의 분비 단백질은 분비 경로로 들어가기 위해 이동되어야 한다.새롭게 합성된 단백질은 신호 펩타이드에 의해 진핵생물 Sec61 또는 원핵생물 SecYEG 전위 채널로 유도된다.진핵생물에서 단백질 분비 효율은 [28]사용된 신호 펩타이드에 따라 매우 달라진다.
단백질 수송
많은 단백질은 세포질이 아닌 세포의 다른 부분을 대상으로 하며 광범위한 신호 배열 또는 (신호 펩타이드)는 단백질을 그들이 있어야 할 곳으로 유도하기 위해 사용된다.원핵생물에서 이것은 일반적으로 세포의 제한된 구획화 때문에 단순한 과정이다.하지만, 진핵 생물에는 단백질이 올바른 소기관에 도착하도록 보장하는 매우 다양한 표적화 과정이 있습니다.
모든 단백질이 세포 내에 남아 있는 것은 아니며 소화 효소, 호르몬, 세포외 기질 단백질 등 많은 단백질이 수출된다.진핵생물에서는 수출경로가 잘 개발되어 이들 단백질의 수출의 주요 메커니즘은 소포체로의 전위, 골지기구를 통한 수송이다.[29][30]
유전자 발현 조절
유전자 발현 조절은 유전자의 기능 산물의 출현의 양과 타이밍을 조절하는 것이다.발현 조절은 세포가 필요할 때 필요한 유전자 제품을 생산하도록 하기 위해 필수적이다; 차례로, 이것은 세포들이 다양한 환경, 외부 신호, 세포의 손상, 그리고 다른 자극에 적응할 수 있는 유연성을 준다.보다 일반적으로, 유전자 조절은 세포에 모든 구조와 기능에 대한 제어를 제공하며, 세포 분화, 형태 형성, 그리고 모든 유기체의 다용성과 적응성의 기초가 된다.
조절 방법에 따라 유전자의 유형을 설명하는 데 다음과 같은 여러 용어가 사용됩니다.
- 구성 유전자는 필요할 때만 전사되는 통성 유전자와 반대로 지속적으로 전사되는 유전자이다.
- 하우스키핑 유전자는 기본적인 세포 기능을 유지하기 위해 필요한 유전자이며, 그래서 전형적으로 유기체의 모든 세포 유형에서 발현된다.예를 들어 액틴, GAPDH 및 유비퀴틴이 있습니다.일부 하우스키핑 유전자는 비교적 일정한 속도로 전사되며 이 유전자들은 다른 유전자의 발현 속도를 측정하기 위한 실험의 기준점으로 사용될 수 있다.
- 통성 유전자는 구성 유전자와는 대조적으로 필요할 때만 전사되는 유전자이다.
- 유도유전자는 환경변화에 반응하거나 세포주기의 위치에 의존하는 발현을 가진 유전자이다.
유전자 발현 단계는 DNA-RNA 전사 단계부터 단백질의 번역 후 수식 단계까지 변조할 수 있다.RNA든 단백질이든 최종 유전자 생성물의 안정성 또한 유전자의 발현 수준에 기여합니다. 불안정한 생성물은 낮은 발현 수준을 초래합니다.일반적으로 유전자 발현은 DNA의 전사[33] 및 [34]RNA의 번역에 집합적으로 영향을 미치는 분자 간의[32] 상호작용 수와 유형의 변화를 통해[31] 조절된다.
유전자 발현이 중요한 몇 가지 간단한 예는 다음과 같습니다.
- 인슐린 발현을 제어하여 혈당 조절 신호를 제공합니다.
- 암컷 포유동물에서 X염색체 불활성화는 그것이 함유된 유전자의 "과다량"을 방지하기 위한 것이다.
- 사이클린 발현 수준은 진핵 세포 사이클을 통한 진행을 조절합니다.
전사 규제
전사의 조절은 세 가지 주요 영향 경로로 나눌 수 있다: 유전자(유전자와의 제어 인자의 직접적인 상호작용), 전사 기계와의 제어 인자의 변조 상호작용 및 후생 유전자(전사에 영향을 미치는 DNA 구조의 비순서 변화).
DNA와의 직접적인 상호작용은 단백질이 전사 수준을 변화시키는 가장 간단하고 직접적인 방법이다.유전자는 종종 전사를 조절하는 특정한 기능을 가진 코딩 영역 주위에 여러 개의 단백질 결합 부위가 있습니다.조절 DNA 결합 부위에는 강화제, 절연체 및 소음제로 알려진 많은 종류가 있습니다.전사를 조절하는 메커니즘은 RNA 중합효소에 대한 DNA의 주요 결합 부위를 차단하는 것에서부터 활성제 역할을 하고 RNA 중합효소 결합을 보조함으로써 전사를 촉진하는 것까지 다양하다.
전사 인자의 활성은 인산화, 아세틸화 또는 글리코실화를 포함한 번역 후 단백질 변형을 일으키는 세포 내 신호에 의해 더욱 조절된다.이러한 변화는 전사인자가 직간접적으로 프로모터 DNA에 결합하거나 RNA 중합효소를 모집하거나 새로 합성된 RNA 분자의 신장을 선호하는 능력에 영향을 미친다.
진핵 생물의 핵막은 핵에서 존재하는 기간에 의해 전사 인자의 추가적인 조절을 가능하게 하며, 이는 구조의 가역적 변화와 다른 [35]단백질의 결합에 의해 조절된다.환경 자극 또는 내분비 신호는[36] 조절 단백질의[37] 변형을 일으킬 수 있으며, [38]이는 유전자 발현을 조절하는 결과를 초래한다.
최근에는 전사에 대한 비DNA-시퀀스 특이적 효과가 유의미한 영향을 미친다는 것이 명백해졌다.이러한 효과는 후생유전학이라고 하며 DNA의 고차 구조, 비배열 특이 DNA 결합 단백질 및 DNA의 화학적 변형을 포함한다.일반적으로 후생유전학적 효과는 단백질에 대한 DNA의 접근성을 변화시켜 전사를 조절한다.
진핵생물에서 히스톤 코드에 의해 제어되는 염색질의 구조는 에우크로마틴과 헤테로크로마틴 영역의 유전자 발현에 큰 영향을 미치며 DNA에 대한 접근을 조절한다.
포유동물 전사의 인핸서, 전사 인자, 매개체 복합체 및 DNA 루프

포유동물에서의 유전자 발현은 DNA 상류의 (감각 가닥의 5' 영역을 향해) 유전자의 전사 시작 지점 근처에 위치한 핵심 촉진제 및 프로모터-근접 요소를 포함한 많은 시스 조절 요소에 의해 조절된다.다른 중요한 시스 조절 모듈은 전사 시작 부위에서 멀리 떨어진 DNA 영역에 국소화된다.여기에는 강화제, 소음기, 절연체 및 테더링 [39]요소가 포함됩니다.인핸서 및 그와 관련된 전사 인자는 유전자 발현 [40]조절에 주도적인 역할을 한다.
증강제는 유전자를 조절하는 게놈 영역이다.인핸서는 세포에 특정한 유전자 발현 프로그램을 제어하는데, 대부분의 경우 그들의 [41]목표 유전자의 촉진자와 물리적으로 근접하기 위해 먼 거리를 순환합니다.각각 종종 그들의 표적 유전자와 멀리 떨어져 있는 수 만 또는 수 십만 개의 뉴클레오티드가 그들의 표적 유전자 프로모터에 루프하고 유전자 [41]발현을 조절하기 위해 서로 협력합니다.
이 그림은 표적 유전자의 프로모터와 근접하기 위해 루프를 돌고 있는 인핸서를 보여준다.루프는 커넥터 단백질의 이합체(예: CTCF 또는 YY1)에 의해 안정화됩니다.조광체의 한쪽 부재는 인핸서상의 결속 모티브에 고정하고, 다른 한쪽 부재는 프로모터상의 결속 모티브에 고정한다(그림 [42]중 빨간색 지그재그로 표시).여러 세포 기능 특이적 전사 인자(인간 세포의 [43]약 1,600개의 전사 인자 중)는 일반적으로 인핸서의 [44]특정 모티브와 결합한다.이들 인핸서 결합 전사인자의 소량 조합은 DNA 루프를 통해 프로모터에 근접했을 때 표적 유전자의 전사 수준을 지배한다.중개자(통상 상호작용 구조에서 약 26개의 단백질로 이루어진 복합체)는 촉진제 DNA 결합 전사인자에서 프로모터에 결합하는 [45]RNA 중합효소 II(pol II) 효소로 직접 조절 신호를 전달한다.
활성 상태일 때 RNA 중합효소가 [46]두 가지 다른 방향으로 작용하여 그림에 표시된 것과 같이 두 개의 eRNA를 생성하면서 일반적으로 양쪽 DNA 가닥에서 전사됩니다.불활성전사인자는 불활성전사인자에 의해 결합되어 있어도 된다.전사 인자의 인산화는 그것을 활성화 시킬 수 있고, 그 활성화된 전사 인자는 그것이 결합되어 있는 인핸서를 활성화시킬 수 있다([47]그림에서 전사 인자의 인산화를 나타내는 작은 붉은 별 참조).활성 증강제는 표적 유전자에서 [48]메신저 RNA의 전사를 활성화하기 전에 RNA의 전사를 시작합니다.
전사조절에서의 DNA메틸화 및 탈메틸화
DNA 메틸화는 유전자 발현에 후생유전적 영향을 미치는 광범위한 메커니즘으로 박테리아와 진핵생물에서 볼 수 있으며 유전자의 전사 소음과 전사 조절에 역할을 한다.메틸화는 시토신에서 가장 자주 발생합니다(그림 참조).시토신의 메틸화는 주로 시토신이 CpG 부위인 구아닌에 이어지는 디뉴클레오티드 배열에서 발생한다.인간 게놈의 CpG 사이트 수는 약 2800만 [49]개입니다.세포 유형에 따라 CpG 부위의 약 70%가 메틸화 시토신을 [50]가지고 있다.
DNA에서 시토신의 메틸화는 유전자 발현을 조절하는 데 중요한 역할을 한다.유전자의 프로모터 영역에서의 CpGs의 메틸화는 보통 유전자[51] 전사를 억제하는 반면 유전자의 체내에서의 CpGs의 메틸화는 [52]발현을 증가시킨다.TET 효소는 메틸화된 세포신의 탈메틸화에 중심적인 역할을 한다.TET 효소 활성에 의한 유전자 프로모터의 CpGs의 탈메틸화는 유전자의 [53]전사를 증가시킨다.
학습 및 기억의 전사 규제
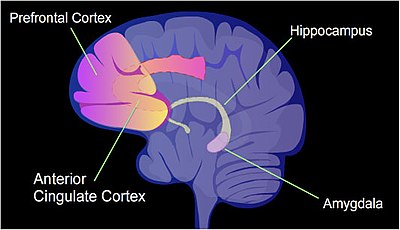
쥐에게 상황별 공포 조절(CFC)은 고통스러운 학습 경험이다.단 한 번의 프레온 중독으로 평생의 두려운 [54]기억이 생길 수 있다.CFC 에피소드 후, 쥐의 해마 뉴런 DNA [55]내 모든 유전자의 약 9.17%의 프로모터 영역에서 시토신 메틸화가 변화한다.해마는 새로운 기억이 처음 저장되는 곳이다.CFC 이후 약 500개의 유전자가 전사를 증가시키고(종종 프로모터 영역의 CpG 부위의 탈메틸화로 인해), 약 1,000개의 유전자가 전사를 감소시킨다(종종 프로모터 영역의 CpG 부위에서 새롭게 형성된 5-메틸시토신 때문에).뉴런 내의 유도 및 억제된 유전자의 패턴은 쥐 [55]뇌의 해마에서 이 훈련 사건의 첫 번째 일시적인 기억을 형성하기 위한 분자 기반을 제공하는 것으로 보입니다.
기억력 확립 중 해마의 새로운 DNA 메틸화 및 새로운 DNA 탈메틸화를 유도하는 몇 가지 특정 메커니즘이 최근에 확립되었습니다(요약 참조).하나의 메커니즘은 TET1 DNA 탈메틸화효소인 TET1s의 짧은 아이소폼을 게놈 상의 약 600개 위치로 안내하는 것을 포함한다.이 가이던스는 메모리 형성에 중요한 전사 인자인 EGR1 단백질과 TET1의 연관성에 의해 수행됩니다.이러한 장소에 TET1을 가져오는 것은 관련 유전자를 상향 조절하면서 그러한 부위에서 DNA 탈메틸화를 시작합니다.두 번째 메커니즘은 DNA의 세포신에 메틸기를 첨가하는 DNA메틸전달효소 DNMT3A의 스플라이스 이소형인 DNMT3A2를 포함한다.이 아이소폼은 시냅스 활성에 의해 유도되며, 그 작용 위치는 번역 후 히스톤 수정(히스톤 코드)에 의해 결정되는 것으로 보인다.그 결과 생성된 새로운 메신저 RNA는 메신저 RNP 입자에 의해 뉴런의 시냅스로 운반되고, 시냅스의 [56]활동에 영향을 미치는 단백질로 번역될 수 있습니다.
특히, 뇌에서 파생된 신경영양인자 유전자는 "학습 유전자"[57]로 알려져 있다.CFC 이후 유전자의 특정 내부 프로모터의 CpG 메틸화 감소와 관련된 BDNF 유전자 발현 상향조절이 있었으며,[57] 이는 학습과 관련이 있었다.
암의 전사 조절
대부분의 유전자 촉진제는 수많은 CpG [58]부위가 있는 CpG 섬을 포함하고 있다.유전자의 프로모터 CpG 부위의 많은 부분이 메틸화되면 유전자는 [59]침묵하게 된다.대장암은 일반적으로 3~6명의 운전자 돌연변이와 33~66명의 히치하이커 또는 승객 [60]돌연변이가 있다.그러나 전사 소음은 암으로의 진행을 유발하는데 돌연변이보다 더 중요할 수 있다.예를 들어 대장암의 경우 약 600~800개의 유전자가 CpG 섬 메틸화에 의해 전사적으로 침묵된다(암에서의 전사 조절 참조).암에서의 전사 억제는 마이크로RNA의 [61]발현 변화와 같은 다른 후생유전 메커니즘에 의해서도 발생할 수 있다.유방암에서 BRCA1의 전사 억제는 BRCA1 프로모터의 과메틸화보다 과잉 발현된 마이크로RNA-182에 의해 더 자주 발생할 수 있다(유방암과 난소암에서 BRCA1의 저발현 참조).
전사 후 규정
번역이 가능하기 전에 RNA의 수출이 필요한 진핵생물에서, 핵 수출은 유전자 발현에 대한 추가적인 제어를 제공하는 것으로 생각된다.핵을 드나드는 모든 수송은 핵의 모공을 통해 이루어지며, 수송은 광범위한 임포틴 및 수출 단백질에 의해 제어된다.
단백질을 코드하는 유전자의 발현은 코드를 운반하는 메신저 RNA가 번역될 수 있을 정도로 오래 생존해야 가능하다.전형적인 세포에서 RNA 분자는 분해로부터 특별히 보호되어야만 안정적이다.RNA 분해는 mRNA가 번역되기 전에 상당한 거리를 이동해야 하는 진핵 세포에서 발현 조절에 특히 중요하다.진핵생물에서 RNA는 특정 전사 후 변형, 특히 5' 캡과 폴리 아데닐화 꼬리에 의해 안정화된다.
mRNA의 의도적인 분해는 외래 RNA(일반적으로 바이러스로부터)의 방어 메커니즘뿐만 아니라 mRNA 불안정화의 경로로도 사용된다.mRNA 분자가 작은 간섭 RNA에 대한 상보적 배열을 가지고 있다면, 그것은 RNA 간섭 경로를 통해 파괴를 목표로 한다.
3개의 주요 미번역 영역과 마이크로RNA
메신저 RNA(mRNA)의 3가지 주요 미번역 영역(3'UTR)은 종종 전사 후 유전자 발현에 영향을 미치는 조절 서열을 포함한다.이러한 3'-UTR은 종종 마이크로RNA(miRNA)와 조절 단백질의 결합 부위를 모두 포함한다.miRNA는 3μ-UTR 내의 특정 부위에 결합함으로써 번역이 저해되거나 전사물의 분해를 직접 유발함으로써 다양한 mRNA의 유전자 발현을 감소시킬 수 있다.또한 3'-UTR은 mRNA의 발현을 억제하는 억제 단백질과 결합하는 소음기 영역을 가질 수 있다.
3'-UTR에는 많은 경우 microRNA response element(MRE; 마이크로RNA 응답 요소)가 포함되어 있습니다.MRE는 miRNA가 바인드되는 시퀀스입니다.이것들은 3'-UTR 내에서 널리 사용되고 있는 모티브입니다.3µ-UTR(예를 들어 소음기 영역 포함) 내의 모든 규제 모티브 중 MRE는 모티브의 약 절반을 차지한다.
2014년 현재, miRNA 염기서열 및 주석의 아카이브인 miRBase [62]웹사이트에는 233종의 생물종이 28,645개의 항목을 열거했다.이 중 1,881개의 miRNA가 인간의 miRNA 위치에 있었으며, miRNA는 평균 약 400개의 표적 mRNA(수백개의 [63]유전자에 영향을 미치는 발현)를 가질 것으로 예측되었다.Friedman [63]등은 인간의 mRNA 3µUTR 내에서 45,000개 이상의 miRNA 표적 부위가 배경 수준 이상으로 보존되며, 인간 단백질 코드 유전자의 60% 이상이 miRNA와의 쌍을 유지하기 위해 선택적 압력을 받고 있다고 추정했다.
직접 실험에 따르면 단일 miRNA가 수백 [64]개의 고유한 mRNA의 안정성을 감소시킬 수 있습니다.다른 실험들은 단일 miRNA가 수백 개의 단백질 생성을 억제할 수 있지만, 이러한 억제 효과는 종종 비교적 경미하다는 것을 보여준다.[65][66]
miRNA 유전자 발현 조절 장애의 영향은 [67]암에서 중요한 것으로 보인다.예를 들어, 위장암에서 9개의 miRNA는 후생적으로 변화하고 DNA 복구 [68]효소의 하향 조절에 효과적인 것으로 확인되었다.
miRNA 유전자 발현 조절 장애의 영향은 정신 분열증, 조울증, 주요 우울증, 파킨슨병, 알츠하이머병, 자폐 스펙트럼 [69][70]장애와 같은 신경 정신 질환에서도 중요한 것으로 보인다.
번역 규제
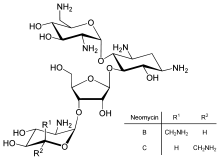
번역의 직접 조절은 전사 또는 mRNA 안정성의 조절보다 덜 보편적이지만 때때로 사용됩니다.단백질 번역의 억제는 독소와 항생제의 주요 표적이므로, 그들은 정상적인 유전자 발현 제어를 무시함으로써 세포를 죽일 수 있다.단백질 합성 억제제는 항생제 네오마이신 및 독소 리신을 포함한다.
번역 후 수정
번역 후 수정(PTM)은 단백질에 대한 공유 결합 수정이다.RNA 스플라이싱과 마찬가지로 프로테옴을 상당히 다양화하는 데 도움이 됩니다.이러한 변형은 보통 효소에 의해 촉매된다.또한 아미노산 측쇄 잔기에 대한 공유 첨가와 같은 과정은 종종 다른 효소에 의해 역전될 수 있다.그러나 단백질 골격의 단백질 분해 분열과 같은 일부는 되돌릴 [71]수 없다.
PTM은 [72]세포 내에서 많은 중요한 역할을 합니다.예를 들어, 인산화 작용은 주로 단백질의 활성화 및 비활성화 및 신호 [73]전달 경로에 관여한다.PTM은 전사 조절에 관여합니다: 아세틸화와 메틸화의 중요한 기능은 히스톤 꼬리 변형입니다. 이것은 [71]전사를 위해 DNA에 접근하는 방법을 변화시킵니다.그들은 또한 글리코실화가 중요한 역할을 [74]하는 면역 체계에서도 볼 수 있다.유비퀴티네이션이 단백질 [71]분해를 통해 단백질을 분해하는 방법에서 알 수 있듯이, 한 유형의 PTM은 다른 유형의 PTM을 시작할 수 있습니다.단백질 분해는 단백질 분해에 관여하는 것 외에 단백질의 활성화와 비활성화, 그리고 DNA 전사 및 세포 [75]사멸과 같은 생물학적 과정을 조절하는 데에도 중요하다.
측정.
특정 유전자가 세포, 조직 또는 유기체 내에서 발현되는 수준을 정량화하는 능력은 많은 귀중한 정보를 제공할 수 있기 때문에 유전자 발현을 측정하는 것은 많은 생명과학의 중요한 부분이다.예를 들어, 유전자 발현을 측정하면 다음과 같이 할 수 있습니다.
마찬가지로 단백질 발현 위치의 분석은 강력한 도구이며, 이것은 생물적 또는 세포적 규모로 이루어질 수 있다.국소화 연구는 다세포 유기체에서의 발달 연구와 단세포에서의 단백질 기능의 지표로서 특히 중요하다.이상적으로는 발현 측정은 최종 유전자 생성물(많은 유전자의 경우 이것이 단백질)을 검출함으로써 이루어지지만, 종종 전구체 중 하나(일반적으로 mRNA)를 검출하고 이러한 측정에서 유전자 발현 수준을 추론하는 것이 더 쉽다.
mRNA 정량
mRNA의 수준은 mRNA 분자에 대한 크기와 배열 정보를 제공하는 노던 블로팅을 통해 정량적으로 측정할 수 있다.RNA 시료를 아가로스겔상에서 분리하여 목표 배열을 상보하는 방사성 라벨이 부착된 RNA 프로브에 하이브리드화한다.그런 다음 방사선 라벨이 부착된 RNA가 자동 방사선 촬영기에 의해 검출됩니다.방사성 시약의 사용은 시술에 시간이 많이 걸리고 잠재적으로 위험하기 때문에 디옥시게닌과 비오틴 화학과 같은 대체 라벨링 및 검출 방법이 개발되었습니다.Northern Blotting의 인식된 단점은 대량의 RNA가 필요하며 겔 이미지에서 밴드 강도를 측정해야 하므로 정량화가 완전히 정확하지 않을 수 있다는 것입니다.한편, 노던 블롯의 추가 mRNA 크기 정보에 의해 교대로 스플라이스된 전사물을 구별할 수 있다.
mRNA 풍요도를 측정하는 또 다른 접근방식은 RT-qPCR이다.이 기술에서는 역전사 후에 정량적 PCR이 뒤따른다.역전사를 통해 먼저 mRNA에서 DNA 템플릿이 생성됩니다. 이 단일 가닥 템플릿은 cDNA라고 합니다.cDNA 템플릿은 정량적 단계에서 증폭되며, 그 동안 라벨 부착 하이브리드화 프로브 또는 중간 염료에 의해 방출되는 형광은 DNA 증폭 과정이 진행됨에 따라 변화한다.신중하게 구성된 표준 곡선을 사용하여 qPCR은 일반적으로 균질화된 조직의 나노리터당 복사본 단위 또는 세포당 복사본 단위로 원래 mRNA의 복사본 수를 절대 측정할 수 있습니다. qPCR은 매우 민감합니다(이론적으로 단일 mRNA 분자의 탐지가 가능하지만, 재유형에 따라 비용이 많이 들 수 있습니다).사용된 포터; 형광 라벨이 부착된 올리고뉴클레오티드 프로브는 비특이적 중간 형광 염료보다 더 비싸다.
발현 프로파일링 또는 샘플 내의 많은 유전자의 높은 throughput 분석을 위해 저밀도 배열의 경우 수백 개의 유전자에 대해 정량 PCR을 동시에 실행할 수 있다.두 번째 접근 방식은 하이브리드화 마이크로 어레이입니다.단일 어레이 또는 "칩"은 하나 이상의 유기체의 게놈에 있는 모든 알려진 유전자에 대한 전사 수준을 결정하기 위한 프로브를 포함할 수 있다.또는 서로 다른 mRNA의 세포 농도를 상대적으로 측정할 수 있는 유전자 발현 직렬 분석(SAGE) 및 RNA-Seq와 같은 "태그 기반" 기술을 사용할 수 있다.태그 기반 방식의 장점은 "오픈 아키텍처"로, 알려진 시퀀스 또는 알려지지 않은 시퀀스로 모든 스크립트를 정확하게 측정할 수 있습니다.RNA-Seq와 같은 차세대 염기서열결정(NGS)은 참조 게놈과 일치할 수 있는 방대한 양의 염기서열 데이터를 생성하는 또 다른 접근법이다.NGS는 비교적 시간이 많이 걸리고, 비용이 많이 들고, 자원 집약적이지만, 단핵 다형성, 스플라이스 변이체 및 새로운 유전자를 식별할 수 있으며, 배열 정보가 거의 또는 전혀 없는 유기체의 발현 프로파일링에도 사용될 수 있다.
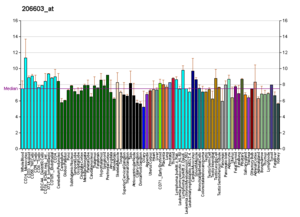
위키피디아 RNA 프로파일
이런 프로파일은 위키피디아에 등재된 거의 모든 단백질에서 발견됩니다.그것들은 노바티스 연구 재단의 유전체학 연구소나 유럽 생물 정보학 연구소 같은 단체들에 의해 생성되었다.추가 정보는 해당 데이터베이스를 검색하여 찾을 수 있습니다(이 그림에 표시된 GLUT4 전송기의 예에 대해서는 [76]인용 참조).이러한 프로파일은 특정 조직에서 특정 단백질의 DNA 발현 수준(그리고 생성된 RNA)을 나타내며, 각 위키피디아 페이지의 오른쪽에 있는 단백질 상자에 있는 이미지에 따라 색상으로 구분됩니다.
단백질 정량
단백질을 코드하는 유전자의 경우, 발현 수준은 mRNA 정량화 기술에 대한 명확한 유사성을 가진 많은 방법으로 직접 평가할 수 있다.
가장 일반적으로 사용되는 방법 중 하나는 관심 [77]있는 단백질에 대해 웨스턴 블롯을 수행하는 것입니다.이것은 단백질의 크기와 더불어 단백질의 크기에 대한 정보를 제공한다.시료(종 세포 용해액)는 폴리아크릴아미드겔 위에서 분리되어 막에 전달된 후 해당 단백질에 대한 항체로 프로빙된다.항체는 불소포자 또는 영상 및/또는 정량화를 위해 고추냉이 과산화효소에 결합될 수 있다.이 분석의 겔 기반 특성은 정량화를 덜 정확하게 만들지만, 단백질 분해나 유비퀴티네이션과 같은 단백질에 대한 나중에 크기 변화를 확인할 수 있다는 장점이 있습니다.
mRNA단백질상관
전사가 유전자 발현을 직접적으로 반영하는 반면, mRNA 분자의 복사 수는 mRNA에서 번역된 단백질 분자의 수와 직접적으로 상관하지 않습니다. 단백질과 mRNA의 정량화는 두 수준의 상관 관계를 허용합니다.유전자[19] 발현 단계별 조절은 번역 조절 또는 단백질 [78]안정성에 나타나듯이 상관관계에 영향을 미칠 수 있다.극지방 [79]세포에서 단백질 운반과 같은 번역 후 인자는 측정된 mRNA-단백질 상관 관계에도 영향을 미칠 수 있습니다.
현지화
mRNA는 적절히 라벨이 부착된 상보적인 mRNA 가닥으로 검출할 수 있으며 단백질은 라벨이 부착된 항체를 통해 검출할 수 있다.그런 다음 mRNA 또는 단백질이 어디에 있는지 식별하기 위해 프로브된 샘플을 현미경으로 관찰합니다.
이 유전자를 녹색 형광 단백질(또는 유사한) 마커에 융합된 새로운 버전으로 치환함으로써 살아있는 세포에서 발현을 직접 정량화할 수 있다.이것은 형광 현미경을 이용한 영상촬영으로 이루어진다.유전자 발현 수준에 영향을 주지 않고 GFP 융합 단백질을 게놈의 고유 위치에 복제하는 것은 매우 어렵기 때문에 이 방법은 종종 내인성 유전자 발현을 측정하는데 사용될 수 없다.단, 예를 들어 발현 벡터를 통해 세포에 인공적으로 도입된 유전자의 발현을 측정하는데 널리 사용된다.형광 리포터에 표적 단백질을 융합시킴으로써 세포의 국재화와 발현 수준을 포함한 단백질의 거동이 크게 변할 수 있다는 점에 유의해야 한다.
효소결합면역흡수제 어세이에서는 마이크로미터 플레이트에 고정된 항체를 사용하여 우물에 첨가된 샘플에서 관심 단백질을 포착합니다.효소 또는 불소체와 결합된 검출 항체를 이용하여 형광계 또는 측색계 검출에 의해 결합 단백질의 양을 정확하게 측정할 수 있다.검출 과정은 웨스턴 블롯과 매우 유사하지만 겔 단계를 피함으로써 보다 정확한 정량화를 달성할 수 있습니다.
표현 체계
발현 시스템은 선택된 유전자 산물의 생산을 위해 특별히 설계된 시스템이다.이것은 보통 단백질이지만 tRNA나 리보자임과 같은 RNA일 수도 있다.발현 시스템은 보통 DNA에 의해 부호화된 유전자와 해당 DNA를 mRNA로 전사하고 제공된 시약을 사용하여 mRNA를 단백질로 변환하는 데 필요한 분자 기계로 구성된다.가장 넓은 의미에서 이것은 모든 살아있는 세포를 포함하지만, 이 용어는 실험 도구로서의 발현을 언급하기 위해 더 일반적으로 사용됩니다.그러므로 표현 체계는 종종 어떤 식으로든 인위적이다.그러나 표현 체계는 기본적으로 자연스러운 과정입니다.바이러스는 숙주 세포를 바이러스 단백질과 게놈의 발현 체계로 사용함으로써 복제되는 훌륭한 예이다.
유도식
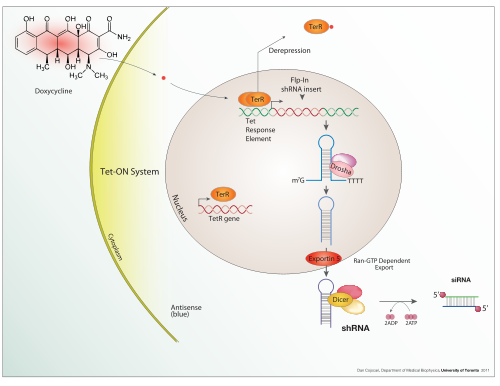
독시사이클린은 또한 유기체 및 세포 배양에서 트랜스젠 발현을 조절하기 위해 "테트온" 및 "테트오프" 테트라사이클린 제어 전사 활성화에 사용된다.
자연에서
이러한 생물학적 도구 외에도, 자연적으로 관찰된 DNA의 특정 구성(유전자, 촉진제, 증강제, 억제제) 및 관련 기계 자체를 발현 시스템이라고 한다.이 용어는 일반적으로 잘 정의된 조건, 예를 들어 람다 파지의 단순 억제기 스위치 발현 시스템 및 박테리아의 락 연산자 시스템 하에서 유전자 또는 유전자 세트가 켜지는 경우에 사용됩니다.Tet-on 및 Tet-off 표현 시스템과 같은 인공 표현 시스템에 몇 가지 자연 표현 시스템이 직접 사용되거나 수정되어 사용됩니다.
유전자 네트워크
유전자는 때때로 네트워크의 노드로 간주되어 왔으며 입력은 전사 인자와 같은 단백질이며 출력은 유전자 발현 수준입니다.노드 자체가 기능을 수행하며, 이러한 기능의 작동은 세포 내에서 정보처리를 수행하고 세포 동작을 결정하는 것으로 해석되어 왔다.
유전자 네트워크는 명시적 인과 모델을 형성하지 않고도 구성될 수 있다.이는 대규모 식 데이터 [80]세트에서 네트워크를 조립할 때 자주 발생합니다.발현에 대한 공변화와 상관관계는 사례와 측정(대개 전사체 또는 프로테옴 데이터)의 대규모 샘플에 걸쳐 계산됩니다.변동의 원인은 실험적이거나 자연(관측적)일 수 있습니다.유전자 발현 네트워크를 구성하는 방법에는 여러 가지가 있지만, 한 가지 일반적인 접근법은 조건, 시점 또는 개인에 걸친 발현 쌍별 상관의 모든 행렬을 계산하고 (일부 컷오프 값으로 임계값화한 후) 그 행렬을 노드가 유전자, 전사물 또는 프로를 나타내는 그래픽 표현으로 변환하는 것이다.이러한 노드를 연결하는 주석과 가장자리는 연관성의 강도를 나타냅니다(GeneNetwork GeneNetwork [81]2 참조).
기술 및 도구
다음의 실험 기술은 유전자 발현을 측정하기 위해 사용되며, 더 오래되고 확립된 기술부터 시작하여 대략 연대순으로 나열되어 있습니다.다중성의 정도에 따라 두 그룹으로 나뉩니다.
- 로우플렉스부터 미드플렉스까지의 기술:
- 고플렉스 기술:
유전자 발현 데이터베이스
- NCBI[87] 유전자 발현 옴니버스(GEO)
- EBI에서의 Expression Atlas
- 잭슨 연구소의 마우스 유전자 발현 데이터베이스
- CollecTF:[88] 박테리아에서 실험적으로 검증된 전사 인자 결합 부위의 데이터베이스.
- COLLOBOS:[89] 박테리아 발현 개요 수집.
- 많은 Microbe Microarrays 데이터베이스: 미생물 Affymetrix 데이터[90]
「 」를 참조해 주세요.
레퍼런스
- ^ Crick FH (1958). "On protein synthesis". Symposia of the Society for Experimental Biology. 12: 138–63. PMID 13580867.
- ^ Crick F (August 1970). "Central dogma of molecular biology". Nature. 227 (5258): 561–3. Bibcode:1970Natur.227..561C. doi:10.1038/227561a0. PMID 4913914. S2CID 4164029.
- ^ "Central dogma reversed". Nature. 226 (5252): 1198–9. June 1970. Bibcode:1970Natur.226.1198.. doi:10.1038/2261198a0. PMID 5422595. S2CID 4184060.
- ^ Temin HM, Mizutani S (June 1970). "RNA-dependent DNA polymerase in virions of Rous sarcoma virus". Nature. 226 (5252): 1211–3. doi:10.1038/2261211a0. PMID 4316301. S2CID 4187764.
- ^ Baltimore D (June 1970). "RNA-dependent DNA polymerase in virions of RNA tumour viruses". Nature. 226 (5252): 1209–11. doi:10.1038/2261209a0. PMID 4316300. S2CID 4222378.
- ^ Iyer LM, Koonin EV, Aravind L (January 2003). "Evolutionary connection between the catalytic subunits of DNA-dependent RNA polymerases and eukaryotic RNA-dependent RNA polymerases and the origin of RNA polymerases". BMC Structural Biology. 3: 1. doi:10.1186/1472-6807-3-1. PMC 151600. PMID 12553882.
- ^ Brueckner F, Armache KJ, Cheung A, Damsma GE, Kettenberger H, Lehmann E, Sydow J, Cramer P (February 2009). "Structure-function studies of the RNA polymerase II elongation complex". Acta Crystallographica D. 65 (Pt 2): 112–20. doi:10.1107/S0907444908039875. PMC 2631633. PMID 19171965.
- ^ Krebs, Jocelyn E. (2017-03-02). Lewin's genes XII. Goldstein, Elliott S.,, Kilpatrick, Stephen T. Burlington, MA. ISBN 978-1-284-10449-3. OCLC 965781334.
- ^ Sirri V, Urcuqui-Inchima S, Roussel P, Hernandez-Verdun D (January 2008). "Nucleolus: the fascinating nuclear body". Histochemistry and Cell Biology. 129 (1): 13–31. doi:10.1007/s00418-007-0359-6. PMC 2137947. PMID 18046571.
- ^ Frank DN, Pace NR (1998). "Ribonuclease P: unity and diversity in a tRNA processing ribozyme". Annual Review of Biochemistry. 67: 153–80. doi:10.1146/annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). "tRNase Z". Protein and Peptide Letters. 14 (2): 137–45. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Weiner AM (October 2004). "tRNA maturation: RNA polymerization without a nucleic acid template". Current Biology. 14 (20): R883–5. doi:10.1016/j.cub.2004.09.069. PMID 15498478.
- ^ Köhler A, Hurt E (October 2007). "Exporting RNA from the nucleus to the cytoplasm". Nature Reviews. Molecular Cell Biology. 8 (10): 761–73. doi:10.1038/nrm2255. PMID 17786152. S2CID 10836137.
- ^ Jambhekar A, Derisi JL (May 2007). "Cis-acting determinants of asymmetric, cytoplasmic RNA transport". RNA. 13 (5): 625–42. doi:10.1261/rna.262607. PMC 1852811. PMID 17449729.
- ^ Amaral PP, Dinger ME, Mercer TR, Mattick JS (March 2008). "The eukaryotic genome as an RNA machine". Science. 319 (5871): 1787–9. Bibcode:2008Sci...319.1787A. doi:10.1126/science.1155472. PMID 18369136. S2CID 206511756.
- ^ Hansen TM, Baranov PV, Ivanov IP, Gesteland RF, Atkins JF (May 2003). "Maintenance of the correct open reading frame by the ribosome". EMBO Reports. 4 (5): 499–504. doi:10.1038/sj.embor.embor825. PMC 1319180. PMID 12717454.
- ^ Berk V, Cate JH (June 2007). "Insights into protein biosynthesis from structures of bacterial ribosomes". Current Opinion in Structural Biology. 17 (3): 302–9. doi:10.1016/j.sbi.2007.05.009. PMID 17574829.
- ^ Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, Chen W, Selbach M (May 2011). "Global quantification of mammalian gene expression control" (PDF). Nature. 473 (7347): 337–42. Bibcode:2011Natur.473..337S. doi:10.1038/nature10098. PMID 21593866. S2CID 205224972.
- ^ a b Schwanhäusser B, Busse D, Li N, Dittmar G, Schuchhardt J, Wolf J, Chen W, Selbach M (March 2013). "Corrigendum: Global quantification of mammalian gene expression control". Nature. 495 (7439): 126–7. Bibcode:2013Natur.495..126S. doi:10.1038/nature11848. PMID 23407496.
- ^ Hegde RS, Kang SW (July 2008). "The concept of translocational regulation". The Journal of Cell Biology. 182 (2): 225–32. doi:10.1083/jcb.200804157. PMC 2483521. PMID 18644895.
- ^ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). "The Shape and Structure of Proteins". Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 978-0-8153-3218-3.
- ^ Anfinsen CB (July 1972). "The formation and stabilization of protein structure". The Biochemical Journal. 128 (4): 737–49. doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ^ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer; Web content by Neil D. Clarke (2002). "3. Protein Structure and Function". Biochemistry. San Francisco: W. H. Freeman. ISBN 978-0-7167-4684-3.
{{cite book}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Selkoe DJ (December 2003). "Folding proteins in fatal ways". Nature. 426 (6968): 900–4. Bibcode:2003Natur.426..900S. doi:10.1038/nature02264. PMID 14685251. S2CID 6451881.
- ^ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2010). "Protein Structure and Function". Essential Cell Biology (3rd ed.). New York: Garland Science, Taylor and Francis Group, LLC. pp. 120–170. ISBN 978-0815341291.
- ^ Hebert DN, Molinari M (October 2007). "In and out of the ER: protein folding, quality control, degradation, and related human diseases". Physiological Reviews. 87 (4): 1377–408. doi:10.1152/physrev.00050.2006. PMID 17928587.
- ^ Russell R (January 2008). "RNA misfolding and the action of chaperones". Frontiers in Bioscience. 13 (13): 1–20. doi:10.2741/2557. PMC 2610265. PMID 17981525.
- ^ Kober L, Zehe C, Bode J (April 2013). "Optimized signal peptides for the development of high expressing CHO cell lines". Biotechnology and Bioengineering. 110 (4): 1164–73. doi:10.1002/bit.24776. PMID 23124363. S2CID 449870.
- ^ Moreau P, Brandizzi F, Hanton S, Chatre L, Melser S, Hawes C, Satiat-Jeunemaitre B (2007). "The plant ER-Golgi interface: a highly structured and dynamic membrane complex". Journal of Experimental Botany. 58 (1): 49–64. doi:10.1093/jxb/erl135. PMID 16990376.
- ^ Prudovsky I, Tarantini F, Landriscina M, Neivandt D, Soldi R, Kirov A, Small D, Kathir KM, Rajalingam D, Kumar TK (April 2008). "Secretion without Golgi". Journal of Cellular Biochemistry. 103 (5): 1327–43. doi:10.1002/jcb.21513. PMC 2613191. PMID 17786931.
- ^ Zaidi SK, Young DW, Choi JY, Pratap J, Javed A, Montecino M, Stein JL, Lian JB, van Wijnen AJ, Stein GS (October 2004). "Intranuclear trafficking: organization and assembly of regulatory machinery for combinatorial biological control". The Journal of Biological Chemistry. 279 (42): 43363–6. doi:10.1074/jbc.R400020200. PMID 15277516.
- ^ Mattick JS, Amaral PP, Dinger ME, Mercer TR, Mehler MF (January 2009). "RNA regulation of epigenetic processes". BioEssays. 31 (1): 51–9. doi:10.1002/bies.080099. PMID 19154003.
- ^ Martinez NJ, Walhout AJ (April 2009). "The interplay between transcription factors and microRNAs in genome-scale regulatory networks". BioEssays. 31 (4): 435–45. doi:10.1002/bies.200800212. PMC 3118512. PMID 19274664.
- ^ Tomilin NV (April 2008). "Regulation of mammalian gene expression by retroelements and non-coding tandem repeats". BioEssays. 30 (4): 338–48. doi:10.1002/bies.20741. PMID 18348251.
- ^ Veitia RA (November 2008). "One thousand and one ways of making functionally similar transcriptional enhancers". BioEssays. 30 (11–12): 1052–7. doi:10.1002/bies.20849. PMID 18937349.
- ^ Nguyen T, Nioi P, Pickett CB (May 2009). "The Nrf2-antioxidant response element signaling pathway and its activation by oxidative stress". The Journal of Biological Chemistry. 284 (20): 13291–5. doi:10.1074/jbc.R900010200. PMC 2679427. PMID 19182219.
- ^ Paul S (November 2008). "Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches". BioEssays. 30 (11–12): 1172–84. doi:10.1002/bies.20852. PMID 18937370. S2CID 29422790.
- ^ Los M, Maddika S, Erb B, Schulze-Osthoff K (May 2009). "Switching Akt: from survival signaling to deadly response". BioEssays. 31 (5): 492–5. doi:10.1002/bies.200900005. PMC 2954189. PMID 19319914.
- ^ Verheul TC, van Hijfte L, Perenthaler E, Barakat TS (2020). "The Why of YY1: Mechanisms of Transcriptional Regulation by Yin Yang 1". Front Cell Dev Biol. 8: 592164. doi:10.3389/fcell.2020.592164. PMC 7554316. PMID 33102493.
- ^ Spitz F, Furlong EE (September 2012). "Transcription factors: from enhancer binding to developmental control". Nat Rev Genet. 13 (9): 613–26. doi:10.1038/nrg3207. PMID 22868264. S2CID 205485256.
- ^ a b Schoenfelder S, Fraser P (August 2019). "Long-range enhancer-promoter contacts in gene expression control". Nat Rev Genet. 20 (8): 437–455. doi:10.1038/s41576-019-0128-0. PMID 31086298. S2CID 152283312.
- ^ Weintraub AS, Li CH, Zamudio AV, Sigova AA, Hannett NM, Day DS, Abraham BJ, Cohen MA, Nabet B, Buckley DL, Guo YE, Hnisz D, Jaenisch R, Bradner JE, Gray NS, Young RA (December 2017). "YY1 Is a Structural Regulator of Enhancer-Promoter Loops". Cell. 171 (7): 1573–1588.e28. doi:10.1016/j.cell.2017.11.008. PMC 5785279. PMID 29224777.
- ^ Lambert SA, Jolma A, Campitelli LF, Das PK, Yin Y, Albu M, Chen X, Taipale J, Hughes TR, Weirauch MT (February 2018). "The Human Transcription Factors". Cell. 172 (4): 650–665. doi:10.1016/j.cell.2018.01.029. PMID 29425488.
- ^ Grossman SR, Engreitz J, Ray JP, Nguyen TH, Hacohen N, Lander ES (July 2018). "Positional specificity of different transcription factor classes within enhancers". Proc Natl Acad Sci U S A. 115 (30): E7222–E7230. doi:10.1073/pnas.1804663115. PMC 6065035. PMID 29987030.
- ^ Allen BL, Taatjes DJ (March 2015). "The Mediator complex: a central integrator of transcription". Nat Rev Mol Cell Biol. 16 (3): 155–66. doi:10.1038/nrm3951. PMC 4963239. PMID 25693131.
- ^ Mikhaylichenko O, Bondarenko V, Harnett D, Schor IE, Males M, Viales RR, Furlong EE (January 2018). "The degree of enhancer or promoter activity is reflected by the levels and directionality of eRNA transcription". Genes Dev. 32 (1): 42–57. doi:10.1101/gad.308619.117. PMC 5828394. PMID 29378788.
- ^ Li QJ, Yang SH, Maeda Y, Sladek FM, Sharrocks AD, Martins-Green M (January 2003). "MAP kinase phosphorylation-dependent activation of Elk-1 leads to activation of the co-activator p300". EMBO J. 22 (2): 281–91. doi:10.1093/emboj/cdg028. PMC 140103. PMID 12514134.
- ^ Carullo NV, Phillips I RA, Simon RC, Soto SA, Hinds JE, Salisbury AJ, Revanna JS, Bunner KD, Ianov L, Sultan FA, Savell KE, Gersbach CA, Day JJ (September 2020). "Enhancer RNAs predict enhancer-gene regulatory links and are critical for enhancer function in neuronal systems". Nucleic Acids Res. 48 (17): 9550–9570. doi:10.1093/nar/gkaa671. PMC 7515708. PMID 32810208.
- ^ Lövkvist C, Dodd IB, Sneppen K, Haerter JO (June 2016). "DNA methylation in human epigenomes depends on local topology of CpG sites". Nucleic Acids Research. 44 (11): 5123–32. doi:10.1093/nar/gkw124. PMC 4914085. PMID 26932361.
- ^ Jabbari K, Bernardi G (May 2004). "Cytosine methylation and CpG, TpG (CpA) and TpA frequencies". Gene. 333: 143–9. doi:10.1016/j.gene.2004.02.043. PMID 15177689.
- ^ Weber M, Hellmann I, Stadler MB, Ramos L, Pääbo S, Rebhan M, Schübeler D (April 2007). "Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome". Nat. Genet. 39 (4): 457–66. doi:10.1038/ng1990. PMID 17334365. S2CID 22446734.
- ^ Yang X, Han H, De Carvalho DD, Lay FD, Jones PA, Liang G (October 2014). "Gene body methylation can alter gene expression and is a therapeutic target in cancer". Cancer Cell. 26 (4): 577–90. doi:10.1016/j.ccr.2014.07.028. PMC 4224113. PMID 25263941.
- ^ Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Bernstein BE, Costello JF, Wilkinson MF, Joung JK (December 2013). "Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins". Nat. Biotechnol. 31 (12): 1137–42. doi:10.1038/nbt.2726. PMC 3858462. PMID 24108092.
- ^ Kim JJ, Jung MW (2006). "Neural circuits and mechanisms involved in Pavlovian fear conditioning: a critical review". Neuroscience and Biobehavioral Reviews. 30 (2): 188–202. doi:10.1016/j.neubiorev.2005.06.005. PMC 4342048. PMID 16120461.
- ^ a b Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (July 2017). "Experience-dependent epigenomic reorganization in the hippocampus". Learning & Memory. 24 (7): 278–288. doi:10.1101/lm.045112.117. PMC 5473107. PMID 28620075.
- ^ a b Bernstein C (2022). "DNA Methylation and Establishing Memory". Epigenet Insights. 15: 25168657211072499. doi:10.1177/25168657211072499. PMC 8793415. PMID 35098021.
- ^ a b Keifer J (February 2017). "Primetime for Learning Genes". Genes (Basel). 8 (2): 69. doi:10.3390/genes8020069. PMC 5333058. PMID 28208656.
- ^ Saxonov S, Berg P, Brutlag DL (January 2006). "A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters". Proceedings of the National Academy of Sciences of the United States of America. 103 (5): 1412–7. Bibcode:2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC 1345710. PMID 16432200.
- ^ Bird A (January 2002). "DNA methylation patterns and epigenetic memory". Genes & Development. 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Tessitore A, Cicciarelli G, Del Vecchio F, Gaggiano A, Verzella D, Fischietti M, Vecchiotti D, Capece D, Zazzeroni F, Alesse E (2014). "MicroRNAs in the DNA Damage/Repair Network and Cancer". International Journal of Genomics. 2014: 1–10. doi:10.1155/2014/820248. PMC 3926391. PMID 24616890.
- ^ miRBase.org
- ^ a b Friedman RC, Farh KK, Burge CB, Bartel DP (January 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (September 2008). "Widespread changes in protein synthesis induced by microRNAs". Nature. 455 (7209): 58–63. Bibcode:2008Natur.455...58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (September 2008). "The impact of microRNAs on protein output". Nature. 455 (7209): 64–71. Bibcode:2008Natur.455...64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ Palmero EI, de Campos SG, Campos M, de Souza NC, Guerreiro ID, Carvalho AL, Marques MM (July 2011). "Mechanisms and role of microRNA deregulation in cancer onset and progression". Genetics and Molecular Biology. 34 (3): 363–70. doi:10.1590/S1415-47572011000300001. PMC 3168173. PMID 21931505.
- ^ Bernstein C, Bernstein H (May 2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World Journal of Gastrointestinal Oncology. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ Mellios N, Sur M (2012). "The Emerging Role of microRNAs in Schizophrenia and Autism Spectrum Disorders". Frontiers in Psychiatry. 3: 39. doi:10.3389/fpsyt.2012.00039. PMC 3336189. PMID 22539927.
- ^ Geaghan M, Cairns MJ (August 2015). "MicroRNA and Posttranscriptional Dysregulation in Psychiatry". Biological Psychiatry. 78 (4): 231–9. doi:10.1016/j.biopsych.2014.12.009. PMID 25636176.
- ^ a b c Walsh CT, Garneau-Tsodikova S, Gatto GJ (December 2005). "Protein posttranslational modifications: the chemistry of proteome diversifications". Angewandte Chemie. 44 (45): 7342–72. doi:10.1002/anie.200501023. PMID 16267872. S2CID 32157563.
- ^ Khoury GA, Baliban RC, Floudas CA (September 2011). "Proteome-wide post-translational modification statistics: frequency analysis and curation of the swiss-prot database". Scientific Reports. 1 (90): 90. Bibcode:2011NatSR...1E..90K. doi:10.1038/srep00090. PMC 3201773. PMID 22034591.
- ^ Mann M, Jensen ON (March 2003). "Proteomic analysis of post-translational modifications". Nature Biotechnology. 21 (3): 255–61. doi:10.1038/nbt0303-255. PMID 12610572. S2CID 205266061.
- ^ Seo J, Lee KJ (January 2004). "Post-translational modifications and their biological functions: proteomic analysis and systematic approaches". Journal of Biochemistry and Molecular Biology. 37 (1): 35–44. doi:10.5483/bmbrep.2004.37.1.035. PMID 14761301.
- ^ Rogers LD, Overall CM (December 2013). "Proteolytic post-translational modification of proteins: proteomic tools and methodology". Molecular & Cellular Proteomics. 12 (12): 3532–42. doi:10.1074/mcp.M113.031310. PMC 3861706. PMID 23887885.
- ^ "Search results < Expression Atlas < EMBL-EBI". www.ebi.ac.uk.
- ^ Moritz, CP (10 February 2020). "40 years Western blotting: A scientific birthday toast". Journal of Proteomics. 212: 103575. doi:10.1016/j.jprot.2019.103575. PMID 31706026.
- ^ Burkhart JM, Vaudel M, Gambaryan S, Radau S, Walter U, Martens L, Geiger J, Sickmann A, Zahedi RP (October 2011). "The first comprehensive and quantitative analysis of human platelet protein composition allows the comparative analysis of structural and functional pathways". Blood. 120 (15): e73–82. doi:10.1182/blood-2012-04-416594. PMID 22869793.
- ^ Moritz CP, Mühlhaus T, Tenzer S, Schulenborg T, Friauf E (June 2019). "Poor transcript-protein correlation in the brain: negatively correlating gene products reveal neuronal polarity as a potential cause" (PDF). Journal of Neurochemistry. 149 (5): 582–604. doi:10.1111/jnc.14664. PMID 30664243. S2CID 58667771.
- ^ Banf M, Rhee SY (January 2017). "Computational inference of gene regulatory networks: Approaches, limitations and opportunities". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1860 (1): 41–52. doi:10.1016/j.bbagrm.2016.09.003. PMID 27641093.
- ^ Chesler EJ, Lu L, Wang J, Williams RW, Manly KF (May 2004). "WebQTL: rapid exploratory analysis of gene expression and genetic networks for brain and behavior". Nature Neuroscience. 7 (5): 485–6. doi:10.1038/nn0504-485. PMID 15114364. S2CID 20241963.
- ^ Song Y, Wang W, Qu X, Sun S (February 2009). "Effects of hypoxia inducible factor-1alpha (HIF-1alpha) on the growth & adhesion in tongue squamous cell carcinoma cells". The Indian Journal of Medical Research. 129 (2): 154–63. PMID 19293442.
- ^ Hanriot L, Keime C, Gay N, Faure C, Dossat C, Wincker P, Scoté-Blachon C, Peyron C, Gandrillon O (September 2008). "A combination of LongSAGE with Solexa sequencing is well suited to explore the depth and the complexity of transcriptome". BMC Genomics. 9: 418. doi:10.1186/1471-2164-9-418. PMC 2562395. PMID 18796152.
- ^ Wheelan SJ, Martínez Murillo F, Boeke JD (July 2008). "The incredible shrinking world of DNA microarrays". Molecular BioSystems. 4 (7): 726–32. doi:10.1039/b706237k. PMC 2535915. PMID 18563246.
- ^ Miyakoshi M, Nishida H, Shintani M, Yamane H, Nojiri H (January 2009). "High-resolution mapping of plasmid transcriptomes in different host bacteria". BMC Genomics. 10: 12. doi:10.1186/1471-2164-10-12. PMC 2642839. PMID 19134166.
- ^ Denoeud F, Aury JM, Da Silva C, Noel B, Rogier O, Delledonne M, Morgante M, Valle G, Wincker P, Scarpelli C, Jaillon O, Artiguenave F (2008). "Annotating genomes with massive-scale RNA sequencing". Genome Biology. 9 (12): R175. doi:10.1186/gb-2008-9-12-r175. PMC 2646279. PMID 19087247.
- ^ Clough E, Barrett T (2016). "The Gene Expression Omnibus Database". Statistical Genomics. Methods in Molecular Biology. Vol. 1418. pp. 93–110. doi:10.1007/978-1-4939-3578-9_5. ISBN 978-1-4939-3576-5. PMC 4944384. PMID 27008011.
- ^ Kiliç S, White ER, Sagitova DM, Cornish JP, Erill I (January 2014). "CollecTF: a database of experimentally validated transcription factor-binding sites in Bacteria". Nucleic Acids Research. 42 (Database issue): D156–60. doi:10.1093/nar/gkt1123. PMC 3965012. PMID 24234444.
- ^ Moretto M, Sonego P, Dierckxsens N, Brilli M, Bianco L, Ledezma-Tejeida D, et al. (January 2016). "COLOMBOS v3.0: leveraging gene expression compendia for cross-species analyses". Nucleic Acids Research. 44 (D1): D620–3. doi:10.1093/nar/gkv1251. PMC 4702885. PMID 26586805.
- ^ Faith JJ, Driscoll ME, Fusaro VA, Cosgrove EJ, Hayete B, Juhn FS, et al. (January 2008). "Many Microbe Microarrays Database: uniformly normalized Affymetrix compendia with structured experimental metadata". Nucleic Acids Research. 36 (Database issue): D866–70. doi:10.1093/nar/gkm815. PMC 2238822. PMID 17932051.