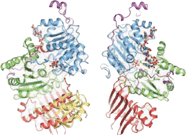
엑손접합단지
Exon junction complexeJC(Exon Junction Complex)는 RNA 스플라이싱 중에 함께 결합한 두 exon의 접합부에 있는 사전 메시지 RNA Strand에 형성되는 단백질 복합체다. EJC는 분할된 mRNA의 번역, 감시, 국산화 등에 큰 영향을 미친다.[1] 스플라이싱 도중 mRNA에 먼저 침전된 후 세포질 속으로 운반된다. 거기서 그것은 mRNA의 번역 후 규제에 큰 역할을 한다. 엑손 접합 단지는 스플라이싱 이벤트에 대한 위치별 메모리를 제공하는 것으로 생각된다. EJC는 mRNA 경로에 필요한 다른 요인에 대한 바인딩 플랫폼 역할을 하는 안정적인 헤테로테트라머 코어로 구성된다.[1] EJC의 코어는 아데노신 3인산염(ATP) 아날로그에 바인딩된 단백질 진핵생물 시작 인자 4A-III(eIF4A-III, DEAD-box RNA 헬리코아제)와 더불어 추가 단백질인 마고와 Y14를 포함한다.[2]이러한 단백질의 핵 점박이 영역으로의 결합은 최근에 측정되었으며 PI3K/AKT/mTOR 신호 경로에 의해 조절될 수 있다.[3] 복합체를 mRNA에 바인딩하기 위해 eIF4AIII 인자가 억제되어 ATP의 가수분해를 막는다.[2] 이것은 EJC를 ATP 종속단지로 인식한다. EJC는 또한 많은 추가 단백질과 상호작용한다.[4] 특히 SR 단백질이다. 이러한 상호작용은 mRNA 압축에 중요한 것으로 제안된다.[4] mRNA 수출에서 EJC의 역할이 논란이 되고 있다.
단백질 성분
EJC는 RNPS1, Y14, SRM160, Aly/REF, Magoh 등 몇 가지 주요 단백질 성분으로 구성되어 있다.[5][6][7] RNPS1은 스플라이싱의 공동 활성제로서 기능할 수 있지만, Y14와 함께 진핵생물의 난센스 매개 붕괴 과정에도 참여한다.[8][9] SRM160은 mRNA 3의 최종 처리를 개선하기 위해 제안된 또 다른 공동 활성제다.[10][11] 단백질 성분 마고는 알리가 핵 mRNA 수출에 종사하는 동안 mRNA의 아세포질 국산화 작용을 촉진하는 것으로 생각된다.[12][13][14] 알리는 단백질 UAP56에 의해 엑손 접합단지에 채용된 것으로 추정되며,[15] UAP56은 RNA 헬리코아제로 인식되지만 초기 스플라이소솜 조립에 필요한 스플라이싱 계수의 역할을 한다.[16] EJC 경로에 관련된 또 다른 요인은 DEK이다. 이 성분은 스플라이싱부터 전사조절, 염색체 구조까지 다양한 기능에 참여하는 것으로 알려져 있다.[17][18][19]
구조
엑손접합단지의 결정화로 단백질 성분의 구조적 구성이 드러났다. 단지의 중심은 총 99˚의 전체 치수가 54˚로 길다.[20] eIF4를 중심으로 구성된다.AIII 인자. 요인 자체는 mRNA 주위의 두 가지 다른 유형의 순응으로 구성된다. 즉, 폐쇄형과 개방형이다. 닫힌 상태에서 eIF4의 두 도메인AIII는 5'-addenyl-β, γ-imidodiphosphate(ADPNP) 및 mRNA에 대한 복합 결합 사이트를 형성한다.[20] 개방형 순응에서 두 도메인은 폐쇄 상태18에 비해 160도 회전한다. 단백질 성분 Magoh와 Y14는 EJC의 5' 극에 위치한 이단체를 형성하기 위해 결합된다.[21][22][23] Magoh는 eIF4에 바인딩된다.AIII 영역은 두 개의 C-단자 나선형의 잔류물과 큰 β-시트 한쪽 끝 사이의 상호작용을 통해 생성된다.[20] 두 eIF4 사이의 링커에 보존된 잔류물AIII 영역은 마고에서 특정 잔류물이 있는 염교나 수소 결합을 형성한다.[20] Mago β–시트의 두 번째 루프와 두 eIF4 사이에 다른 결합이 발생한다.AIII 도메인 및 연결 장치.[20] Y14와 eIF4 사이에는 단 하나의 부분적 결합만이 형성되어 있다.AIII. 보존된 잔류물 Y14 Arg108과 eIF4 사이의 염교로 구성된다.AIII 아스프401.[20] 이러한 잔류물에 모두 돌연변이가 일어난다면, EJC와 Magoh-Y14의 연관성은 존재하지 않을 것이다.[24]
메커니즘
진핵 세포에서 스플라이싱의 두 번째 단계 동안 EJC는 애벌레가 형성되고 엑손들이 함께 결합될 때 스플라이스 접합부의 5의 끝 부분(두 개의 엑손들이 결합되는 곳)에서 약 20-24 뉴클레오티드를 침전시킨다.[25][26] EJC를 mRNA에 바인딩하는 것은 성숙한 메신저 리보뉴클레오프로틴(mRNP)을 형성하기 위해 순차적으로 독립적으로 발생한다.[27] EJC는 핵에서 세포질로 내보내질 때 이 mRNP와 안정적으로 결합되어 있다. 단백질 성분은 EJC가 운반할 때 결합되거나 방출된다. 핵공기단지를 통한 mRNA의 번역이 발생하기 위해서는 NXF1/TAP와 NXT1/p15로 구성된 이단계가 대본에 바인딩되어야 한다.[28] NXF1/TAP은 세포질에 mRNA를 수출하는 주요 수용체다. 이는 RNA 결합 어댑터 단백질과 핵공기 복합체의 성분과 모두와 상호작용하기 때문이다.[29]
세포질에서 번역하는 동안 조기종료 코돈의 인식이 발생한다. 아래 보이는 이미지는 이 분야의 일반적인 견해와 달리 이 사건이 핵이라는 것을 암시한다. 핵의 번역은 데이터로 잘 뒷받침되지 않는 매우 논란이 많은 주제라는 것을 독자들은 알아야 한다.
말도 안 되는 매개 부패에서
mRNA 감시에는 엑손접합 단지들이 큰 역할을 하고 있다. 좀 더 구체적으로 말하면, 그것들은 NMD(Nonse mediated decusion way)에서 발견되는데, 여기서 조기 정지 코돈이 있는 mRNA 대본에서는 성능이 저하된다. 정상적인 mRNA 번역에서는 리보솜이 대본에 결합하여 아미노산 사슬의 신장화를 시작한다. 그것은 엑손 접속 단지의 위치에 도달할 때까지 계속되며, 그것은 그것을 대체한다. 다음으로 리보솜이 종료 코돈에 도달하면 번역이 완료된다. NMD에서 mRNA 대본은 말도 안 되는 돌연변이로 인한 조기종료 코돈(PTC)을 포함하고 있다. 이 코돈이 EJC 사이트 이전에 발생하는 경우, EJC는 구속 상태를 유지하여 mRNA 붕괴를 트리거한다.[30] EJC와 그 위치는 성적서의 결함 여부를 결정하는 조정기의 한 종류 역할을 한다.
EJC는 또한 감시 요소 UPF1, UPF2, UPF3의 모집 등 다른 방식으로 NMD에 참여하는 것으로 알려져 있다.[31] 이 단백질들은 NMD 메커니즘의 가장 중요한 구성 요소들이다. EJC 단백질 MAGOH, Y14 및 eIF4AIII는 UPF2와 UPF1 사이에서 트리머 콤플렉스를 형성하는 가교 역할을 하는 UPF3에 바인딩을 제공한다.[32] 이 단지 내에서 UPF2와 UPF3는 UPF1의 ATPase와 RNA 헬리코아제를 촉진하기 위해 협력적으로 작용한다.[32] EJC 코어는 UPF 콤플렉스를 mRNA에 안정적으로 고정시키고 필수 UPF1 단백질의 조절에 도움을 준다.[32] PTC에 정지된 리보솜은 방출 인자 eRF1 및 eRF3와의 상호작용을 통해 UPF1을 모집한다.[32] SMG1, eRF1, eRF3, UPF1이라는 단백질과 함께 복합 SUP를 형성한다. 이 복합체는 리보솜과 UPF3와 UPF2와 연관된 다운스트림 EJC 사이에 다리를 형성한다.[32] 이 상호작용은 SMG1에 의한 UPF1의 인산화 작용을 촉발하여 eRF1과 eRF3의 분리를 유발한다.[32] 생성된 단지는 EJC, UPF3, UPF2, 인광 UPF1, SMG1로 구성되며, 차례로 mRNA의 열화를 유발한다.[32]
참고 및 참조
- ^ a b Tange, TØ; Nott, A; Moore, MJ (June 2004). "The ever-increasing complexities of the exon junction complex". Current Opinion in Cell Biology. 16 (3): 279–84. doi:10.1016/j.ceb.2004.03.012. PMID 15145352.
- ^ a b Ballut L, Marchadier B, Baguet A, Tomasetto C, Séraphin B, Le Hir H (2005). "The exon junction core complex is locked onto RNA by inhibition of eIF4AIII ATPase activity". Nat. Struct. Mol. Biol. 12 (10): 861–9. doi:10.1038/nsmb990. PMID 16170325. S2CID 1359792.
- ^ Quaresma, Alexandre J.; Nickerson, Jeffrey A. (2013), "Regulation of mRNA export by the PI3 kinase/AKT signal transduction pathway", Mol Biol Cell, 24 (8): 1208–21, doi:10.1091/mbc.E12-06-0450, PMC 3623641, PMID 23427269
- ^ a b Singh G, Kucukural A, Cenik C, Leszyk JD, Shaffer SA, Weng Z, Moore MJ (2012). "The cellular EJC interactome reveals higher-order mRNP structure and an EJC-SR protein nexus". Cell. 151 (4): 750–64. doi:10.1016/j.cell.2012.10.007. PMC 3522173. PMID 23084401.
- ^ Kataoka, N.; Yong, J.; Kim, V. N.; Velazquez, F.; Perkinson, R. A.; Wang, F.; Dreyfuss, G. (2000). "Pre-mRNA Splicing Imprints mRNA in the Nucleus with a Novel RNA-Binding Protein that Persists in the Cytoplasm". Mol Cell. 6 (3): 673–682. doi:10.1016/s1097-2765(00)00065-4. PMID 11030346.
- ^ Le Hir H, Gatfield D, Izaurralde E, Moore MJ (2001). "The exon-exon junction complex provides a binding platform for factors involved in mRNA export and nonsense-mediated mRNA decay". EMBO J. 20 (17): 4987–97. doi:10.1093/emboj/20.17.4987. PMC 125616. PMID 11532962.
- ^ Le Hir, H.; Izaurralde, E.; Maquat, L. E.; Moore, M. J. (2000). "The spliceosome deposits multiple proteins 20-24 nucleotides upstream of mRNA exon-exon junctions". EMBO J. 19 (24): 6860–6869. doi:10.1093/emboj/19.24.6860. PMC 305905. PMID 11118221.
- ^ Lykke-Andersen, J.; Shu, M.-D.; Steitz, J. A. (2001). "Communication of the Position of Exon-Exon Junctions to the mRNA Surveillance Machinery by the Protein RNPS1". Science. 293 (5536): 1836–1839. doi:10.1126/science.1062786. PMID 11546874. S2CID 389385.
- ^ Lejeune, F.; Ishigaki, Y.; Li, X.; Maquat, L. E. (2002). "The exon junction complex is detected on CBP80-bound but not eIF4E-bound mRNA in mammalian cells: dynamics of mRNP remodeling". EMBO J. 21 (13): 3536–3545. doi:10.1093/emboj/cdf345. PMC 126094. PMID 12093754.
- ^ Mayeda, A.; Badolato, J.; Kobayashi, R.; Zhang, M. Q.; Gardiner, E. M.; Krainer, A. R. (1999). "Purification and characterization of human RNPS1: a general activator of pre-mRNA splicing". EMBO J. 18 (16): 4560–4570. doi:10.1093/emboj/18.16.4560. PMC 1171530. PMID 10449421.
- ^ McCracken, S.; Lambermon, M.; Blencowe, B. J. (2002). "SRm160 Splicing Coactivator Promotes Transcript 3'-End Cleavage". Mol. Cell. Biol. 22 (1): 148–160. doi:10.1128/mcb.22.1.148-160.2002. PMC 134228. PMID 11739730.
- ^ Le Hir, H.; Gatfield, D.; Braun, I. C.; Forler, D.; Izaurralde, E. (2001). "The protein Mago provides a link between splicing and mRNA localization". EMBO Rep. 2 (12): 1119–1124. doi:10.1093/embo-reports/kve245. PMC 1084163. PMID 11743026.
- ^ Zhou, Z.; Luo, M.-J.; Straesser, K.; Katahira, J.; Hurt, E.; Reed, R. (2000). "The protein Aly links pre-messenger-RNA splicing to nuclear export in metazoans". Nature. 407 (6802): 401–405. doi:10.1038/35030160. PMID 11014198. S2CID 312262.
- ^ Rodrigues, J. P.; Rode, M.; Gatfield, D.; Blencowe, B. J.; Carmo-Fonseca, M.; Izaurralde, E. (2001). "REF proteins mediate the export of spliced and unspliced mRNAs from the nucleus". Proc. Natl. Acad. Sci. USA. 98 (3): 1030–1035. doi:10.1073/pnas.98.3.1030. PMC 14703. PMID 11158589.
- ^ Cullen, B. R. (2003). "Nuclear RNA export". J. Cell Sci. 116 (4): 587–597. doi:10.1242/jcs.00268. PMID 12538759.
- ^ Gatfield, D.; Izaurralde, E. (2002). "REF1/Aly and the additional exon junction complex proteins are dispensable for nuclear mRNA export". J. Cell Biol. 159 (4): 579–588. doi:10.1083/jcb.200207128. PMC 2173090. PMID 12438415.
- ^ Alexiadis V, Waldmann T, Andersen J, Mann M, Knippers R, Gruss C (2000). "The protein encoded by the proto-oncogene DEK changes the topology of chromatin and reduces the efficiency of DNA replication in a chromatin-specific manner". Genes Dev. 14 (11): 1308–12. PMC 316669. PMID 10837023.
- ^ McGarvey, T.; Rosonina, E.; McCracken, S.; Li, Q.; Arnaout, R.; Mientjes, E.; Nickerson, J.A.; Awrey, D.; Greenblatt, J.; Grosveld, G.; Blencowe, BJ (2000). "The acute myeloid leukemia-associated protein, DEK, forms a splicing-dependent interaction with exon-product complexes". J. Cell Biol. 150 (2): 309–320. doi:10.1083/jcb.150.2.309. PMC 2180225. PMID 10908574.
- ^ Faulkner, N.E.; Hilfinger, J.M.; Markovitz, D.M. (2001). "Protein phosphatase 2A activates the HIV-2 promoter through enhancer elements that include the pets site". J. Biol. Chem. 276 (28): 25804–25812. doi:10.1074/jbc.m006454200. PMID 11320078.
- ^ a b c d e f Andersen CB, Ballut L, Johansen JS, Chamieh H, Nielsen KH, Oliveira CL, Pedersen JS, Séraphin B, Le Hir H, Andersen GR (2006). "Structure of the exon junction core complex with a trapped DEAD-box ATPase bound to RNA". Science. 313 (5795): 1968–72. doi:10.1126/science.1131981. PMID 16931718. S2CID 26409491.
- ^ Lau, C. K.; Diem, M. D.; Dreyfuss, G.; Van Duyne, G. D. (2003). "Structure of the Y14-Magoh Core of the Exon Junction Complex". Curr. Biol. 13 (11): 933–941. doi:10.1016/s0960-9822(03)00328-2. PMID 12781131.
- ^ Fribourg, S.; Gatfield, D.; Izaurralde, E. Conti (2003). "A novel mode of RBD-protein recognition in the Y14–Mago complex". Nat. Struct. Biol. 10 (6): 433–439. doi:10.1038/nsb926. PMID 12730685. S2CID 40116577.
- ^ Shi H, Xu RM (2003). "Crystal structure of the Drosophila Mago nashi-Y14 complex". Genes Dev. 17 (8): 971–6. doi:10.1101/gad.260403. PMC 196043. PMID 12704080.
- ^ Gehring, Niels H.; Kunz, Joachim B.; Neu-Yilik, Gabriele; Breit, Stephen; Viegas, Marcelo H.; Hentze, Matthias W.; Kulozik, Andreas E. (2005). "Exon-Junction Complex Components Specify Distinct Routes of Nonsense-Mediated mRNA Decay with Differential Cofactor Requirements". Molecular Cell. 20 (1): 65–75. doi:10.1016/j.molcel.2005.08.012. ISSN 1097-2765. PMID 16209946.
- ^ Reichert, V.L.; Le Hir, H.; Jurica, M.S.; Moore, M.J. (2002). "5' exon interactions within the spliceosome establish a framework for exon junction complex structure and assembly". Genes Dev. 16 (21): 2778–2791. doi:10.1101/gad.1030602. PMC 187475. PMID 12414731.
- ^ Shibuya, T.; Tange, T.O.; Sonenberg, N.; Moore, M.J. (2004). "eIF4AIII binds spliced mRNA in the exon junction complex and is essential for nonsense mediated decay". Nat. Struct. Mol. Biol. 11 (4): 346–351. doi:10.1038/nsmb750. PMID 15034551. S2CID 30171314.
- ^ Le Hir, H.; Izaurralde, E.; Maquat, L.E.; Moore, M.J. (2000). "The spliceosome deposits multiple proteins 20-24 nucleotides upstream of mRNA exon-exon junctions". EMBO J. 19 (24): 6860–6869. doi:10.1093/emboj/19.24.6860. PMC 305905. PMID 11118221.
- ^ Reed, R.; Hurt, E. (2002). "A conserved mRNA export machinery coupled to pre-mRNA splicing". Cell. 108 (4): 523–531. doi:10.1016/s0092-8674(02)00627-x. PMID 11909523.
- ^ Izaurralde, E (2002). "A novel family of nuclear transport receptors mediates the export of messenger RNA to the cytoplasm. Eur". J. Cell Biol. 81 (11): 577–584. doi:10.1078/0171-9335-00273. PMID 12498157.
- ^ Chang YF, Imam JS, Wilkinson MF (2007). "The nonsense-mediated decay RNA surveillance pathway". Annu Rev Biochem. 76: 51–74. doi:10.1146/annurev.biochem.76.050106.093909. PMID 17352659.
- ^ Conti, E.; Izaurralde, E. (2005). "Nonsense-mediated mRNA decay: molecular insights and mechanistic variations across species". Curr. Opin. Cell Biol. 17 (3): 316–325. doi:10.1016/j.ceb.2005.04.005. PMID 15901503.
- ^ a b c d e f g Chamieh, Hala; Ballut, Lionel; Bonneau, Fabien; Le Hir, Hervé (2007). "NMD factors UPF2 and UPF3 bridge UPF1 to the exon junction complex and stimulate its RNA helicase activity". Nature Structural & Molecular Biology. 15 (1): 85–93. doi:10.1038/nsmb1330. ISSN 1545-9993. PMID 18066079. S2CID 6268216.