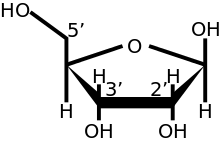
파이브 프라임 캡
Five-prime cap분자생물학에서 5-프라임 캡(5' cap)은 전구체 메신저 RNA와 같은 일부 1차 전사체의 5' 말단에서 특별히 변경된 뉴클레오티드이다.mRNA 캡핑으로 알려진 이 과정은 단백질 합성 동안 번역을 겪을 수 있는 안정적이고 성숙한 메신저 RNA의 생성에 매우 조절되고 필수적입니다.미토콘드리아 mRNA와 엽록체 mRNA는 제한되지 않는다.
구조.
진핵생물에서 mRNA 분자의 5' 말단에서 발견되는 5' 캡(cap-0)은 특이한 5' ~ 5' 삼인산 결합을 통해 mRNA에 연결된 구아닌 뉴클레오티드로 구성된다.이 구아노신은 메틸전달효소([3][4][5][6]methyl transferase)에 의해 생체내 캡핑 직후 7위치로 메틸화된다.이는 7-메틸구아닐산 캡(mG7)으로 약칭된다.
다세포 진핵생물 및 일부 [7]바이러스에서는 mRNA의 5' 말단의 첫 번째 2' 리보스당의 2' 히드록시기의 메틸화를 포함한 추가 변형이 존재하며, cap-1은 첫 번째 리보스당의 메틸화 2'-히드록시기를 가지며, cap-2는 오른쪽의 첫 번째 2'-히드록시기를 가진다.5' 캡은 RNA 분자의 3' 말단과 화학적으로 유사하다(캡 리보스의 5' 탄소는 결합되고 3' 비결합).이는 5' 엑소핵산가수분해효소에 [citation needed]상당한 내성을 제공한다.
작은 핵 RNA에는 고유한 5µ-caps가 포함되어 있습니다.Sm-클래스 snRNA는 5µ-트리메틸구아노신 캡에서 발견되며, LSM-클래스 snRNA는 5µ-모노메틸인산 [8]캡에서 발견됩니다.
박테리아에서, 그리고 잠재적으로 고등 생물에서, 일부 RNA는 NAD, NADH 또는 3'-디포스포-코엔자임 [9][10]A로+ 덮인다.
모든 유기체에서 mRNA 분자는 메신저 RNA 디캡핑이라고 알려진 과정에서 디캡핑될 수 있다.
캡핑 프로세스
7-메틸구아닐산염으로 캡핑하는 시작점은 RNA 분자의 변경되지 않은 5µ 말단이며, 이는 삼인산기로 종단된다.이는 최종 뉴클레오티드를 거쳐 5' [3]탄소에 결합된 세 개의 인산기가 특징입니다.초기 pre-mRNA가 합성되고 있기 때문에 전사가 완료되기 전에 캡핑 과정이 시작됩니다.
- 말단 인산기 중 하나는 RNA 트리포스파타아제에 의해 제거되고, 이인산기(즉, 5µ(ppN)[pN])n가 남는다.
- GTP는 mRNA 구아닐전달효소(guanyl transferase)에 의해 말단 2인산에 첨가되며, 이 과정에서 GTP 기질로부터 피로인산을 잃는다.그 결과 5µ(Gp)(ppN)[pN]n이 생성되는 5µ-5' 삼인산 결합이 생성된다.
- 구아닌의 7-질소는 mRNA(구아닌-N7-)-메틸전달효소에 의해 메틸화되고, S-아데노실-L-메티오닌은 탈메틸화되어 S-아데노실-L-호모시스테인이 생성되며, 5µ(m7Gp)(ppN)[nPN](cap-0)이 된다.
- 캡 인접 변형은 일반적으로 첫 번째 및 두 번째 뉴클레오티드에 대해 최대 5µ(m7Gp)(ppN*)(pN*)[npN](캡-1 및 캡-2)[7]까지 생성될 수 있다.
- 가장 가까운 캡인접 뉴클레오티드가 2μ-O-리보스 메틸아데노신(즉, 5μ(m7Gp)(ppAm)[pN])n인 경우, N6 메틸 위치에서 더욱 메틸화되어 N-메틸아데노신을6 형성할 수 있으며, 결과적으로 5μ(m7Gp)(pp6Am)[P]n[3]가 된다.
NAD, NADH 또는 3'-디포스포-코엔자임 A로 캡핑하는+ 메커니즘은 다르다.NAD, NADH 또는 3'-디포스포-코엔자임A에 의한+ 캡핑은 NAD, NADH 또는 3'-데스포스포-코엔자임A가 전사를 개시하기 위한 '비카노니컬 개시 뉴클레오티드(NCIN)'가 되는+ 'ab initio 캡핑 기구'를 통해 이루어지며, 이를 통해 RNA아제에 의해 직접 결합된다.박테리아 RNA 중합효소 및 진핵생물 RNA 중합효소 II는 모두 이 "ab initio caping 메커니즘"[9]을 수행할 수 있다.
타겟팅
7-메틸구아닐산염으로 캡핑하기 위해 캡핑효소복합체(CEC)는 전사가 시작되기 전에 RNA 중합효소 II에 결합한다.RNA 중합효소 II에서 새로운 전사체의 5' 말단이 나오는 즉시 CEC는 캡핑 프로세스를 수행합니다(이러한 메커니즘은 [11][12][13][14]폴리아데닐화와 마찬가지로 캡핑을 보장합니다).캡을 씌우는 효소는 RNA 중합효소 II에만 결합할 수 있으며, 거의 전적으로 [12][14]mRNA인 이러한 전사물에만 특이성을 보장합니다.
NAD, NADH 또는 3'-디포스포-코엔자임 A에 의한+ 캡핑은 프로모터 [9]배열에 의해 표적화된다.NAD+, NADH 또는 3'-디포스포-코엔자임 A에 의한 캡핑은 전사 시작 부위의 바로 상류에 특정 서열을 가진 프로모터에서만 발생하므로 [9]특정 프로모터에서 합성된 RNA에서만 발생합니다.
기능.
5인치 캡에는 4가지 주요 기능이 있습니다.
- 원자력 [15][16]수출 규제
- 엑소핵산가수분해효소에 [9][17][18][19]의한 분해방지
- 번역 촉진(리보솜 및 번역 참조)[3][4][5]
- 5인치 근위부 [20]내 절제 촉진.
RNA의 핵수출은 캡결합복합체(CBC)에 의해 조절되며, 캡결합복합체는 7-메틸구아닐산캡 RNA에만 결합한다.CBC는 핵공 복합체에 의해 인식되어 수출된다.번역의 선구자 라운드 후 세포질에 들어가면 CBC는 eIF4F [6]복합체의 변환 계수 eIF4E 및 eIF4G로 대체됩니다.이 복합체는 리보솜을 [21]포함한 다른 번역 개시 기계에 의해 인식됩니다.
7-메틸구아닐산염으로 캡핑하면 두 가지 방법으로 5µ 분해가 방지됩니다.첫째, 기능적으로 3' 말단처럼 보이므로 (이상과 같이) 5' 엑소핵산가수분해효소에 의한 mRNA의 분해를 방지할 수 있다.둘째, CBC와 eIF4E/eIF4G는 캡에 대한 디캡핑 효소의 접근을 차단합니다.이것은 진핵생물에서 필수적인 mRNA의 반감기를 증가시키는데, 이는 수출과 번역 과정이 상당한 시간이 걸리기 때문이다.
캡을 결합하기 위해 eIF4E와 경합해야 하는 적어도 Dcp1 및 Dcp2로 이루어진 탈캡핑 복합체에 의해 7-메틸구아닐레이트 캡핑 mRNA의 탈캡핑을 촉매한다.따라서 7-메틸구아닐산 캡은 능동적으로 번역되는 mRNA의 마커이며 새로운 자극에 반응하여 mRNA 반감기를 조절하는 데 세포에 의해 사용된다.바람직하지 않은 mRNA는 P-body로 보내져 임시 저장 또는 디캡핑이 이루어지며, 자세한 내용은 아직 [22]해결 중입니다.
5' 근위부 인트론 절제 촉진 메커니즘은 잘 알려져 있지 않지만 7-메틸구아닐레이트 캡은 스플라이싱 과정에서 스플라이싱과 상호 작용하여 인트론 절제를 촉진하는 것으로 보인다.
「 」를 참조해 주세요.
레퍼런스
- ^ Temperley RJ, Wydro M, Lightowlers RN, Chrzanowska-Lightowlers ZM (June 2010). "Human mitochondrial mRNAs—like members of all families, similar but different". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1797 (6–7): 1081–1085. doi:10.1016/j.bbabio.2010.02.036. PMC 3003153. PMID 20211597.
- ^ Monde RA, Schuster G, Stern DB (7 June 2000). "Processing and degradation of chloroplast mRNA". Biochimie. 82 (6–7): 573–582. doi:10.1016/S0300-9084(00)00606-4. PMID 10946108.
- ^ a b c d Shatkin, A (December 1976). "Capping of eucaryotic mRNAs". Cell. 9 (4): 645–653. doi:10.1016/0092-8674(76)90128-8. PMID 1017010. S2CID 26743858.
- ^ a b Banerjee AK (June 1980). "5′-terminal cap structure in eucaryotic messenger ribonucleic acids". Microbiological Reviews. 44 (2): 175–205. doi:10.1128/mmbr.44.2.175-205.1980. PMC 373176. PMID 6247631.
- ^ a b Sonenberg N, Gingras AC (April 1998). "The mRNA 5′ cap-binding protein eIF4E and control of cell growth". Current Opinion in Cell Biology. 10 (2): 268–275. doi:10.1016/S0955-0674(98)80150-6. PMID 9561852.
- ^ a b Marcotrigiano J, Gingras AC, Sonenberg N, Burley SK (June 1997). "Cocrystal structure of the messenger RNA 5′ cap-binding protein (eIF4E) bound to 7-methyl-GDP". Cell. 89 (6): 951–961. doi:10.1016/S0092-8674(00)80280-9. PMID 9200613. S2CID 15200116.
- ^ a b Fechter P, Brownlee GG (May 2005). "Recognition of mRNA cap structures by viral and cellular proteins". The Journal of General Virology. 86 (Pt 5): 1239–1249. doi:10.1099/vir.0.80755-0. PMID 15831934.
- ^ Matera AG, Terns RM, Terns MP (March 2007). "Non-coding RNAs: lessons from the small nuclear and small nucleolar RNAs". Nature Reviews. Molecular Cell Biology. 8 (3): 209–220. doi:10.1038/nrm2124. PMID 17318225. S2CID 30268055.
- ^ a b c d e f Bird JG, Zhang Y, Tian Y, Panova N, Barvík I, Greene L, Liu M, Buckley B, Krásný L, Lee JK, Kaplan CD, Ebright RH, Nickels BE (July 2016). "The mechanism of RNA 5′ capping with NAD+, NADH and desphospho-CoA". Nature. 535 (7612): 444–447. Bibcode:2016Natur.535..444B. doi:10.1038/nature18622. PMC 4961592. PMID 27383794.
- ^ Cahová H, Winz ML, Höfer K, Nübel G, Jäschke A (March 2015). "NAD captureSeq indicates NAD as a bacterial cap for a subset of regulatory RNAs". Nature. 519 (7543): 374–377. Bibcode:2015Natur.519..374C. doi:10.1038/nature14020. PMID 25533955. S2CID 4446837.
- ^ Cho EJ, Takagi T, Moore CR, Buratowski S (December 1997). "mRNA capping enzyme is recruited to the transcription complex by phosphorylation of the RNA polymerase II carboxy-terminal domain". Genes & Development. 11 (24): 3319–3326. doi:10.1101/gad.11.24.3319. PMC 316800. PMID 9407025.
- ^ a b Fabrega C, Shen V, Shuman S, Lima CD (June 2003). "Structure of an mRNA capping enzyme bound to the phosphorylated carboxy-terminal domain of RNA polymerase II". Molecular Cell. 11 (6): 1549–1561. doi:10.1016/S1097-2765(03)00187-4. PMID 12820968.
- ^ Ho CK, Lehman K, Shuman S (December 1999). "An essential surface motif (WAQKW) of yeast RNA triphosphatase mediates formation of the mRNA capping enzyme complex with RNA guanylyltransferase". Nucleic Acids Research. 27 (24): 4671–4678. doi:10.1093/nar/27.24.4671. PMC 148765. PMID 10572165.
- ^ a b Hirose Y, Manley JL (June 2000). "RNA polymerase II and the integration of nuclear events". Genes & Development. 14 (12): 1415–1429. doi:10.1101/gad.14.12.1415. PMID 10859161. S2CID 43589702. Retrieved 23 November 2014.
- ^ Visa N, Izaurralde E, Ferreira J, Daneholt B, Mattaj IW (April 1996). "A nuclear cap-binding complex binds Balbiani ring pre-mRNA cotranscriptionally and accompanies the ribonucleoprotein particle during nuclear export". The Journal of Cell Biology. 133 (1): 5–14. doi:10.1083/jcb.133.1.5. PMC 2120770. PMID 8601613.
- ^ Lewis JD, Izaurralde E (July 1997). "The role of the cap structure in RNA processing and nuclear export". European Journal of Biochemistry. 247 (2): 461–469. doi:10.1111/j.1432-1033.1997.00461.x. PMID 9266685.
- ^ Evdokimova V, Ruzanov P, Imataka H, Raught B, Svitkin Y, Ovchinnikov LP, Sonenberg N (October 2001). "The major mRNA-associated protein YB-1 is a potent 5′ cap-dependent mRNA stabilizer". The EMBO Journal. 20 (19): 5491–5502. doi:10.1093/emboj/20.19.5491. PMC 125650. PMID 11574481.
- ^ Gao M, Fritz DT, Ford LP, Wilusz J (March 2000). "Interaction between a poly(A)-specific ribonuclease and the 5′ cap influences mRNA deadenylation rates in vitro". Molecular Cell. 5 (3): 479–488. doi:10.1016/S1097-2765(00)80442-6. PMC 2811581. PMID 10882133.
- ^ Burkard KT, Butler JS (January 2000). "A nuclear 3′–5′ exonuclease involved in mRNA degradation interacts with Poly(A) polymerase and the hnRNA protein Npl3p". Molecular and Cellular Biology. 20 (2): 604–616. doi:10.1128/MCB.20.2.604-616.2000. PMC 85144. PMID 10611239.
- ^ Konarska MM, Padgett RA, Sharp PA (October 1984). "Recognition of cap structure in splicing in vitro of mRNA precursors". Cell. 38 (3): 731–736. doi:10.1016/0092-8674(84)90268-X. PMID 6567484. S2CID 10721149.
- ^ Kapp LD, Lorsch JR (2004). "The molecular mechanics of eukaryotic translation". Annual Review of Biochemistry. 73 (1): 657–704. doi:10.1146/annurev.biochem.73.030403.080419. PMID 15189156.
- ^ Parker R, Sheth U (March 2007). "P bodies and the control of mRNA translation and degradation". Molecular Cell. 25 (5): 635–646. doi:10.1016/j.molcel.2007.02.011. PMID 17349952.
외부 링크
- "RNA Caps". PubMed Medical Subject Heading (MeSH). National Institutes of Health.