헤테로크로마틴
Heterochromatin헤테로크로마틴은 여러 가지 종류가 있는 단단하게 포장된 DNA 또는 응축된 DNA의 형태이다.이러한 변종들은 구성 헤테로크로마틴과 통성 헤테로크로마틴의 두 극단 사이의 연속체 위에 있다.둘 다 유전자 발현에 역할을 한다.그러나 Volpe 등 연구진에 따르면, 그것은 촘촘하게 포장되어 있기 때문에 중합효소에 접근할 수 없는 것으로 생각되어 전사되지 않았다.(2002)[1] 및 그 이후의 [2]많은 논문들은 사실 이 DNA의 많은 부분이 전사되지만, RNA 유도 전사 사일런싱(RITS)을 통해 지속적으로 전사된다.전자현미경 검사와4 OsO 염색에 대한 최근의 연구는 조밀한 패킹이 염색질에 [3]의한 것이 아니라는 것을 보여준다.
구성 헤테로크로마틴은 그 주변의 유전자에 영향을 미칠 수 있다(예: 위치 효과 정맥류).그것은 보통 반복적이며 다른 유전자 발현이나 억제 신호의 유인체 역할을 할 뿐만 아니라 동원체나 텔로미어와 같은 구조적 기능을 형성한다.
통성 헤테로크로마틴은 히스톤 탈아세틸화 또는 RNAi를 통한 PiRNA(Piwi-Interacting RNA)와 같은 메커니즘을 통해 침묵되는 유전자의 결과물이다.그것은 반복적이지 않고 구성 헤테로크로마틴의 콤팩트한 구조를 공유한다.그러나 특정 발달적 또는 환경적 시그널링 신호에서는 응축된 구조가 상실되고 문자 변환이 [4]활성화될 수 있습니다.
헤테로크로마틴은 인간 [5]게놈의 특정 부분에서 H3K9의 디메틸화 및 트리메틸화와 관련이 있다.H3K9me3 관련 메틸전달효소는 유기형성 시작 시 혈통공약 중 헤테로크로마틴을 수정하고 혈통 [6]충실도를 유지하는 데 중추적인 역할을 하는 것으로 보인다.
구조.
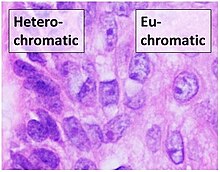
염색질은 에크로마틴과 헤테로크로마틴의 [7]두 가지 종류가 있습니다.원래, 두 가지 형태는 세포학적으로 얼마나 심하게 염색되는지에 따라 구별되었다. 즉, 에우크로마틴은 덜 강렬하지만 헤테로크로마틴은 강하게 염색되어 더 엄격한 포장 상태를 나타낸다.헤테로크로마틴은 보통 핵 주변에 국소화된다.이러한 초기의 이분법에도 불구하고, 동물과 식물 모두에서[9] 최근[8] 증거는 두 개 이상의 뚜렷한 헤테로크로마틴 상태가 존재하며, 사실 그것은 후생유전학적 마크의 다른 조합으로 특징지어지는 4~5개의 '상태'에 존재할 수 있다는 것을 시사했다.
헤테로크로마틴은 주로 유전적으로 비활성화된 위성 [10]배열로 구성되며, 많은 유전자들이 다양한 범위로 억제되지만, 일부는 [11]에크로마틴으로 발현될 수 없다.동원체와 텔로미어는 둘 다 헤테로크로미컬이며, 두 번째 여성 불활성 X염색체의 Bar체이다.
기능.
헤테로크로마틴은 유전자 조절에서 염색체 [12]무결성의 보호까지 여러 기능과 연관되어 왔다; 이러한 역할들 중 일부는 DNA의 조밀한 패킹에 기인할 수 있으며, 이것은 보통 DNA 또는 그와 관련된 요소들과 결합하는 단백질 인자에 덜 접근하게 만든다.예를 들어, 벌거벗은 이중 가닥 DNA 말단은 보통 세포에 의해 손상되거나 바이러스 DNA로 해석되어 세포 주기 정지, DNA 복구 또는 박테리아의 핵산 분해 효소에 의해 파편의 파괴를 유발합니다.
염색질의 일부 영역은 유사분열 시 염색체와 비슷한 상태를 보이는 섬유로 매우 촘촘히 채워져 있다.헤테로크로마틴은 일반적으로 복제적으로 유전된다; 세포가 분열할 때, 두 딸 세포는 전형적으로 DNA의 같은 영역 내에 헤테로크로마틴을 포함하고, 후생유전적인 유전을 초래한다.변이는 헤테로크로마틴이 인접한 유전자를 잠식하거나 영역의 극단에서 유전자에서 멀어지게 한다.전사 가능한 재료는 이들 경계 영역에 위치함으로써 억제할 수 있다.이로 인해 [13]세포마다 다른 발현 수준이 발생하며,[14] 위치 효과 변동에 의해 입증될 수 있습니다.단열재 배열은 구성 헤테로크로마틴과 고활성 유전자가 나란히 있는 드문 경우 장벽으로 작용할 수 있다(예: 5').닭의 β-글로빈 [15]궤적 상류 HS4 절연체 및 2개의 사카로미세스의 궤적).[16][17]
구성 헤테로크로마틴
특정 종의 모든 세포는 구성 헤테로크로마틴에서 DNA의 동일한 영역을 패키징하기 때문에 모든 세포에서 구성 헤테로크로마틴에 포함된 유전자는 제대로 발현되지 않는다.예를 들어 인간의 모든 염색체 1, 9, 16 및 Y염색체는 구성 헤테로크로마틴의 큰 영역을 포함한다.대부분의 유기체에서, 구성 헤테로크로마틴은 염색체 동원체 주변과 텔로미어 근처에서 발생한다.
통성 헤테로크로마틴
통성 헤테로크로마틴으로 포장된 DNA의 영역은 종 내 세포 유형 간에 일관되지 않을 것이고, 따라서 통성 헤테로크로마틴으로 포장된 한 세포의 배열은 다른 세포의 에크로마틴으로 포장될 수 있다(그리고 그 안의 유전자는 더 이상 침묵되지 않는다).단, 통성 헤테로크로마틴의 형성은 조절되며, 종종 형태형성 또는 분화와 관련된다.통성 헤테로크로마틴의 예는 암컷 포유동물에서의 X염색체 불활성화이다.한쪽 X염색체는 통성 헤테로크로마틴으로 포장되어 침묵되고, 다른쪽 X염색체는 에우크로마틴으로 포장되어 발현된다.
헤테로크로마틴의 확산을 조절하는 것으로 보이는 분자 성분 중에는 폴리콤 그룹 단백질과 Xist와 같은 비코드 유전자가 있다.이러한 확산의 메커니즘은 여전히 [18]논란의 대상이다.폴리콤 억제 복합체 PRC1과 PRC2는 크로마틴 압축과 유전자 발현을 조절하고 발달 과정에서 기본적인 역할을 한다.PRC 매개 후생유전자 이상은 게놈 불안정성 및 악성종양과 관련이 있으며 DNA 손상 반응, DNA 복구 [19]및 복제의 충실도에서 역할을 한다.
효모헤테로크로마틴
사카로미세스 세레비시아에(Saccharomyes cerevisiae) 또는 발아 효모는 진핵생물 모델이며 그 헤테로크로마틴은 철저히 정의되어 있다.비록 그것의 게놈의 대부분이 에크로마틴으로 특징지어질 수 있지만, 세레비시아에는 매우 적게 전사되는 DNA 영역이 있습니다.이러한 위치는 이른바 무성 결합형 위치(HML 및 HMR), rDNA(리보솜 RNA 인코딩) 및 서브 텔로미어 영역입니다.핵분열 효모(시조당류 폼베)는 동원체에서 헤테로크로마틴 형성을 위한 또 다른 메커니즘을 사용한다.이 위치에서 유전자 소음은 RNAi 경로의 구성요소에 따라 달라집니다.이중가닥 RNA는 일련의 단계를 통해 영역의 침묵을 초래하는 것으로 여겨진다.
헤테로크로마틴 조립체의 개시, 전파 및 유지에 관여하는 RNAi 기계의 일부인 RITS 착체와 RNA 지향 RNA 중합효소 착체(RDRC)의 2개의 RNAi 착체.이 두 가지 복합체는 헤테로크로마틴 조립 부위에서 siRNA 의존적인 방식으로 염색체에 국재한다.RNA 중합효소 II는 RITS, RDRC 및 헤테로크로마틴 [20][21]조립에 필요한 다른 복합체를 모집하기 위한 플랫폼 역할을 하는 전사체를 합성한다.RNAi와 엑소좀 의존성 RNA 분해 과정은 모두 헤테로크로마 유전자 소음화에 기여한다.이러한 분열당류 폼브의 메커니즘은 다른 [22]진핵생물에서 발생할 수 있다.RevCen이라고 불리는 큰 RNA 구조는 또한 일부 핵분열 [23]효모에서 헤테로크로마틴 형성을 중재하기 위한 siRNA의 생산과 관련이 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Volpe TA, Kidner C, Hall IM, Teng G, Grewal SI, Martienssen RA (September 2002). "Regulation of heterochromatic silencing and histone H3 lysine-9 methylation by RNAi". Science. 297 (5588): 1833–7. Bibcode:2002Sci...297.1833V. doi:10.1126/science.1074973. PMID 12193640. S2CID 2613813.
- ^ "What is the current evidence showing active transcription withinin..." www.researchgate.net. Retrieved 2016-04-30.
- ^ Ou HD, Phan S, Deerinck TJ, Thor A, Ellisman MH, O'Shea CC (July 2017). "ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells". Science. 357 (6349): eaag0025. doi:10.1126/science.aag0025. PMC 5646685. PMID 28751582.
- ^ Oberdoerffer P, Sinclair DA (September 2007). "The role of nuclear architecture in genomic instability and ageing". Nature Reviews. Molecular Cell Biology. 8 (9): 692–702. doi:10.1038/nrm2238. PMID 17700626. S2CID 15674132.
- ^ Rosenfeld JA, Wang Z, Schones DE, Zhao K, DeSalle R, Zhang MQ (March 2009). "Determination of enriched histone modifications in non-genic portions of the human genome". BMC Genomics. 10 (1): 143. doi:10.1186/1471-2164-10-143. PMC 2667539. PMID 19335899.
- ^ Nicetto D, Donahue G, Jain T, Peng T, Sidoli S, Sheng L, et al. (January 2019). "H3K9me3-heterochromatin loss at protein-coding genes enables developmental lineage specification". Science. 363 (6424): 294–297. Bibcode:2019Sci...363..294N. doi:10.1126/science.aau0583. PMC 6664818. PMID 30606806.
- ^ Elgin, S.C. (1996). "Heterochromatin and gene regulation in Drosophila". Current Opinion in Genetics & Development. 6 (2): 193–202. doi:10.1016/S0959-437X(96)80050-5. ISSN 0959-437X. PMID 8722176.
- ^ van Steensel B (May 2011). "Chromatin: constructing the big picture". The EMBO Journal. 30 (10): 1885–95. doi:10.1038/emboj.2011.135. PMC 3098493. PMID 21527910.
- ^ Roudier F, Ahmed I, Bérard C, Sarazin A, Mary-Huard T, Cortijo S, et al. (May 2011). "Integrative epigenomic mapping defines four main chromatin states in Arabidopsis". The EMBO Journal. 30 (10): 1928–38. doi:10.1038/emboj.2011.103. PMC 3098477. PMID 21487388.
- ^ Lohe AR, Hilliker AJ, Roberts PA (August 1993). "Mapping simple repeated DNA sequences in heterochromatin of Drosophila melanogaster". Genetics. 134 (4): 1149–74. doi:10.1093/genetics/134.4.1149. PMC 1205583. PMID 8375654.
- ^ Lu BY, Emtage PC, Duyf BJ, Hilliker AJ, Eissenberg JC (June 2000). "Heterochromatin protein 1 is required for the normal expression of two heterochromatin genes in Drosophila". Genetics. 155 (2): 699–708. doi:10.1093/genetics/155.2.699. PMC 1461102. PMID 10835392.
- ^ Grewal SI, Jia S (January 2007). "Heterochromatin revisited". Nature Reviews. Genetics. 8 (1): 35–46. doi:10.1038/nrg2008. PMID 17173056. S2CID 31811880.
An up-to-date account of the current understanding of repetitive DNA, which usually doesn't contain genetic information. If evolution makes sense only in the context of the regulatory control of genes, we propose that heterochromatin, which is the main form of chromatin in higher eukaryotes, is positioned to be a deeply effective target for evolutionary change. Future investigations into assembly, maintenance and the many other functions of heterochromatin will shed light on the processes of gene and chromosome regulation.
- ^ Fisher AG, Merkenschlager M (April 2002). "Gene silencing, cell fate and nuclear organisation". Current Opinion in Genetics & Development. 12 (2): 193–7. doi:10.1016/S0959-437X(02)00286-1. PMID 11893493.
- ^ Zhimulev, I.F.; et al. (December 1986). "Cytogenetic and molecular aspects of position effect variegation in Drosophila melanogaster". Chromosoma. 94 (6): 492–504. doi:10.1007/BF00292759. ISSN 1432-0886. S2CID 24439936.
- ^ Burgess-Beusse B, Farrell C, Gaszner M, Litt M, Mutskov V, Recillas-Targa F, et al. (December 2002). "The insulation of genes from external enhancers and silencing chromatin". Proceedings of the National Academy of Sciences of the United States of America. 99 Suppl 4 (Suppl 4): 16433–7. Bibcode:2002PNAS...9916433B. doi:10.1073/pnas.162342499. PMC 139905. PMID 12154228.
- ^ Allis CD, Grewal SI (August 2001). "Transitions in distinct histone H3 methylation patterns at the heterochromatin domain boundaries". Science. 293 (5532): 1150–5. doi:10.1126/science.1064150. PMID 11498594. S2CID 26350729.
- ^ Donze D, Kamakaka RT (February 2001). "RNA polymerase III and RNA polymerase II promoter complexes are heterochromatin barriers in Saccharomyces cerevisiae". The EMBO Journal. 20 (3): 520–31. doi:10.1093/emboj/20.3.520. PMC 133458. PMID 11157758.
- ^ Talbert PB, Henikoff S (October 2006). "Spreading of silent chromatin: inaction at a distance". Nature Reviews. Genetics. 7 (10): 793–803. doi:10.1038/nrg1920. PMID 16983375. S2CID 1671107.
- ^ Veneti Z, Gkouskou KK, Eliopoulos AG (July 2017). "Polycomb Repressor Complex 2 in Genomic Instability and Cancer". International Journal of Molecular Sciences. 18 (8): 1657. doi:10.3390/ijms18081657. PMC 5578047. PMID 28758948.
- ^ Kato H, Goto DB, Martienssen RA, Urano T, Furukawa K, Murakami Y (July 2005). "RNA polymerase II is required for RNAi-dependent heterochromatin assembly". Science. 309 (5733): 467–9. Bibcode:2005Sci...309..467K. doi:10.1126/science.1114955. PMID 15947136. S2CID 22636283.
- ^ Djupedal I, Portoso M, Spåhr H, Bonilla C, Gustafsson CM, Allshire RC, Ekwall K (October 2005). "RNA Pol II subunit Rpb7 promotes centromeric transcription and RNAi-directed chromatin silencing". Genes & Development. 19 (19): 2301–6. doi:10.1101/gad.344205. PMC 1240039. PMID 16204182.
- ^ Vavasseur; et al. (2008). "Heterochromatin Assembly and Transcriptional Gene Silencing under the Control of Nuclear RNAi: Lessons from Fission Yeast". RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Caister Academic Press. ISBN 978-1-904455-25-7.
- ^ Djupedal I, Kos-Braun IC, Mosher RA, Söderholm N, Simmer F, Hardcastle TJ, et al. (December 2009). "Analysis of small RNA in fission yeast; centromeric siRNAs are potentially generated through a structured RNA". The EMBO Journal. 28 (24): 3832–44. doi:10.1038/emboj.2009.351. PMC 2797062. PMID 19942857.
외부 링크
- 조직 이미지: 20102loa – 보스턴 대학교 조직학 학습 시스템
- Avramova ZV (May 2002). "Heterochromatin in animals and plants. Similarities and differences". Plant Physiology. 129 (1): 40–9. doi:10.1104/pp.010981. PMC 1540225. PMID 12011336.
- Caron H, van Schaik B, van der Mee M, Baas F, Riggins G, van Sluis P, et al. (February 2001). "The human transcriptome map: clustering of highly expressed genes in chromosomal domains". Science. 291 (5507): 1289–92. Bibcode:2001Sci...291.1289C. doi:10.1126/science.1056794. PMID 11181992.
- Cha, Ariana Eunjung; Bernstein, Lenny (April 30, 2015). "Scientists discover an important new driver of aging". New York Times. Retrieved 4 May 2015.