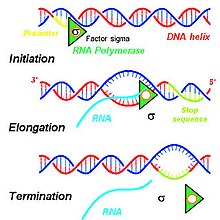
주요 성적표
Primary transcript1차 전사물은 DNA의 전사에 의해 합성되어 mRNA, tRNA 및 rRNA와 같은 다양한 성숙한 RNA 생성물을 생성하도록 가공된 단일사슬 리보핵산(RNA) 생성물이다.mRNA로 지정된 주요 트랜스크립트는 번역 준비를 위해 수정됩니다.예를 들어 전구체 mRNA(pre-mRNA)는 처리 후 메신저 RNA(mRNA)가 되는 1차 전사체의 일종이다.
전사에 의해 세포핵 내의 DNA 템플릿에서 pre-mRNA를 합성한다.프리mRNA는 이종핵RNA(hnRNA)의 대부분을 포함한다.pre-mRNA가 완전히 처리되면, 그것은 "성숙한 메신저 RNA" 또는 간단히 "메신저 RNA"라고 불립니다.hnRNA라는 용어는 종종 pre-mRNA의 동의어로 사용되지만, 엄밀한 의미에서 hnRNA는 세포질 mRNA로 끝나지 않는 핵 RNA 전사를 포함할 수 있다.
1차 성적표 작성에는 몇 가지 단계가 있습니다.이 모든 단계는 진핵생물 핵에서 DNA 전사를 시작하고 완료하기 위한 일련의 상호작용을 포함한다.특정 인자는 1차 전사 생성을 조절하는 전사의 활성화 및 저지에 중요한 역할을 합니다.문자 변환은 몇 가지 프로세스에 의해 추가로 수정된 기본 문자 변환을 생성합니다.이러한 과정에는 5' 캡, 3' 폴리아데닐화 및 대체 스플라이싱이 포함됩니다.특히, 대체 스플라이싱은 세포에서 발견되는 mRNA의 다양성에 직접적으로 기여한다.1차 성적표의 수정은 이러한 성적표의 역할과 중요성에 대한 더 많은 지식을 추구하는 연구에서 더 많이 연구되었다.1차 전사물에 대한 분자 변화와 전사 전후의 과정을 바탕으로 한 실험 연구는 1차 전사물과 관련된 질병에 대한 이해를 증진시켰다.
생산.
1차 전사의 생성에 기여하는 단계는 세포의 핵 내에서 DNA의 전사를 시작하는 일련의 분자 상호작용을 포함한다.특정 세포의 요구에 근거하여 특정 DNA 배열을 전사하여 세포 사용을 위한 기능성 단백질로 번역되는 다양한 RNA 생성물을 생성한다.세포의 핵에서 전사 과정을 시작하기 위해, DNA 이중 나선은 풀리고 DNA의 호환 가능한 핵산을 연결하는 수소 결합은 두 개의 연결되지 않은 단일 DNA [1]가닥을 생성하기 위해 파괴된다.DNA 템플릿의 1가닥은 1차 전사 mRNA의 전사에 사용된다.이 DNA 가닥은 [2]DNA의 프로모터 영역에서 RNA 중합효소에 의해 결합됩니다.
진핵생물에서, 세 가지 종류의 RNA, 즉 rNA, tRNA, mRNA는 세 가지 다른 RNA 중합효소의 활성에 기초하여 생산되는 반면, 원핵생물에서는 모든 종류의 RNA [3]분자를 만들기 위해 오직 하나의 RNA 중합효소만이 존재한다.RNA중합 효소 II생물은 안티 센스 DNA는 5'에 템플릿 3의 방향, 새로 합성된 1차전 사물 DNA.[1]RNA중합 효소 II의 1차전 사물 4사양의 집합을 사용하여 건설하는 안티 센스 가닥을 보완하기 위한 것은 1차전 사물, 성적 증명서 mRNA로 가공될 운명, transcribes.한다면성장 중인 MRNA의 [1]3' 말단에서 3' 하이드록실기에 연속적으로 첨가되는 ic 리보뉴클레오시드 1인산(AMP), 시티딘 1인산(CMP), 구아노신 1인산(GMP) 및 우리딘 1인산(UMP) 잔류물.
RNA 중합효소 II에 의해 생성된 1차 전사물에 대한 연구는 평균 1차 전사물의 길이가 7,000 뉴클레오티드이며,[2] 일부는 길이가 20,000 뉴클레오티드까지 자란다는 것을 보여준다.1차 전사물에 엑손과 인트론 배열이 모두 포함된 것은 단백질로 변환될 준비가 된 더 큰 1차 전사물과 더 작고 성숙한 mRNA 사이의 크기 차이를 설명한다.
규정
많은 인자가 전사의 활성화 및 저지에 기여하고, 따라서 주어진 DNA 템플릿에서 1차 전사물의 생성을 조절한다.
1차 전사를 생성하기 위한 RNA 중합효소 활성의 활성화는 종종 증강제라고 불리는 DNA 배열에 의해 제어된다.전사인자, 전사를 활성화하거나 억제하기 위해 DNA 원소에 결합하는 단백질은 강화제에 결합하고 뉴클레오솜 성분을 변화시키는 효소를 모집하여 DNA가 RNA 중합효소에 어느 정도 접근할 수 있도록 합니다.인핸서 DNA 영역에 결합하는 활성화 또는 억제 전사인자의 독특한 조합은 인핸서가 전사를 위해 상호작용하는 유전자가 활성화되는지 여부를 결정합니다.[4]전사의 활성화는 다양한 전사 인자로 구성된 전사 신장 복합체가 RNA 중합효소를 [4]촉진제 영역에 연결하는 매개체 복합체에서 분리하도록 유도할 수 있는지 여부에 따라 달라집니다.
RNA 중합효소 활성의 억제는 또한 소음기라고 불리는 DNA 배열에 의해 조절될 수 있다.증강제처럼 소음제는 그들이 조절하는 유전자보다 더 위쪽 또는 아래쪽 위치에 있을 수 있습니다.이러한 DNA 배열은 RNA 중합효소를 활성화하는 데 필요한 시작 복합체의 불안정화에 기여하는 인자와 결합하고,[5] 따라서 전사를 억제합니다.
전사 인자에 의한 히스톤 변형은 RNA 중합효소에 의한 전사에 대한 또 다른 핵심 조절 인자이다.일반적으로 히스톤 아세틸화를 일으키는 인자는 전사를 활성화하는 반면, 히스톤 탈아세틸화를 일으키는 인자는 [6]전사를 억제한다.히스톤의 아세틸화는 뉴클레오솜 내의 음의 성분들 사이에서 거부감을 유발하여 RNA 중합효소 접근을 가능하게 한다.히스톤의 탈아세틸화는 단단히 감긴 뉴클레오솜을 안정화시켜 RNA 중합효소 접근을 억제한다.히스톤의 아세틸화 패턴과 더불어 DNA의 프로모터 영역에서의 메틸화 패턴은 주어진 템플릿에 대한 RNA 중합효소 접근을 조절할 수 있다.RNA 중합효소는 표적 유전자의 프로모터 영역이 특정 메틸화 사이토신(전사 활성화 인자의 결합을 방해하고 RNA 중합효소에 대한 접근을 배제하고 프로를 막는 다른 효소)을 포함하는 경우 종종 1차 전사를 합성할 수 없다.1차 [4]성적증명서 복제.
R루프
R-루프는 전사 중에 형성됩니다.R-루프는 DNA-RNA 하이브리드 영역과 관련된 비템플릿 단일사슬 DNA를 포함하는 3가닥 핵산 구조이다.활발하게 전사된 DNA 영역은 종종 DNA 손상에 취약한 R-루프를 형성합니다.인트론은 고도로 발현된 효모 [7]유전자의 R-루프 형성과 DNA 손상을 감소시킨다.
RNA 처리
유전자 발현에서 고도로 조절된 단계인 전사는 1차 전사를 생성한다.그러나 전사는 RNA의 [8]기능적 형태를 생성하는 많은 수정이 뒤따를 첫 번째 단계일 뿐이다.달리 명시되지 않은 경우, 새로 합성된 1차 전사물은 mRNA, tRNA 및 rRNA와 같은 다른 단백질과 RNA를 생성하기 위해 성숙하고 기능적인 형태로 변환되기 위해 여러 가지 방법으로 수정된다.
처리.
기본적인 1차 전사 수정 과정은 진핵세포와 원핵세포 모두에서 tRNA와 rRNA에 대해 유사하다.한편, 1차 전사 처리는 원핵세포와 진핵세포의 [8]mRNA에서 다르다.예를 들어, 일부 원핵 세균 mRNA는 전사를 통해 생산되는 동시에 단백질 합성을 위한 템플릿 역할을 한다.또는 진핵세포의 전mRNA는 성숙한 형태가 번역되는 [8]핵에서 세포질로 이동하기 전에 광범위한 변형을 겪는다.이러한 변경은 다양한 유형의 제품을 번역할 수 있는 다양한 유형의 인코딩된 메시지의 원인이 됩니다.또한 1차 전사처리는 유전자 발현 제어 및 mRNA의 분해율 조절기구를 제공한다.진핵세포에서 pre-mRNA의 처리에는 5' caping, 3' polyadenylation, 그리고 대체 스플라이싱이 포함된다.
5인치 캡핑
진핵생물에서 전사가 시작된 직후, mRNA의 5' 전단은 5' [8]캡으로도 알려진 7-메틸구아노신 캡의 첨가에 의해 수정된다.5' 캡 수정은 역방향으로 사전 mRNA의 5' 말단 뉴클레오티드에 GTP를 추가한 후 G [8]잔기에 메틸기를 추가함으로써 시작된다.5' 캡은 번역 [8]중 mRNA와 리보솜을 정렬하는 역할을 하므로 기능성 mRNA 생산에 필수적이다.
폴리아데닐화
진핵생물에서 폴리아데닐화는 폴리A 꼬리라고 불리는 구조가 [8]추가되는 동안 사전 mRNA를 더욱 수정한다.폴리A 꼬리(길이 약 200 뉴클레오티드)의 첨가를 신호로 하는 단백질 그룹에 의해, 복수의 RNA 배열 요소를 포함한 폴리아데닐화 신호가 검출된다.폴리아데닐화 반응은 전사의 종료를 알리는 신호를 제공하며, 이 반응은 폴리 A 꼬리 [8]위치로부터 약 수백 개의 뉴클레오티드로 끝납니다.
대체 스플라이싱
진핵생물 사전 mRNA는 작은 핵 [9][10]리보핵단백질로 이루어진 스플라이소좀에 의해 그들의 인트론을 분리한다.
복잡한 진핵 세포에서, 하나의 1차 전사물은 대체 스플라이싱으로 인해 다량의 성숙한 mRNA를 준비할 수 있다.대체 스플라이싱은 성숙한 각 mRNA가 단백질의 다수를 부호화할 수 있도록 조절된다.
유전자 발현에서의 대체 접합의 효과는 게놈에 고정된 수의 유전자를 가지고 있지만 훨씬 더 많은 다른 유전자 [8]생성물을 생산하는 복잡한 진핵 생물에서 볼 수 있습니다.대부분의 진핵생물 사전 mRNA 전사물은 다중 인트론 및 엑손(exon)을 포함한다.사전 mRNA에서 5'와 3' 스플라이스 부위의 다양한 가능한 조합은 성숙한 mRNA에서 인트론이 제거되는 동안 엑손의 다른 절제 및 조합을 초래할 수 있다.따라서 다양한 종류의 성숙한 mRNA가 [8]생성된다.대체 스플라이싱은 스플라이싱이라고 불리는 큰 단백질 복합체에서 일어난다.대체 스플라이싱은 유전자 [8]발현에 있어 조직 특이적이고 발달적인 조절에 중요하다.대체 스플라이싱은 염색체 전위와 같은 돌연변이를 포함한 다양한 요인에 의해 영향을 받을 수 있다.
원핵생물에서 스플라이싱은 자기촉매분열 또는 내용해분열로 이루어진다.단백질이 관여하지 않는 자가 촉매 분열은 보통 rRNA를 코드하는 부분에 저장되는 반면, 내용해 분열은 tRNA 전구체에 해당한다.
실험
뉴욕 버팔로의 로즈웰 파크 종합 암 센터 실험 치료학과(당시 로즈웰 파크 기념 연구소라고 알려져 있음)의 신디 L. 윌스와 브루스 J. 돌닉의 연구는 위스콘신 대학의 세포와 분자 생물학 프로그램에서 이해되었다.주요 녹취록과 관련이 있습니다.연구자들은 암 치료에 사용되는 것으로 알려진 5-플루오로라실(FURA)이 메토트렉세이트 내성 KB 세포에서 디히드로폴산 환원효소(DHFR) 사전 mRNA 처리 및/또는 핵 mRNA 안정성을 억제하거나 차단하는지 여부를 파악하고자 했다.FURA에 대한 장기 피폭은 처리의 일부로 보통 배열에서 제외되는 사전 mRNA 섹션인 특정 인트론을 포함하는 DHFR 사전 mRNA 수준에 영향을 미치지 않았다.그러나 1.0μM FURA에 피폭된 세포에서는 총 DHFR mRNA 수치가 2배 감소하였다.FURA에 피폭된 세포에서 관찰된 총 DHFR mRNA 또는 사전 mRNA의 50%가 붕괴되는 데 걸리는 시간을 의미하는 반감기에는 유의미한 변화가 없었다.그리고 핵/세포질 RNA 라벨링 실험은 FURA로 처리된 세포에서 핵 DHFR RNA가 세포질 DHFR mRNA로 변화하는 속도가 감소했음을 보여주었다.이러한 결과는 FURA가 mRNA 전구체 처리에 도움이 될 수 있고 핵 DHFR mRNA의 [11]안정성에 영향을 미칠 수 있다는 추가 증거를 제공한다.
매사추세츠 캠브리지에 있는 매사추세츠공과대학(MIT)의 생물학과 주디스 렝기엘과 셸던 펜만은 두 개의 날개를 가진 곤충의 유전자에 관련된 한 종류의 주요 전사물에 관한 기사를 썼다.드로소필라와 에데스.이 기사는 연구자들이 두 종류의 곤충에서 hnRNA, 즉 기본적으로 mRNA 이전의 1차 전사물을 어떻게 봤는지를 설명하고 있다.Drosophila melanogaster와 Aedes albopictus의 hnRNA 전사 크기와 세포주 또는 단일 세포에서 mRNA로 변환되는 hnRNA의 비율을 비교했다.두 곤충 모두 쌍떡잎식물이지만, Aedes는 Drosophila보다 더 큰 게놈을 가지고 있다.이것은 Aedes가 더 많은 유전자를 가지고 있다는 것을 의미한다.두 세포주가 유사한 조건에서 성장하여 동일한 크기와 배열 복잡도의 성숙 또는 가공된 mRNA를 생성했음에도 불구하고 Aedes 라인은 Drosophila 라인보다 더 큰 hnRNA를 생성한다.이러한 데이터는 게놈 크기가 증가함에 따라 hnRNA의 크기가 증가한다는 것을 시사하는데,[12] 이는 Aedes에 의해 명백하게 나타난다.
프라하의 체코 과학 아카데미 실험의학연구소 세포생물학과 이보 멜카코바, 스테판카 멜카코바, 보이텍 콥스키, 자로미라 베세로바, 이반 라스카는 핵 반점이 전-RNA에 미치는 영향을 연구했다.핵 반점(스펙클)은 세포핵의 일부이며 mRNA 처리에 관여하는 것으로 알려진 스플라이싱 인자로 농축된다.핵 반점은 이러한 스플라이싱 인자의 저장 장소로서 인접한 활성 유전자에 도움이 되는 것으로 나타났습니다.이 연구에서 연구자들은 자궁경부암에 걸린 사람의 세포에서 파생되어 실험에 유용한 것으로 입증된 Hela 세포에서 mRNA 이전의 스플라이소좀의 첫 번째 그룹이 이 반점에서 나온다는 것을 보여주었다.연구자들은 다른 그룹을 만들기 위해 스플라이싱 인자 결합을 가진 스플라이싱 수용과 돌연변이 아데노바이러스 사전 mRNA의 미세 주입을 사용한 후 그들이 많이 존재하는 부위를 추적했다.스플라이세오솜 수용 전 mRNA는 빠르게 스펙클에 표적화 되었지만, 표적화는 온도에 의존하는 것으로 밝혀졌다.mRNA의 폴리피리미딘 트랙터 배열은 스플라이세오솜 그룹의 구성을 촉진하고 표적화에 필요하지만, 그것만으로는 충분하지 않았다.다운스트림 플랭크 시퀀스는 반점에서 돌연변이 프리 mRNA의 표적에 특히 중요했다.보조 실험에서, 반센스 디옥시올리고리보뉴클레오티드(특정 배열에 대한 DNA 및 RNA의 상보적 배열)의 미세 주입 후에 반점의 거동이 뒤따랐으며, 이 경우, snRNA의 특정 배열은 사전 MRNA의 처리에도 도움이 되는 것으로 알려져 있다.이러한 조건에서 스플라이세오솜 그룹은 내인성 사전 mRNA에서 형성되었다.연구자들은 미세 주입된 사전 mRNA 위의 스플라이세오솜 그룹이 반점 안에서 형성된다는 결론을 내렸다.pre-mRNA 타겟팅 및 specle에서의 축적은 pre-mRNA에 대한 스플라이싱 인자의 부하에 의한 것으로,[13] spliceosome 그룹은 관찰된 specle 패턴을 발생시켰다.
관련 질병
연구는 또한 주요 기록물 내의 변화와 관련된 특정 질병에 대한 더 많은 지식을 이끌어냈다.한 연구는 에스트로겐 수용체와 차등 스플라이싱을 포함했다.이탈리아 제노바 국립암연구소 분자종양학 연구소의 파올라 페로, 알레산드라 포를라니, 마르코 무셀리, 울리히 프페퍼의 "인간 에스트로겐 수용체 알파 1차 성분의 대체 스플라이싱: 엑손 건너뛰기 메커니즘"이라는 제목의 논문은 D에 있는 1785개의 뉴클레오타이드가 있다고 설명한다.에스트로겐 수용체 알파(ER-alpha)를 코드하는 NA는 1차 전사물에 300,000개 이상의 뉴클레오티드가 있는 영역에 퍼져 있습니다.이 프리mRNA의 스플라이싱은 단백질 코딩에 필요한 하나 이상의 엑손 또는 영역이 결여된 변형 또는 다른 종류의 mRNA로 이어진다.이러한 변종들은 유방암의 [14]진행과 관련이 있다.레트로바이러스의 라이프 사이클에서 프로바이러스 DNA는 감염되는 세포의 DNA 전사에 포함된다.레트로바이러스는 이 DNA가 영향을 미치는 숙주의 DNA 안에 통합될 수 있도록 그들의 mRNA를 DNA로 바꿀 필요가 있기 때문에, 그 DNA 템플릿의 형성은 레트로바이러스 복제를 위한 중요한 단계이다.세포 유형, 세포의 분화 또는 변화된 상태, 세포의 생리적인 상태에 따라 전사에 필요한 특정 인자의 가용성과 활동이 크게 변화한다.이러한 변수들은 광범위한 바이러스 유전자 발현을 생성한다.예를 들어, 조류 또는 쥐 백혈병 바이러스의 감염성 바이러스(ASLV 또는 MLV)를 능동적으로 생성하는 조직 배양 세포는 세포 내 mRNA의 5~10%가 바이러스 기원이 될 수 있을 정도로 높은 수준의 바이러스 RNA를 포함하고 있다.이것은 이러한 레트로바이러스에 의해 생성된 1차 전사물이 항상 단백질 생산의 정상적인 경로를 따라 증식하고 [15]확장하기 위해 DNA로 다시 변환되는 것은 아니라는 것을 보여준다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c T. Strachan; Andrew P. Read (January 2004). Human Molecular Genetics 3. Garland Science. pp. 16–17. ISBN 978-0-8153-4184-0.
- ^ a b Alberts B. "Molecular Biology of the Cell". NCBI (3rd ed.). New York: Garland Science.
- ^ Griffiths AJ. "An Introduction to Genetic Analysis". NCBI. New York: W.H. Freeman.
- ^ a b c Scott F. Gilbert (15 July 2013). Developmental Biology. Sinauer Associates, Incorporated. pp. 38–39, 50. ISBN 978-1-60535-173-5.
- ^ Brown TA. "Genomes" (2nd ed.). Oxford: Wiley-Liss.
- ^ Harvey Lodish (2008). Molecular Cell Biology. W. H. Freeman. pp. 303–306. ISBN 978-0-7167-7601-7.
- ^ Bonnet A, Grosso AR, Elkaoutari A, Coleno E, Presle A, Sridhara SC, Janbon G, Géli V, de Almeida SF, Palancade B (August 2017). "Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability". Molecular Cell. 67 (4): 608–621.e6. doi:10.1016/j.molcel.2017.07.002. PMID 28757210.
- ^ a b c d e f g h i j k Cooper GM. "The Cell: A Molecular Approach" (2nd ed.). Sunderland (MA): Sinauer Associates; 2000.
- ^ 위버, 로버트 F. (2005)분자생물학, 페이지 432-448맥그로힐, 뉴욕, ISBN 0-07-284611-9.
- ^ Wahl MC, Will CL, Lührmann R (February 2009). "The spliceosome: design principles of a dynamic RNP machine". Cell. 136 (4): 701–18. doi:10.1016/j.cell.2009.02.009. hdl:11858/00-001M-0000-000F-9EAB-8. PMID 19239890.
- ^ Will CL, Dolnick BJ (December 1989). "5-Fluorouracil inhibits dihydrofolate reductase precursor mRNA processing and/or nuclear mRNA stability in methotrexate-resistant KB cells". The Journal of Biological Chemistry. 264 (35): 21413–21. PMID 2592384.
- ^ Lengyel J, Penman S (July 1975). "hnRNA size and processing as related to different DNA content in two dipterans: Drosophila and Aedes". Cell. 5 (3): 281–90. doi:10.1016/0092-8674(75)90103-8. PMID 807333.
- ^ Melcák I, Melcáková S, Kopský V, Vecerová J, Raska I (February 2001). "Prespliceosomal assembly on microinjected precursor mRNA takes place in nuclear speckles". Molecular Biology of the Cell. 12 (2): 393–406. CiteSeerX 10.1.1.324.8865. doi:10.1091/mbc.12.2.393. PMC 30951. PMID 11179423.
- ^ Ferro P, Forlani A, Muselli M, Pfeffer U (September 2003). "Alternative splicing of the human estrogen receptor alpha primary transcript: mechanisms of exon skipping". International Journal of Molecular Medicine. 12 (3): 355–63. PMID 12883652.
- ^ Coup JM, Hughes SH, Varmus HE 편집장레트로바이러스.콜드 스프링 하버(NY): 콜드 스프링 하버 연구소 프레스; 1997.입수처: https://www.ncbi.nlm.nih.gov/books/NBK19441/