CpG 사이트
CpG siteCpG 사이트 또는 CG 사이트는 5' → 3' 방향으로 베이스의 선형 순서에서 구아닌 뉴클레오티드가 뒤따르는 DNA 영역이다. CpG 사이트는 CpG 섬(또는 CG 섬)이라고 불리는 게놈 지역에서 높은 빈도로 발생한다.
CpG 디뉴클레오티드의 시토신은 메틸화하여 5메틸시토신을 형성할 수 있다. 메틸 그룹을 첨가하는 효소를 DNA 메틸전달효소라고 한다. 포유류에서는 CpG 시토신의 70~80%가 메틸화된다.[1] 유전자 내 시토신을 메틸화하면 후생유전학이라고 불리는 유전자 조절을 연구하는 더 큰 과학 분야의 한 부분인 메커니즘인 그 표현이 바뀔 수 있다. 메틸화 된 시토신은 종종 흉선에 변이된다.
인간의 경우, 유전자(최소 발기인)의 전사 시작 지점 근처에 위치한 발기인 중 약 70%가 CpG 섬을 포함하고 있다.[2][3]
CpG 특성
정의
CpG는 5'—C—인산염—G—3'의 속칭이다. 즉, 인산염과 구아닌은 한 개의 인산염 그룹으로 분리된다. 인산염은 DNA에서 두 개의 뉴클레오시드를 서로 연결한다. CpG 표기법은 이 단일 가닥 선형 시퀀스를 이중 가닥 시퀀스에 대한 시토신과 구아닌의 CG 베이스 페어링과 구별하기 위해 사용된다. 따라서 CpG 표기법은 구아닌 베이스에 시토신이 5 prime인 것으로 해석된다. CpG는 GpC와 혼동해서는 안 되는데,후자는구아닌이단일 가닥뒤따른다는 뜻이다 시토신이 5→ 3'방향으로 시퀀스의.
높은 돌연변이율로 인한 과소표현
CpG 디뉴클레오티드(dinucleotide)는 척추동물 게놈의 수열에서 무작위적인 우연으로 인해 예상되는 것보다 훨씬 낮은 빈도로 발생하는 것으로 오랫동안 관찰되어 왔다. 예를 들어 GC 함량이 42%인 인간 게놈에서 구아닌에 이어 시토신(cytosine)으로 구성된 뉴클레오티드의 쌍은 0.0.= 0.의 시간 동안 할 것으로 예상된다.[4] 인간 게놈에서 CpG 디뉴클레오티드 발생 빈도는 예상 빈도의 5분의 1에도 미치지 못한다.[5]
이러한 과소 표현은 메틸화 CpG 사이트의 높은 돌연변이 비율의 결과로서, 즉 메틸화 시토신의 자연발생적인 디아밍은 티민(Tymmine)을 초래하고, 그로 인한 G:T 불일치 베이스는 종종 A에게 부적절하게 해결된다.T; 반면 비메틸화 시토신을 탈염하면 요람이 생기는데, 요람은 외국 기지로써 염기 분리 수리 메커니즘에 의해 빠르게 시토신으로 대체된다. 메틸화 CpG 현장의 C to T 전환율은 미메틸화 현장보다 최대 10배 높다.[6][7][8][9]
게놈 분포
| CpG 사이트 | GPC 사이트 |
|---|---|
 |  |
| 인간 APRT 유전자에서 CpG 사이트(왼쪽: 빨간색)와 GpC 사이트(오른쪽: 녹색)의 분포. CpG는 CpG섬을 형성하는 유전자의 상류지역이 더 풍부한데 비해 GpC는 더 고르게 분포한다. APRT 유전자의 5 exon은 (파란색)으로 표시되며, 시작(ATG)과 중지(TGA) 코돈은 강조(볼드 블루)로 표시된다. | |
CpG 디뉴클레오티드는 CpG 섬에서 자주 발생한다(아래 CpG 섬 정의 참조). 인간 게놈에는 2만8890개의 CpG 섬이 있다(CpG 섬이 반복 시퀀스에 포함되는 경우 50,267개).[10] 이는 벤터 외 게놈 서열에는 매우 유사한 반복 원소들의 내부와 센트롬 근처의 극히 밀도 있는 반복 영역이 포함되지 않았기 때문에 벤터 외 연구진이 발견한 28,519 CpG 섬들과 일치한다.[11][12] CpG 섬은 여러 CpG 디뉴클레오티드 시퀀스를 포함하고 있기 때문에 인간 게놈에는 2,000만 CpG 디뉴클레오티드(dinucleotide)가 넘는 것으로 보인다.
CpG 섬
CpG섬(또는 CG섬)은 CpG 사이트 빈도가 높은 지역이다. CpG 섬에 대한 객관적 정의는 제한적이지만, 일반적인 공식 정의는 최소 200bp, GC 백분율이 50% 이상, 관측 대 예상 CpG 비율이 60% 이상인 지역이다. 어디가 관찰한(C의 수 p G신규){\displaystyle({\text{}의 수}CpGs)}:와 순서{시퀀스의\displaystyle({\text{}의 수}C*{\text{}의 수}G)/{\text{길이}의(G의 C∗ 번호의 수)/길이 예상대로 계산한다"CpG비율 observed-to-expected"}}[13]또는 파생될 수 있다. ( + )/ 2) / [14]
포유류 게놈의 많은 유전자는 유전자[15](프로모터 지역)의 시작과 연관된 CpG 섬을 가지고 있다. 이 때문에 CpG섬의 존재는 유전자의 예측과 주석을 돕는 데 사용된다.
포유류 게놈에서 CpG 섬은 일반적으로 300~3,000개의 염기쌍이며, 포유류 유전자의 약 40%에서 발견되었다.[16] 인간의 60% 이상의 유전자와 거의 모든 집기 유전자는 CpG 섬에 그들의 촉진제를 내장하고 있다.[17] GC 2-뉴클레오티드 시퀀스의 빈도를 고려할 때,[14] CpG 디뉴클레오티드 수는 예상보다 훨씬 낮다.
2002년 연구에서는 CpG 섬 예측 규칙을 수정하여 Alu 반복과 같은 GC가 풍부한 유전체 시퀀스를 제외시켰다. 인간 염색체 21과 22의 전체 염기서열에 대한 광범위한 조사를 바탕으로, 500bp 이상의 DNA 영역은 GC 함량이 55% 이상이고 관측된 예상 CpG 비율이 [18]65% 이상인 경우 5'유전자 영역과 연관된 "진정한" CpG 섬일 가능성이 더 높은 것으로 밝혀졌다.
CpG 섬은 CpG 디뉴클레오티드 함량이 통계적으로 예상(약 4~6%)할 수 있는 CpG 디뉴클레오티드 함량이 60%(약 4~6%)인 반면, 나머지 게놈은 CpG 주파수(~1%)가 훨씬 낮아 CpG 억제라는 현상이 특징이다. 유전자의 부호화 영역에 있는 CpG 사이트와 달리, 대부분의 경우 CpG 촉진자 섬에 있는 CpG 사이트는 유전자가 발현될 경우 메틸화되지 않는다. 이러한 관찰은 유전자의 촉진자 내 CpG 사이트의 메틸화가 유전자 발현을 억제할 수 있다는 추측으로 이어졌다. 히스톤 수정과 함께 메틸레이션이 각인의 중심이다.[19] 조직 간의 메틸화 차이 또는 정상 검체와 암 검체 간의 대부분의 메틸화 차이는 섬 자체보다는 CpG 섬("CpG 섬 해안")에서 단거리에서 발생한다.[20]
CpG 섬은 일반적으로 척추동물의 유전자, 특히 하우스키핑 유전자의 전사 시작 지점 또는 근처에서 발생한다.[14] 척추동물의 DNA에는 바로 G(구아닌) 염기(CpG)가 뒤따르는 C(시토신) 염기(Cytosine) 염기(Cytosine) 염기(CpG)가 드물다. 왜냐하면 그러한 배열의 시토신은 메틸화되기 때문이다. 이 메틸레이션은 새로 합성된 DNA 가닥과 부모 가닥을 구별하는 데 도움을 주며, 복제 후 DNA 교정의 최종 단계에 도움을 준다. 그러나 시간이 흐르면서 자연적인 탈염으로 인해 메틸화 된 시토신은 흉선으로 변하는 경향이 있다. T/G 불일치로부터 T를 특별히 대체하는 특별한 효소(Tymine-DNA glycosylase, 또는 TDG)가 인간에게 있다. 그러나 CpGs의 희귀성 때문에, 이뉴클레오티드의 빠른 돌연변이를 방지하는데 불충분하다는 이론이 있다. CpG섬의 존재는 대개 상대적으로 높은 CpG 함량에 대한 선택적 힘 또는 그 게놈 영역에서의 낮은 메틸화 수준의 존재로 설명되며, 아마도 유전자 발현 조절과 관련이 있을 것이다. 2011년의 한 연구는 대부분의 CpG 섬이 비선택적 힘의 결과라는 것을 보여주었다.[21]
메틸화, 음소거, 암, 노화
프로모터 내의 CpG 섬
인간의 경우, 유전자(최소 발기인)의 전사 시작 지점 근처에 위치한 발기인 중 약 70%가 CpG 섬을 포함하고 있다.[2][3]
원위 프로모터 요소에는 CpG 섬도 자주 포함된다. 예를 들어, DNA 수리 유전자 ERCC1이 있는데, 여기서 CpG 섬 함유 원소는 ERCC1 유전자의 전사 시작 부지의 업스트림 약 5,400개의 뉴클레오티드가 위치한다.[22] CpG 섬은 마이크로RNA와 같은 기능 비코딩 RNA의 프로모터에서도 자주 발생한다.[23]
CpG섬의 메틸레이션 안정적 침묵유전자
인간에서 DNA 메틸화는 CpG 사이트 내 시토신 잔류물의 피리미딘 링의 5개 위치에서 발생하여 5-메틸시토신을 형성한다. CpG 촉진자 섬에 메틸화 CpG 사이트가 여러 개 존재하면 유전자의 안정적인 음소거가 발생한다.[24] 유전자의 음소거는 다른 메커니즘에 의해 시작될 수 있지만, 이는 종종 촉진자 CpG섬의 CpG 사이트 메틸레이션에 따라 유전자의 안정적인 음소거가 발생한다.[24]
촉진자 CpG 하이퍼/하이포 메틸화(암
암에서는 유전자 발현상실이 돌연변이보다 촉진제 CpG섬의 하이퍼메틸화에 의해 약 10배 더 자주 발생한다. 예를 들어 대장암에서는 보통 약 3~6명의 운전자 돌연변이가 발생하고 33~66명의 히치하이커나 승객 돌연변이가 발생한다.[25] 대조적으로, 인접한 정상으로 나타나는 대장 점막에 비해 대장 종양에 대한 한 연구에서 1,734개의 CpG 섬이 종양에서 심하게 메틸화 된 반면, 이러한 CpG 섬들은 인접한 점막에서 메틸화되지 않았다.[26] CpG섬의 절반은 주석 처리된 단백질 부호화 유전자의 촉진제였으며,[26] 이는 대장종양에서 약 867개의 유전자가 CpG섬 메틸화로 인해 발현력을 잃었음을 시사한다. 다른 연구에서는 6개의 대장암(주변 점막과 비교) 게놈에서 평균 1,549개의 차등 메틸화 부위(하이퍼메틸화 또는 하이포메틸화)가 발견되었으며, 그 중 629개는 유전자의 알려진 촉진 부위였다.[27] 세 번째 연구는 2,000개 이상의 유전자가 대장암과 인접한 점막 사이에 차등 메틸화되었다는 것을 발견했다. 유전자 세트 농축 분석을 이용해 938개 유전자 세트 중 569개가 과메틸화, 369개가 암에서 저메틸화됐다.[28] 촉진자 내 CpG섬의 저산화는 영향을 받는 유전자나 유전자 세트의 과도한 억압을 초래한다.
2012년 한 연구에서는[29] 대장암과 관련된 하이퍼메틸화 촉진제를 가진 147개의 특정 유전자와 이러한 하이퍼메틸화가 대장암에서 발견된 빈도를 함께 열거했다. 그 유전자 중 적어도 10개는 거의 100%의 대장암에 고메틸화 촉진제를 가지고 있었다. 그들은 또한 발기인이 대장암에서 50%에서 100% 사이의 빈도로 과메틸화 된 11개의 마이크로RNA를 나타냈다. miRNA(microRNA)는 작은 내생성 RNA로, 메신저 RNA의 시퀀스와 짝을 지어 전달 후 억압을 지시한다. 평균적으로 각 마이크로RNA는 수백 개의 표적 유전자를 억제한다.[30] 따라서 초메틸화 촉진제를 사용하는 마이크로RNA는 암에서 수백에서 수천 개의 유전자를 과다하게 노출시킬 수 있다.
위의 정보는 암에서 촉진자 CpG 하이퍼/하이포 메틸화 유전자와 마이크로RNA가 돌연변이보다 훨씬 더 많은 유전자의 발현 손실(또는 때로는 발현 증가)을 일으킨다는 것을 보여준다.
DNA는 암에서 초/초산 메틸화 촉진제로 유전자를 치료한다.
DNA 수리 유전자는 발기인 내 CpG섬의 과몰입으로 암에서 자주 억제된다. In head and neck squamous cell carcinomas at least 15 DNA repair genes have frequently hypermethylated promoters; these genes are XRCC1, MLH3, PMS1, RAD51B, XRCC3, RAD54B, BRCA1, SHFM1, GEN1, FANCE, FAAP20, SPRTN, SETMAR, HUS1, and PER1.[31] 약 17종류의 암은 발기인의 과메틸화로 인해 하나 이상의 DNA 수리 유전자가 결핍되는 경우가 많다.[32] 예를 들어, DNA 수리 유전자 MGMT의 촉진자 하이퍼메틸화는 방광암의 93%, 위암의 88%, 갑상선암의 74%, 대장암의 40~90%, 뇌암의 50%에서 발생한다. LIG4의 촉진자 과몰입은 대장암의 82%에서 발생한다. NEI1의 촉진자 과몰입은 머리와 목암의 62%와 비소세포 폐암의 42%에서 발생한다. ATM의 촉진자 과몰입은 비소세포 폐암의 47%에서 발생한다. MLH1의 촉진자 과몰입은 비소세포 폐암 편평세포암의 48%에서 발생한다. FANCB의 촉진자 하이퍼메틸화는 머리와 목암의 46%에서 발생한다.
한편, PARP1과 FEN1이라는 두 유전자의 발기인은 저메틸화되었고 이러한 유전자들은 수많은 암에서 과잉 발현되었다. PARP1과 FEN1은 오류가 발생하기 쉽고 돌연변이 DNA 수리 경로 미생물 매개 엔드 결합에 필수적인 유전자다. 만약 이 통로가 과도한 돌연변이를 과도하게 표출한다면 암으로 이어질 수 있다. PARP1은 티로신 키나제 활성 백혈병,[33] 신경블라스토마,[34] 고환 및 기타 세균 세포종양에서 과다하게 발현되며,[35] 유동의 육종에서 [36]FEN1은 유방암,[37] 전립선암,[38] 위암,[39][40][41] 신경블라스토마, [42]췌장암, 폐암에서 대부분 과다 발현된다.[43]
DNA 손상이 암의 주요 원인인 것으로 보인다.[44][45] 정확한 DNA 수리가 부족하면 DNA 손상이 누적되는 경향이 있다. 이러한 과도한 DNA 손상은 오류 발생 가능성이 높은 전이 합성 때문에 DNA 복제 중 돌연변이 오류를 증가시킬 수 있다. 과도한 DNA 손상은 또한 DNA 수리 중 오류로 인한 후생유전적 변화를 증가시킬 수 있다.[46][47] 그러한 돌연변이와 후생유전적 변화는 암을 유발할 수 있다(악성 신엽 참조). 따라서, DNA 수리 유전자의 발기인에서 CpG 섬 하이퍼/하이포 메틸화는 암으로 진행하는데 중심적일 가능성이 높다.
연령별 CpG 사이트 메틸화
나이가 수만 개의 CpG 사이트에서 DNA 메틸레이션 수준에 강한 영향을 미치기 때문에 인간과 침팬지에게 매우 정확한 생체시계(후유전학적 시계 또는 DNA 메틸레이션 시대라고 한다)를 정의할 수 있다.[48]
비메틸화 사이트
비메틸화 CpG 디뉴클레오티드 사이트는 플라스마시토이드 덴드리틱 세포, 단세포, 자연 킬러(NK) 세포, 인간 내 B세포에서 톨러 유사 수용체 9(TLR 9)[49]가 검출할 수 있다. 이것은 세포내 바이러스 감염을 감지하는 데 사용된다.
메모리 내 CpG 사이트 역할
포유류에서 DNA 메틸전달효소(DNA 베이스에 메틸 그룹을 추가하는 것)는 CpG 사이트 내 시토신에 대한 염기서열 선호도를 나타낸다.[50] 마우스 뇌에서, 모든 시토신의 4.2%가 메틸화되며, 주로 CpG 사이트 맥락에서 5mCpG를 형성한다.[51] 대부분의 과메틸화 5mCpG 사이트는 관련 유전자의 억제를 증가시킨다.[51]
듀크 외 연구진이 검토한 바와 같이 뉴런 DNA 메틸레이션(특정 유전자의 억압적 표현)은 뉴런 활동에 의해 변형된다. 시냅스 가소성에 뉴런 DNA 메틸화가 필요하며, 경험에 의해 변형되며, 기억 형성 및 유지보수에 적극적인 DNA 메틸화와 디메틸화가 필요하다.[52]
2016년 [53]핼더 외 마우스 사용,[52] 그리고 2017년 듀크 외 쥐를 사용하여 설치류에 문맥적 공포 조절을 적용하여 특히 강한 장기 기억을 형성한다. 조건화 후 24시간 후 쥐의 해마뇌영역에서는 1,048개의 유전자의 발현이 하향 조절되었고(일반적으로 유전자 발기인에서는 5mCpG와 연관됨), 564개의 유전자의 발현이 상향 조절되었다(흔히 유전자 발기인에서는 CpG 사이트의 저계화 작용과 관련됨). 훈련 24시간 후 해마 뉴런의 쥐 게놈 유전자의 9.2%가 차등 메틸화됐다. 그러나 해마는 새로운 정보를 배우는 데 필수적이지만, 그것은 정보 자체를 저장하지는 않는다. 할더의 쥐 실험에서 1,206개의 차등 메틸화 유전자가 상황별 공포 조절 후 1시간 후에 해마에서 관찰되었지만, 이러한 변화된 메틸화 유전자는 역전되어 4주가 지나도 보이지 않았다. 해마에서 장기간 CpG 메틸레이션 변화가 없는 것과 대조적으로 기억력 유지 시 피질 뉴런에서 상당한 양의 CpG 메틸레이션이 검출될 수 있었다. 상황별 공포 조건화 4주 후 생쥐의 전방 응고 피질에는 1,223개의 미분 메틸화 유전자가 있었다.
CpG 사이트에서 디메틸화를 수행하려면 ROS 활동이 필요함

성인 체세포에서 DNA 메틸화는 일반적으로 CpG 디뉴클레오티드(CpG 사이트)의 맥락에서 발생하며, 5-메틸시토신-pG 또는 5mCpG를 형성한다. 반응성 산소종(ROS)은 디뉴클레오티드 부위에서 구아닌을 공격하여 8-hydroxy-2'-deoxyguanosine(8-OHDG)을 형성하고 5mCp-8-OHDG 디뉴클레오티드 부지를 형성할 수 있다. 염기분리보수효소 OGG1은 8-OHDG를 대상으로 하며 즉각적인 절연 없이 병변에 결합한다. OGG1은 5mCp-8-OHDG 사이트에 있으며 TET1은 8-OHDG에 인접한 5mC를 산화시킨다. 이것은 5mC의 데메틸화를 시작한다.[54]

2018년 [55]검토된 바와 같이 뇌신경세포에서 5mC는 이산화지질소(TET1, TET2, TET3)의 10개 일레븐 변환(TET) 계열에 의해 산화되어 5hmC(5hydroxymethylcytosine)를 생성한다. 연속적인 단계에서 TET 효소는 추가로 5hmC를 히드록시하여 5-포밀시토신(5fC)과 5-카르복실시토신(5caC)을 생성한다. 티민-DNA 글리코실라아제(TDG)는 중간 염기 5fC와 5caC를 인식하고, 유리 코실린 결합을 분해하여 아피트리미딘 사이트(AP 사이트)를 만든다. 대체 산화제 탈염 경로에서 5hmC는 활성 유도 시티딘 탈아미노제/아폴리포프로테이트 B mRNA 편집 콤플렉스(AD/APOBEC) 탈아민제를 통해 산화 탈아민화하여 5-하이드록시메틸우라실(5hMU) 또는 5mC를 티민(Thy)으로 변환할 수 있다. 5hmU는 TDG, 1-선택적 무오작성 우라실-DNA 글리코실라아제 1(SMUG1) Nei-Like DNA 글리코실라아제 1(NEIL1) 또는 메틸-CpG 결합 단백질 4(MBD4)로 분해할 수 있다. 그런 다음 AP 사이트와 T:G 불일치를 베이스 절연수리(BER) 효소에 의해 수리하여 시토신(Cyt)을 산출한다.
두 개의[56][57] 리뷰는 메모리 형성에 있어서 ROS의 중요하고 필수적인 역할에 대한 많은 증거들을 요약한다. 메모리 형성 중 수천 개의 CpG 부위의 DNA 디메틸화는 ROS의 시작에 따라 달라진다. 2016년 저우 외 [54]연구진은 ROS가 DNA 디메틸화의 중심 역할을 한다는 것을 보여주었다.
TET1은 5mCpG 제염에 관여하는 핵심 효소다. 그러나 TET1은 ROS가 구아닌에 작용하여 8-히드록시-2'-데옥시구아노신(8-OHDG)을 처음 형성한 경우에만 5mCpG에 작용하여 5mCp-8-OHDG 디뉴클레오타이드(이 절의 첫 번째 그림 참조)를 발생시킨 경우에만 5mCpG에 작용할 수 있다.[54] 5mCp-8-OHDG 형성 후 베이스 절연 복구 효소 OGG1은 즉각적인 절연 없이 8-OHDG 병변에 결합한다. OGG1을 5mCp-8-OHDG 사이트에 고수하면 TET1을 모집하여 TET1이 이 섹션의 첫 번째 그림에서와 같이 8-OHDG에 인접한 5mC를 산화시킬 수 있다. 이렇게 하면 이 섹션의 두 번째 그림에 표시된 디메틸화 경로가 시작된다.
뉴런 DNA 내 유전자 촉진제 내 CpG 사이트의 ROS 의존적 디메틸화에 의해 제어되는 뉴런의 단백질 발현 변화는 기억 형성의 중심이다.[58]
CPG손실
TE가 게놈 팽창뿐만 아니라 숙주 DNA에서 CpG 손실까지 담당하는 TE(Transposable Elements, Transposable Elements)의 DNA 메틸화 과정에서 CPG 고갈이 관찰된 바 있다. TE는 "methylation centers"라고 알려져 있는데, 여기서 Methylation process에 의해 TEs는 숙주 DNA에서 한 번 옆구리 DNA로 퍼진다. 이러한 확산은 이후 진화 시간에 걸쳐 CPG 손실을 초래할 수 있다. 더 오래된 진화 시간은 젊은 진화 시간에 비해 옆구리 DNA에서 CpG 손실이 더 높다는 것을 보여준다. 따라서 DNA 메틸레이션은 결국 이웃 DNA에서 눈에 띄게 CpG 부지를 잃게 할 수 있다.
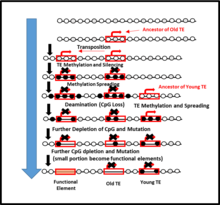
이전의 연구들은 무척추동물과 척추동물이 인간에 비해 작고 큰 게놈을 가지고 있는 게놈 크기의 다양성을 확인했다. 게놈 크기는 전이 가능한 원소의 수와 강하게 연결되어 있다. 단, TEs 메틸화 수 대 CPG 양 사이에는 상관관계가 있다. 이러한 부정적인 상관관계는 결과적으로 유전자간 DNA 메틸화로 인한 CPG의 고갈을 유발하는데, 이는 대부분 TE의 메틸화로 기인한다. 전반적으로, 이것은 다른 게놈 종에서 CPG 손실의 현저한 양에 기여한다. [59]
CPG 손실의 촉진자로서 알루 요소
알루 원소는 전이 가능한 원소의 가장 풍부한 유형으로 알려져 있다. 어떤 연구들은 알루 원소를 게놈 확장에 어떤 요인이 작용하는지 연구하기 위한 방법으로 이용했다. 알루 원소는 LINEs나 EERV와는 달리 더 긴 수의 시퀀스에서 CPG가 풍부하다. 알러스는 메틸레이션 센터로 일할 수 있으며, 숙주 DNA에 삽입하면 DNA 메틸화가 생성되어 플랭킹 DNA 영역으로 확산될 수 있다. 이러한 확산은 CPG 손실이 상당히 크고 게놈 확장이 상당히 증가하는 이유다.[59] 다만 오래된 알루스 원소가 젊은 것에 비해 인접 DNA 부위에서 CPG 손실이 더 많이 나타나기 때문에 시간이 지남에 따라 분석되는 결과다.
참고 항목
- TLR9, 비메틸 CpG 사이트 검출기
- DNA 메틸화 시대
참조
- ^ Jabbari K, Bernardi G (May 2004). "Cytosine methylation and CpG, TpG (CpA) and TpA frequencies". Gene. 333: 143–9. doi:10.1016/j.gene.2004.02.043. PMID 15177689.
- ^ a b Saxonov S, Berg P, Brutlag DL (2006). "A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters". Proc. Natl. Acad. Sci. U.S.A. 103 (5): 1412–7. Bibcode:2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC 1345710. PMID 16432200.
- ^ a b Deaton AM, Bird A (2011). "CpG islands and the regulation of transcription". Genes Dev. 25 (10): 1010–22. doi:10.1101/gad.2037511. PMC 3093116. PMID 21576262.
- ^ Lander, Eric S.; Linton, Lauren M.; Birren, Bruce; Nusbaum, Chad; Zody, Michael C.; Baldwin, Jennifer; Devon, Keri; Dewar, Ken; Doyle, Michael (15 February 2001). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. ISSN 1476-4687. PMID 11237011.
- ^ International Human Genome Sequencing Consortium (2001-02-15). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860–921. doi:10.1038/35057062. ISSN 0028-0836. PMID 11237011.
- ^ Hwang DG, Green P (2004). "Bayesian Markov chain Monte Carlo sequence analysis reveals varying neutral substitution patterns in mammalian evolution". Proc Natl Acad Sci U S A. 101 (39): 13994–4001. Bibcode:2004PNAS..10113994H. doi:10.1073/pnas.0404142101. PMC 521089. PMID 15292512.
- ^ Walsh CP, Xu GL (2006). "Cytosine methylation and DNA repair". Curr Top Microbiol Immunol. Current Topics in Microbiology and Immunology. 301: 283–315. doi:10.1007/3-540-31390-7_11. ISBN 3-540-29114-8. PMID 16570853.
- ^ Arnheim N, Calabrese P (2009). "Understanding what determines the frequency and pattern of human germline mutations". Nat Rev Genet. 10 (7): 478–488. doi:10.1038/nrg2529. PMC 2744436. PMID 19488047.
- ^ Ségurel L, Wyman MJ, Przeworski M (2014). "Understanding what determines the frequency and pattern of human germline mutations". Annu Rev Genom Hum Genet. 15: 47–70. doi:10.1146/annurev-genom-031714-125740. PMID 25000986.
- ^ Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (February 2001). "Initial sequencing and analysis of the human genome". Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. (February 2001). "The sequence of the human genome". Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ Myers EW, Sutton GG, Smith HO, Adams MD, Venter JC (April 2002). "On the sequencing and assembly of the human genome". Proc. Natl. Acad. Sci. U.S.A. 99 (7): 4145–6. Bibcode:2002PNAS...99.4145M. doi:10.1073/pnas.092136699. PMC 123615. PMID 11904395.
- ^ Gardiner-Garden M, Frommer M (1987). "CpG islands in vertebrate genomes". Journal of Molecular Biology. 196 (2): 261–282. doi:10.1016/0022-2836(87)90689-9. PMID 3656447.
- ^ a b c Saxonov S, Berg P, Brutlag DL (2006). "A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters". Proc Natl Acad Sci USA. 103 (5): 1412–1417. Bibcode:2006PNAS..103.1412S. doi:10.1073/pnas.0510310103. PMC 1345710. PMID 16432200.
- ^ Hartl DL, Jones EW (2005). Genetics: Analysis of Genes and Genomes (6th ed.). Missisauga: Jones & Bartlett, Canada. p. 477. ISBN 978-0-7637-1511-3.
- ^ Fatemi M, Pao MM, Jeong S, Gal-Yam EN, Egger G, Weisenberger DJ, et al. (2005). "Footprinting of mammalian promoters: use of a CpG DNA methyltransferase revealing nucleosome positions at a single molecule level". Nucleic Acids Res. 33 (20): e176. doi:10.1093/nar/gni180. PMC 1292996. PMID 16314307.
- ^ Alberts, Bruce (18 November 2014). Molecular biology of the cell (Sixth ed.). New York, NY. p. 406. ISBN 978-0-8153-4432-2. OCLC 887605755.
- ^ Takai D, Jones PA (2002). "Comprehensive analysis of CpG islands in human chromosomes 21 and 22". Proc Natl Acad Sci USA. 99 (6): 3740–5. Bibcode:2002PNAS...99.3740T. doi:10.1073/pnas.052410099. PMC 122594. PMID 11891299.
- ^ Feil R, Berger F (2007). "Convergent evolution of genomic imprinting in plants and mammals". Trends Genet. 23 (4): 192–199. doi:10.1016/j.tig.2007.02.004. PMID 17316885.
- ^ Irizarry RA, Ladd-Acosta C, Wen B, Wu Z, Montano C, Onyango P, et al. (2009). "The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores". Nature Genetics. 41 (2): 178–186. doi:10.1038/ng.298. PMC 2729128. PMID 19151715.
- ^ Cohen N, Kenigsberg E, Tanay A (2011). "Primate CpG Islands Are Maintained by Heterogeneous Evolutionary Regimes Involving Minimal Selection". Cell. 145 (5): 773–786. doi:10.1016/j.cell.2011.04.024. PMID 21620139. S2CID 14856605.
- ^ Chen HY, Shao CJ, Chen FR, Kwan AL, Chen ZP (2010). "Role of ERCC1 promoter hypermethylation in drug resistance to cisplatin in human gliomas". Int. J. Cancer. 126 (8): 1944–54. doi:10.1002/ijc.24772. PMID 19626585.
- ^ Kaur S, Lotsari-Salomaa JE, Seppänen-Kaijansinkko R, Peltomäki P (2016). "MicroRNA Methylation in Colorectal Cancer". Adv. Exp. Med. Biol. Advances in Experimental Medicine and Biology. 937: 109–22. doi:10.1007/978-3-319-42059-2_6. ISBN 978-3-319-42057-8. PMID 27573897.
- ^ a b Bird A (2002). "DNA methylation patterns and epigenetic memory". Genes Dev. 16 (1): 6–21. doi:10.1101/gad.947102. PMID 11782440.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ a b Illingworth RS, Gruenewald-Schneider U, Webb S, Kerr AR, James KD, Turner DJ, Smith C, Harrison DJ, Andrews R, Bird AP (2010). "Orphan CpG islands identify numerous conserved promoters in the mammalian genome". PLOS Genet. 6 (9): e1001134. doi:10.1371/journal.pgen.1001134. PMC 2944787. PMID 20885785.
- ^ Wei J, Li G, Dang S, Zhou Y, Zeng K, Liu M (2016). "Discovery and Validation of Hypermethylated Markers for Colorectal Cancer". Dis. Markers. 2016: 1–7. doi:10.1155/2016/2192853. PMC 4963574. PMID 27493446.
- ^ Beggs AD, Jones A, El-Bahrawy M, El-Bahwary M, Abulafi M, Hodgson SV, et al. (2013). "Whole-genome methylation analysis of benign and malignant colorectal tumours". J. Pathol. 229 (5): 697–704. doi:10.1002/path.4132. PMC 3619233. PMID 23096130.
- ^ Schnekenburger M, Diederich M (2012). "Epigenetics Offer New Horizons for Colorectal Cancer Prevention". Curr Colorectal Cancer Rep. 8 (1): 66–81. doi:10.1007/s11888-011-0116-z. PMC 3277709. PMID 22389639.
- ^ Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Rieke DT, Ochsenreither S, Klinghammer K, Seiwert TY, Klauschen F, Tinhofer I, et al. (2016). "Methylation of RAD51B, XRCC3 and other homologous recombination genes is associated with expression of immune checkpoints and an inflammatory signature in squamous cell carcinoma of the head and neck, lung and cervix". Oncotarget. 7 (46): 75379–75393. doi:10.18632/oncotarget.12211. PMC 5342748. PMID 27683114.
- ^ Jin B, Robertson KD (2013). "DNA methyltransferases, DNA damage repair, and cancer". Adv. Exp. Med. Biol. Advances in Experimental Medicine and Biology. 754: 3–29. doi:10.1007/978-1-4419-9967-2_1. ISBN 978-1-4419-9966-5. PMC 3707278. PMID 22956494.
- ^ Muvarak N, Kelley S, Robert C, Baer MR, Perrotti D, Gambacorti-Passerini C, et al. (2015). "c-MYC Generates Repair Errors via Increased Transcription of Alternative-NHEJ Factors, LIG3 and PARP1, in Tyrosine Kinase-Activated Leukemias". Mol. Cancer Res. 13 (4): 699–712. doi:10.1158/1541-7786.MCR-14-0422. PMC 4398615. PMID 25828893.
- ^ Newman EA, Lu F, Bashllari D, Wang L, Opipari AW, Castle VP (2015). "Alternative NHEJ Pathway Components Are Therapeutic Targets in High-Risk Neuroblastoma". Mol. Cancer Res. 13 (3): 470–82. doi:10.1158/1541-7786.MCR-14-0337. PMID 25563294.
- ^ Mego M, Cierna Z, Svetlovska D, Macak D, Machalekova K, Miskovska V, et al. (2013). "PARP expression in germ cell tumours". J. Clin. Pathol. 66 (7): 607–12. doi:10.1136/jclinpath-2012-201088. PMID 23486608. S2CID 535704.
- ^ Newman RE, Soldatenkov VA, Dritschilo A, Notario V (2002). "Poly(ADP-ribose) polymerase turnover alterations do not contribute to PARP overexpression in Ewing's sarcoma cells". Oncol. Rep. 9 (3): 529–32. doi:10.3892/or.9.3.529. PMID 11956622.
- ^ Singh P, Yang M, Dai H, Yu D, Huang Q, Tan W, Kernstine KH, Lin D, Shen B (2008). "Overexpression and hypomethylation of flap endonuclease 1 gene in breast and other cancers". Mol. Cancer Res. 6 (11): 1710–7. doi:10.1158/1541-7786.MCR-08-0269. PMC 2948671. PMID 19010819.
- ^ Lam JS, Seligson DB, Yu H, Li A, Eeva M, Pantuck AJ, Zeng G, Horvath S, Belldegrun AS (2006). "Flap endonuclease 1 is overexpressed in prostate cancer and is associated with a high Gleason score". BJU Int. 98 (2): 445–51. doi:10.1111/j.1464-410X.2006.06224.x. PMID 16879693. S2CID 22165252.
- ^ Kim JM, Sohn HY, Yoon SY, Oh JH, Yang JO, Kim JH, et al. (2005). "Identification of gastric cancer-related genes using a cDNA microarray containing novel expressed sequence tags expressed in gastric cancer cells". Clin. Cancer Res. 11 (2 Pt 1): 473–82. PMID 15701830.
- ^ Wang K, Xie C, Chen D (2014). "Flap endonuclease 1 is a promising candidate biomarker in gastric cancer and is involved in cell proliferation and apoptosis". Int. J. Mol. Med. 33 (5): 1268–74. doi:10.3892/ijmm.2014.1682. PMID 24590400.
- ^ Krause A, Combaret V, Iacono I, Lacroix B, Compagnon C, Bergeron C, et al. (2005). "Genome-wide analysis of gene expression in neuroblastomas detected by mass screening" (PDF). Cancer Lett. 225 (1): 111–20. doi:10.1016/j.canlet.2004.10.035. PMID 15922863.
- ^ Iacobuzio-Donahue CA, Maitra A, Olsen M, Lowe AW, van Heek NT, Rosty C, et al. (2003). "Exploration of global gene expression patterns in pancreatic adenocarcinoma using cDNA microarrays". Am. J. Pathol. 162 (4): 1151–62. doi:10.1016/S0002-9440(10)63911-9. PMC 1851213. PMID 12651607.
- ^ Nikolova T, Christmann M, Kaina B (2009). "FEN1 is overexpressed in testis, lung and brain tumors". Anticancer Res. 29 (7): 2453–9. PMID 19596913.
- ^ Kastan MB (2008). "DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture". Mol. Cancer Res. 6 (4): 517–24. doi:10.1158/1541-7786.MCR-08-0020. PMID 18403632.
- ^ Bernstein, C; Prasad, AR; Nfonsam, V; Bernstein, H. (2013). "Chapter 16: DNA Damage, DNA Repair and Cancer". In Chen, Clark (ed.). New Research Directions in DNA Repair. p. 413. ISBN 978-953-51-1114-6.
- ^ O'Hagan HM, Mohammad HP, Baylin SB (2008). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. 4 (8): e1000155. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ^ Cuozzo C, Porcellini A, Angrisano T, et al. (July 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ Horvath S (2013). "DNA methylation age of human tissues and cell types". Genome Biology. 14 (10): R115. doi:10.1186/gb-2013-14-10-r115. PMC 4015143. PMID 24138928.
- ^ Ramirez-Ortiz ZG, Specht CA, Wang JP, Lee CK, Bartholomeu DC, Gazzinelli RT, Levitz SM (2008). "Toll-like receptor 9-dependent immune activation by unmethylated CpG motifs in Aspergillus fumigatus DNA". Infect. Immun. 76 (5): 2123–2129. doi:10.1128/IAI.00047-08. PMC 2346696. PMID 18332208.
- ^ Ziller MJ, Müller F, Liao J, Zhang Y, Gu H, Bock C, et al. (December 2011). "Genomic distribution and inter-sample variation of non-CpG methylation across human cell types". PLOS Genet. 7 (12): e1002389. doi:10.1371/journal.pgen.1002389. PMC 3234221. PMID 22174693.
- ^ a b Fasolino M, Zhou Z (May 2017). "The Crucial Role of DNA Methylation and MeCP2 in Neuronal Function". Genes (Basel). 8 (5): 141. doi:10.3390/genes8050141. PMC 5448015. PMID 28505093.
- ^ a b Duke CG, Kennedy AJ, Gavin CF, Day JJ, Sweatt JD (July 2017). "Experience-dependent epigenomic reorganization in the hippocampus". Learn. Mem. 24 (7): 278–288. doi:10.1101/lm.045112.117. PMC 5473107. PMID 28620075.
- ^ Halder R, Hennion M, Vidal RO, Shomroni O, Rahman RU, Rajput A, et al. (January 2016). "DNA methylation changes in plasticity genes accompany the formation and maintenance of memory". Nat. Neurosci. 19 (1): 102–10. doi:10.1038/nn.4194. PMC 4700510. PMID 26656643.
- ^ a b c Zhou X, Zhuang Z, Wang W, He L, Wu H, Cao Y, Pan F, Zhao J, Hu Z, Sekhar C, Guo Z (September 2016). "OGG1 is essential in oxidative stress induced DNA demethylation". Cell. Signal. 28 (9): 1163–71. doi:10.1016/j.cellsig.2016.05.021. PMID 27251462.
- ^ Bayraktar G, Kreutz MR (2018). "The Role of Activity-Dependent DNA Demethylation in the Adult Brain and in Neurological Disorders". Front Mol Neurosci. 11: 169. doi:10.3389/fnmol.2018.00169. PMC 5975432. PMID 29875631.
- ^ Massaad CA, Klann E (May 2011). "Reactive oxygen species in the regulation of synaptic plasticity and memory". Antioxid. Redox Signal. 14 (10): 2013–54. doi:10.1089/ars.2010.3208. PMC 3078504. PMID 20649473.
- ^ Beckhauser TF, Francis-Oliveira J, De Pasquale R (2016). "Reactive Oxygen Species: Physiological and Physiopathological Effects on Synaptic Plasticity". J Exp Neurosci. 10 (Suppl 1): 23–48. doi:10.4137/JEN.S39887. PMC 5012454. PMID 27625575.
- ^ Day JJ, Sweatt JD (November 2010). "DNA methylation and memory formation". Nat. Neurosci. 13 (11): 1319–23. doi:10.1038/nn.2666. PMC 3130618. PMID 20975755.
- ^ a b c Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 August 2020). "DNA methylation enables transposable element-driven genome expansion". Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. doi:10.1073/pnas.1921719117. ISSN 1091-6490. PMC 7431005. PMID 32719115.
{{cite journal}}: CS1 maint: 날짜 및 연도(링크) - ^ Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 August 2020). "DNA methylation enables transposable element-driven genome expansion". Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. doi:10.1073/pnas.1921719117. ISSN 1091-6490. PMC 7431005. PMID 32719115.







