내장미생물
Gut microbiota
내장 마이크로바이옴(gut microbiota)은 박테리아, 고균, 곰팡이, 바이러스 등 동물의 소화관에 서식하는 미생물을 말합니다.[1][2]위장 메타게놈은 장내 미생물 무리의 모든 게놈의 집합체입니다.[3][4]내장은 인간 마이크로바이옴의 주요 위치입니다.[5]장내 미생물 무리는 식민지화에 대한 영향, 병원균에 대한 저항, 장 상피 유지, 식이 및 약학적 화합물 대사, 면역 기능 조절 및 심지어 장-뇌 축을 통한 행동을 포함한 광범위한 영향을 미칩니다.
장내 미생물의 구성은 소화관의 영역에 따라 다릅니다.대장은 지금까지 연구된 인간 관련 미생물 군집 중 가장 높은 미생물 밀도를 가지고 있으며, 이는 300종에서 1000종 사이를 대표합니다.[6]박테리아는 가장 크고 현재까지 가장 잘 연구된 구성 요소이며 내장 박테리아의 99%는 약 30종 또는 40종에서 비롯됩니다.[7]대변의 건조한 덩어리의 60%가 세균입니다.[8]장내 세균의 99% 이상이 혐기성 세균이지만 맹장에서는 호기성 세균이 높은 밀도에 도달합니다.[5]인간 내장 미생물은 인간 게놈보다 약 백 배 많은 유전자를 가지고 있는 것으로 추정됩니다.
개요
인간의 경우, 내장 미생물은 몸의 다른 영역에 비해 가장 많은 수와 종류의 박테리아를 가지고 있습니다.[9]인간의 경우, 내장 세균은 출생 후 1년에서 2년에 형성되는데, 이때까지 내장 상피와 이를 분비하는 장점막 장벽이 내장 세균에 내성을 갖고 심지어는 이를 지지하는 방식으로 함께 발전하게 됩니다.[10][11]
일부 내장 미생물과 인간의 관계는 단순히 상식적인 것(해롭지 않은 공존)이 아니라 상호주의적인 관계입니다.[5]: 700 일부 인간 내장 미생물은 식이 섬유를 아세트산과 부티르산과 같은 짧은 사슬 지방산(SCFA)으로 발효시킴으로써 숙주에게 이익을 주고, 숙주에 의해 흡수됩니다.[9][12]장내세균은 또한 비타민 B와 비타민 K를 합성하고 담즙산, 스테롤, 제노바이오틱스를 대사하는 역할을 합니다.[5][12]SCFAs와 그들이 만들어내는 다른 화합물들의 시스템적인 중요성은 호르몬과 같고 내장 식물 자체는 내분비 기관처럼 작용하는 것으로 보입니다.[12]내장 세균의 조절 장애는 다수의 염증 및 자가 면역 질환과 관련이 있습니다.[9][13]
인간 내장 미생물의 구성은 시간이 지남에 따라, 식단이 변화할 때, 그리고 전반적인 건강 변화에 따라 바뀝니다.[9][13]2016년의 체계적인 검토는 상업적으로 이용 가능한 특정 종류의 프로바이오틱스 박테리아로 수행된 전임상 및 소규모 인간 실험을 조사하고 특정 중추신경계 질환에 가장 유용할 가능성이 있는 실험을 확인했습니다.[14]
분류
장내 미생물의 구성은 소화관에 따라 다양합니다.위와 소장에는 일반적으로 비교적 적은 종의 박테리아가 존재합니다.[6][15]대조적으로 대장은 지금까지[16] 연구된 인간 관련 미생물 군집 중 가장 높은 미생물 밀도를 가지고 있으며, 장내 함량은 1g당 최대 10개의12 세포를 가지고 있습니다.[6]이 박테리아들은 300종에서 1000종 사이의 다른 종을 나타냅니다.[6][15]하지만, 박테리아의 99%는 약 30종 또는 40종에서 비롯됩니다.[7]장내에 풍부하게 존재하는 결과로, 박테리아는 또한 대변의 건조한 덩어리의 60%를 차지합니다.[8]곰팡이, 원생동물, 고균, 바이러스 등도 내장 속에 존재하지만, 그들의 활동에 대해서는 알려진 바가 적습니다.[17]
장내 세균의 99% 이상이 혐기성 세균이지만 맹장에서는 호기성 세균이 높은 밀도에 도달합니다.[5]이 내장 세균들은 인간 게놈에 있는 유전자보다 총 100배 가량 많은 유전자를 가지고 있는 것으로 추정됩니다.[18]
내장 속의 많은 종들은 배양이 불가능하기 때문에 숙주 밖에서 연구되지 않았습니다.[15][7][19]대부분의 개체들이 공유하는 소수의 핵심 미생물 종들이 있지만, 미생물의 개체수는 매우 다양할 수 있습니다.[20]개인 내에서, 그들의 미생물 집단은 시간이 지남에 따라 상당히 일정하게 유지되며, 생활 양식, 식습관 그리고 나이의 변화로 인해 약간의 변화가 발생합니다.[6][21]인간 마이크로바이옴 프로젝트는 인간의 내장과 다른 신체 위치의 마이크로바이옴을 더 잘 설명하기 시작했습니다.[citation needed]
사람의 내장에 있는 4개의 지배적인 박테리아 계통은 바실로타(Bacillota, Firmicutes), 박테로이도타(Bacteroidota), 방선균(Actinomycetota), 녹농균(Pseudomonadota)입니다.[22]대부분의 박테리아는 박테로이데스속, 클로스트리디움속, 페이칼리박테리움속,[6][7] 유박테리움속, 루미노코커스속, 펩토코커스속, 펩토스트렙토코커스속, 비피도박테리움속에 속합니다.[6][7]에세리키아(Escherichia)와 락토바실러스(Lactobacillus)와 같은 다른 속들은 덜 존재합니다.[6]박테로이데스 속의 종들만 해도 내장에 있는 모든 박테리아의 약 30%를 구성하고 있는데, 이 속은 숙주의 기능에 있어서 특히 중요함을 시사합니다.[15]
장에서 검출된 곰팡이속으로는 칸디다, 사카로마이세스, 아스페르길루스, 페니실륨, 로도토룰라, 트라메테스, 플레오스포라, 스크레로티니아, 불레라, 갈락토마이세스 등이 있습니다.[23][24]호도토룰라는 염증성 장질환 환자에서 가장 많이 발견되고 칸디다는 B형 간염 간경변증과 만성 B형 간염 환자에서 가장 많이 발견됩니다.[23]
고균은 발효의 세균 생성물의 대사에 중요한 또 다른 큰 종류의 내장 세균총을 구성합니다.
산업화는 미생물 무리의 변화와 관련이 있으며 다양성의 감소는 특정 종을 멸종으로 몰고 갈 수 있습니다. 2018년에 연구원들은 인간 미생물 무리의 바이오뱅크 저장소를 제안했습니다.[25]
엔터타입
엔테로타입은 나이, 성별, 체중, 국가 구분에 의해 지시되지 않는 인간 내장 마이크로바이옴의 박테리아 생태계에 기초하여 생물을 분류하는 것입니다.[26]장기간의 식이요법이 장형에 영향을 준다는 징후가 있습니다.[27]세 가지 인간 엔테로타입이 제안되었지만, [26][28]그 가치에 대해서는 의문이 제기되고 있습니다.[29]
구성.
박테리오메
배
위의 산성도가 높기 때문에 대부분의 미생물은 그곳에서 살아남을 수 없습니다.위의 주요 세균 거주자는 다음과 같습니다.스트렙토코커스, 포도상구균, 락토바실러스, 펩토스트렙토코커스.[5]: 720 헬리코박터 파일로리균은 만성 위염과 소화성 궤양 질환을 일으키는 위점막에 생기는 그람 음성 나선균으로 위암의 발암물질입니다.[5]: 904
창자
| 사람의 대장에서 흔히 발견되는 박테리아[30] | |
| 박테리아 | 발생률(%) |
|---|---|
| 박테로이데스프라길리스 | 100 |
| 박테로이데스 멜라닌 생성균 | 100 |
| 박테로이데스 오랄리스 | 100 |
| 장구균성 대변 | 100 |
| 대장균 | 100 |
| bacter sp를 입력합니다. | 40–80 |
| 클렙시엘라 sp. | 40–80 |
| 비피도박테리움 비피덤 | 30–70 |
| 황색포도상구균 | 30–50 |
| 유산균 | 20–60 |
| 클로스트리듐 퍼프링겐스 | 25–35 |
| 미라빌리스 | 5–55 |
| 클로스트리듐테타니 | 1–35 |
| 클로스트리듐세균 | 5–25 |
| 아루기노사 | 3–11 |
| 살모넬라 장중첩증 | 3–7 |
| 프라우스니츠이속 | ?공통의 |
| 펩토스트렙토코커스 sp. | ?공통의 |
| 펩토코커스 sp. | ?공통의 |
소장에는 위의 근접성과 영향으로 미량의 미생물이 들어 있습니다.그람 양성 cocci와 막대모양 박테리아는 소장에서 발견되는 주요 미생물입니다.[5]그러나 소장의 알칼리성 원위부에서는 장내세균과의 그람 음성 박테리아를 지지합니다.[5]소장의 세균총은 광범위한 장 기능에 도움을 줍니다.세균총은 내장의 발달과 유용성을 가능하게 하는 조절 신호를 제공합니다.소장의 세균이 과도하게 증식하면 장 기능 부전으로 이어질 수 있습니다.[31]게다가 대장은 인체에서 가장 큰 박테리아 생태계를 가지고 있습니다.[5]대장과 대변 세균총의 약 99%는 Bacteroides와 Bifidobacterium과 같은 의무 혐기성 세균으로 이루어져 있습니다.[32]대장 미생물 개체수를 방해하는 요인으로는 항생제, 스트레스, 기생충 등이 있습니다.[5]
세균은 대장에[33] 있는 세균의 대부분을 차지하고 대변의 건조한 덩어리의 60%를 차지합니다.[6]이 사실은 대변 표본으로부터 핵산을 추출함으로써 대변을 어떤 실험과 실험을 위한 이상적인 내장 식물의 원천으로 만들고, 박테리아 16S rRNA 유전자 배열은 박테리아 프라이머로 생성됩니다.이러한 형태의 테스트는 또한 종종 생체검사와 같은 더 침습적인 기술보다 선호됩니다.
장내 미생물 무리는 박테로이도타, 바실로타(Firmicutes), 방선균(Actinomycota), 슈도모나도타(Pseudomonadota), 베루코미로보타(Verrucomicrobiota) 등 5개 계통이 주를 이루고 있으며, 박테로이도타와 바실로타는 구성 성분의 90%를 차지하고 있습니다.[34]300[6]~1000종의 다른 종들이 내장에 살고 있으며,[15] 대부분의 종들은 약 500마리 정도로 추정됩니다.[35][36]그러나, 박테리아의 99%가 약 30종 또는 40종에서 온 것일 가능성이 있으며, Faecalibacterium prausnitzii (문명 firmicutes)는 건강한 성인에게 가장 흔한 종입니다.[7][37]
연구에 따르면 내장 세균과 인간의 관계는 단순히 상식적인 것(해롭지 않은 공존)이 아니라, 상호주의적이고 공생적인 관계라고 합니다.[15]비록 사람들이 내장 세균이 없어도 생존할 수 있지만,[35] 미생물들은 사용되지 않은 에너지 기질을 발효시키고, 프로피오네이트와 아세트산과 같은 대사의 최종 산물을 통해 면역 체계를 훈련시키고, 해로운 종의 성장을 방지하고, 내장의 발달을 조절하고, 숙주를 위한 비타민을 생산하는 등의 유용한 기능을 수행합니다.비오틴과 비타민 K), 그리고 숙주가 지방을 저장하도록 지시하는 호르몬을 생산합니다.[5]내장 마이크로바이옴과 마이크로바이옴 또는 유전자 수집의 광범위한 변형과 불균형은 비만과 관련이 있습니다.[38]하지만, 특정한 상황에서, 어떤 종은 숙주에게 감염을 일으키거나 암 위험을 증가시킴으로써 질병을 일으킬 수 있다고 여겨집니다.[6][33]
마이코비옴
곰팡이와 원생동물 또한 내장 세균의 한 부분을 차지하지만, 그들의 활동에 대해서는 알려진 바가 적습니다.[39]
바이롬
변화
나이
생활 중 마이크로바이옴 조성 진화의 일반적인 패턴이 있다는 것이 증명되었습니다.[41]일반적으로 분변 샘플의 미생물 구성의 다양성은 성인이 어린이보다 상당히 높지만 대인관계의 차이는 어린이가 성인보다 더 높습니다.[42]마이크로바이오타가 성체와 같은 형태로 성숙하는 많은 부분은 생애 첫 3년 동안에 일어납니다.[42]
마이크로바이옴의 구성이 변하면서 내장에서 생성되는 박테리아 단백질의 구성도 변합니다.성인 마이크로바이옴에서 발효, 메탄 생성 및 아르기닌, 글루타메이트, 아스파르트산 및 라이신의 대사에 관여하는 효소의 높은 보급이 발견되었습니다.대조적으로, 유아 마이크로바이옴에서 우세한 효소는 시스테인 대사와 발효 경로에 관여합니다.[42]
지리학
내장 마이크로바이옴 조성은 인구의 지리적 기원에 따라 달라집니다.프레보텔라(Prevotella)의 트레이드 오프, 우레아제 유전자의 표현, 글루탐산 합성효소/분해효소 또는 아미노산 분해 또는 비타민 생합성에 관여하는 다른 효소를 코딩하는 유전자의 표현의 변화는 미국, 말라위 또는 미국 기원의 인구 사이에서 상당한 차이를 보여줍니다.[42]
미국 인구는 글루타민의 분해를 암호화하는 효소와 비타민 및 리포산 생합성에 관여하는 효소의 높은 표현을 가지고 있는 반면 말라위와 아메린디아 인구는 글루탐산 합성효소를 암호화하는 효소의 높은 표현을 가지고 있으며 또한 그들의 마이크로바이옴에서 α-아밀라아제의 과잉 표현을 가지고 있습니다.미국 인구는 옥수수가 풍부한 식단을 가진 미국인이나 말라위인 인구보다 지방이 풍부한 식단을 가지고 있기 때문에, 아마도 식단이 내장 세균 구성의 주요 결정 요인일 것입니다.[42]
추가적인 연구들은 유럽과 아프리카 시골 어린이들 사이에 미생물 무리의 구성에 큰 차이가 있음을 나타냈습니다.플로렌스에서 온 아이들의 분변 세균은 부르키나파소의 작은 시골 마을인 불폰에서 온 아이들의 분변 세균과 비교되었습니다.이 마을에 사는 전형적인 아이의 식단은 지방과 동물성 단백질이 많이 부족하고 다당류와 식물성 단백질이 풍부합니다.유럽 어린이의 분변 세균은 피르미쿠테스가 지배적이었고 생물 다양성의 현저한 감소를 나타냈으며, 불폰 어린이의 분변 세균은 박테로이데스가 지배적이었습니다.아프리카 인구에서 증가된 생물 다양성과 내장 마이크로바이옴의 다른 구성은 정상적으로 소화되지 않는 식물 다당류의 소화를 도울 수 있고 또한 감염되지 않는 대장 질환의 발생을 감소시킬 수 있습니다.[43]
더 작은 규모로, 가족 내에서 수많은 공통적인 환경 노출을 공유하는 것이 개별 마이크로바이옴 구성의 강력한 결정 요인임이 밝혀졌습니다.이 효과는 유전적인 영향을 미치지 않으며 문화적으로 다른 집단에서 지속적으로 관찰됩니다.[42]
영양실조
영양실조에 걸린 아이들은 건강한 아이들에 비해 성숙도가 낮고 내장 미생물의 다양성이 떨어지며, 영양소 부족과 관련된 마이크로바이옴의 변화는 다시 영양실조의 병태생리학적 원인이 될 수 있습니다.[44][45]영양실조에 걸린 어린이들은 또한 전형적으로 더 병원성이 있는 내장 세균총을 가지고 있으며 입과 목에 더 많은 효모를 가지고 있습니다.[46]식단을 바꾸는 것은 장내 미생물의 구성과 다양성의 변화를 초래할 수 있습니다.[47]
인종과 민족
아메리칸 굿 프로젝트와 휴먼 마이크로바이옴 프로젝트의 연구원들은 12개의 마이크로바이옴 과가 개인의 인종이나 민족성에 따라 다양하다는 것을 발견했습니다.이러한 연관성의 강도는 작은 표본 크기에 의해 제한됩니다. American Gut Project는 1,375명의 데이터를 수집했으며, 그 중 90%가 백인이었습니다.[48]암스테르담의 HELIUS(Healthy Life in an Urban Setting) 연구는 네덜란드 혈통의 사람들이 내장 미생물의 다양성이 가장 높은 반면 남아시아와 수리남 혈통의 사람들은 가장 낮은 다양성을 가지고 있다는 것을 발견했습니다.연구 결과는 같은 인종이나 민족의 사람들이 다른 인종 배경을 가진 사람들보다 더 유사한 마이크로바이옴을 가지고 있다는 것을 보여주었습니다.[48]
사회경제적 지위
2020년 현재, 최소 두 개의 연구에서 개인의 사회경제적 지위(SES)와 내장 미생물 무리 사이의 관련성을 입증했습니다.시카고의 한 연구는 고등 SES 지역에 사는 사람들이 더 큰 미생물 무리의 다양성을 가지고 있다는 것을 발견했습니다.고등 SES 지역의 사람들은 또한 더 풍부한 박테로이데스 박테리아를 가지고 있었습니다.마찬가지로, 영국의 쌍둥이에 대한 연구는 높은 SES 또한 더 큰 내장의 다양성과 연관이 있다는 것을 발견했습니다.[48]
유아에서 획득
소화관의 형성은 성인의 건강과 위장관의 기능에 매우 중요합니다.[49]사람의 경우, 마이크로바이오타가 음식물, 물 및 기타 환경원으로부터 전달 및 전달을 통해 획득됨에 따라 출생 후 1년에서 2년 이내에 성인과 유사한 내장 식물이 형성됩니다.[50][10]
정상적인 태아의 위장관에 대한 전통적인 관점은 무균이라는 것이지만, 이 관점은 지난 몇 년간 도전을 받아왔습니다.[timeframe?][51]자궁내 환경에 박테리아가 있을 수도 있다는 증거가 여러 줄 나오기 시작했습니다.사람의 경우 태반 생검에 락토바실러스와 비피도박테리움 종이 존재했다는 연구 결과가 나와 태아에게[52] 미생물의 식민지화가 일어날 수 있다는 연구 결과가 나왔습니다.[53]몇몇 설치류 연구는 무균 제왕절개로 태어난 아기의 태반뿐만 아니라 양수와 태반에 박테리아가 존재한다는 것을 증명했습니다.[54][55]또 다른 연구에서, 연구원들은 임신한 쥐에게 박테리아 배양액을 경구로 투여했고, 혈류를 통해 소화관과 양수 사이에 전염되는 것으로 보이는 자손에게서 박테리아를 발견했습니다.[56]하지만, 연구원들은 자궁 내 박테리아의 출처, 그것들이 살아있는지, 그리고 그것들의 역할은 아직 밝혀지지 않았다고 경고합니다.[57][53]
출생 시와 그 이후에 급속하게 산모와 주변 환경에서 나온 박테리아가 아기의 내장을 식민지로 만듭니다.[10]박테리아의 정확한 공급원은 완전히 이해되지는 않지만, 산도, 다른 사람들(부모, 형제, 병원 직원), 모유, 음식, 그리고 유아가 상호작용하는 일반적인 환경을 포함할 수 있습니다.[58]연구에 따르면 질적으로 태어난 아기들의 마이크로바이옴은 제왕절개로 분만된 아기들의 마이크로바이옴과 현저한 차이가 있으며, 질적으로 태어난 아기들은 대부분의 내장 박테리아를 어머니로부터 얻은 반면,[59] 제왕절개로 태어난 아기들의 마이크로바이옴은 병원 환경과 연관된 박테리아를 더 많이 가지고 있습니다.
생애 첫 해 동안, 내장 식물의 구성은 일반적으로 단순하고 시간에 따라 크게 변화하며 개체 간에 동일하지 않습니다.[10]초기 박테리아 개체군은 일반적으로 기능성 혐기성 유기체이며, 연구자들은 이러한 초기 식민지가 장내의 산소 농도를 감소시키고, 이는 다시 박테로이도타, 방선균, 바실로타와 같은 의무적으로 혐기성 박테리아가 정착하고 번성하도록 한다고 생각합니다.[10]모유를 먹은 아기들은 비피도박테리아에 의해 지배되는데, 아마도 모유의 비피도박테리아 성장 인자의 함량 때문일 것이고, 모유가 프리바이오틱 성분을 가지고 있어서 건강한 박테리아 성장을 가능하게 합니다.[53][60]모유는 또한 아기의 면역 체계에 대한 내성과 조절을 돕기 위해 높은 수준의 면역글로불린 A를 포함하고 있습니다.[61]이와 대조적으로, 분유를 먹은 유아의 미생물 무리는 더 다양하며, 장내세균, 장구균, 비피도박테리아, 박테로이데스, 클로스트리디아의 수가 많습니다.[62]
내장 마이크로바이옴의 조성은 제왕절개, 항생제 및 조제분유 공급에 의해 변경될 수 있습니다.[53]항생제 치료를 받은 아이들은 안정성이 떨어지고 꽃 군락의 다양성이 떨어집니다.[63]제왕절개는 셀리악병, 천식, 그리고 제1형 당뇨병과 같은 질병의 위험을 높임으로써 자손의 전반적인 건강에 영향을 미치는 박테리아의 어미로부터 오는 전염에 지장을 주는 것으로 밝혀졌습니다.[53]이것은 건강한 내장 마이크로바이옴의 중요성을 더욱 증명합니다.마이크로바이옴 복원의 다양한 방법이 연구되고 있으며, 전형적으로 유아를 산모의 질 내용물에 노출시키는 것과 경구 프로바이오틱스를 포함하고 있습니다.[53]
기능들
1995년 장내 세균총에 대한 연구가 시작되었을 때,[64] 그것은 병원균에 대한 직접적인 방어, 장내 상피의 발달과 유지, 그리고 그곳에서의 항체 생성을 유도하는 역할에 의한 숙주 방어의 강화, 그리고 음식물에서 소화되지 않는 화합물을 대사하는 것과 같은 세 가지 중요한 역할을 하는 것으로 생각되었습니다; 그 후의 연구는 그것의 역할을 발견하였습니다.발달하는 면역 체계를 훈련하는 데 있어, 그리고 더 나아가 그것의 내장-뇌 축에서의 역할에 초점을 맞춘 연구.[65]
병원체 직접억제
장내 세균 군집은 그 공간을 완전히 식민지화하고, 이용 가능한 모든 영양소를 사용함으로써 병원균으로부터 방어하는 직접적인 역할을 하고, 그것과 영양분을 경쟁할 환영받지 못하는 유기체를 죽이거나 억제하는 화합물을 분비함으로써, 이 화합물들은 사이토카인이라고 알려져 있습니다.[66]다른 종류의 내장 박테리아는 다른 사이토카인의 생성을 일으킵니다.사이토카인은 감염에 대한 염증 반응을 시작하기 위해 우리 면역체계에 의해 생성되는 화학적 화합물입니다.장내 세균총의 파괴는 클로스트리듐 디피실과 같은 경쟁 생물체가 자리잡게 하고 그렇지 않으면 보류 상태로 유지되게 합니다.[66]
장 보호 및 면역체계의 발달
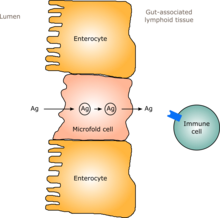
인간의 경우, 성인과 비슷한 내장 식물체가 태어난 지 1년에서 2년 이내에 형성됩니다.[10]장내 세균총이 형성됨에 따라, 장내의 안감인 장상피와 그것이 분비하는 장점막 장벽 또한 어느 정도 상식적인 미생물에 내성을 갖고 심지어 지지하는 방식으로 발전하고 병원성 미생물에도 장벽을 제공합니다.[10]구체적으로, 점막을 생성하는 유리잔 세포가 증식하고, 점막층이 두꺼워져 "친한" 미생물이 닻을 내리고 먹이를 줄 수 있는 외부 점막층과 이 유기체들조차 침투할 수 없는 내부층을 제공합니다.[10][11]또한 장상피의 일부를 형성하고 병원균을 검출하고 반응하는 장연관림프조직(GALT)의 발달은 장상피의 일부를 형성하며 장연관림프조직(Gut-associated lymphoid tissue,[10] GALT)이 발달하고 형성되는 시기에 발생합니다.발달하는 GALT는 내장 세균종에는 내성이 있지만 다른 미생물에는 내성이 없습니다.[10]GALT는 또한 일반적으로 유아가 노출된 음식, 음식의 소화물, 그리고 음식으로부터 생성된 내장 식물의 대사 물질(대사로부터 형성된 분자)에 대해 내성을 갖습니다.[10]
인간의 면역체계는 스스로를 보호하기 위해 면역체계가 염증을 일으키도록 유도할 수 있는 사이토카인을 만들어내고, 면역반응을 억제하여 항상성을 유지하고 모욕이나 부상 후에도 치유될 수 있도록 합니다.[10]장내 세균총에서 나타나는 다양한 박테리아 종들이 면역 체계를 이끌어 사이토카인을 선택적으로 생성할 수 있는 것으로 나타났습니다. 예를 들어, 박테로이데스 프라길리스와 일부 클로스트리디아 종들은 항염증 반응을 일으키는 것으로 보이며, 반면에 일부 분절된 필라멘트 박테리아는 염증성 사이토카인의 생성을 촉진합니다.[10][67]장 세균총은 또한 면역체계에 의한 항체 생성을 조절할 수 있습니다.[10][68]이 규정의 한 가지 기능은 B 세포가 IgA로 클래스 전환되도록 하는 것입니다.대부분의 경우 B 세포는 클래스 전환을 유도하기 위해 T 도우미 세포로부터의 활성화가 필요합니다; 그러나 다른 경로에서, 내장 세균은 장내 상피 세포에 의한 NF-kB 신호 전달을 야기하고, 이는 신호 분자가 추가로 분비되는 결과를 낳습니다.[69]이러한 신호전달 분자는 B 세포와 상호작용하여 IgA로 클래스 전환을 유도합니다.[69]IgA는 내장과 같은 점막 환경에서 사용되는 중요한 항체 유형입니다.IgA는 내장의 다양화를 돕고 염증 반응을 일으키는 박테리아를 없애는 데 도움을 주는 것으로 나타났습니다.[70]궁극적으로, IgA는 숙주와 내장 박테리아 사이의 건강한 환경을 유지합니다.[70]이러한 사이토카인과 항체는 내장 밖, 폐 및 기타 조직에 영향을 미칠 수 있습니다.[10]
면역체계는 또한 면역체계의 세포에 영향을 줄 수 있는 대사물질을 생산하는 내장 박테리아의 능력으로 인해 바뀔 수 있습니다.예를 들어, 짧은 사슬 지방산(SCFA)은 발효를 통해 일부 내장 세균에 의해 생성될 수 있습니다.[71]SCFA는 호중구, 호산구 그리고 호산구와 같은 선천적인 면역 세포의 생산의 급격한 증가를 자극합니다.[71]이 세포들은 감염의 확산을 제한하려고 노력하는 선천적 면역체계의 일부입니다.
신진대사
내장 세균총이 없다면 인체는 소화되지 않은 탄수화물 중 일부를 사용할 수 없을 것입니다. 왜냐하면 어떤 종류의 내장 세균총은 인체 세포가 특정 다당류를 분해하는 데 부족한 효소를 가지고 있기 때문입니다.[12]무균 환경에서 자라고 내장이 부족한 설치류는 정상 체중과 같은 무게를 유지하기 위해 30%의 칼로리를 더 섭취해야 합니다.[12]사람이 세균의 도움 없이 소화할 수 없는 탄수화물에는 유당불내증과 당알코올의 경우 유당처럼 몸이 소화해 흡수하지 못한 특정 녹말, 섬유질, 올리고당, 당류 등이 있으며, 내장에서 분비되는 점액, 단백질 등이 있습니다.[9][12]
박테리아는 당 분해 발효라고 불리는 발효의 한 형태에 의해 탄수화물을 짧은 사슬 지방산으로 바꿉니다.[36]제품에는 아세트산, 프로피온산 및 부티르산이 포함됩니다.[7][36]이러한 물질들은 숙주 세포에 의해 사용될 수 있으며, 에너지와 영양분의 주요 공급원을 제공합니다.[36]발효는 (신호전달에[76] 관여하여 평탄성을 야기할 수 있는) 가스와 젖산과 같은 유기산 또한 생성합니다.[7]아세트산은 근육에 의해 사용되고, 프로피온산은 ATP의 간 생성을 용이하게 하며, 부티르산은 내장 세포에 에너지를 공급합니다.[36]
또한 장 식물은 비오틴과 엽산과 같은 비타민을 합성하고 마그네슘, 칼슘, 철분을 포함한 식이 무기질의 흡수를 촉진합니다.[6][21]메타노브레비박터 스미스ii는 박테리아의 한 종이 아니라 도메인 고세균의 일원이기 때문에 독특하며, 인간 위장관 미생물 무리에서 메탄을 가장 많이 생성하는 고세균 종입니다.[77]
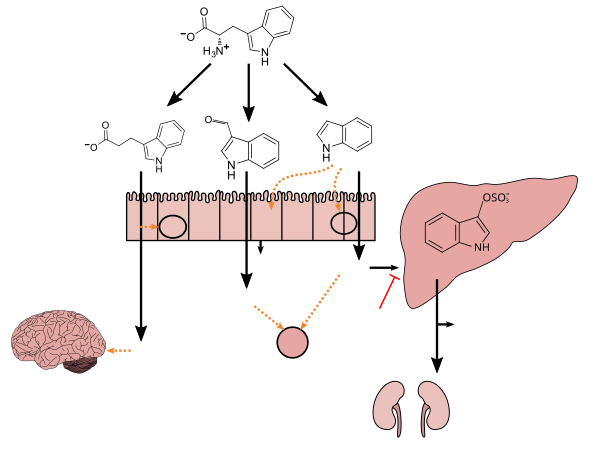
장내 미생물은 또한 몸에서 생성되지 않거나 소량 생성되는 비타민 K와 B12의 공급원 역할을 합니다.[78][79]
약미생물학
인간 메타게놈(즉, 한 개체의 유전적 구성과 그 개체의 몸 위 또는 몸 안에 존재하는 모든 미생물)은 개체 간에 상당한 차이가 있습니다.[80][81]인체 내 미생물 세포의 총 수(100조 이상)가 호모 사피엔스 세포(수십조 이상)를 크게 능가하기 때문에,[note 1][80][82] 약물과 개인의 마이크로바이옴 사이의 상호작용에 대한 상당한 가능성이 있습니다: 인간 마이크로바이옴의 구성을 변경하는 약물, 미생물 효소를 변경하는 약물에 의한 약물 대사.약물의 약동학적 프로파일, 그리고 약물의 임상적 효능과 독성 프로파일에 영향을 미치는 미생물 약물 대사.[80][81][83]
탄수화물 이외에도, 장내 미생물 무리는 또한 약물, 식물화학물질, 그리고 음식 독성 물질과 같은 다른 이종 생물체들을 대사시킬 수 있습니다.30개 이상의 약물이 장내 미생물 무리에 의해 대사되는 것으로 나타났습니다.[84]약물의 미생물 대사는 때때로 약물을 불활성화시킬 수 있습니다.[85]
약물대사에 기여
장내 미생물 무리는 약물, 특히 입으로 섭취하는 것들을 변형시키는 거대한 생화학적 능력을 가진 다양한 유전자들을 포함하는 농축된 공동체입니다.[86]장내 미생물 무리는 직간접적인 메커니즘을 통해 약물 대사에 영향을 미칠 수 있습니다.[87]직접적인 메커니즘은 투여된 약물의 화학적 구조를 변형시킬 수 있는 미생물 효소에 의해 매개됩니다.[88]반대로, 간접 경로는 사이토크롬 P450과 같은 숙주 대사 효소의 발현에 영향을 미치는 미생물 대사 물질에 의해 매개됩니다.[89][87]장내 미생물 무리가 약물의 약동학과 생체이용률에 미치는 영향은 수십 년 전에 조사되었습니다.[90][91][92]이러한 효과는 다양할 수 있는데, 예를 들어 로바스타틴과 같은 비활성 약물을 활성화시키거나,[93] 디곡신과[94] 같은 활성 약물을 비활성화시키거나, 이리노테칸과 같은 약물 독성을 유도할 수 있습니다.[95]그 이후로 장내 미생물 무리가 많은 약물의 약동학에 미치는 영향에 대해 집중적으로 연구되었습니다.[96][86]
사람의 내장 미생물은 투여된 약물이 사람에게 미치는 영향을 조절하는 데 중요한 역할을 합니다.직접적으로 장내 미생물 무리는 탈카복실화효소 및 탈크록실화효소에 의한 L-도파의 미생물 생물 전환과 같은 약물의 대사 능력을 가진 일련의 효소를 합성하여 방출할 수 있습니다.[88]반대로, 장내 미생물 무리는 숙주 약물 대사를 조절함으로써 약물의 대사를 변화시킬 수도 있습니다.이 메커니즘은 미생물 대사산물에 의해 매개되거나 숙주 대사효소의 발현을 변화시키는 숙주 대사산물을 변형시킴으로써 매개될 수 있습니다.[89]
많은 연구에서 장내 미생물 무리에 의한 50가지 이상의 약물의 대사를 증명했습니다.[96][87]예를 들어, 락톤 프로드러그인 로바스타틴(콜레스테롤 강하제)은 활성 산 하이드록실화된 대사산물을 형성하는 인간 내장 마이크로바이오타에 의해 부분적으로 활성화됩니다.[93]반대로, 디곡신(울혈성 심부전을 치료하는 데 사용되는 약물)은 장내 미생물 무리(즉, 에그셀라 란타)에 의해 비활성화됩니다.[97]에거셀란타는 디곡신에 의해 상향 조절되고 디곡신 비활성화와 관련된 사이토크롬-인코딩 오페론을 가지고 있습니다.[97]장내 미생물 무리는 또한 이리노테칸과 같은 화학 치료제의 효능과 독성을 조절할 수 있습니다.[98]이 효과는 위장 독성을 유발하는 이리노테칸의 활성 형태를 회복하는 마이크로바이옴 암호화된 β-글루쿠로니다아제 효소로부터 유도됩니다.[99]
이차대사물질
장내 미생물 무리를 구성하는 대략적인 박테리아의 수는 10-10개1314 정도로 인간보다 방대한 화학 반응을 할 수 있는 유전적 잠재력을 가지고 있습니다.[100]이 공동체는 섬유와 같은 식이 식품, 인돌 또는 담즙산과 같은 내인성 생물학적 화합물의 대사 전환으로부터 때때로 생성되는 뚜렷한 2차 대사 물질을 생산하는 거대한 생화학적 능력을 가지고 있습니다.[101][102][103]미생물 대사산물, 특히 단쇄 지방산(SCFAs)과 2차 담즙산(BAs)은 건강과 질병 상태에서 인간에게 중요한 역할을 합니다.[104][105][106]
장내 미생물 무리에 의해 생성되는 가장 중요한 세균 대사 물질 중 하나는 2차 담즙산(BAs)입니다.[107]이러한 대사 물질은 콜산(CA) 및 체노데옥시콜산(CDCA)과 같은 1차 담즙산이 각각 2차 담즙산(BA) 리토콜산(LCA) 및 디옥시콜산(DCA)으로 세균성 생물 변환에 의해 생성됩니다.[108]간세포에 의해 합성되어 담낭에 저장되는 일차 담즙산은 소수성 특징을 갖습니다.이러한 대사 물질들은 이어서 장내 미생물 무리에 의해 소수성이 증가된 2차 대사 물질로 대사됩니다.[108]Bacteroides, Firmicutes 및 Actinobacteria와 같은 내장 미생물 계통에 걸쳐 보존되는 담즙 염 가수분해효소(BSH)는 2차 담즙산 대사의 첫 단계를 담당합니다.[108]DCA 및 LCA와 같은 2차 담즙산(BA)은 클로스트리듐 디피실 발아 및 발아를 모두 억제하는 것으로 입증되었습니다.[106]
이생체증
장내 미생물 무리는 장내 항상성 유지에 중요합니다.장암의 발생은 천연 미세식물의 불균형과 관련이 있습니다.[109]2차 담즙산 디옥시콜산은 장내 발암을 증가시키는 미생물 군집의 변화와 관련이 있습니다.[109]대장이 2차 담즙산에 노출되는 정도가 증가하면 DNA 손상을 일으킬 수 있고, 이러한 손상은 대장의 세포에 발암성 돌연변이를 일으킬 수 있습니다.[110]소장에 비해 상대적으로 밀도가 낮은 대장(ml당 10개122 내외)에 비해 대장 내 세균의 밀도가 높은 것이 대장 내 암 발생률이 소장에 비해 10배 이상 높은 원인일 수 있습니다.[110]
장뇌축
장뇌축은 위장관과 중추신경계 사이에서 일어나는 생화학적 신호입니다.[65]이 용어는 상호작용에서 내장 세균총의 역할을 포함하도록 확장되었습니다. "마이크로바이옴-장-뇌 축"이라는 용어는 때때로 내장 세균총을 포함하는 패러다임을 명시적으로 설명하는 데 사용됩니다.[65][111][112]광범위하게 정의된, 내장-뇌 축은 중추 신경계, 시상하부-뇌하수체-부신축(HPA 축)을 포함한 신경 내분비 및 신경 면역계, 장 신경계를 포함한 자율 신경계의 교감 및 부교감 팔, 미주 신경 및 내장 미생물을 포함합니다.[65][112]
2016년부터 체계적인 검토를 통해 상업적으로 이용 가능한 특정 종류의 프로바이오틱스 박테리아로 수행된 전임상 및 소규모 인간 실험을 조사한 결과 시험된 실험 중 비피도박테리움 및 락토바실러스 속(B. longum, B. breve, B. infartis, L. helveticus, L. rhamnosus, L. plantarum, L. casei),특정 중추신경계 질환에 유용할 수 있는 [14]가장 잠재력이 있었습니다
미생물 균형의 변화
항생제 사용의 영향
예를 들어 광범위 항생제를 복용함으로써 내장 박테리아의 수를 바꾸는 것은 숙주의 건강과 음식 소화 능력에 영향을 미칠 수 있습니다.[113]항생제는 장을 직접 자극하거나, 미생물의 수준을 바꾸거나, 병원성 박테리아가 자라게 함으로써 항생제와 관련된 설사를 유발할 수 있습니다.[7]항생제의 또 다른 해로운 영향은 항생제 사용 후 발견되는 항생제 내성균의 수가 증가하는 것인데, 이것들은 숙주에 침입했을 때 항생제로 치료하기 어려운 병을 유발합니다.[113]
장내 미생물 무리의 수와 종을 바꾸면 탄수화물을 발효시키고 담즙산을 대사하는 신체의 능력을 감소시키고 설사를 유발할 수 있습니다.분해되지 않은 탄수화물은 너무 많은 물을 흡수하여 콧물이 나거나 장내 미생물에 의해 생성된 SCFA가 부족하면 설사를 유발할 수 있습니다.[7]
토종 박테리아 종의 감소는 또한 C. difficile와 Salmonella kedougou와 같은 해로운 종의 성장을 억제하는 능력을 방해하고, 비록 그들의 과도한 성장은 부수적이고 설사의 진정한 원인이 아닐 수 있지만, 이 종들은 감당할 수 없게 될 수 있습니다.[6][7][113]C. difficile 감염에 대한 새로운 치료 프로토콜은 기증자 대변의 대변 미생물 이식을 포함합니다(변 이식 참조).[114]치료에 대한 초기 보고에서는 성공률이 90%이며 부작용은 거의 없습니다.효능은 박테로이드의 박테리아 균형을 회복하고 박테리아 등급을 굳히는 것에서 비롯된다고 추측됩니다.[115]
장내 마이크로바이옴의 구성은 항생제 사용뿐만 아니라 장내 허혈, 식사 실패, 면역 손상 등의 요인으로 인해 심각한 질병에서도 변화합니다.이로 인한 부정적인 영향으로 병원성 박테리아만 죽이고 건강한 박테리아를 다시 만들 수 있는 치료법인 선택적인 소화관 오염 제거에 관심을 갖게 되었습니다.[116]
항생제는 위장관의 미생물 무리를 변화시키고, 이것은 지역사회 내 대사 상호작용을 변화시키고, 탄수화물을 사용함으로써 칼로리 섭취를 변화시키며, 전세계적으로 숙주 대사, 호르몬 및 면역 항상성에 영향을 미칠 수 있습니다.[117]
락토바실러스 종(Lactobacillus species)이 포함된 프로바이오틱스를 복용하는 것이 항생제 관련 설사를 예방하는 데 도움이 될 수 있다는 합리적인 증거가 있으며, 사카로마이세스(Saccharomyces boulardii)와 함께 프로바이오틱스를 복용하는 것이 전신 항생제 치료 후 클로스트리디움 디피실 감염을 예방하는 데 도움이 될 수 있습니다.[118]
임신
임신이 진행됨에 따라 여성의 장내 미생물 무리가 변하는데, 당뇨병과 같은 대사증후군과 유사한 변화가 나타납니다.장내 미생물 무리의 변화는 나쁜 영향을 주지 않습니다.신생아의 내장 미생물은 산모의 초산 샘플과 비슷합니다.마이크로바이옴의 다양성은 특정 종의 수가 증가함에 따라 1기부터 3기까지 감소합니다.[53][119]
프로바이오틱스, 프리바이오틱스, 신바이오틱스, 제약바이오틱스
생균제는 섭취했을 때 건강상의 이점을 제공한다고 믿어지는 미생물입니다.[120][121]장내 미생물 무리(microbiota)와 관련하여, 프리바이오틱스(prebiotics)는 전형적으로 소화되지 않는, 섬유 화합물로서 위장관의 상부를 통과하고, 이들에 대한 기질로서 작용함으로써 유리한 장내 세균 무리의 성장 또는 활성을 자극하는 것입니다.[36][122]
신바이오틱스란 프로바이오틱스와 프리바이오틱스가 시너지 효과를 내는 형태로 결합된 식품 성분이나 식이보충제를 말합니다.[123]
"약생생물"이란 다양한 의미로 사용되는 용어로 프로바이오틱스, 프리바이오틱스 또는 시노틱스의 약학적 제제(의약품으로서 규제 승인을 받을 수 있는 표준화된 제조);[124] 유전공학적으로 설계되었거나 최상의 성능을 발휘할 수 있도록 최적화된 프로바이오틱스(선반 수명, 소화관에서의 생존 등);[125]내장 세균 대사의 자연적 생성물(vitamins 등).
과민성 장증후군과[127] 만성 특발성 변비에 프로바이오틱스 균종을 사용한 치료가 효과적일 수 있다는 증거가 있습니다.증상 감소를 초래할 가능성이 가장 높은 생물은 다음과 같습니다.
- Bifidobacterium breve
- 비피도박테리움 인판티스
- 장구균 페이시움
- 락토바실러스 플란타룸
- 락토바실러스 류테리
- 락토바실러스 람노수스
- 유산균 살리바리우스
- 프로피오니박테리움 프로이덴라이치
- 스트렙토코커스테로필루스[128][129][130]
분변부동
약 10-15%의 사람들의 배설물은 화장실 물('떠다니는 물')에 지속적으로 떠 있고, 나머지는 가라앉는 배설물('싱크대')을 생성하고 가스가 생성되어 배설물이 떠다니게 됩니다.[131]기존의 쥐들이 종종 '떠다니는 동물'을 생산하는 반면, 무균생식 세균성 쥐는 (무균 격리기에서 번식한) 내장 미생물 무리가 '싱크대'를 생산하고, 무균생식 쥐의 내장 미생물 무리는 미생물 바이오매스로의 음식 변형과 '싱크대'를 '떠다니는 동물'로 바꾸는 다수의 가스 생성 박테리아 종의 농축으로 이어집니다.[132]
조사.
비항생제 약물이 인간의 내장 관련 박테리아에 영향을 미칠 수 있는지 여부에 대한 테스트는 40개의 내장 박테리아 균주를 대상으로 1000개 이상의 시판된 약물에 대한 체외 분석을 통해 수행되었으며, 이는 약물의 24%가 적어도 하나의 박테리아 균주의 성장을 억제했음을 보여줍니다.[133]
운동의 효과
장내 미생물 무리와 운동은 최근 상호 연관성이 있는 것으로 나타났습니다.적당한 운동과 격렬한 운동 모두 일반적으로 지구력 운동 선수들의 훈련 방법의 일부이지만, 그것들은 건강에 다른 영향을 줍니다.장내 미생물 무리와 지구력 스포츠의 상호 연관성은 운동 강도와 훈련 상태에 따라 달라집니다.[134]
질병의 역할
소화관의 박테리아는 다양한 방법으로 질병의 원인이 되고 영향을 받을 수 있습니다.어떤 종류의 박테리아의 존재 또는 과량은 염증성 장질환과 같은 염증성 질환의 원인이 될 수 있습니다.[6]게다가, 내장 세균총의 특정 구성원들로부터의 대사산물들은 비만이나 대장암과 같은 장애들에 기여하면서 숙주 신호 전달 경로에 영향을 미칠 수 있습니다.[6]대안적으로, 내장 상피가 파괴되는 경우, 내장 세균총 성분이 다른 숙주 구획으로 침입하는 것은 패혈증으로 이어질 수 있습니다.[6]
궤양
헬리코박터 파일로리 감염은 박테리아가 위 상피 내벽에 침투할 때 위궤양의 형성을 시작할 수 있고, 그 후 염증성 식세포 반응을 일으킬 수 있습니다.[135]결과적으로, 그 염증은 위에 과도한 염산을 방출하고 보호 점액을 덜 생성하는 두정 세포를 손상시킵니다.[136]위산이 세포의 방어성을 압도하여 내인성 프로스타글란딘 합성을 억제하고 점액과 중탄산염 분비를 감소시키며 점막 혈류를 감소시키고 손상에 대한 저항력을 감소시킴으로써 궤양을 유발하는 위벽 손상이 발생합니다.[136]위 라이닝의 감소된 보호 특성은 위산, 펩신 및 담즙 염에 의한 추가 부상 및 궤양 형성에 대한 취약성을 증가시킵니다.[135][136]
장천공
일반적으로 일반적인 박테리아는 내장에서 추출할 경우 숙주에게 해를 끼칠 수 있습니다.[10][11]세균이 점막 내벽을 통해 내장을 떠날 때 발생하는 전위는 다양한 질병에서 발생할 수 있습니다.[11]내장에 구멍이 뚫리면 세균이 간질을 침범해 치명적인 감염을 일으킬 수 있습니다.[5]: 715
염증성 장질환
염증성 장질환의 두 가지 주요 유형인 크론병과 궤양성 대장염은 내장의 만성 염증성 질환입니다. 이 질환들의 원인은 알려지지 않았고 내장 세균과 숙주와의 관계에 문제가 있습니다.[13][137][138][139]또한, 장-뇌 축과 장-뇌 축의 상호작용이 IBD에 영향을 미치는 것으로 보입니다.시상하부-뇌하수체-adren축을 통해 생리적 스트레스가 매개되어 장상피와 내장 세균총에 대한 변화를 차례로 방출하며 장신경계와 미주신경에서 신호전달을 유발하는 인자와 대사물질을 차례로 방출합니다.
염증성 장질환이 있는 사람은 건강한 사람에 비해 내장 세균의 다양성이 현저히 감소하는 것으로 나타나고, 궤양성 대장염이 있는 사람은 프로테오박테리아와 액티노박테리아가 우세한 것으로 보이며, 크론병이 있는 사람은 엔테로코커스 페시움과 여러 프로테오박테리아가 과다하게 발현되는 것으로 보입니다.[4]
락토바실리와 비피도박테리아로 활생균을 복용하여 장내 세균 불균형을 교정하는 것이 IBD의 내장통증과 내장염증을 감소시킬 수 있다는 합리적인 증거가 있습니다.[118]
과민성장증후군
과민성 장증후군은 스트레스와 만성적인 HPA축 활성화의 결과입니다; 그것의 증상은 복통, 배변의 변화, 그리고 염증성 사이토카인의 증가를 포함합니다.전체적으로, 연구들은 과민성 장증후군 개체들에서 내강과 점막 미생물 무리가 변화한다는 것을 발견했고, 이러한 변화는 설사나 변비와 같은 자극의 종류와 관련이 있을 수 있습니다.또한 분변 락토바실리와 비피도박테리아의 수준이 낮고 대장균과 같은 기능성 혐기성 세균의 수준이 높으며 피리큐테스의 비율이 증가하여 마이크로바이옴의 다양성이 감소합니다.박테로이데스.[112]
기타 염증성 또는 자가면역 상태
알레르기, 천식, 당뇨병은 원인을 알 수 없는 자가면역 및 염증성 질환이지만 내장 세균의 불균형 및 숙주와의 관계와 관련이 있습니다.[13]2016년 기준으로 내장 세균총의 변화가 이러한 자가면역 및 염증성 질환을 유발하는지 또는 이에 대한 적응의 산물인지는 명확하지 않았습니다.[13][140]
천식
선진국에서 천식의 유병률이 증가하는 것을 설명하기 위해 두 가지 가설이 제기되었습니다.위생 가설은 선진국의 어린이들이 충분한 미생물에 노출되지 않기 때문에 보호 역할을 하는 특정 박테리아 분류군의 유병률이 더 낮을 수 있다고 가정합니다.[141]두 번째 가설은 통곡물과 섬유질이 부족하고 단당류가 과다한 서양식 식단에 초점을 맞추고 있습니다.[13]두 가설 모두 면역 조절에서 짧은 사슬 지방산(SCFA)의 역할에 수렴합니다.이러한 박테리아 발효 대사 물질은 천식의 유발을 막는 면역 신호 전달에 관련되어 있고 SCFA 수치가 낮으면 질병과 관련이 있습니다.[141][142]Lachnospira, Veillonella, Rothia 및 Faecalibacterium과 같은 보호가 부족한 속은 SCFA 수준 감소와 관련이 있습니다.[141]게다가, SCFAs는 서양의 패턴 식단에서 낮은 섬유의 박테리아 발효의 산물입니다.[13][142]SCFAs는 내장 세균과 면역 장애 사이의 연관성을 제공하며, 2016년 현재 이는 활발한 연구 분야였습니다.[13]음식과 다른 알레르기의 증가에 대해서도 비슷한 가설들이 제기되고 있습니다.[143]
제1형 당뇨병
장내 미생물 무리와 제1형 당뇨병 사이의 연관성은 또한 부틸레이트와 아세트산과 같은 SCFAs와도 연관되어 있습니다.박테리아 발효로 인한 부틸산염과 초산염을 생산하는 식단은 T 발현을reg 증가시킵니다.[144]T세포는reg 이펙터 T세포를 과소 조절하고, 이것은 결과적으로 내장의 염증 반응을 감소시킵니다.[145]부틸산염은 대장 세포의 에너지원입니다.따라서 뷰틸레이트 yielding 다이어트는 팽팽한 접합부 형성을 위한 충분한 에너지를 제공함으로써 내장 투과성을 감소시킵니다.또한, 부틸산염은 인슐린 저항성을 감소시키는 것으로 밝혀졌으며, 부틸산염을 생성하는 미생물이 적은 내장 공동체가 제2형 당뇨병에 걸릴 가능성을 증가시킬 수 있음을 시사합니다.[147]하지만 젖산염을 생산하는 식단은 또한 잠재적인 대장암 억제 효과를 가질 수 있습니다.[146]
비만대사증후군
소화 과정에서 중요한 역할을 하기 때문에 장내 세균총은 비만과 대사증후군에도 관여되어 있습니다. 서양식 식단은 장내 세균총의 변화를 주도하고 유지하는 것으로 보이며, 이는 결국 음식에서 얼마나 많은 에너지가 파생되고 어떻게 에너지가 사용되는지를 변화시킵니다.[139][148]서양식 식단에서 종종 부족한 건강 식단의 한 측면은 섬유질과 다른 복합 탄수화물로 건강한 내장 식물체가 번성해야 합니다. 서양식 식단에 반응하여 내장 식물체의 변화는 비만과 대사 증후군에 기여할 수 있는 내장 식물체에 의해 생성되는 에너지의 양을 증가시키는 것으로 보입니다.[118]또한 미생물 무리가 미생물 무리의 선호에 따라 먹는 행동에 영향을 미치고, 이는 숙주가 더 많은 음식을 소비하게 되어 결국 비만을 초래할 수 있다는 증거도 있습니다.일반적으로 내장 마이크로바이옴 다양성이 높아지면 마이크로바이옴은 다른 마이크로바이옴과 경쟁하는 데 에너지와 자원을 소비하고 숙주를 조작하는 데는 적게 소비할 것으로 관찰되었습니다.그 반대는 내장 마이크로바이옴의 다양성이 더 낮고, 이 마이크로바이오타들은 숙주의 음식에 대한 갈망을 만들어 내기 위해 함께 일할 수 있습니다.[47]
또한, 간은 당생성과 포도당신생성의 대사경로를 통해 포도당의 흡수와 저장 사이의 균형을 유지함으로써 혈당 항상성에 있어 중요한 역할을 합니다.장 지질은 내장-뇌-간 축을 포함하는 포도당 항상성을 조절합니다.지질을 직접적으로 상부에 투여하는 것은 상부에 있는 긴 사슬의 지방 아실-조효소 A(LCFA-CoA) 수치를 증가시키고, 대하변막하 질개술 또는 내장 탈카페인화 하에서도 포도당 생성을 억제합니다.이것은 뇌와 내장 사이의 신경 연결을 방해하고 상부 장 지질의 포도당 생성을 억제하는 능력을 차단합니다.내장-뇌-간 축 및 내장 마이크로바이오타 조성물은 간에서의 포도당 항상성을 조절할 수 있고 비만 및 당뇨병을 치료할 수 있는 잠재적인 치료 방법을 제공할 수 있습니다.[149]
장 세균총이 비만의 발달을 촉진할 수 있는 피드백 루프에서 기능할 수 있는 것처럼, 칼로리 섭취를 제한하는 것(즉, 다이어트)이 장 세균총의 구성에 변화를 가져올 수 있다는 증거가 있습니다.[139]
간질환
간은 문맥에 의해 직접 공급되기 때문에 장상피와 장점막 장벽을 통과하는 것은 무엇이든 간으로 들어가고, 거기에서 생성된 사이토카인도 마찬가지입니다.[150]장내 세균총의 이질은 간경변증 및 비알코올성 지방간 질환의 발생과 관련이 있습니다.[150]
암
박테로이데스(Bacteroides)와 클로스트리디움(Clostridium)과 같은 박테리아의 일부 속은 종양 성장 속도의 증가와 관련이 있는 반면, 락토바실러스(Lactobacillus)와 비피도박테리아(Bifidobacteria)와 같은 다른 속은 종양 형성을 막는 것으로 알려져 있습니다.[6]2017년 12월 현재 장내 미생물 무리가 PD-1 억제제에 대한 반응을 매개할 수 있다는 예비적이고 간접적인 증거가 있었습니다; 그 메커니즘은 알려지지 않았습니다.[151]
비만한 사람들은 간세포암을 포함한 여러 종류의 암에 걸릴 위험이 더 높습니다.이러한 위험의 증가는 내장 미생물에 의한 발암성 담즙산의 증가와 관련이 있을 수 있습니다.[152][153]식이요법이나 유전적 요인에 의한 비만은 DNA 손상을 일으키는 대사산물인 담즙산 디옥시콜산의 수치를 증가시키는 내장 미생물의 변화와 관련이 있습니다.[152][153]
신경정신과
내장 세균총과 신경 정신적 문제의 관계에 대한 관심은 2014년 연구에서 세균이 없는 쥐가 비GF 실험실 쥐에 비해 스트레스에 대해 과장된 HPA축 반응을 나타냈다는 것을 보여줌으로써 촉발되었습니다.[65]2016년 1월 현재, 내장-뇌 축에서 내장 세균의 역할에 대한 연구는 대부분 동물을 대상으로 이루어졌거나, 내장 세균이 생성할 수 있는 다양한 신경 활성 화합물을 특성화하거나, 다양한 정신 의학적 및 신경학적 차이를 가진 사람들 간의 차이를 측정하는 인간과의 연구 또는 내장의 변화에 대한 연구가 이루어졌습니다.스트레스에 반응하는 세균총, 또는 다양한 생균제의 효과를 측정하는 dubbed "이러한 맥락에서 psych바이오틱스"는 일반적으로 작았고 일반화될 수 없었습니다. 내장 세균총의 변화가 질병의 결과인지, 질병의 원인인지, 아니면 내장-뇌 축의 가능한 피드백 루프의 몇 가지 모두에서 발생하는지는 불분명했습니다.
내장-뇌 축은 정신 건강에도 영향을 미칠 수 있는 역동적인 관계입니다.내장 마이크로바이옴은 일부 신경 전달 물질, 트립토판(아미노산)의 대사, 그리고 짧은 사슬 지방산의 생성을 담당합니다.내장 마이크로바이옴이 불균형해질 수 있는 이유는 식사, 지리, 스트레스(HPA축), 그리고 의약품 등 여러 가지가 있습니다.불균형은 이질과 염증을 유발할 수 있고, 이것은 우울증과 불안으로 이어질 수 있습니다.[154]
2016년의 체계적인 검토는 상업적으로 이용 가능한 특정 종류의 프로바이오틱스 박테리아로 수행된 전임상 및 소규모 인간 실험을 조사했으며 시험된 실험들 중에서 Bifidobacterium과 Lactobacillus속(B. longum, B. breve, B. infantis, L. helveticus, L. rhamnosus, L. plantarum, L. casei)이 m을 가지고 있음을 발견했습니다.ost potential은 특정한 중추신경계 장애에 유용합니다.[14]
기타동물
인간 내장 마이크로바이옴의 구성은 다른 유인원들의 그것과 비슷합니다.하지만, 인간의 내장 생물체는 판에서 진화적으로 분리된 이후 다양성이 감소하고 구성이 변화했습니다.[155]인간은 동물성 단백질과 지방이 많은 식단과 관련된 박테리아 문인 박테로이데테스(Bacteroidetes)가 증가하고 복합 식물 다당류를 발효시키는 그룹인 메타노브레비박터(Methanobrevibacter)와 섬유로박터(Fibrobacter)가 감소하는 것을 보여줍니다.[155]이러한 변화들은 판에서 진화적으로 분리된 이후 인류가 겪어온 복합적인 식생활, 유전적, 문화적 변화들의 결과입니다.[citation needed]
인간과 척추동물 외에도 일부 곤충들은 영양학적으로 중요한 역할을 하는 복잡하고 다양한 내장 미생물을 가지고 있습니다.[2]흰개미와 관련된 미생물 군집은 개체 무게의 대부분을 구성할 수 있으며 리그노셀룰로오스의 소화와 질소 고정에 중요한 역할을 수행합니다.[156]이들 군집은 숙주에 특화되어 있으며, 밀접하게 관련된 곤충 종들은 내장 미생물 구성에서 유사한 유사점을 공유합니다.[157][158]바퀴벌레에서 장내 미생물 무리는 접종체와 관계없이 결정론적인 방식으로 조립되는 것으로 나타났습니다.[159] 이 숙주 특이적 조립의 이유는 아직 명확하지 않습니다.흰개미와 바퀴벌레와 같은 곤충과 관련된 박테리아 군집은 주로 먹이인 힘의 조합에 의해 결정되지만, 숙주 계통발생도 계통발생 선택에 역할을 할 수 있다는 징후가 있습니다.[157][158]
51년 이상 동안, 저용량의 항균제의 투여는 체중 증가를 증가시키기 위해 농장 동물의 성장을 촉진한다고 알려져 왔습니다.[117]
쥐를 대상으로 수행된 연구에서 다양한 항생제의 치료 미만 용량으로 치료된 동물에서 피르미쿠테스와 라흐노스피라과의 비율이 크게 증가했습니다.GI tract에서 대변의 열량과 소쇄지방산(SCFAs)의 농도를 분석함으로써, 미생물의 구성 변화가 소화되지 않는 구성 성분으로부터 열량을 추출하는 능력의 증가와 SCFAs의 생산 증가로 이어진다는 결론을 내렸습니다.이러한 발견은 항생제가 GI 마이크로바이옴의 구성뿐만 아니라 특히 SCFA와 관련하여 대사 능력을 교란시킨다는 증거를 제공합니다.[117]
참고 항목
메모들
참고문헌
- ^ Moszak, M; Szulińska, M; Bogdański, P (15 April 2020). "You Are What You Eat-The Relationship between Diet, Microbiota, and Metabolic Disorders-A Review". Nutrients. 12 (4): 1096. doi:10.3390/nu12041096. PMC 7230850. PMID 32326604. S2CID 216108564.
- ^ a b Engel, P.; Moran, N. (2013). "The gut microbiota of insects–diversity in structure and function". FEMS Microbiology Reviews. 37 (5): 699–735. doi:10.1111/1574-6976.12025. PMID 23692388.
- ^ Segata, N; Boernigen, D; Tickle, TL; Morgan, XC; Garrett, WS; Huttenhower, C (14 May 2013). "Computational meta'omics for microbial community studies". Molecular Systems Biology. 9: 666. doi:10.1038/msb.2013.22. PMC 4039370. PMID 23670539.
- ^ a b c Saxena, R.; Sharma, V.K (2016). "A Metagenomic Insight Into the Human Microbiome: Its Implications in Health and Disease". In Kumar, D.; S. Antonarakis (eds.). Medical and Health Genomics. Elsevier Science. p. 117. doi:10.1016/B978-0-12-420196-5.00009-5. ISBN 978-0-12-799922-7.
- ^ a b c d e f g h i j k l m Sherwood, Linda; Willey, Joanne; Woolverton, Christopher (2013). Prescott's Microbiology (9th ed.). New York: McGraw Hill. pp. 713–21. ISBN 9780073402406. OCLC 886600661.
- ^ a b c d e f g h i j k l m n o p q Guarner, F; Malagelada, J (2003). "Gut flora in health and disease". The Lancet. 361 (9356): 512–19. doi:10.1016/S0140-6736(03)12489-0. PMID 12583961. S2CID 38767655.
- ^ a b c d e f g h i j k Beaugerie, Laurent; Petit, Jean-Claude (2004). "Antibiotic-associated diarrhoea". Best Practice & Research Clinical Gastroenterology. 18 (2): 337–52. doi:10.1016/j.bpg.2003.10.002. PMID 15123074.
- ^ a b Stephen, A. M.; Cummings, J. H. (1980). "The Microbial Contribution to Human Faecal Mass". Journal of Medical Microbiology. 13 (1): 45–56. doi:10.1099/00222615-13-1-45. PMID 7359576.
- ^ a b c d e Quigley, E. M (2013). "Gut bacteria in health and disease". Gastroenterology & Hepatology. 9 (9): 560–9. PMC 3983973. PMID 24729765.
- ^ a b c d e f g h i j k l m n o p Sommer, Felix; Bäckhed, Fredrik (2013). "The gut microbiota — masters of host development and physiology". Nature Reviews Microbiology. 11 (4): 227–38. doi:10.1038/nrmicro2974. PMID 23435359. S2CID 22798964.
- ^ a b c d Faderl, Martin; Noti, Mario; Corazza, Nadia; Mueller, Christoph (2015). "Keeping bugs in check: The mucus layer as a critical component in maintaining intestinal homeostasis". IUBMB Life. 67 (4): 275–85. doi:10.1002/iub.1374. PMID 25914114. S2CID 25878594.
- ^ a b c d e f Clarke, Gerard; Stilling, Roman M; Kennedy, Paul J; Stanton, Catherine; Cryan, John F; Dinan, Timothy G (2014). "Minireview: Gut Microbiota: The Neglected Endocrine Organ". Molecular Endocrinology. 28 (8): 1221–38. doi:10.1210/me.2014-1108. PMC 5414803. PMID 24892638.
- ^ a b c d e f g h Shen, Sj; Wong, Connie HY (2016). "Bugging inflammation: Role of the gut microbiota". Clinical & Translational Immunology. 5 (4): e72. doi:10.1038/cti.2016.12. PMC 4855262. PMID 27195115.
- ^ a b c Wang, Huiying; Lee, In-Seon; Braun, Christoph; Enck, Paul (2016). "Effect of Probiotics on Central Nervous System Functions in Animals and Humans: A Systematic Review". Journal of Neurogastroenterology and Motility. 22 (4): 589–605. doi:10.5056/jnm16018. PMC 5056568. PMID 27413138.
- ^ a b c d e f Sears, Cynthia L. (2005). "A dynamic partnership: Celebrating our gut flora". Anaerobe. 11 (5): 247–51. doi:10.1016/j.anaerobe.2005.05.001. PMID 16701579.
- ^ Shapira, Michael (2016-07-01). "Gut Microbiotas and Host Evolution: Scaling Up Symbiosis". Trends in Ecology & Evolution. 31 (7): 539–549. doi:10.1016/j.tree.2016.03.006. ISSN 0169-5347. PMID 27039196.
- ^ Lozupone, Catherine A.; Stombaugh, Jesse I.; Gordon, Jeffrey I.; Jansson, Janet K.; Knight, Rob (2012). "Diversity, stability and resilience of the human gut microbiota". Nature. 489 (7415): 220–30. Bibcode:2012Natur.489..220L. doi:10.1038/nature11550. PMC 3577372. PMID 22972295.
- ^ Qin, Junjie; Li, Ruiqiang; Raes, Jeroen; Arumugam, Manimozhiyan; Burgdorf, Kristoffer Solvsten; Manichanh, Chaysavanh; Nielsen, Trine; Pons, Nicolas; Levenez, Florence; Yamada, Takuji; Mende, Daniel R.; Li, Junhua; Xu, Junming; Li, Shaochuan; Li, Dongfang; Cao, Jianjun; Wang, Bo; Liang, Huiqing; Zheng, Huisong; Xie, Yinlong; Tap, Julien; Lepage, Patricia; Bertalan, Marcelo; Batto, Jean-Michel; Hansen, Torben; Le Paslier, Denis; Linneberg, Allan; Nielsen, H. Bjørn; Pelletier, Eric; Renault, Pierre (2010). "A human gut microbial gene catalogue established by metagenomic sequencing". Nature. 464 (7285): 59–65. Bibcode:2010Natur.464...59.. doi:10.1038/nature08821. PMC 3779803. PMID 20203603.
- ^ Shanahan, Fergus (2002). "The host–microbe interface within the gut". Best Practice & Research Clinical Gastroenterology. 16 (6): 915–31. doi:10.1053/bega.2002.0342. PMID 12473298.
- ^ Tap, Julien; Mondot, Stanislas; Levenez, Florence; Pelletier, Eric; Caron, Christophe; Furet, Jean-Pierre; Ugarte, Edgardo; Muñoz-Tamayo, Rafael; Paslier, Denis L. E.; Nalin, Renaud; Dore, Joel; Leclerc, Marion (2009). "Towards the human intestinal microbiota phylogenetic core". Environmental Microbiology. 11 (10): 2574–84. doi:10.1111/j.1462-2920.2009.01982.x. PMID 19601958.
- ^ a b O'Hara, Ann M; Shanahan, Fergus (2006). "The gut flora as a forgotten organ". EMBO Reports. 7 (7): 688–93. doi:10.1038/sj.embor.7400731. PMC 1500832. PMID 16819463.
- ^ Khanna, Sahil; Tosh, Pritish K (2014). "A Clinician's Primer on the Role of the Microbiome in Human Health and Disease". Mayo Clinic Proceedings. 89 (1): 107–14. doi:10.1016/j.mayocp.2013.10.011. PMID 24388028.
- ^ a b Cui, Lijia; Morris, Alison; Ghedin, Elodie (2013). "The human mycobiome in health and disease". Genome Medicine. 5 (7): 63. doi:10.1186/gm467. PMC 3978422. PMID 23899327.
- ^ Erdogan, Askin; Rao, Satish S. C (2015). "Small Intestinal Fungal Overgrowth". Current Gastroenterology Reports. 17 (4): 16. doi:10.1007/s11894-015-0436-2. PMID 25786900. S2CID 3098136.
- ^ Bello, Maria G. Dominguez; Knight, Rob; Gilbert, Jack A.; Blaser, Martin J. (4 October 2018). "Preserving microbial diversity". Science. 362 (6410): 33–34. Bibcode:2018Sci...362...33B. doi:10.1126/science.aau8816. PMID 30287652. S2CID 52919917.
- ^ a b Arumugam, Manimozhiyan; Raes, Jeroen; Pelletier, Eric; Le Paslier, Denis; Yamada, Takuji; Mende, Daniel R.; Fernandes, Gabriel R.; Tap, Julien; Bruls, Thomas; Batto, Jean-Michel; Bertalan, Marcelo; Borruel, Natalia; Casellas, Francesc; Fernandez, Leyden; Gautier, Laurent; Hansen, Torben; Hattori, Masahira; Hayashi, Tetsuya; Kleerebezem, Michiel; Kurokawa, Ken; Leclerc, Marion; Levenez, Florence; Manichanh, Chaysavanh; Nielsen, H. Bjørn; Nielsen, Trine; Pons, Nicolas; Poulain, Julie; Qin, Junjie; Sicheritz-Ponten, Thomas; Tims, Sebastian (2011). "Enterotypes of the human gut microbiome". Nature. 473 (7346): 174–80. Bibcode:2011Natur.473..174.. doi:10.1038/nature09944. PMC 3728647. PMID 21508958.
- ^ Wu, G. D.; Chen, J.; Hoffmann, C.; Bittinger, K.; Chen, Y.-Y.; Keilbaugh, S. A.; Bewtra, M.; Knights, D.; Walters, W. A.; Knight, R.; Sinha, R.; Gilroy, E.; Gupta, K.; Baldassano, R.; Nessel, L.; Li, H.; Bushman, F. D.; Lewis, J. D. (2011). "Linking Long-Term Dietary Patterns with Gut Microbial Enterotypes". Science. 334 (6052): 105–08. Bibcode:2011Sci...334..105W. doi:10.1126/science.1208344. PMC 3368382. PMID 21885731.
- ^ Zimmer, Carl (April 20, 2011). "Bacteria Divide People Into 3 Types, Scientists Say". The New York Times. Retrieved April 21, 2011.
a group of scientists now report just three distinct ecosystems in the guts of people they have studied.
- ^ Knights, Dan; Ward, Tonya; McKinlay, Christopher; Miller, Hannah; Gonzalez, Antonio; McDonald, Daniel; Knight, Rob (8 October 2014). "Rethinking "Enterotypes"". Cell Host & Microbe. 16 (4): 433–37. doi:10.1016/j.chom.2014.09.013. PMC 5558460. PMID 25299329.
- ^ Todar, Kenneth (2012). "The Normal Bacterial Flora of Humans". Todar's Online Textbook of Bacteriology. Retrieved June 25, 2016.
- ^ Quigley, Eamonn M.M; Quera, Rodrigo (2006). "Small Intestinal Bacterial Overgrowth: Roles of Antibiotics, Prebiotics, and Probiotics". Gastroenterology. 130 (2): S78–90. doi:10.1053/j.gastro.2005.11.046. PMID 16473077. S2CID 16904501.
- ^ Adams, M. R.; Moss, M. O. (2007). Food Microbiology. doi:10.1039/9781847557940. ISBN 978-0-85404-284-5. S2CID 241261974.
- ^ a b "The normal gut flora" (PDF) (slideshow). 2004. Archived from the original (PDF) on 2004-05-26. Retrieved 2023-01-02 – via University of Glasgow.
- ^ Braune A, Blaut M (2016). "Bacterial species involved in the conversion of dietary flavonoids in the human gut". Gut Microbes. 7 (3): 216–234. doi:10.1080/19490976.2016.1158395. PMC 4939924. PMID 26963713.
- ^ a b Steinhoff, U (2005). "Who controls the crowd? New findings and old questions about the intestinal microflora". Immunology Letters. 99 (1): 12–16. doi:10.1016/j.imlet.2004.12.013. PMID 15894105.
- ^ a b c d e f Gibson, Glenn R (2004). "Fibre and effects on probiotics (the prebiotic concept)". Clinical Nutrition Supplements. 1 (2): 25–31. doi:10.1016/j.clnu.2004.09.005.
- ^ Miquel, S; Martín, R; Rossi, O; Bermúdez-Humarán, LG; Chatel, JM; Sokol, H; Thomas, M; Wells, JM; Langella, P (2013). "Faecalibacterium prausnitzii and human intestinal health". Current Opinion in Microbiology. 16 (3): 255–61. doi:10.1016/j.mib.2013.06.003. PMID 23831042.
- ^ Ley, Ruth E (2010). "Obesity and the human microbiome". Current Opinion in Gastroenterology. 26 (1): 5–11. doi:10.1097/MOG.0b013e328333d751. PMID 19901833. S2CID 23329156.
- ^ Nash, Andrea K; Auchtung, Thomas A; Wong, Matthew C; Smith, Daniel P; Gesell, Jonathan R; Ross, Matthew C; Stewart, Christopher J; Metcalf, Ginger A; Muzny, Donna M; Gibbs, Richard A; Ajami, Nadim J; Petrosino, Joseph F (2017). "The gut mycobiome of the Human Microbiome Project healthy cohort". Microbiome. 5 (1): 153. doi:10.1186/s40168-017-0373-4. PMC 5702186. PMID 29178920.
- ^ Scarpellini, Emidio; Ianiro, Gianluca; Attili, Fabia; Bassanelli, Chiara; De Santis, Adriano; Gasbarrini, Antonio (2015). "The human gut microbiota and virome: Potential therapeutic implications". Digestive and Liver Disease. 47 (12): 1007–12. doi:10.1016/j.dld.2015.07.008. PMC 7185617. PMID 26257129.
- ^ Gerritsen, Jacoline; Smidt, Hauke; Rijkers, Ger; de Vos, Willem (27 May 2011). "Intestinal microbiota in human health and disease: the impact of probiotics". Genes & Nutrition. 6 (3): 209–40. doi:10.1007/s12263-011-0229-7. PMC 3145058. PMID 21617937.
- ^ a b c d e f Yatsunenko, T.; Rey, F. E.; Manary, M. J.; Trehan, I.; Dominguez-Bello, M. G.; Contreras, M.; Magris, M.; Hidalgo, G.; Baldassano, R. N.; Anokhin, A. P.; Heath, A. C.; Warner, B.; Reeder, J.; Kuczynski, J.; Caporaso, J. G.; Lozupone, C. A.; Lauber, C.; Clemente, J. C.; Knights, D.; Knight, R.; Gordon, J. I. (2012). "Human gut microbiome viewed across age and geography". Nature. 486 (7402): 222–27. Bibcode:2012Natur.486..222Y. doi:10.1038/nature11053. PMC 3376388. PMID 22699611.
- ^ De Filippo, C; Cavalieri, D; Di Paola, M; Ramazzotti, M; Poullet, J. B; Massart, S; Collini, S; Pieraccini, G; Lionetti, P (2010). "Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa". Proceedings of the National Academy of Sciences. 107 (33): 14691–6. Bibcode:2010PNAS..10714691D. doi:10.1073/pnas.1005963107. PMC 2930426. PMID 20679230.
- ^ Jonkers, Daisy M.A.E. (2016). "Microbial perturbations and modulation in conditions associated with malnutrition and malabsorption". Best Practice & Research Clinical Gastroenterology. 30 (2): 161–72. doi:10.1016/j.bpg.2016.02.006. PMID 27086883.
- ^ Million, Matthieu; Diallo, Aldiouma; Raoult, Didier (May 2017). "Gut microbiota and malnutrition" (PDF). Microbial Pathogenesis. 106: 127–138. doi:10.1016/j.micpath.2016.02.003. PMID 26853753. S2CID 20381329.
- ^ Rytter, Maren Johanne Heilskov; Kolte, Lilian; Briend, André; Friis, Henrik; Christensen, Vibeke Brix (2014). "The Immune System in Children with Malnutrition—A Systematic Review". PLOS ONE. 9 (8): e105017. Bibcode:2014PLoSO...9j5017R. doi:10.1371/journal.pone.0105017. PMC 4143239. PMID 25153531.
- ^ a b Alcock, Joe; Maley, Carlo C; Aktipis, C. Athena (2014). "Is eating behavior manipulated by the gastrointestinal microbiota? Evolutionary pressures and potential mechanisms". BioEssays. 36 (10): 940–9. doi:10.1002/bies.201400071. PMC 4270213. PMID 25103109.
- ^ a b c Renson, Audrey; Herd, Pamela; Dowd, Jennifer B. (2020). "Sick Individuals and Sick (Microbial) Populations: Challenges in Epidemiology and the Microbiome". Annual Review of Public Health. 41: 63–80. doi:10.1146/annurev-publhealth-040119-094423. PMC 9713946. PMID 31635533.
- ^ Turroni, Francesca; Peano, Clelia; Pass, Daniel A; Foroni, Elena; Severgnini, Marco; Claesson, Marcus J; Kerr, Colm; Hourihane, Jonathan; Murray, Deirdre; Fuligni, Fabio; Gueimonde, Miguel; Margolles, Abelardo; De Bellis, Gianluca; o'Toole, Paul W; Van Sinderen, Douwe; Marchesi, Julian R; Ventura, Marco (2012). "Diversity of Bifidobacteria within the Infant Gut Microbiota". PLOS ONE. 7 (5): e36957. Bibcode:2012PLoSO...736957T. doi:10.1371/journal.pone.0036957. PMC 3350489. PMID 22606315.
- ^ Davenport, Emily R.; Sanders, Jon G.; Song, Se Jin; Amato, Katherine R.; Clark, Andrew G.; Knight, Rob (2017-12-27). "The human microbiome in evolution". BMC Biology. 15 (1): 127. doi:10.1186/s12915-017-0454-7. ISSN 1741-7007. PMC 5744394. PMID 29282061.
- ^ Perez-Muñoz, Maria Elisa; Arrieta, Marie-Claire; Ramer-Tait, Amanda E; Walter, Jens (2017). "A critical assessment of the 'sterile womb' and 'in utero colonization' hypotheses: Implications for research on the pioneer infant microbiome". Microbiome. 5 (1): 48. doi:10.1186/s40168-017-0268-4. PMC 5410102. PMID 28454555.
- ^ Matamoros, Sebastien; Gras-Leguen, Christele; Le Vacon, Françoise; Potel, Gilles; de la Cochetiere, Marie-France (2013). "Development of intestinal microbiota in infants and its impact on health". Trends in Microbiology. 21 (4): 167–73. doi:10.1016/j.tim.2012.12.001. PMID 23332725.
- ^ a b c d e f g Mueller, Noel T.; Bakacs, Elizabeth; Combellick, Joan; Grigoryan, Zoya; Dominguez-Bello, Maria G. (2015). "The infant microbiome development: mom matters". Trends in Molecular Medicine. 21 (2): 109–17. doi:10.1016/j.molmed.2014.12.002. PMC 4464665. PMID 25578246.
- ^ Jiménez, Esther; Fernández, Leonides; Marín, María L; Martín, Rocío; Odriozola, Juan M; Nueno-Palop, Carmen; Narbad, Arjan; Olivares, Mónica; Xaus, Jordi; Rodríguez, Juan M (2005). "Isolation of Commensal Bacteria from Umbilical Cord Blood of Healthy Neonates Born by Cesarean Section". Current Microbiology. 51 (4): 270–4. doi:10.1007/s00284-005-0020-3. PMID 16187156. S2CID 43438656.
- ^ Collado, Maria Carmen; Rautava, Samuli; Aakko, Juhani; Isolauri, Erika; Salminen, Seppo (2016). "Human gut colonisation may be initiated in utero by distinct microbial communities in the placenta and amniotic fluid". Scientific Reports. 6: 23129. Bibcode:2016NatSR...623129C. doi:10.1038/srep23129. PMC 4802384. PMID 27001291.
- ^ Jiménez, Esther; Marín, María L.; Martín, Rocío; Odriozola, Juan M.; Olivares, Mónica; Xaus, Jordi; Fernández, Leonides; Rodríguez, Juan M. (2008). "Is meconium from healthy newborns actually sterile?". Research in Microbiology. 159 (3): 187–93. doi:10.1016/j.resmic.2007.12.007. PMID 18281199.
- ^ Perez-Muñoz, Maria Elisa; Arrieta, Marie-Claire; Ramer-Tait, Amanda E; Walter, Jens (2017). "A critical assessment of the "sterile womb" and "in utero colonization" hypotheses: Implications for research on the pioneer infant microbiome". Microbiome. 5 (1): 48. doi:10.1186/s40168-017-0268-4. PMC 5410102. PMID 28454555.
- ^ Adlerberth, I; Wold, AE (2009). "Establishment of the gut microbiota in Western infants". Acta Paediatrica. 98 (2): 229–38. doi:10.1111/j.1651-2227.2008.01060.x. PMID 19143664. S2CID 205859933.
- ^ "Babies' gut bacteria affected by delivery method: Vaginal delivery promotes mother's gut bacteria in babies' gut". ScienceDaily. 18 September 2019. Archived from the original on 24 November 2021. Retrieved 31 May 2022.
- ^ Coppa, G. V.; Zampini, L.; Galeazzi, T.; Gabrielli, O. (2006). "Prebiotics in human milk: A review". Digestive and Liver Disease. 38: S291–4. doi:10.1016/S1590-8658(07)60013-9. PMID 17259094.
- ^ Mady, Eman A.; Doghish, Ahmed S.; El-Dakroury, Walaa A.; Elkhawaga, Samy Y.; Ismail, Ahmed; El-Mahdy, Hesham A.; Elsakka, Elsayed G. E.; El-Husseiny, Hussein M. (2023-07-01). "Impact of the mother's gut microbiota on infant microbiome and brain development". Neuroscience & Biobehavioral Reviews. 150: 105195. doi:10.1016/j.neubiorev.2023.105195. ISSN 0149-7634. PMID 37100161. S2CID 258302702.
- ^ Fanaro, S; Chierici, R; Guerrini, P; Vigi, V (2007). "Intestinal microflora in early infancy: Composition and development". Acta Paediatrica. 92 (441): 48–55. doi:10.1111/j.1651-2227.2003.tb00646.x. PMID 14599042. S2CID 10316311.
- ^ Yassour, Moran; Vatanen, Tommi; Siljander, Heli; Hämäläinen, Anu-Maaria; Härkönen, Taina; Ryhänen, Samppa J; Franzosa, Eric A; Vlamakis, Hera; Huttenhower, Curtis; Gevers, Dirk; Lander, Eric S; Knip, Mikael; Xavier, Ramnik J (2016). "Natural history of the infant gut microbiome and impact of antibiotic treatment on bacterial strain diversity and stability". Science Translational Medicine. 8 (343): 343ra81. doi:10.1126/scitranslmed.aad0917. PMC 5032909. PMID 27306663.
- ^ Gibson, G. R.; Roberfroid, M. B. (1995). "Dietary modulation of the human colonic microbiota: Introducing the concept of prebiotics". The Journal of Nutrition. 125 (6): 1401–1412. doi:10.1093/jn/125.6.1401. PMID 7782892.
- ^ a b c d e f Wang, Yan; Kasper, Lloyd H (2014). "The role of microbiome in central nervous system disorders". Brain, Behavior, and Immunity. 38: 1–12. doi:10.1016/j.bbi.2013.12.015. PMC 4062078. PMID 24370461.
- ^ a b Yoon, My Young; Lee, Keehoon; Yoon, Sang Sun (2014). "Protective role of gut commensal microbes against intestinal infections". Journal of Microbiology. 52 (12): 983–9. doi:10.1007/s12275-014-4655-2. PMID 25467115. S2CID 54622675.
- ^ Reinoso Webb, Cynthia; Koboziev, Iurii; Furr, Kathryn L; Grisham, Matthew B (2016). "Protective and pro-inflammatory roles of intestinal bacteria". Pathophysiology. 23 (2): 67–80. doi:10.1016/j.pathophys.2016.02.002. PMC 4867289. PMID 26947707.
- ^ Mantis, N J; Rol, N; Corthésy, B (2011). "Secretory IgA's complex roles in immunity and mucosal homeostasis in the gut". Mucosal Immunology. 4 (6): 603–11. doi:10.1038/mi.2011.41. PMC 3774538. PMID 21975936.
- ^ a b Peterson, Lance W; Artis, David (2014). "Intestinal epithelial cells: Regulators of barrier function and immune homeostasis". Nature Reviews Immunology. 14 (3): 141–53. doi:10.1038/nri3608. PMID 24566914. S2CID 3351351.
- ^ a b Honda, Kenya; Littman, Dan R (2016). "The microbiota in adaptive immune homeostasis and disease". Nature. 535 (7610): 75–84. Bibcode:2016Natur.535...75H. doi:10.1038/nature18848. PMID 27383982. S2CID 4461492.
- ^ a b Levy, M.; Thaiss, C.A.; Elinav, E. (2016). "Metabolites: messengers between the microbiota and the immune system". Genes & Development. 30 (14): 1589–97. doi:10.1101/gad.284091.116. PMC 4973288. PMID 27474437.
- ^ a b c d e f g h i Zhang LS, Davies SS (April 2016). "Microbial metabolism of dietary components to bioactive metabolites: opportunities for new therapeutic interventions". Genome Med. 8 (1): 46. doi:10.1186/s13073-016-0296-x. PMC 4840492. PMID 27102537.
Lactobacillus spp. convert tryptophan to indole-3-aldehyde (I3A) through unidentified enzymes [125]. Clostridium sporogenes convert tryptophan to IPA [6], likely via a tryptophan deaminase. ... IPA also potently scavenges hydroxyl radicals
표 2: 미생물 대사산물: 그 합성, 작용기전, 건강 및 질병에 미치는 영향
그림 1: 인돌과 그 대사물이 숙주 생리 및 질병에 미치는 분자적 작용기전 - ^ Wikoff WR, Anfora AT, Liu J, Schultz PG, Lesley SA, Peters EC, Siuzdak G (March 2009). "Metabolomics analysis reveals large effects of gut microflora on mammalian blood metabolites". Proc. Natl. Acad. Sci. U.S.A. 106 (10): 3698–3703. Bibcode:2009PNAS..106.3698W. doi:10.1073/pnas.0812874106. PMC 2656143. PMID 19234110.
Production of IPA was shown to be completely dependent on the presence of gut microflora and could be established by colonization with the bacterium Clostridium sporogenes.
IPA 대사도 - ^ "3-Indolepropionic acid". Human Metabolome Database. University of Alberta. Retrieved 12 June 2018.
- ^ Chyan YJ, Poeggeler B, Omar RA, Chain DG, Frangione B, Ghiso J, Pappolla MA (July 1999). "Potent neuroprotective properties against the Alzheimer beta-amyloid by an endogenous melatonin-related indole structure, indole-3-propionic acid". J. Biol. Chem. 274 (31): 21937–21942. doi:10.1074/jbc.274.31.21937. PMID 10419516. S2CID 6630247.
[Indole-3-propionic acid (IPA)] has previously been identified in the plasma and cerebrospinal fluid of humans, but its functions are not known. ... In kinetic competition experiments using free radical-trapping agents, the capacity of IPA to scavenge hydroxyl radicals exceeded that of melatonin, an indoleamine considered to be the most potent naturally occurring scavenger of free radicals. In contrast with other antioxidants, IPA was not converted to reactive intermediates with pro-oxidant activity.
- ^ Hopper, Christopher P.; De La Cruz, Ladie Kimberly; Lyles, Kristin V.; Wareham, Lauren K.; Gilbert, Jack A.; Eichenbaum, Zehava; Magierowski, Marcin; Poole, Robert K.; Wollborn, Jakob; Wang, Binghe (2020-12-23). "Role of Carbon Monoxide in Host–Gut Microbiome Communication". Chemical Reviews. 120 (24): 13273–13311. doi:10.1021/acs.chemrev.0c00586. ISSN 0009-2665. PMID 33089988. S2CID 224824871.
- ^ Rajilić-Stojanović, Mirjana; De Vos, Willem M (2014). "The first 1000 cultured species of the human gastrointestinal microbiota". FEMS Microbiology Reviews. 38 (5): 996–1047. doi:10.1111/1574-6976.12075. PMC 4262072. PMID 24861948.
- ^ Hill, M. J. (March 1997). "Intestinal flora and endogenous vitamin synthesis". European Journal of Cancer Prevention. 6 (Suppl 1): S43–45. doi:10.1097/00008469-199703001-00009. ISSN 0959-8278. PMID 9167138. S2CID 8740364.
- ^ "The Microbiome". Tufts Now. 2013-09-17. Retrieved 2020-12-09.
- ^ a b c d e ElRakaiby M, Dutilh BE, Rizkallah MR, Boleij A, Cole JN, Aziz RK (July 2014). "Pharmacomicrobiomics: the impact of human microbiome variations on systems pharmacology and personalized therapeutics". Omics. 18 (7): 402–414. doi:10.1089/omi.2014.0018. PMC 4086029. PMID 24785449.
The hundred trillion microbes and viruses residing in every human body, which outnumber human cells and contribute at least 100 times more genes than those encoded on the human genome (Ley et al., 2006), offer an immense accessory pool for inter-individual genetic variation that has been underestimated and largely unexplored (Savage, 1977; Medini et al., 2008; Minot et al., 2011; Wylie et al., 2012). ... Meanwhile, a wealth of literature has long been available about the biotransformation of xenobiotics, notably by gut bacteria (reviewed in Sousa et al., 2008; Rizkallah et al., 2010; Johnson et al., 2012; Haiser and Turnbaugh, 2013). This valuable information is predominantly about drug metabolism by unknown human-associated microbes; however, only a few cases of inter-individual microbiome variations have been documented [e.g., digoxin (Mathan et al., 1989) and acetaminophen (Clayton et al., 2009)].
- ^ a b c Cho I, Blaser MJ (March 2012). "The human microbiome: at the interface of health and disease". Nature Reviews. Genetics. 13 (4): 260–270. doi:10.1038/nrg3182. PMC 3418802. PMID 22411464.
The composition of the microbiome varies by anatomical site (Figure 1). The primary determinant of community composition is anatomical location: interpersonal variation is substantial23,24 and is higher than the temporal variability seen at most sites in a single individual25. ... How does the microbiome affect the pharmacology of medications? Can we "micro-type" people to improve pharmacokinetics and/or reduce toxicity? Can we manipulate the microbiome to improve pharmacokinetic stability?
- ^ Hutter T, Gimbert C, Bouchard F, Lapointe FJ (2015). "Being human is a gut feeling". Microbiome. 3: 9. doi:10.1186/s40168-015-0076-7. PMC 4359430. PMID 25774294.
Some metagenomic studies have suggested that less than 10% of the cells that comprise our bodies are Homo sapiens cells. The remaining 90% are bacterial cells. The description of this so-called human microbiome is of great interest and importance for several reasons. For one, it helps us redefine what a biological individual is. We suggest that a human individual is now best described as a super-individual in which a large number of different species (including Homo sapiens) coexist.
- ^ Kumar K, Dhoke GV, Sharma AK, Jaiswal SK, Sharma VK (January 2019). "Mechanistic elucidation of amphetamine metabolism by tyramine oxidase from human gut microbiota using molecular dynamics simulations". Journal of Cellular Biochemistry. 120 (7): 11206–11215. doi:10.1002/jcb.28396. PMID 30701587. S2CID 73413138.
Particularly in the case of the human gut, which harbors a large diversity of bacterial species, the differences in microbial composition can significantly alter the metabolic activity in the gut lumen.4 The differential metabolic activity due to the differences in gut microbial species has been recently linked with various metabolic disorders and diseases.5-12 In addition to the impact of gut microbial diversity or dysbiosis in various human diseases, there is an increasing amount of evidence which shows that the gut microbes can affect the bioavailability and efficacy of various orally administrated drug molecules through promiscuous enzymatic metabolism.13,14 ... The present study on the atomistic details of amphetamine binding and binding affinity to the tyramine oxidase along with the comparison with two natural substrates of this enzyme namely tyramine and phenylalanine provides strong evidence for the promiscuity‐based metabolism of amphetamine by the tyramine oxidase enzyme of E. coli. The obtained results will be crucial in designing a surrogate molecule for amphetamine that can help either in improving the efficacy and bioavailability of the amphetamine drug via competitive inhibition or in redesigning the drug for better pharmacological effects. This study will also have useful clinical implications in reducing the gut microbiota caused variation in the drug response among different populations.
- ^ Sousa, Tiago; Paterson, Ronnie; Moore, Vanessa; Carlsson, Anders; Abrahamsson, Bertil; Basit, Abdul W (2008). "The gastrointestinal microbiota as a site for the biotransformation of drugs". International Journal of Pharmaceutics. 363 (1–2): 1–25. doi:10.1016/j.ijpharm.2008.07.009. PMID 18682282.
- ^ Haiser, H. J; Gootenberg, D. B; Chatman, K; Sirasani, G; Balskus, E. P; Turnbaugh, P. J (2013). "Predicting and Manipulating Cardiac Drug Inactivation by the Human Gut Bacterium Eggerthella lenta". Science. 341 (6143): 295–8. Bibcode:2013Sci...341..295H. doi:10.1126/science.1235872. PMC 3736355. PMID 23869020.
- ^ a b Koppel, Nitzan; Maini Rekdal, Vayu; Balskus, Emily P. (2017-06-23). "Chemical transformation of xenobiotics by the human gut microbiota". Science. 356 (6344). doi:10.1126/science.aag2770. ISSN 0036-8075. PMC 5534341. PMID 28642381.
- ^ a b c Spanogiannopoulos, Peter; Bess, Elizabeth N.; Carmody, Rachel N.; Turnbaugh, Peter J. (2016-03-14). "The microbial pharmacists within us: a metagenomic view of xenobiotic metabolism". Nature Reviews Microbiology. 14 (5): 273–287. doi:10.1038/nrmicro.2016.17. ISSN 1740-1526. PMC 5243131. PMID 26972811.
- ^ a b Maini Rekdal, Vayu; Bess, Elizabeth N.; Bisanz, Jordan E.; Turnbaugh, Peter J.; Balskus, Emily P. (2019-06-14). "Discovery and inhibition of an interspecies gut bacterial pathway for Levodopa metabolism". Science. 364 (6445). doi:10.1126/science.aau6323. ISSN 0036-8075. PMC 7745125. PMID 31196984.
- ^ a b Dempsey, Joseph L.; Cui, Julia Yue (2019-10-19). "Microbiome Is a Functional Modifier of P450 Drug Metabolism". Current Pharmacology Reports. 5 (6): 481–490. doi:10.1007/s40495-019-00200-w. ISSN 2198-641X. PMC 7731899. PMID 33312848.
- ^ Boerner, Udo; Abbott, Seth; Roe, Robert L. (January 1975). "The Metabolism of Morphine and Heroin in Man". Drug Metabolism Reviews. 4 (1): 39–73. doi:10.3109/03602537508993748. ISSN 0360-2532. PMID 1204496.
- ^ Dobkin, Jay F.; Saha, Jnan R.; Butler, Vincent P.; Neu, Harold C.; Lindenbaum, John (1983-04-15). "Digoxin-Inactivating Bacteria: Identification in Human Gut Flora". Science. 220 (4594): 325–327. doi:10.1126/science.6836275. ISSN 0036-8075. PMID 6836275.
- ^ Sahota, S. S.; Bramley, P. M.; Menzies, I. S. (1982-02-01). "The Fermentation of Lactulose by Colonic Bacteria". Microbiology. 128 (2): 319–325. doi:10.1099/00221287-128-2-319. ISSN 1350-0872. PMID 6804597.
- ^ a b Yoo, Dae-Hyoung; Kim, In Sook; Van Le, Thi Kim; Jung, Il-Hoon; Yoo, Hye Hyun; Kim, Dong-Hyun (2014-06-19). "Gut Microbiota-Mediated Drug Interactions between Lovastatin and Antibiotics". Drug Metabolism and Disposition. 42 (9): 1508–1513. doi:10.1124/dmd.114.058354. ISSN 0090-9556. PMID 24947972. S2CID 7524335.
- ^ Haiser, Henry J.; Gootenberg, David B.; Chatman, Kelly; Sirasani, Gopal; Balskus, Emily P.; Turnbaugh, Peter J. (2013-07-19). "Predicting and Manipulating Cardiac Drug Inactivation by the Human Gut Bacterium Eggerthella lenta". Science. 341 (6143): 295–298. Bibcode:2013Sci...341..295H. doi:10.1126/science.1235872. ISSN 0036-8075. PMC 3736355. PMID 23869020.
- ^ Parvez, Md Masud; Basit, Abdul; Jariwala, Parth B.; Gáborik, Zsuzsanna; Kis, Emese; Heyward, Scott; Redinbo, Matthew R.; Prasad, Bhagwat (2021-06-01). "Quantitative Investigation of Irinotecan Metabolism, Transport, and Gut Microbiome Activation". Drug Metabolism and Disposition. 49 (8): 683–693. doi:10.1124/dmd.121.000476. ISSN 0090-9556. PMC 8407663. PMID 34074730.
- ^ a b Sousa, Tiago; Paterson, Ronnie; Moore, Vanessa; Carlsson, Anders; Abrahamsson, Bertil; Basit, Abdul W. (November 2008). "The gastrointestinal microbiota as a site for the biotransformation of drugs". International Journal of Pharmaceutics. 363 (1–2): 1–25. doi:10.1016/j.ijpharm.2008.07.009. ISSN 0378-5173. PMID 18682282.
- ^ a b Koppel, Nitzan; Bisanz, Jordan E; Pandelia, Maria-Eirini; Turnbaugh, Peter J; Balskus, Emily P (2018-05-15). Ley, Ruth Emily (ed.). "Discovery and characterization of a prevalent human gut bacterial enzyme sufficient for the inactivation of a family of plant toxins". eLife. 7: e33953. doi:10.7554/eLife.33953. ISSN 2050-084X. PMC 5953540. PMID 29761785.
- ^ Alexander, James L.; Wilson, Ian D.; Teare, Julian; Marchesi, Julian R.; Nicholson, Jeremy K.; Kinross, James M. (2017-03-08). "Gut microbiota modulation of chemotherapy efficacy and toxicity". Nature Reviews Gastroenterology & Hepatology. 14 (6): 356–365. doi:10.1038/nrgastro.2017.20. ISSN 1759-5045. PMID 28270698. S2CID 9654170.
- ^ Brandi, Giovanni; Dabard, Jean; Raibaud, Pierre; Di Battista, Monica; Bridonneau, Chantal; Pisi, Anna Maria; Morselli Labate, Antonio Maria; Pantaleo, Maria Abbondanza; De Vivo, Antonello; Biasco, Guido (2006-02-15). "Intestinal microflora and digestive toxicity of irinotecan in mice". Clinical Cancer Research. 12 (4): 1299–1307. doi:10.1158/1078-0432.ccr-05-0750. ISSN 1078-0432. PMID 16489087. S2CID 26655779.
- ^ Turnbaugh, Peter J.; Ley, Ruth E.; Hamady, Micah; Fraser-Liggett, Claire M.; Knight, Rob; Gordon, Jeffrey I. (October 2007). "The Human Microbiome Project". Nature. 449 (7164): 804–810. Bibcode:2007Natur.449..804T. doi:10.1038/nature06244. ISSN 0028-0836. PMC 3709439. PMID 17943116.
- ^ Koh, Ara; De Vadder, Filipe; Kovatcheva-Datchary, Petia; Bäckhed, Fredrik (June 2016). "From Dietary Fiber to Host Physiology: Short-Chain Fatty Acids as Key Bacterial Metabolites". Cell. 165 (6): 1332–1345. doi:10.1016/j.cell.2016.05.041. ISSN 0092-8674. PMID 27259147. S2CID 8562345.
- ^ Konopelski, Piotr; Ufnal, Marcin (2018-09-14). "Indoles - Gut Bacteria Metabolites of Tryptophan with Pharmacotherapeutic Potential". Current Drug Metabolism. 19 (10): 883–890. doi:10.2174/1389200219666180427164731. ISSN 1389-2002. PMID 29708069. S2CID 13979774.
- ^ Collins, Stephanie L.; Stine, Jonathan G.; Bisanz, Jordan E.; Okafor, C. Denise; Patterson, Andrew D. (2022-10-17). "Bile acids and the gut microbiota: metabolic interactions and impacts on disease". Nature Reviews Microbiology. 21 (4): 236–247. doi:10.1038/s41579-022-00805-x. ISSN 1740-1526. PMID 36253479. S2CID 252970168.
- ^ Yang, Wenjing; Yu, Tianming; Huang, Xiangsheng; Bilotta, Anthony J.; Xu, Leiqi; Lu, Yao; Sun, Jiaren; Pan, Fan; Zhou, Jia; Zhang, Wenbo; Yao, Suxia; Maynard, Craig L.; Singh, Nagendra; Dann, Sara M.; Liu, Zhanju (2020-09-08). "Intestinal microbiota-derived short-chain fatty acids regulation of immune cell IL-22 production and gut immunity". Nature Communications. 11 (1): 4457. Bibcode:2020NatCo..11.4457Y. doi:10.1038/s41467-020-18262-6. ISSN 2041-1723. PMC 7478978. PMID 32901017.
- ^ Murugesan, Selvasankar; Nirmalkar, Khemlal; Hoyo-Vadillo, Carlos; García-Espitia, Matilde; Ramírez-Sánchez, Daniela; García-Mena, Jaime (2017-12-02). "Gut microbiome production of short-chain fatty acids and obesity in children". European Journal of Clinical Microbiology & Infectious Diseases. 37 (4): 621–625. doi:10.1007/s10096-017-3143-0. ISSN 0934-9723. PMID 29196878. S2CID 254132108.
- ^ a b Thanissery, Rajani; Winston, Jenessa A.; Theriot, Casey M. (June 2017). "Inhibition of spore germination, growth, and toxin activity of clinically relevant C. difficile strains by gut microbiota derived secondary bile acids". Anaerobe. 45: 86–100. doi:10.1016/j.anaerobe.2017.03.004. ISSN 1075-9964. PMC 5466893. PMID 28279860.
- ^ Collins, Stephanie L.; Stine, Jonathan G.; Bisanz, Jordan E.; Okafor, C. Denise; Patterson, Andrew D. (2022-10-17). "Bile acids and the gut microbiota: metabolic interactions and impacts on disease". Nature Reviews Microbiology. 21 (4): 236–247. doi:10.1038/s41579-022-00805-x. ISSN 1740-1526. PMID 36253479. S2CID 252970168.
- ^ a b c Jones, Brian V.; Begley, Máire; Hill, Colin; Gahan, Cormac G. M.; Marchesi, Julian R. (2008-09-09). "Functional and comparative metagenomic analysis of bile salt hydrolase activity in the human gut microbiome". Proceedings of the National Academy of Sciences. 105 (36): 13580–13585. Bibcode:2008PNAS..10513580J. doi:10.1073/pnas.0804437105. ISSN 0027-8424. PMC 2533232. PMID 18757757.
- ^ a b 카오에이치, 쉬엠, 동더블유, 덩비, 왕에스, 장이, 왕에스, 루오에스, 왕더블유, 치이, 가오제이, 카오엑스, 옌에프, 왕비.2차 담즙산에 의한 생체이상증은 장내 발암을 촉진합니다.Int J Cancer. 2017 Jun 1;140(11):2545-2556. Doi:10.1002/ijc.30643.Epub 2017 3월 1일 PMID : 28187526
- ^ a b 번스타인 H, 번스타인 C.대장 및 위장계의 다른 부위에 발암물질로서 담즙산.Exp Biol Med(메이우드).2023년 1월;248(1):79-89. 도이:10.1177/15353702221318582022년 11월 19일 에펍.PMID: 36408538; PMCID: PMC9989147
- ^ Mayer, E. A; Knight, R; Mazmanian, S. K; Cryan, J. F; Tillisch, K (2014). "Gut Microbes and the Brain: Paradigm Shift in Neuroscience". Journal of Neuroscience. 34 (46): 15490–6. doi:10.1523/JNEUROSCI.3299-14.2014. PMC 4228144. PMID 25392516.
- ^ a b c Dinan, Timothy G; Cryan, John F (2015). "The impact of gut microbiota on brain and behaviour". Current Opinion in Clinical Nutrition and Metabolic Care. 18 (6): 552–8. doi:10.1097/MCO.0000000000000221. PMID 26372511. S2CID 21424690.
- ^ a b c Carman, Robert J.; Simon, Mary Alice; Fernández, Haydée; Miller, Margaret A.; Bartholomew, Mary J. (2004). "Ciprofloxacin at low levels disrupts colonization resistance of human fecal microflora growing in chemostats". Regulatory Toxicology and Pharmacology. 40 (3): 319–26. doi:10.1016/j.yrtph.2004.08.005. PMID 15546686.
- ^ Hvas, Christian Lodberg; Baunwall, Simon Mark Dahl; Erikstrup, Christian (2020-07-01). "Faecal microbiota transplantation: A life-saving therapy challenged by commercial claims for exclusivity". eClinicalMedicine. 24: 100436. doi:10.1016/j.eclinm.2020.100436. ISSN 2589-5370. PMC 7334803. PMID 32642633.
- ^ Brandt, Lawrence J.; Borody, Thomas Julius; Campbell, Jordana (2011). "Endoscopic Fecal Microbiota Transplantation". Journal of Clinical Gastroenterology. 45 (8): 655–57. doi:10.1097/MCG.0b013e3182257d4f. PMID 21716124. S2CID 2508836.
- ^ Knight, DJW; Girling, KJ (2003). "Gut flora in health and disease". The Lancet. 361 (9371): 512–19. doi:10.1016/S0140-6736(03)13438-1. PMID 12781578. S2CID 40683723.
- ^ a b c Cho, I.; Yamanishi, S.; Cox, L.; Methé, B. A.; Zavadil, J.; Li, K.; Gao, Z.; Mahana, D.; Raju, K.; Teitler, I.; Li, H.; Alekseyenko, A. V.; Blaser, M. J. (2012). "Antibiotics in early life alter the murine colonic microbiome and adiposity". Nature. 488 (7413): 621–26. Bibcode:2012Natur.488..621C. doi:10.1038/nature11400. PMC 3553221. PMID 22914093.
- ^ a b c d Schneiderhan, J; Master-Hunter, T; Locke, A (2016). "Targeting gut flora to treat and prevent disease". The Journal of Family Practice. 65 (1): 34–8. PMID 26845162.
- ^ Baker, Monya (2012). "Pregnancy alters resident gut microbes". Nature. doi:10.1038/nature.2012.11118. S2CID 87078157.
- ^ Hill, Colin; Guarner, Francisco; Reid, Gregor; Gibson, Glenn R; Merenstein, Daniel J; Pot, Bruno; Morelli, Lorenzo; Canani, Roberto Berni; Flint, Harry J; Salminen, Seppo; Calder, Philip C; Sanders, Mary Ellen (2014). "The International Scientific Association for Probiotics and Prebiotics consensus statement on the scope and appropriate use of the term probiotic". Nature Reviews Gastroenterology & Hepatology. 11 (8): 506–14. doi:10.1038/nrgastro.2014.66. PMID 24912386.
- ^ Rijkers, Ger T; De Vos, Willem M; Brummer, Robert-Jan; Morelli, Lorenzo; Corthier, Gerard; Marteau, Philippe (2011). "Health benefits and health claims of probiotics: Bridging science and marketing". British Journal of Nutrition. 106 (9): 1291–6. doi:10.1017/S000711451100287X. PMID 21861940.
- ^ Hutkins, Robert W; Krumbeck, Janina A; Bindels, Laure B; Cani, Patrice D; Fahey, George; Goh, Yong Jun; Hamaker, Bruce; Martens, Eric C; Mills, David A; Rastal, Robert A; Vaughan, Elaine; Sanders, Mary Ellen (2016). "Prebiotics: Why definitions matter". Current Opinion in Biotechnology. 37: 1–7. doi:10.1016/j.copbio.2015.09.001. PMC 4744122. PMID 26431716.
- ^ Pandey, Kavita. R; Naik, Suresh. R; Vakil, Babu. V (2015). "Probiotics, prebiotics and synbiotics- a review". Journal of Food Science and Technology. 52 (12): 7577–87. doi:10.1007/s13197-015-1921-1. PMC 4648921. PMID 26604335.
- ^ Broeckx, Géraldine; Vandenheuvel, Dieter; Claes, Ingmar J.J; Lebeer, Sarah; Kiekens, Filip (2016). "Drying techniques of probiotic bacteria as an important step towards the development of novel pharmabiotics" (PDF). International Journal of Pharmaceutics. 505 (1–2): 303–18. doi:10.1016/j.ijpharm.2016.04.002. hdl:10067/1328840151162165141. PMID 27050865.
- ^ Sleator, Roy D; Hill, Colin (2009). "Rational Design of Improved Pharmabiotics". Journal of Biomedicine and Biotechnology. 2009: 275287. doi:10.1155/2009/275287. PMC 2742647. PMID 19753318.
- ^ Patterson, Elaine; Cryan, John F; Fitzgerald, Gerald F; Ross, R. Paul; Dinan, Timothy G; Stanton, Catherine (2014). "Gut microbiota, the pharmabiotics they produce and host health". Proceedings of the Nutrition Society. 73 (4): 477–89. doi:10.1017/S0029665114001426. PMID 25196939.
- ^ Guandalini, Stefano; Magazzù, Giuseppe; Chiaro, Andrea; La Balestra, Valeria; Di Nardo, Giovanni; Gopalan, Sarath; Sibal, A.; Romano, Claudio; Canani, Roberto Berni; Lionetti, Paolo; Setty, Mala (July 2010). "VSL#3 Improves Symptoms in Children with Irritable Bowel Syndrome: A Multicenter, Randomized, Placebo-Controlled, Double-Blind, Crossover Study". Journal of Pediatric Gastroenterology and Nutrition. 51 (1): 24–30. doi:10.1097/MPG.0b013e3181ca4d95. PMID 20453678. S2CID 33659736.
- ^ Ford, Alexander C; Quigley, Eamonn M M; Lacy, Brian E; Lembo, Anthony J; Saito, Yuri A; Schiller, Lawrence R; Soffer, Edy E; Spiegel, Brennan M R; Moayyedi, Paul (2014). "Efficacy of Prebiotics, Probiotics and Synbiotics in Irritable Bowel Syndrome and Chronic Idiopathic Constipation: Systematic Review and Meta-analysis". The American Journal of Gastroenterology. 109 (10): 1547–61, quiz 1546, 1562. doi:10.1038/ajg.2014.202. PMID 25070051. S2CID 205100508.
- ^ Dupont, Andrew; Richards; Jelinek, Katherine A; Krill, Joseph; Rahimi, Erik; Ghouri, Yezaz (2014). "Systematic review of randomized controlled trials of probiotics, prebiotics, and synbiotics in inflammatory bowel disease". Clinical and Experimental Gastroenterology. 7: 473–87. doi:10.2147/CEG.S27530. PMC 4266241. PMID 25525379.
- ^ Yu, Cheng Gong; Huang, Qin (2013). "Recent progress on the role of gut microbiota in the pathogenesis of inflammatory bowel disease". Journal of Digestive Diseases. 14 (10): 513–7. doi:10.1111/1751-2980.12087. PMID 23848393. S2CID 26982085.
- ^ Levitt, Michael D.; Duane, William C. (1972-05-04). "Floating Stools — Flatus versus Fat". New England Journal of Medicine. 286 (18): 973–975. doi:10.1056/NEJM197205042861804. ISSN 0028-4793. PMID 5015442.
- ^ Aalam, Syed Mohammed Musheer; Crasta, Daphne Norma; Roy, Pooja; Miller, A. Lee; Gamb, Scott I.; Johnson, Stephen; Till, Lisa M.; Chen, Jun; Kashyap, Purna; Kannan, Nagarajan (2022-10-27). "Genesis of fecal floatation is causally linked to gut microbial colonization in mice". Scientific Reports. 12 (1): 18109. Bibcode:2022NatSR..1218109A. doi:10.1038/s41598-022-22626-x. ISSN 2045-2322. PMC 9613883. PMID 36302811.
- ^ Maier, Lisa; Pruteanu, Mihaela; Kuhn, Michael; Zeller, Georg; Telzerow, Anja; Anderson, Exene Erin; Brochado, Ana Rita; Fernandez, Keith Conrad; Dose, Hitomi; Mori, Hirotada; Patil, Kiran Raosaheb; Bork, Peer; Typas, Athanasios (2018). "Extensive impact of non-antibiotic drugs on human gut bacteria". Nature. 555 (7698): 623–628. Bibcode:2018Natur.555..623M. doi:10.1038/nature25979. PMC 6108420. PMID 29555994.
- ^ Clauss, Matthieu; Gérard, Philippe; Mosca, Alexis; Leclerc, Marion (2021). "Interplay Between Exercise and Gut Microbiome in the Context of Human Health and Performance". Frontiers in Nutrition. 8: 637010. doi:10.3389/fnut.2021.637010. PMC 8222532. PMID 34179053.
- ^ a b Kamboj, AK; Cotter, TG; Oxentenko, AS (2017). "Helicobacter pylori: The Past, Present, and Future in Management". Mayo Clinic Proceedings. 92 (4): 599–604. doi:10.1016/j.mayocp.2016.11.017. ISSN 0025-6196. PMID 28209367.
- ^ a b c "Peptic ulcer disease" (PDF). The Johns Hopkins University School of Medicine. 2013. Retrieved 21 October 2020.
- ^ Burisch, Johan; Jess, Tine; Martinato, Matteo; Lakatos, Peter L (2013). "The burden of inflammatory bowel disease in Europe". Journal of Crohn's and Colitis. 7 (4): 322–37. doi:10.1016/j.crohns.2013.01.010. PMID 23395397.
- ^ Blandino, G; Inturri, R; Lazzara, F; Di Rosa, M; Malaguarnera, L (2016). "Impact of gut microbiota on diabetes mellitus". Diabetes & Metabolism. 42 (5): 303–315. doi:10.1016/j.diabet.2016.04.004. PMID 27179626.
- ^ a b c Boulangé, Claire L; Neves, Ana Luisa; Chilloux, Julien; Nicholson, Jeremy K; Dumas, Marc-Emmanuel (2016). "Impact of the gut microbiota on inflammation, obesity, and metabolic disease". Genome Medicine. 8 (1): 42. doi:10.1186/s13073-016-0303-2. PMC 4839080. PMID 27098727.
- ^ Spiller, Robin (2016). "Irritable bowel syndrome: New insights into symptom mechanisms and advances in treatment". F1000Research. 5: 780. doi:10.12688/f1000research.7992.1. PMC 4856111. PMID 27158477.
- ^ a b c Arrieta, Marie-Claire; Stiemsma, Leah T; Dimitriu, Pedro A; Thorson, Lisa; Russell, Shannon; Yurist-Doutsch, Sophie; Kuzeljevic, Boris; Gold, Matthew J; Britton, Heidi M; Lefebvre, Diana L; Subbarao, Padmaja; Mandhane, Piush; Becker, Allan; McNagny, Kelly M; Sears, Malcolm R; Kollmann, Tobias; Mohn, William W; Turvey, Stuart E; Brett Finlay, B (2015). "Early infancy microbial and metabolic alterations affect risk of childhood asthma". Science Translational Medicine. 7 (307): 307ra152. doi:10.1126/scitranslmed.aab2271. PMID 26424567. S2CID 206687974.
- ^ a b Stiemsma, Leah T; Turvey, Stuart E (2017). "Asthma and the microbiome: Defining the critical window in early life". Allergy, Asthma & Clinical Immunology. 13: 3. doi:10.1186/s13223-016-0173-6. PMC 5217603. PMID 28077947.
- ^ Ipci, Kagan; Altıntoprak, Niyazi; Muluk, Nuray Bayar; Senturk, Mehmet; Cingi, Cemal (2016). "The possible mechanisms of the human microbiome in allergic diseases". European Archives of Oto-Rhino-Laryngology. 274 (2): 617–626. doi:10.1007/s00405-016-4058-6. PMID 27115907. S2CID 27328940.
- ^ Mariño, Eliana; Richards, James L.; McLeod, Keiran H.; Stanley, Dragana; Yap, Yu Anne; Knight, Jacinta; McKenzie, Craig; Kranich, Jan; Oliveira, Ana Carolina; Rossello, Fernando J.; Krishnamurthy, Balasubramanian; Nefzger, Christian M.; Macia, Laurence; Thorburn, Alison; Baxter, Alan G. (May 2017). "Gut microbial metabolites limit the frequency of autoimmune T cells and protect against type 1 diabetes". Nature Immunology. 18 (5): 552–562. doi:10.1038/ni.3713. ISSN 1529-2916. PMID 28346408. S2CID 30078908.
- ^ Bettelli E, Carrier Y, Gao W, Korn T, Strom TB, Oukka M, Weiner HL, Kuchrou VK (2006년 5월)"병원성 이펙터 TH17 및 조절 T세포 생성을 위한 호혜적 발달 경로"자연.441 (7090): 235–8.비브코드:2006 Natur.441..235B. 도이:10.1038/natural04753PMID 16648838.
- ^ a b Säemann, Marcus D.; Böhmig, Georg A.; Österreicher, Christoph H.; Burtscher, Helmut; Parolini, Ornella; Diakos, Christos; Stöckl, Johannes; Hörl, Walter H.; Zlabinger, Gerhard J. (December 2000). "Anti‐inflammatory effects of sodium butyrate on human monocytes: potent inhibition of IL‐12 and up‐regulation of IL‐10 production". The FASEB Journal. 14 (15): 2380–2382. doi:10.1096/fj.00-0359fje. ISSN 0892-6638. PMID 11024006. S2CID 41553220.
- ^ Gao, Z; Yin, J; Zhang, J; Ward, R. E; Martin, R. J; Lefevre, M; Cefalu, W. T; Ye, J (2009). "Butyrate Improves Insulin Sensitivity and Increases Energy Expenditure in Mice". Diabetes. 58 (7): 1509–17. doi:10.2337/db08-1637. PMC 2699871. PMID 19366864.
- ^ Mazidi, Mohsen; Rezaie, Peyman; Kengne, Andre Pascal; Mobarhan, Majid Ghayour; Ferns, Gordon A (2016). "Gut microbiome and metabolic syndrome". Diabetes & Metabolic Syndrome: Clinical Research & Reviews. 10 (2): S150–7. doi:10.1016/j.dsx.2016.01.024. PMID 26916014.
- ^ Chen, Xiao; d'Souza, Roshan; Hong, Seong-Tshool (2013). "The role of gut microbiota in the gut-brain axis: Current challenges and perspectives". Protein & Cell. 4 (6): 403–14. doi:10.1007/s13238-013-3017-x. PMC 4875553. PMID 23686721.
- ^ a b Minemura, Masami (2015). "Gut microbiota and liver diseases". World Journal of Gastroenterology. 21 (6): 1691–702. doi:10.3748/wjg.v21.i6.1691. PMC 4323444. PMID 25684933.
- ^ Syn, Nicholas L; Teng, Michele W L; Mok, Tony S K; Soo, Ross A (2017). "De-novo and acquired resistance to immune checkpoint targeting". The Lancet Oncology. 18 (12): e731–41. doi:10.1016/s1470-2045(17)30607-1. PMID 29208439.
- ^ a b 데브코타 S, 턴보 PJ. 암:산성 고리.자연.2013년 7월 4일;499(7456):37-8. 도이:10.1038/자연 12404.Epub 2013 Jun 26. PMID 23803768
- ^ a b 요시모토 S, 루TM, 아타라시 K, 간다 H, 사토 S, 오야도마리 S, 이와쿠라 Y, 오시마 K, 모리타 H, 핫토리 M, 혼다 K, 이시카와 Y, 하라 E, 오타니 N. 비만 유도 내장 미생물 대사산물은 노화 세크리돔을 통해 간암을 촉진합니다.자연.2013년 7월 4일;499(7456):97-101. 도이:10.1038/자연 12347.2013년 6월 26일 epub.에라툼 인: 자연.2014년 2월
- ^ "How Your Gut Microbiome is Linked to Depression and Anxiety". CAS. 2 February 2022. Retrieved 12 December 2022.
- ^ a b Moeller, Andrew H.; Li, Yingying; Ngole, Eitel Mpoudi; Ahuka-Mundeke, Steve; Lonsdorf, Elizabeth V.; Pusey, Anne E.; Peeters, Martine; Hahn, Beatrice H.; Ochman, Howard (2014-11-18). "Rapid changes in the gut microbiome during human evolution". Proceedings of the National Academy of Sciences. 111 (46): 16431–16435. Bibcode:2014PNAS..11116431M. doi:10.1073/pnas.1419136111. ISSN 0027-8424. PMC 4246287. PMID 25368157.
- ^ Brune, A. (2014). "Symbiotic digestion of lignocellulose in termite guts". Nature Reviews Microbiology. 12 (3): 168–80. doi:10.1038/nrmicro3182. PMID 24487819. S2CID 5220210.
- ^ a b Dietrich, C.; Köhler, T.; Brune, A. (2014). "The cockroach origin of the termite gut microbiota: patterns in bacterial community structure reflect major evolutionary events". Applied and Environmental Microbiology. 80 (7): 2261–69. Bibcode:2014ApEnM..80.2261D. doi:10.1128/AEM.04206-13. PMC 3993134. PMID 24487532.
- ^ a b Mikaelyan, A.; Dietrich, C.; Köhler, T.; Poulsen, M.; Sillam-Dussès, D.; Brune, A. (2015). "Diet is the primary determinant of bacterial community structure in the guts of higher termites". Molecular Ecology. 24 (20): 5824–95. doi:10.1111/mec.13376. PMID 26348261. S2CID 206182668.
- ^ Mikaelyan, A.; Thompson, C.; Hofer, M.; Brune, A. (2016). "The deterministic assembly of complex bacterial communities in germ-free cockroach guts". Applied and Environmental Microbiology. 82 (4): 1256–63. doi:10.1128/AEM.03700-15. PMC 4751828. PMID 26655763.
추가열람
- 기사 검토
- De Preter, Vicky; Hamer, Henrike M; Windey, Karen; Verbeke, Kristin (2011). "The impact of pre- and/or probiotics on human colonic metabolism: Does it affect human health?". Molecular Nutrition & Food Research. 55 (1): 46–57. doi:10.1002/mnfr.201000451. PMID 21207512.
- Maranduba, Carlos Magno da Costa; De Castro, Sandra Bertelli Ribeiro; Souza, Gustavo Torres de; Rossato, Cristiano; Da Guia, Francisco Carlos; Valente, Maria Anete Santana; Rettore, João Vitor Paes; Maranduba, Claudinéia Pereira; Souza, Camila Maurmann de; Carmo, Antônio Márcio Resende do; MacEdo, Gilson Costa; Silva, Fernando de Sá (2015). "Intestinal Microbiota as Modulators of the Immune System and Neuroimmune System: Impact on the Host Health and Homeostasis". Journal of Immunology Research. 2015: 931574. doi:10.1155/2015/931574. PMC 4352473. PMID 25759850.
- Prakash, Satya; Rodes, Laetitia; Coussa-Charley, Michael; Tomaro-Duchesneau, Catherine; Tomaro-Duchesneau, Catherine; Coussa-Charley; Rodes (2011). "Gut microbiota: Next frontier in understanding human health and development of biotherapeutics". Biologics: Targets and Therapy. 5: 71–86. doi:10.2147/BTT.S19099. PMC 3156250. PMID 21847343.
- Wu, G. D.; Chen, J.; Hoffmann, C.; Bittinger, K.; Chen, Y.-Y.; Keilbaugh, S. A.; Bewtra, M.; Knights, D.; Walters, W. A.; Knight, R.; Sinha, R.; Gilroy, E.; Gupta, K.; Baldassano, R.; Nessel, L.; Li, H.; Bushman, F. D.; Lewis, J.D. (2011). "Linking Long-Term Dietary Patterns with Gut Microbial Enterotypes". Science. 334 (6052): 105–08. Bibcode:2011Sci...334..105W. doi:10.1126/science.1208344. PMC 3368382. PMID 21885731.