마이크로바이오타
Microbiota마이크로바이오타는 식물을 포함한 모든 다세포 생물에서 발견되는 공생 또는 병원성 미생물의 범위이다.미생물은 박테리아, 고세균, 원생동물, 곰팡이,[2][3] 바이러스를 포함하며 숙주의 면역학적, 호르몬적, 대사적 항상성에 매우 중요한 것으로 밝혀졌다.
마이크로바이옴이라는 용어는 생태학적 틈새에 존재하는 미생물들의 집단 게놈이나 미생물들 [4][5][6]자체에 존재하는 미생물들의 집단 게놈을 말한다.
마이크로바이옴과 숙주는 진화 과정에서 후생유전학과 유전적 특성으로부터 상승작용을 하는 단위로 나타났고, 때로는 총칭하여 홀로비온트라고 [7][8]불린다.인간과 다른 메타조아 내장에 있는 마이크로바이오타의 존재는 메타조아와 [9][10]박테리아 사이의 공진화를 이해하는 데 있어 매우 중요합니다.마이크로바이오타는 발효산물(짧은 사슬 지방산)인 [11]아세테이트를 통해 장내 면역 및 대사 반응에 중요한 역할을 합니다.
서론
단순한 생명체에서 인간에 이르기까지 모든 식물과 동물은 미생물과 [12]밀접하게 연계되어 살고 있다.다음과 같은 몇 가지 발전이 마이크로바이옴에 대한 인식을 주도했다.
- 메타게노믹스와 메타트랜스크립토믹스[13] 분야에서 단세포와 미생물 군집 전체의 게놈과 유전자 발현 분석을 수행하는 능력
- 여러 분야의[13] 연구자가 접근할 수 있는 데이터베이스
- 복잡한 데이터[13] 세트에 적합한 수학적 분석 방법
생물학자들은 미생물이 유기체의 표현형에서 중요한 부분을 구성한다는 것을 인식하게 되었고, 이는 때때로 공생하는 사례 [13]연구를 훨씬 넘어선다.
microbe-host 관계 유형
19세기 동안 루뱅 대학의 벨기에 교수였던 피에르 조제프 반 베네덴에 의해 개발된 개념인 Commensalism은 미생물군이 무해한 공존 속에서 숙주를 식민지로 만드는 마이크로바이옴의 중심이다.그들의 숙주와의 관계는 유기체가 숙주에게 불리할 때,[15]: 700 [16] 기생충에게 유용한 것으로 알려진 작업을 수행할 때 상호주의라고 불립니다.다른 저자들은 상황을 상호주의로 정의하는데, 상호주의에서는 둘 다 이익을 얻으며, 상호주의에서는 영향을 받지 않는 숙주가 [17]심비온트에게 이익을 준다.영양소 교환은 양방향 또는 단방향일 수 있으며 맥락에 따라 달라질 수 있으며 다양한 [17]방법으로 발생할 수 있다.존재할 것으로 예상되며 정상적인 상황에서 질병을 일으키지 않는 미생물군은 정상적인 미생물군 또는 정상적인 미생물군으로 [15]간주됩니다.정상적인 미생물군은 무해할 뿐만 아니라 [18]숙주를 보호할 수 있습니다.
취득 및 변경
포유동물에서 해면동물까지 미생물의 최초 획득은 태어날 때이며, 심지어 생식세포주를 통해서도 발생할 수 있다.식물에서, 군집화 과정은 뿌리 영역의 땅 아래, 발아 종자, 정자권 주변, 또는 위 지상 부분, 엽층, 꽃 구역 또는 [19]앤서피어에서 시작될 수 있다.세대별 뿌리권 미생물군의 안정성은 식물 유형에 따라 달라지지만 토양 구성, 즉 생활환경과 [20]비생활환경에 따라 더욱 달라진다.임상적으로, 새로운 마이크로바이오타는 만성 C. 디피실 [21]감염과 같은 감염을 치료하기 위해 분변 마이크로바이오타 이식을 통해 획득될 수 있다.
호스트별 마이크로바이오타
인간
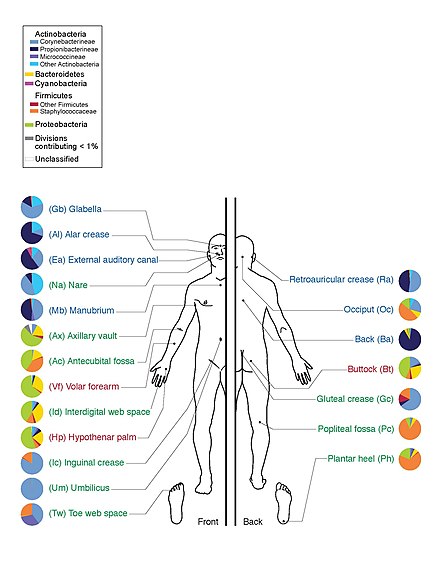
인간의 마이크로바이오타에는 박테리아, 곰팡이, 고세균, 바이러스가 포함된다.인체에 사는 미생물은 제외된다.인간 마이크로바이옴은 그들의 집단 [15]게놈을 말한다.
인간은 많은 미생물에 의해 식민지화되어 있다; 전통적인 추정치는 인간이 인간 세포보다 10배나 더 많은 비인간 세포를 가지고 산다는 것이었다; 더 최근의 추정치는 이것을 3:1로, 심지어 [22][23][24][25]1:1로 낮췄다.
사실, 이것들은 너무 작아서 인간의 [26]몸에는 약 100조 개의 미생물이 있습니다.
Human Microbiom Project는 특히 피부, 입, 코, 소화관, [15]질에 주로 서식하는 미생물군에 초점을 맞추어 인간 미생물군의 게놈을 배열했다.초기 결과를 [27]발표했던 2012년에는 이정표에 도달했다.
인간이 아닌 동물
- 양서류는 [28]피부에 미생물군을 가지고 있다.어떤 종들은 바트라코치티움 덴드로바티디스라는 이름의 곰팡이를 옮길 수 있고, 다른 종들은 병원체 군집에 저항하거나 항균성 피부 [29]펩타이드로 그들의 성장을 억제하면서, 그들의 미생물군에 따라 치명적인 감염을 일으킬 수 있습니다.
- 포유동물에서, 소와 같은 초식동물은 셀룰로오스를 단백질, 짧은 사슬 지방산, 그리고 기체로 바꾸기 위해 그들의 반추미생물체에 의존합니다.배양법은 존재하는 모든 미생물에 대한 정보를 제공할 수 없다.비교 메타제노믹스 연구는 각각의 소가 현저하게 다른 군집 구조를 가지고 있고, 예측된 표현형, [30]대사 잠재력이 동일한 식단을 제공받았음에도 불구하고 함께 수용되었고, 식물 세포벽 자원의 활용에서 명백히 동일한 것으로 보인다는 놀라운 결과를 도출했다.
- 쥐는 그들의 마이크로바이옴에 관해 가장 많이 연구된 포유동물이 되었다.장내 미생물군은 알레르기 기도 질환, 비만, 위장 질환, 당뇨병과 관련하여 연구되어 왔다.저용량 항생제를 통한 마이크로바이오타 주산기 이동은 향후 알레르기 기도 질환에 대한 민감성에 장기간 영향을 미칠 수 있다.미생물의 특정 하위 집합의 빈도는 질병의 심각성과 관련이 있다.산후 생활 초기에 특정 미생물의 존재는 미래의 면역 [31][32]반응을 지시합니다.관능생물 생쥐에서 특정 내장 박테리아가 수용체 무균 생쥐에게 특정 표현형을 전달하는 것으로 밝혀졌으며, 이는 대장 조절 T세포의 축적을 촉진하고 생쥐 지방과 맹장 대사물 [33]농도를 조절하는 균주를 촉진했다.이 조합적 접근방식을 통해 인간 [34]생물학에 대한 미생물의 기여에 대한 시스템 차원의 이해를 할 수 있습니다.그러나 천식, 알레르기,[35] 질병과 같은 질병과 관련하여 폐와 질과 같은 다른 점막 조직들도 연구되어 왔다.
- 곤충들은 그들만의 미생물을 가지고 있다.예를 들어, 잎을 깎는 개미는 매년 수백 킬로그램의 잎을 수확하는 거대한 땅속 군락을 형성하고 잎 속의 셀룰로오스를 직접 소화시킬 수 없다.그들은 곰팡이 정원을 식민지의 주요 식량원으로 유지하고 있다.곰팡이 자체가 셀룰로오스를 소화시키지 않는 반면, 다양한 박테리아를 포함한 미생물 집단은 그렇게 하고 있다.미생물 집단의 게놈 분석 결과 셀룰로오스 소화에 역할을 하는 많은 유전자가 밝혀졌다.이 마이크로바이옴의 예측된 탄수화물 분해 효소 프로필은 소 루멘의 그것과 비슷하지만, 종 구성은 거의 완전히 다릅니다.[36]초파리의 내장 마이크로바이오타는 상피 재생률, 세포 간격, 그리고 [37]상피에서 다른 종류의 세포 구성에 영향을 줌으로써 내장 모양에 영향을 미칠 수 있습니다.나방이 바쿨로바이러스 면역 관련 유전자에 감염되면 내장의 미생물량이 증가한다.[38]디프테란 장에서 장내분비세포는 내장미생물유래대사물을 검출하여 항균,[39] 기계적, 대사분지를 대응미생물소에 대한 숙주장의 선천적 면역반응으로 조정한다.
- 물고기는 단명종인 Nothobranchius furzeri를 포함한 그들만의 마이크로바이옴을 가지고 있습니다.어린 킬피쉬에서 중년의 킬피쉬로 내장 마이크로바이오타를 옮기는 것은 중년의 [40]킬피쉬의 수명을 크게 연장시킨다.
식물

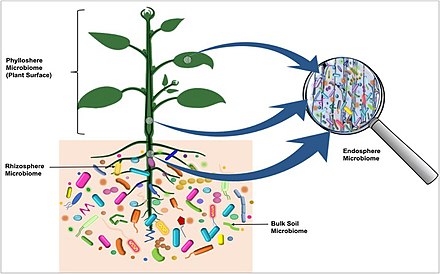
식물 마이크로바이옴은 최근 [42]씨앗에서 유래한 것으로 밝혀졌다.씨앗을 통해 전염되는 미생물은 특정 집단이 잎으로 이동하고 다른 집단은 [42]뿌리로 이동하는 특정한 경로로 발달하는 묘목 속으로 이동한다.오른쪽 그림에서는 뿌리권을 군집하는 미생물군이 뿌리권에 들어가 스톨론을 통해 다음 결핵세대를 군집하는 모습이 빨간색으로 시각화되어 있다.어미 덩이줄기에 존재하는 박테리아는 스톨론을 통과해 식물과 차세대 덩이줄기로 이동한다.[41]
- 토양은 감자 덩이줄기를 군집시키는 박테리아를 위한 주요 저장고이다.
- 박테리아는 감자종과는 다소 무관하게 토양에서 채취된다.
- 박테리아는 주로 식물 내부에서 스토론을 통해 덩이줄기에 서식할 수 있다.
- 감자 덩이줄기의 세균성 마이크로바이오타(microbiota)는 한 덩어리 세대에서 다음 세대로 전염되는 박테리아와 토양에서 채취되는 박테리아가 [41]뿌리를 통해 감자식물을 군집화한다.
식물은 다양한 영양소를 공급하기 때문에 미생물의 매력적인 숙주이다.식물의 미생물은 착생식물일 수도 있고 내생식물일 수도 있다.[43][44]균류와 균류는 수렴 진화를 통해 유사한 형태학을 발달시켜 유사한 생태학적 틈새를 차지하고 있다.그들은 숙주 세포를 관통하는 균사 같은 구조를 발달시킨다.상호주의적 상황에서 식물은 종종 헥소스 당을 곰팡이 심비온트의 무기 인산염과 교환한다.그러한 매우 오래된 연관성은 식물이 처음 [17][45]땅을 식민지로 만들었을 때 도움을 준 것으로 추측된다.식물성장촉진세균(PGPB)은 질소고정, 인과 같은 미네랄의 가용화, 식물호르몬의 합성, 미네랄 섭취의 직접적인 증가, 그리고 [46][47]병원체로부터의 보호와 같은 필수적인 서비스를 식물에 제공합니다.PGPB는 생태적 틈새 또는 기질을 위해 병원체와 경쟁하거나 억제성 대립 유전 화학물질을 생성하거나 병원체에[19] 대한 숙주 식물의 전신 저항을 유도하여 병원체로부터 식물을 보호할 수 있다.
조사.
숙주와 그 미생물 사이의 공생 관계는 그것이 [48][49]포유류의 면역 체계를 어떻게 형성할 수 있는지에 대한 실험실 연구 중에 있다.많은 동물에서, 면역 체계와 마이크로바이오타는 화학 신호를 교환함으로써 "교차 대화"에 관여할 수 있으며, 이것은 마이크로바이오타가 면역 반응성과 [50]표적에 영향을 미칠 수 있게 할 수 있습니다.박테리아는 산모에서 아이로 직접 접촉하거나 [51]태어난 후에 전염될 수 있다.신생아의 마이크로바이옴이 형성되면서, 보상 박테리아가 내장에 빠르게 퍼지면서, 다양한 면역 반응을 촉진하고 면역 체계를 오래 지속되는 [50]효과로 "프로그래밍"합니다.이 박테리아는 장 점막과 관련된 림프조직을 자극할 수 있으며,[50] 이는 장에 들어갈 수 있는 병원균에 대한 항체를 만들어 낼 수 있게 해준다.
인간 마이크로바이옴은 장에 있는 톨과 같은 수용체의 활성화에 역할을 할 수 있는데, 이것은 숙주 세포가 위험을 인식하고 손상을 복구하기 위해 사용하는 패턴 인식 수용체의 한 종류이다.병원균은 이러한 공존에 영향을 미쳐 질병, 염증의 메커니즘, 면역 내성,[52][53] 자가면역 질환을 포함한 면역 조절 장애로 이어질 수 있습니다.
마이크로바이오타 공진화

유기체는 생태계 내에서 진화하기 때문에 한 유기체의 변화가 다른 유기체의 변화에 영향을 미친다.홀로게놈 진화론은 자연선택의 대상은 개별 유기체가 아니라 미생물 군집을 포함한 유기체와 함께 유기체라고 주장한다.
산호초.홀로게놈 이론은 산호초에 [54]대한 연구에서 비롯되었다.산호초는 생물에 의해 만들어진 가장 큰 구조물로 풍부하고 매우 복잡한 미생물 군집을 포함하고 있다.지난 수십 년 동안 산호 개체수의 큰 감소가 일어났다.기후 변화, 수질 오염, 남획은 질병에 걸리기 쉬운 것으로 묘사된 세 가지 스트레스 요인이다.20개 이상의 다른 산호병이 설명되었지만, 그 중 소수의 산호병만이 그들의 원인 물질을 분리하여 특징지었습니다.산호 표백은 이러한 질병들 중 가장 심각한 질병이다.지중해에서 오큘리나 파타고니카의 표백은 1994년에 처음 기술되었고 곧 Vibrio Shiloi에 의한 감염으로 판명되었다.1994년부터 2002년까지 동부 지중해에서 매년 여름 O. 파타고니카의 세균 표백이 발생했다.그러나 놀랍게도 2003년 이후 지중해 동부의 파타고니카는 다른 질병들이 표백의 원인이 되고 있음에도 불구하고 V. shiloi 감염에 내성을 가지고 있다.놀라운 것은 산호는 수명이 수십 [55]년 정도이며 적응 [citation needed]면역 체계를 가지고 있지 않다는 사실에서 비롯된다.그들의 타고난 면역 체계는 항체를 생성하지 않으며, 그들은 겉으로 보기에 진화적인 시간 [citation needed]척도를 제외하고는 새로운 도전에 반응할 수 없을 것이다.
산호가 어떻게 특정 병원체에 대한 내성을 얻었는지에 대한 수수께끼는 산호와 그들의 공생 미생물 군집 사이에 역동적인 관계가 존재한다는 2007년 제안을 이끌어냈다.구성을 변경함으로써 홀로비온은 유전적 돌연변이와 선택에 의해서만 적응하는 것보다 훨씬 더 빠르게 변화하는 환경 조건에 적응할 수 있다고 생각된다.이 가설을 고등 식물과 동물을 포함한 다른 유기체들로 추정하는 것은 [54]진화에 대한 홀로게놈 이론의 제안으로 이어졌다.
2007년 현재[update] 홀로그램 이론은 여전히 [56]논의되고 있다.주요 비판은 V. shiloi가 산호 표백의 원인물질로 오인되고 표백된 O. patagonica의 존재는 단순히 기회주의적 [57]식민지화의 것이라는 주장이었다.만약 이것이 사실이라면, 이론으로 이어지는 기본적인 관찰은 무효가 될 것이다.이 이론은 자연선택의 전통적인 메커니즘으로 설명될 수 없는 적응의 빠른 변화를 설명하는 방법으로 상당한 인기를 얻었다.홀로그놈 이론에서 홀로비온은 자연 선택의 주요 단위가 될 뿐만 아니라 세포(상징, 내심근증)와 [7]게놈 수준에서 관찰되는 다른 통합 단계의 결과물이 되었다.
조사 방법
대상 앰프 아이콘 시퀀스 처리
| 시리즈의 일부 |
| 마이크로바이옴 |
|---|
 |
표적 엠프리콘 시퀀싱은 연구 대상 커뮤니티의 구성에 대한 기대가 있어야 합니다.표적 앰프콘 배열에서는 계통학적으로 유익한 마커를 배열 대상으로 한다.이러한 지표는 모든 예상 유기체에 이상적으로 존재해야 한다.그것은 또한 프라이머가 분류학적 수준에서 더 미세한 분해능을 가능하게 할 만큼 충분히 빠르게 진화하면서 광범위한 유기체의 유전자를 목표로 삼을 수 있을 만큼 충분히 보존되는 방식으로 진화해야 한다.인간 마이크로바이옴 연구의 공통적인 지표는 박테리아 16S rRNA의 유전자이다(즉, "16S rDNA, 리보솜 RNA [58]분자를 코드하는 DNA의 배열).리보솜은 모든 살아있는 유기체에 존재하기 때문에, 16S rDNA를 사용하는 것은 다른 마커를 사용하는 것보다 더 많은 유기체로부터 DNA를 증폭시킬 수 있다.16S rRNA 유전자는 느리게 진화하는 영역과 9개의 빠르게 진화하는 영역 모두를 포함합니다. HVRs라고도 알려져 있습니다; HVRs는 [59]넓은 프라이머를 설계하는 데 사용될 수 있는 반면 후자는 더 세밀한 분류학적 구별을 가능하게 합니다.그러나 종 수준의 분해능은 일반적으로 16S rDNA를 사용하여 가능하지 않다.프라이머의 표적이 될 수 없는 것은 증폭되지 않기 때문에 검출되지 않기 때문에 프라이머 선택은 중요한 단계입니다.게다가 유전자의 다른 HVR 또는 그 쌍을 증폭하기 위해 다른 프라이머 세트를 선택할 수 있습니다.서로 다른 대상 영역이 분류학적 [60]분류에 영향을 미치는 것으로 나타났기 때문에 관심 분류 그룹에 따라 HVR을 확대하는 적절한 선택을 해야 한다.
진핵생물 및 바이러스 군집의 표적 연구는 제한적이며[61], 인간 마이크로바이옴에서 [62]숙주 DNA를 증폭 및 감소된 진핵생물 및 바이러스 바이오매스에서 제외해야 하는 과제에 직면한다.
앰피콘 배열 후 분자 계통 발생법을 사용하여 미생물 군집의 조성을 추론한다.이는 앰플리콘을 운용분류단위(OTU)로 클러스터링하는 클러스터링 방법론 또는 앰플리콘 시퀀스 변종(ASV)을 식별하는 노이즈 제거 방법론을 통해 수행될 수 있다.
다음으로 계통발생적인 관계가 배열 사이에 추론된다.데이터의 복잡성으로 인해 일반적으로 UniFrac 거리 등의 거리 측정이 마이크로바이옴 샘플 간에 정의되며 거리 행렬에 대해 하류 다변량 방법이 수행된다.중요한 점은 데이터의 규모가 광범위하며, 이용 가능한 정보에서 패턴을 식별하기 위해 추가 접근법을 취해야 한다는 것이다.데이터 분석에 사용되는 도구에는 VAMPS,[63] QIIME,[64] matur[65] 및 노이즈[66] 제거를 위한 DADA2 또는[67] UNOISE3 등이 있습니다.
메타제노믹 배열
메타제노믹스는 미생물 [68][69][70]군집 연구에도 광범위하게 사용된다.메타게노믹 배열 분석에서 DNA는 커뮤니티 전체 구성원의 모든 유전자로부터 편향되지 않은 샘플을 얻는 것을 목표로 목표 없이 환경 샘플로부터 직접 회수된다.최근의 연구들은 [71]읽기의 시퀀스를 회복하기 위해 샷건 생어 시퀀싱 또는 파이로시퀀싱을 사용한다.그런 다음 판독치를 콘티그(contig)로 조립할 수 있습니다.배열의 계통학적 동일성을 결정하기 위해 BLAST와 같은 방법을 사용하여 사용 가능한 전체 게놈 배열과 비교됩니다.이 접근법의 한 가지 단점은 미생물 군집의 많은 구성원들이 대표적인 배열 게놈을 가지고 있지 않다는 것이지만, 이는 16S rRNA 앰플리콘 배열에도 적용되며 근본적인 [58]문제이다.산탄총 염기서열 분석에서는 미지의 게놈의 커버리지(50~100배)가 높아 효과적으로 de novo 게놈 조립을 함으로써 해결할 수 있다.미지의 유기체의 완전한 게놈을 이용할 수 있게 되면, 그것은 계통학적으로 비교될 수 있고, 새로운 분류군을 만들어 유기체를 생명의 나무에서 그 자리에 놓을 수 있다.새로운 접근법은 샷건 시퀀싱과 근접경계 데이터(Hi-C)를 결합하여 [72]배양 없이 완전한 미생물 게놈을 조립하는 것이다.
메타게노믹스가 참조 배열의 가용성에 의해 제한된다는 사실에도 불구하고 메타게노믹스의 표적 증폭기 배열에 대한 중요한 장점 중 하나는 메타게노믹스 데이터가 군집 [73][74]DNA의 기능적 잠재력을 설명할 수 있다는 것이다.표적 유전자 조사는 다른 유기체의 동일한 유전자 사이의 계통학적 관계만 밝혀내기 때문에 이것을 할 수 없다.기능 분석은 복구된 시퀀스를 KEGG와 같은 메타제노믹 주석의 데이터베이스와 비교함으로써 수행됩니다.이러한 유전자가 관여하는 [75]대사 경로는[76] MG-RAST, CAMERA 및 IMG/[77]M과 같은 도구를 사용하여 예측할 수 있습니다.
RNA 및 단백질 기반 접근법
추출된 [78]RNA의 파이로시퀀싱과 같은 방법을 통해 미생물 군집의 유전자 발현을 연구하기 위해 메타트랜스크립토믹스 연구가 수행되었다.구조 기반 연구는 또한 마이크로바이오타에서 [79]리보자임과 같은 비코드화 RNA(ncRNA)를 식별하였다.메타프로테오믹스는 마이크로바이오타에 의해 발현되는 단백질을 연구하는 접근법으로, 그 기능적 [80]잠재력에 대한 통찰력을 제공한다.
프로젝트
Human Microbiom Project는 2008년 미국 국립보건원(National Institute of Health)의 이니셔티브로, 건강한 사람과 질병이 있는 [81]사람 모두에서 발견되는 미생물을 식별하고 특징짓기 위한 프로젝트입니다.1억 1천 5백만 달러의 예산이 투입된 타당성 조사로 가장 잘 특징지어지는 이 5개년 프로젝트는 인간 마이크로바이옴의 변화가 인간의 건강이나 [81]질병과 어떻게 관련이 있는지를 실험했다.
Earth Microbiom Project(EMP)는 전 세계 미생물 집단을 분석하고 천연 샘플을 수집하기 위한 이니셔티브입니다.미생물은 매우 풍부하고 다양하며 생태계에서 중요한 역할을 한다.그러나 2010년 현재[update], 지구환경의 총 DNA 염기서열 분석 작업은 바닷물 1리터 또는 토양 [82]1그램에서 발견된 총 DNA의 1% 미만을 생산한 것으로 추정되며, 미생물 간의 구체적인 상호작용은 거의 알려지지 않았다.EMP는 서로 다른 바이오옴에서 20만 개에 달하는 샘플을 처리하는 것을 목표로 하고 있으며, 미생물 구성과 상호작용에 의해 환경과 생태계를 특징짓기 위해 지구상의 미생물의 완전한 데이터베이스를 생성하는 것을 목표로 하고 있습니다.이러한 데이터를 사용하여 새로운 생태 및 진화 이론을 제안하고 [83]테스트할 수 있습니다.
내장 미생물군과 제2형 당뇨병
내장 마이크로바이오타는 소화되지 않는 다당류(내성 녹말, 올리고당, 이눌린의 발효)를 분해하여 내장 무결성을 강화하거나 장 상피를 형성하고, 에너지를 수확하고, 병원체로부터 보호하고, 숙주 [84][85]면역력을 조절하는 역할을 하기 때문에 숙주의 건강에 매우 중요합니다.
여러 연구에서 당뇨병 환자의 장내 세균 구성은 Ractobacillus gasseri, Streptococcus mutans 및 Clostridiales 구성원의 수치가 증가하면 변화하고 Roseburia generalis 및 Faecalibacterium prausnitzi와 [86][87]같은 낙산 생성 박테리아가 감소하는 것으로 나타났다.이러한 변화는 항생제 남용, 다이어트, 그리고 나이와 같은 많은 요인들에 기인한다.
낙산염 생성의 감소는 장 투과성의 결함과 관련이 있으며, 이는 그램 음성 세균 세포벽에서 순환하는 리포다당류의 증가 수준인 내독소혈증을 초래할 수 있다.내산소혈증은 인슐린 [86]저항성의 발달과 관련이 있는 것으로 밝혀졌다.
게다가 낙산염 생산은 세로토닌 수치에 [86]영향을 미친다.세로토닌 수치가 높아지면 당뇨병 발병의 위험인자로 알려진 비만도 증가한다.
마이크로바이오타는 의학적인 목적으로 [88]인체에 이식될 수 있다.
내장미생물발달 및 항생제
인간의 내장 미생물군의 군집화는 [89]태어나기 전에 이미 시작되었을지도 모른다.환경에는 미생물의 발달에 영향을 미치는 여러 요인이 있으며, 출생 모드는 가장 영향을 [90]미치는 요소 중 하나이다.
장내 미생물, 특히 어린이들에게 큰 변화를 일으키는 것으로 관찰된 또 다른 요인은 높은 체질량지수(BMI)[91][92]와 같은 건강 문제와 관련된 항생제의 사용,[93] 그리고 비만과 같은 대사성 질환에 대한 더 높은 위험이다.유아에서 아목시실린과 마크로리드가 비피도박테리아, 장내박테리아 및 클로스트리디아 [94]균류의 변화로 특징지어지는 내장 미생물군에 상당한 변화를 일으키는 것으로 관찰되었다.성인의 항생제 한 가지 과정은 세균과 곰팡이 미생물군의 변화를 야기하고, 곰팡이 [95]군집의 더 지속적인 변화를 일으킨다.박테리아와 곰팡이는 내장에 함께 살고 [96][97]영양소 공급원에 대한 경쟁이 있을 것이다.Seelbinder 등 연구진은 장에 있는 보상균이 특히 프로피온산,[95] 아세트산 및 5-도데세노산염에 의한 칸디다 알비칸의 성장과 병원성을 조절한다는 것을 발견했다.칸디다균은 이전에 IBD와 관련되어 있었으며,[99] 또한 IBD가 심한 IBD 환자에게 투여된 생물학적 약물인 infliximab에 대한 비응답자에서 증가된 것으로 관찰되었다.프로피온산 및 아세트산은 모두 내장 마이크로바이오타 건강에 [100][101][102]이로운 것으로 관찰된 짧은 사슬 지방산(SCFA)이다.항생제가 장내 세균의 성장에 영향을 미칠 때, 특정 곰팡이의 과잉 성장이 있을 수 있으며,[95] 이는 조절되지 않을 때 병원성이 있을 수 있습니다.
프라이버시 문제
사람의 몸에 서식하는 미생물 DNA는 그 사람을 독특하게 식별할 수 있다.만약 그 사람이 익명으로 마이크로베 DNA 데이터를 기증한다면 개인의 사생활이 침해될 수 있다.그들의 의학적 상태와 신원이 [103][104][105]밝혀질 수도 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Dastgeer, K.M., Tumpa, F.H., Sultana, A., A., Akter, M.A. 및 Chakraborty, A. (20) "식물 마이크로바이옴 – 공동체 구성과 다양성을 형성하는 요소의 설명"Current Plant Biology, 23: 100161.doi:10.1016/j.cpb.2020.100161.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ De Sordi, Luisa; Lourenço, Marta; Debarbieux, Laurent (2019). "The battle within: interactions of bacteriophages and bacteria in the gastrointestinal tract". Cell Host & Microbe. 25 (2): 210–18. doi:10.1016/j.chom.2019.01.018. PMID 30763535. S2CID 73455329.
- ^ Peterson, J; Garges, S; et al. (2009). "The NIH Human Microbiome Project". Genome Research. NIH HMP Working Group. 19 (12): 2317–23. doi:10.1101/gr.096651.109. PMC 2792171. PMID 19819907.
- ^ Backhed, F.; Ley, R. E.; Sonnenburg, J. L.; Peterson, D. A.; Gordon, J. I. (2005). "Host-Bacterial Mutualism in the Human Intestine". Science. 307 (5717): 1915–20. Bibcode:2005Sci...307.1915B. doi:10.1126/science.1104816. PMID 15790844. S2CID 6332272.
- ^ Turnbaugh, P. J.; Ley, R. E.; Hamady, M.; Fraser-Liggett, C. M.; Knight, R.; Gordon, J. I. (2007). "The Human Microbiome Project". Nature. 449 (7164): 804–10. Bibcode:2007Natur.449..804T. doi:10.1038/nature06244. PMC 3709439. PMID 17943116.
- ^ Ley, R. E.; Peterson, D. A.; Gordon, J. I. (2006). "Ecological and Evolutionary Forces Shaping Microbial Diversity in the Human Intestine". Cell. 124 (4): 837–48. doi:10.1016/j.cell.2006.02.017. PMID 16497592. S2CID 17203181.
- ^ a b Salvucci, E. (2016). "Microbiome, holobiont and the net of life". Critical Reviews in Microbiology. 42 (3): 485–94. doi:10.3109/1040841X.2014.962478. PMID 25430522. S2CID 30677140.
- ^ Guerrero, R.; Margulis, Lynn; Berlanga, M. (2013). "Symbiogenesis: The holobiont as a unit of evolution". International Microbiology. 16 (3): 133–43. doi:10.2436/20.1501.01.188. PMID 24568029.
- ^ 데이븐포트, 에밀리 R 등"진화 중인 인간 마이크로바이옴"BMC Biology. vol. 15,1 127. 2017년 12월 27일, doi:10.1186/s12915-017-0454-7
- ^ "인간의 내장 균의 진화"앤드류 H. 몰러, 잉잉 리, 아이텔 음푸디 응글, 스티브 아후카 문데케, 엘리자베스 5세론스도르프, 앤 E푸세이, 마틴 피터스, 베아트리체 H한, 하워드 오크먼입니다미국 국립과학아카데미의 진행상황입니다.2014년 11월, 111 (46) 16431-35, 도이: 10.1073/pnas.1419136111
- ^ Jugder, Bat-Erdene; Kamareddine, Layla; Watnick, Paula I. (2021). "Microbiota-derived acetate activates intestinal innate immunity via the Tip60 histone acetyltransferase complex". Immunity. 54 (8): 1683–97.e3. doi:10.1016/j.immuni.2021.05.017. ISSN 1074-7613. PMC 8363570. PMID 34107298.
- ^ Mendes, R.; Raaijmakers, J.M. (2015). "Cross-kingdom similarities in microbiome functions". The ISME Journal. 9 (9): 1905–1907. doi:10.1038/ismej.2015.7. PMC 4542044. PMID 25647346.
- ^ a b c d Bosch, T. C. G.; McFall-Ngai, M. J. (2011). "Metaorganisms as the new frontier". Zoology. 114 (4): 185–190. doi:10.1016/j.zool.2011.04.001. PMC 3992624. PMID 21737250.
- ^ Foreau B., Biologicalie et complexité : histoire et modéles du commensalisme.2014년 프랑스 리옹 대학교 박사학위 논문.
- ^ a b c d Sherwood, Linda; Willey, Joanne; Woolverton, Christopher (2013). Prescott's Microbiology (9th ed.). New York: McGraw Hill. pp. 713–721. ISBN 9780073402406. OCLC 886600661.
- ^ Quigley, E. M. (Sep 2013). "Gut bacteria in health and disease". Gastroenterol Hepatol (N Y). 9 (9): 560–9. PMC 3983973. PMID 24729765.
- ^ a b c Remy W, Taylor TN, Hass H, Kerp H (1994). "Four hundred-million-year-old vesicular arbuscular mycorrhizae". Proc. Natl. Acad. Sci. USA. 91 (25): 11841–3. Bibcode:1994PNAS...9111841R. doi:10.1073/pnas.91.25.11841. PMC 45331. PMID 11607500.
- ^ Copeland, CS (Sep–Oct 2017). "The World Within Us". Healthcare Journal of New Orleans.
- ^ a b Compant S, Duffy B, Nowak J, Clément C, Barka EA (2005). "Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects". Appl Environ Microbiol. 71 (9): 4951–9. Bibcode:2005ApEnM..71.4951C. doi:10.1128/AEM.71.9.4951-4959.2005. PMC 1214602. PMID 16151072.
- ^ Tkacz, Andrzej; Cheema, Jitender; Chandra, Govind; Grant, Alastair; Poole, Philip S. (Nov 2015). "Stability and succession of the rhizosphere microbiota depends upon plant type and soil composition". ISME J. 9 (11): 2349–2359. doi:10.1038/ismej.2015.41. PMC 4611498. PMID 25909975.
- ^ Copeland, CS (19 April 2019). "What is Clostridium difficile?". Vitalacy.
- ^ 미국 미생물학회 FAQ: Wayback Machine에서 2016-12-31로 보관된 인간 미생물학 FAQ 2014년 1월
- ^ 2014년 2월, Microbe Magazine의 유다 L. 로스너.인간의 신체 세포보다 10배 많은 미생물 세포?
- ^ 네이처 뉴스의 앨리슨 애벗입니다.2016년 1월 8일 과학자들은 우리 몸에 인간의 세포보다 더 많은 박테리아가 있다는 신화를 깨뜨렸다.
- ^ Sender, R; Fuchs, S; Milo, R (Jan 2016). "Are We Really Vastly Outnumbered? Revisiting the Ratio of Bacterial to Host Cells in Humans". Cell. 164 (3): 337–40. doi:10.1016/j.cell.2016.01.013. PMID 26824647. S2CID 1790146.
- ^ "On and You"마이크로피아, www.micropia.nl/en/discover/stories/on-and-in-you/#~:text=%20on%20you%2C%20in, re%20known%20as%20human%20마이크로바이오타입니다.
- ^ "NIH Human Microbiome Project defines normal bacterial makeup of the body". NIH News. 13 June 2012.
- ^ Bataille, A; Lee-Cruz, L; Tripathi, B; Kim, H; Waldman, B (Jan 2016). "Microbiome Variation Across Amphibian Skin Regions: Implications for Chytridiomycosis Mitigation Efforts". Microb. Ecol. 71 (1): 221–32. doi:10.1007/s00248-015-0653-0. PMID 26271741. S2CID 12951957.
- ^ Woodhams DC, Rollins-Smith LA, Alford RA, Simon MA, Harris RN (2007). "Innate immune defenses of amphibian skin: antimicrobial peptides and more". Animal Conservation. 10 (4): 425–8. doi:10.1111/j.1469-1795.2007.00150.x. S2CID 84293044.
- ^ Brulc JM; Antonopoulos DA; Miller MEB; et al. (2009). "Gene-centric metagenomics of the fiber-adherent bovine rumen microbiome reveals forage specific glycoside hydrolases". Proc. Natl. Acad. Sci. USA. 106 (6): 1948–53. Bibcode:2009PNAS..106.1948B. doi:10.1073/pnas.0806191105. PMC 2633212. PMID 19181843.
- ^ Russell SL, Gold MJ; et al. (May 2012). "Early life antibiotic-driven changes in microbiota enhance susceptibility to allergic asthma". EMBO Rep. 13 (5): 440–7. doi:10.1038/embor.2012.32. PMC 3343350. PMID 22422004.
- ^ Russell SL, Gold MJ, et al. (Aug 2014). "Perinatal antibiotic-induced shifts in gut microbiota have differential effects on inflammatory lung diseases". J Allergy Clin Immunol. 135 (1): 100–9. doi:10.1016/j.jaci.2014.06.027. PMID 25145536.
- ^ Turnbaugh PJ, et al. (Dec 2006). "An obesity-associated gut microbiome with increased capacity for energy harvest". Nature. 444 (7122): 1027–31. Bibcode:2006Natur.444.1027T. doi:10.1038/nature05414. PMID 17183312. S2CID 4400297.
- ^ Faith JJ, Ahern PP, Ridaura VK, et al. (Jan 2014). "Identifying gut microbe-host phenotype relationships using combinatorial communities in gnotobiotic mice". Sci. Transl. Med. 6 (220): 220. doi:10.1126/scitranslmed.3008051. PMC 3973144. PMID 24452263.
- ^ Barfod, KK; Roggenbuck, M; Hansen, LH; Schjørring, S; Larsen, ST; Sørensen, SJ; Krogfelt, KA (2013). "The murine lung microbiome in relation to the intestinal and vaginal bacterial communities". BMC Microbiol. 13: 303. doi:10.1186/1471-2180-13-303. PMC 3878784. PMID 24373613.
- ^ Suen; Scott JJ; Aylward FO; et al. (2010). Sonnenburg, Justin (ed.). "An Insect Herbivore Microbiome with High Plant Biomass-Degrading Capacity". PLOS Genet. 6 (9): e1001129. doi:10.1371/journal.pgen.1001129. PMC 2944797. PMID 20885794.
- ^ Broderick, Nichole A.; Buchon, Nicolas; Lemaitre, Bruno (2014). "Microbiota-Induced Changes in Drosophila melanogaster Host Gene Expression and Gut Morphology". mBio. 5 (3): e01117–14. doi:10.1128/mBio.01117-14. PMC 4045073. PMID 24865556.
- ^ Jakubowska, Agata K.; Vogel, Heiko; Herrero, Salvador (May 2013). "Increase in Gut Microbiota after Immune Suppression in Baculovirus-infected Larvae". PLOS Pathog. 9 (5): e1003379. doi:10.1371/journal.ppat.1003379. PMC 3662647. PMID 23717206.
- ^ Watnick, Paula I.; Jugder, Bat-Erdene (2020-02-01). "Microbial Control of Intestinal Homeostasis via Enteroendocrine Cell Innate Immune Signaling". Trends in Microbiology. 28 (2): 141–149. doi:10.1016/j.tim.2019.09.005. ISSN 0966-842X. PMC 6980660. PMID 31699645.
- ^ Tibbs TN, Lopez LR, Arthur JC (2019). "The influence of the microbiota on immune development, chronic inflammation, and cancer in the context of aging". Microbial Cell. 6 (8): 324–334. doi:10.15698/mic2019.08.685. PMC 6685047. PMID 31403049.
- ^ a b c Buckholz, F., Antonielli, L., Kostich, T., Sessitch, A. 및 Mitter, B.(2019) "감자 내 세균 군집은 토양에서 모집되며 세대를 걸쳐 부분적으로 유전된다."PLOS 1, 14(11): e0223691.doi:10.1371/journal.pone.0223691.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ a b Abdelfattah, Ahmed; Wisniewski, Michael; Schena, Leonardo; Tack, Ayco J. M. (2021). "Experimental evidence of microbial inheritance in plants and transmission routes from seed to phyllosphere and root". Environmental Microbiology. 23 (4): 2199–2214. doi:10.1111/1462-2920.15392. ISSN 1462-2920. PMID 33427409. S2CID 231576517.
- ^ Berlec, Aleš (2012-09-01). "Novel techniques and findings in the study of plant microbiota: Search for plant probiotics". Plant Science. 193–194: 96–102. doi:10.1016/j.plantsci.2012.05.010. PMID 22794922.
- ^ Whipps, J.m.; Hand, P.; Pink, D.; Bending, G.d. (2008-12-01). "Phyllosphere microbiology with special reference to diversity and plant genotype" (PDF). Journal of Applied Microbiology. 105 (6): 1744–1755. doi:10.1111/j.1365-2672.2008.03906.x. ISSN 1365-2672. PMID 19120625. S2CID 35055151.
- ^ Chibucos MC, Tyler BM (2009). "Common themes in nutrient acquisition by plant symbiotic microbes, described by the Gene Ontology". BMC Microbiology. 9(Suppl 1): S6. doi:10.1186/1471-2180-9-S1-S6. PMC 2654666. PMID 19278554.
- ^ Kloepper, J. W (1993). "Plant growth-promoting rhizobacteria as biological control agents". In Metting, F. B. Jr (ed.). Soil microbial ecology: applications in agricultural and environmental management. New York: Marcel Dekker Inc. pp. 255–274. ISBN 978-0-8247-8737-0.
- ^ Bloemberg, G. V.; Lugtenberg, B. J. J. (2001). "Molecular basis of plant growth promotion and biocontrol by rhizobacteria". Current Opinion in Plant Biology. 4 (4): 343–350. doi:10.1016/S1369-5266(00)00183-7. PMID 11418345.
- ^ Palm, Noah W.; de Zoete, Marcel R.; Flavell, Richard A. (30 June 2015). "Immune–microbiota interactions in health and disease". Clinical Immunology. 159 (2): 122–127. doi:10.1016/j.clim.2015.05.014. ISSN 1521-6616. PMC 4943041. PMID 26141651.
- ^ Round, June L.; O'Connell, Ryan M.; Mazmanian, Sarkis K. (2010). "Coordination of tolerogenic immune responses by the commensal microbiota". Journal of Autoimmunity. 34 (3): J220–J225. doi:10.1016/j.jaut.2009.11.007. PMC 3155383. PMID 19963349.
- ^ a b c Cahenzli, Julia; Balmer, Maria L.; McCoy, Kathy D. (2012). "Microbial-immune cross-talk and regulation of the immune system". Immunology. 138 (1): 12–22. doi:10.1111/j.1365-2567.2012.03624.x. PMC 3533697. PMID 22804726.
- ^ Rosenberg, Eugene; Zilber-Rosenberg, Ilana (2016). "Microbes drive evolution of animals and plants: the hologenome concept". mBio. 7 (2): e01395–15. doi:10.1128/mbio.01395-15. PMC 4817260. PMID 27034283.
- ^ Blander, J Magarian; Longman, Randy S; Iliev, Iliyan D; Sonnenberg, Gregory F; Artis, David (19 July 2017). "Regulation of inflammation by microbiota interactions with the host". Nature Immunology. 18 (8): 851–860. doi:10.1038/ni.3780. ISSN 1529-2908. PMC 5800875. PMID 28722709.
- ^ Nikoopour, E; Singh, B (2014). "Reciprocity in microbiome and immune system interactions and its implications in disease and health". Inflamm Allergy Drug Targets. 13 (2): 94–104. doi:10.2174/1871528113666140330201056. PMID 24678760.
- ^ a b Rosenberg E, Koren O, Reshef L, Efrony R, Zilber-Rosenberg I (2007). "The role of microorganisms in coral health, disease and evolution". Nature Reviews Microbiology. 5 (5): 355–362. doi:10.1038/nrmicro1635. PMID 17384666. S2CID 2967190.
- ^ Baird AH, Bhagooli R, Ralph PJ, Takahashi S (2009). "Coral bleaching: the role of the host" (PDF). Trends in Ecology and Evolution. 24 (1): 16–20. doi:10.1016/j.tree.2008.09.005. PMID 19022522.
- ^ Leggat W, Ainsworth T, Bythell J, Dove S, Gates R, Hoegh-Guldberg O, Iglesias-Prieto R, Yellowlees D (2007). "The hologenome theory disregards the coral holobiont". Nature Reviews Microbiology. 5 (10): Online Correspondence. doi:10.1038/nrmicro1635-c1. S2CID 9031305.
- ^ Ainsworth TD, Fine M, Roff G, Hoegh-Guldberg O (2008). "Bacteria are not the primary cause of bleaching in the Mediterranean coral Oculina patagonica". The ISME Journal. 2 (1): 67–73. doi:10.1038/ismej.2007.88. PMID 18059488. S2CID 1032896.
- ^ a b Kuczynski, J.; Lauber, C. L.; Walters, W. A.; Parfrey, L. W.; Clemente, J. C.; Gevers, D.; Knight, R. (2011). "Experimental and analytical tools for studying the human microbiome". Nature Reviews Genetics. 13 (1): 47–58. doi:10.1038/nrg3129. PMC 5119550. PMID 22179717.
- ^ Chakravorty, Soumitesh; Helb, Danica; Burday, Michele; Connell, Nancy; Alland, David (May 2007). "A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria". Journal of Microbiological Methods. 69 (2): 330–339. doi:10.1016/j.mimet.2007.02.005. PMC 2562909. PMID 17391789.
- ^ Soriano-Lerma, Ana; Pérez-Carrasco, Virginia; Sánchez-Marañón, Manuel; Ortiz-González, Matilde; Sánchez-Martín, Victoria; Gijón, Juan; Navarro-Mari, José María; García-Salcedo, José Antonio; Soriano, Miguel (December 2020). "Influence of 16S rRNA target region on the outcome of microbiome studies in soil and saliva samples". Scientific Reports. 10 (1): 13637. Bibcode:2020NatSR..1013637S. doi:10.1038/s41598-020-70141-8. ISSN 2045-2322. PMC 7423937. PMID 32788589.
- ^ Marchesi, J. R. (2010). "Prokaryotic and Eukaryotic Diversity of the Human Gut". Advances in Applied Microbiology Volume 72. Advances in Applied Microbiology. Vol. 72. pp. 43–62. doi:10.1016/S0065-2164(10)72002-5. ISBN 9780123809896. PMID 20602987.
- ^ Vestheim, H.; Jarman, S. N. (2008). "Blocking primers to enhance PCR amplification of rare sequences in mixed samples – a case study on prey DNA in Antarctic krill stomachs". Frontiers in Zoology. 5: 12. doi:10.1186/1742-9994-5-12. PMC 2517594. PMID 18638418.
- ^ "VAMPS: The Visualization and Analysis of Microbial Population Structures". Bay Paul Center, MBL, Woods Hole. Retrieved 11 March 2012.
- ^ Caporaso, J. G.; Kuczynski, J.; Stombaugh, J.; Bittinger, K.; Bushman, F. D.; Costello, E. K.; Fierer, N.; Peña, A. G.; Goodrich, J. K.; Gordon, J. I.; Huttley, G. A.; Kelley, S. T.; Knights, D.; Koenig, J. E.; Ley, R. E.; Lozupone, C. A.; McDonald, D.; Muegge, B. D.; Pirrung, M.; Reeder, J.; Sevinsky, J. R.; Turnbaugh, P. J.; Walters, W. A.; Widmann, J.; Yatsunenko, T.; Zaneveld, J.; Knight, R. (2010). "QIIME allows analysis of high-throughput community sequencing data". Nature Methods. 7 (5): 335–336. doi:10.1038/nmeth.f.303. PMC 3156573. PMID 20383131.
- ^ Schloss, P. D.; Westcott, S. L.; Ryabin, T.; Hall, J. R.; Hartmann, M.; Hollister, E. B.; Lesniewski, R. A.; Oakley, B. B.; Parks, D. H.; Robinson, C. J.; Sahl, J. W.; Stres, B.; Thallinger, G. G.; Van Horn, D. J.; Weber, C. F. (2009). "Introducing mothur: Open-Source, Platform-Independent, Community-Supported Software for Describing and Comparing Microbial Communities". Applied and Environmental Microbiology. 75 (23): 7537–7541. Bibcode:2009ApEnM..75.7537S. doi:10.1128/AEM.01541-09. PMC 2786419. PMID 19801464.
- ^ Callahan, Benjamin J.; McMurdie, Paul J.; Rosen, Michael J.; Han, Andrew W.; Johnson, Amy Jo A.; Holmes, Susan P. (July 2016). "DADA2: High-resolution sample inference from Illumina amplicon data". Nature Methods. 13 (7): 581–583. doi:10.1038/nmeth.3869. ISSN 1548-7105. PMC 4927377. PMID 27214047.
- ^ Edgar, Robert C. (2016-10-15). "UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing": 081257. doi:10.1101/081257. S2CID 784388.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Turnbaugh, P. J.; Hamady, M.; Yatsunenko, T.; Cantarel, B. L.; Duncan, A.; Ley, R. E.; Sogin, M. L.; Jones, W. J.; Roe, B. A.; Affourtit, J. P.; Egholm, M.; Henrissat, B.; Heath, A. C.; Knight, R.; Gordon, J. I. (2008). "A core gut microbiome in obese and lean twins". Nature. 457 (7228): 480–484. Bibcode:2009Natur.457..480T. doi:10.1038/nature07540. PMC 2677729. PMID 19043404.
- ^ Qin, J.; Li, R.; Raes, J.; Arumugam, M.; Burgdorf, K. S.; Manichanh, C.; Nielsen, T.; Pons, N.; Levenez, F.; Yamada, T.; Mende, D. R.; Li, J.; Xu, J.; Li, S.; Li, D.; Cao, J.; Wang, B.; Liang, H.; Zheng, H.; Xie, Y.; Tap, J.; Lepage, P.; Bertalan, M.; Batto, J. M.; Hansen, T.; Le Paslier, D.; Linneberg, A.; Nielsen, H. B. R.; Pelletier, E.; Renault, P. (2010). "A human gut microbial gene catalogue established by metagenomic sequencing". Nature. 464 (7285): 59–65. Bibcode:2010Natur.464...59.. doi:10.1038/nature08821. PMC 3779803. PMID 20203603.
- ^ Tringe, S. G.; Von Mering, C.; Kobayashi, A.; Salamov, A. A.; Chen, K.; Chang, H. W.; Podar, M.; Short, J. M.; Mathur, E. J.; Detter, J. C.; Bork, P.; Hugenholtz, P.; Rubin, E. M. (2005). "Comparative Metagenomics of Microbial Communities". Science. 308 (5721): 554–557. Bibcode:2005Sci...308..554T. CiteSeerX 10.1.1.377.2288. doi:10.1126/science.1107851. PMID 15845853. S2CID 161283.
- ^ Wooley, J. C.; Godzik, A.; Friedberg, I. (2010). Bourne, Philip E. (ed.). "A Primer on Metagenomics". PLOS Computational Biology. 6 (2): e1000667. Bibcode:2010PLSCB...6E0667W. doi:10.1371/journal.pcbi.1000667. PMC 2829047. PMID 20195499.
- ^ Watson, Mick; Roehe, Rainer; Walker, Alan W.; Dewhurst, Richard J.; Snelling, Timothy J.; Ivan Liachko; Langford, Kyle W.; Press, Maximilian O.; Wiser, Andrew H. (2018-02-28). "Assembly of 913 microbial genomes from metagenomic sequencing of the cow rumen". Nature Communications. 9 (1): 870. Bibcode:2018NatCo...9..870S. doi:10.1038/s41467-018-03317-6. ISSN 2041-1723. PMC 5830445. PMID 29491419.
- ^ Muller, J.; Szklarczyk, D.; Julien, P.; Letunic, I.; Roth, A.; Kuhn, M.; Powell, S.; Von Mering, C.; Doerks, T.; Jensen, L. J.; Bork, P. (2009). "EggNOG v2.0: Extending the evolutionary genealogy of genes with enhanced non-supervised orthologous groups, species and functional annotations". Nucleic Acids Research. 38 (Database issue): D190–D195. doi:10.1093/nar/gkp951. PMC 2808932. PMID 19900971.
- ^ Kanehisa, M.; Goto, S.; Furumichi, M.; Tanabe, M.; Hirakawa, M. (2009). "KEGG for representation and analysis of molecular networks involving diseases and drugs". Nucleic Acids Research. 38 (Database issue): D355–D360. doi:10.1093/nar/gkp896. PMC 2808910. PMID 19880382.
- ^ Meyer, F.; Paarmann, D.; d'Souza, M.; Olson, R.; Glass, E. M.; Kubal, M.; Paczian, T.; Rodriguez, A.; Stevens, R.; Wilke, A.; Wilkening, J.; Edwards, R. A. (2008). "The metagenomics RAST server – a public resource for the automatic phylogenetic and functional analysis of metagenomes". BMC Bioinformatics. 9: 386. doi:10.1186/1471-2105-9-386. PMC 2563014. PMID 18803844.
- ^ Sun, S.; Chen, J.; Li, W.; Altintas, I.; Lin, A.; Peltier, S.; Stocks, K.; Allen, E. E.; Ellisman, M.; Grethe, J.; Wooley, J. (2010). "Community cyberinfrastructure for Advanced Microbial Ecology Research and Analysis: The CAMERA resource". Nucleic Acids Research. 39 (Database issue): D546–D551. doi:10.1093/nar/gkq1102. PMC 3013694. PMID 21045053.
- ^ Markowitz, V. M.; Ivanova, N. N.; Szeto, E.; Palaniappan, K.; Chu, K.; Dalevi, D.; Chen, I. M. A.; Grechkin, Y.; Dubchak, I.; Anderson, I.; Lykidis, A.; Mavromatis, K.; Hugenholtz, P.; Kyrpides, N. C. (2007). "IMG/M: A data management and analysis system for metagenomes". Nucleic Acids Research. 36 (Database issue): D534–D538. doi:10.1093/nar/gkm869. PMC 2238950. PMID 17932063.
- ^ Shi, Y.; Tyson, G. W.; Delong, E. F. (2009). "Metatranscriptomics reveals unique microbial small RNAs in the ocean's water column". Nature. 459 (7244): 266–269. Bibcode:2009Natur.459..266S. doi:10.1038/nature08055. PMID 19444216. S2CID 4340144.
- ^ Jimenez, R. M.; Delwart, E.; Luptak, A (2011). "Structure-based Search Reveals Hammerhead Ribozymes in the Human Microbiome". Journal of Biological Chemistry. 286 (10): 7737–7743. doi:10.1074/jbc.C110.209288. PMC 3048661. PMID 21257745.
- ^ Maron, PA; Ranjard, L.; Mougel, C.; Lemanceau, P. (2007). "Metaproteomics: A New Approach for Studying Functional Microbial Ecology". Microbial Ecology. 53 (3): 486–493. doi:10.1007/s00248-006-9196-8. PMID 17431707. S2CID 26953155.
- ^ a b "NIH Human Microbiome Project". US National Institutes of Health, Department of Health and Human Services, US Government. 2016. Archived from the original on 11 June 2016. Retrieved 14 June 2016.
- ^ Gilbert, J. A.; Meyer, F.; Antonopoulos, D.; et al. (2010). "Meeting Report: The Terabase Metagenomics Workshop and the Vision of an Earth Microbiome Project". Standards in Genomic Sciences. 3 (3): 243–248. doi:10.4056/sigs.1433550. PMC 3035311. PMID 21304727.
- ^ Gilbert, J. A.; O'Dor, R.; King, N.; Vogel, T. M. (2011). "The importance of metagenomic surveys to microbial ecology: Or why Darwin would have been a metagenomic scientist". Microbial Informatics and Experimentation. 1 (1): 5. doi:10.1186/2042-5783-1-5. PMC 3348666. PMID 22587826.
- ^ Ibrahim, Nesma (2018-07-01). "Gut Microbiota and Type 2 Diabetes Mellitus : What is The Link ?". Afro-Egyptian Journal of Infectious and Endemic Diseases. 6 (2): 112–119. doi:10.21608/aeji.2018.9950. ISSN 2090-7184. S2CID 3900880.
- ^ Thursby, Elizabeth; Juge, Nathalie (2017-06-01). "Introduction to the human gut microbiota". Biochemical Journal. 474 (11): 1823–1836. doi:10.1042/BCJ20160510. ISSN 0264-6021. PMC 5433529. PMID 28512250.
- ^ a b c Muñoz-Garach, Araceli; Diaz-Perdigones, Cristina; Tinahones, Francisco J. (December 2016). "Microbiota y diabetes mellitus tipo 2". Endocrinología y Nutrición (in Spanish). 63 (10): 560–568. doi:10.1016/j.endonu.2016.07.008. PMID 27633134.
- ^ Blandino, G.; Inturri, R.; Lazzara, F.; Di Rosa, M.; Malaguarnera, L. (2016-11-01). "Impact of gut microbiota on diabetes mellitus". Diabetes & Metabolism. 42 (5): 303–315. doi:10.1016/j.diabet.2016.04.004. ISSN 1262-3636. PMID 27179626.
- ^ https://microbioma.it.
{{cite journal}}: 일기장의 필요성(도움말)을 인용한다.누락 또는 비어 있음(도움말) - ^ Vandenplas, Y., Carnelli, V. P., Ksiazyk, J., Luna, M. S., Migacheva, N., Moselmans, J. M. ... 및 Wabitch, M.(2020), 초기 장내 미생물 발달에 영향을 미치는 요인.영양, 78, 110812
- ^ Korpela K, Helve O, Kolho KL, Saisto T, Skogberg K, Dikareva E, Stefanovic V, Salon A, Andersson S, de Vos WM.제왕절개 신생아의 모체 분변 미생물 이식은 장내 미생물의 정상적인 발달을 빠르게 회복시킨다: 개념 증명 연구.2020년 10월 15일;183 (2):324-334.e5.doi:10.1016/j.cell.2020.08.047.Epub 2020 10월 1일 PMID 33007265.
- ^ K., K., 살로넨, A., Saxen, H., Nikkonen, A., Peltola, V., Jaakkola, T., ... 및 Kolho, L. (20)초기 생애의 항생제는 미래의 세로형 유아 코호트에서 특정 내장 미생물군 시그니처와 관련된다.소아과 연구, 1-6
- ^ Shei, K., Simpson, M. R., Avershina, E., Rudi, K., öien, T., Juliusson, P. B. ... 및 ödegod, R. A. (20)초기 장내 곰팡이균과 세균 미생물 및 소아 성장.소아과 프런티어, 8, 658
- ^ K., K., 살로넨, A., Virta, L. J., Kekkonen, R. A., Forslund, K., Bork, P. 및 De Vos, W. M. (2016년)장내 마이크로바이옴은 핀란드 취학 전 아동들의 평생 항생제 사용과 관련이 있다.네이처 커뮤니케이션, 7, 10410
- ^ K., K., 살로넨, A., Saxen, H., Nikkonen, A., Peltola, V., Jaakkola, T., ... 및 Kolho, L. (20)초기 생애의 항생제는 미래의 세로형 유아 코호트에서 특정 내장 미생물군 시그니처와 관련된다.소아과, 1-6
- ^ a b c Seelbinder, B., Chen, J., Brunke, S., Vazquez-Uribe, R., Santhaman, R., Meyer, A. C., ... 및 Panagiotou, G. (2020)항생제는 박테리아보다 곰팡이에 더 오래 지속되는 영향을 주면서 인간의 내장 공동체에 상호주의에서 경쟁으로 변화를 일으킨다.마이크로바이옴, 8(1), 1-20
- ^ 카브랄, D. J., 페누무추, S., 노리스, Morones-Rires, J. R. 및 Belenky, P. (2018년)대장균과 칸디다 알비칸의 미생물 경쟁은 수용성 곰팡이 인자를 드러낸다.미생물 세포, 5(5), 249
- ^ Peleg, A. Y., Hogan, D. A. 및 Mylonakis, E. (2010).의학적으로 중요한 세균-펑갈 상호작용입니다.Nature Reviews 미생물학, 8(5), 340-349
- ^ Sokol H, Leducq V, Ascard H, Pham H P, Jegou S, Landman C, Cohen D, Liguori G, Bourrier A, Nion-Larmurier I, Cosnes J, Seksik P, Langella ML, Skurn, SkurnIBD의 곰팡이성 미생물 이상증.Gut 2017;66:1039–1048.doi:10.1136/gutjnl-2015-310746
- ^ Reebecka Ventin-Holmberg, Anja Eberl, Schahzad Saqib, Katri Korpela, Seppo Virtanen, Taina Sipponen, Anne Salonen, Paiivi Saavalainen, Eija Nissilae, 세균 및 Foxima의 약제 infliab 표지자
- ^ 엘 헤이지, 헤르난데스 사나브리아, E., 칼라타유드 아로요, 프롭스, R. 및 반 데 비엘레, T.(2019).프로피온산염 생산 컨소시엄은 인체 장내 미생물 생태계의 동적인 체외 모델에서 항생제 유도 이상 생물을 회복시킨다.미생물학 프런티어 10, 1206
- ^ Tian, X., Hellman, J., Horswill, A. R., Crossby, H. A., Francis, K. P. 및 Prakash, A.(2019).장내 마이크로바이옴 유래 프로피온산염 수치 상승은 생쥐의 무균 폐염 및 세균 면역 감소와 관련이 있다.미생물학 분야 프런티어 10, 159호
- ^ Li, Y, Faden, H.S. 및 Zhu, L.(2020).생후 첫 2년 동안 식생활 변화에 대한 내장 미생물군의 반응.약학 분야 프런티어, 11, 334
- ^ magazine, Ewen. "Microbial DNA in Human Body Can Be Used to Identify Individuals". Scientific American. Retrieved 2015-05-17.
- ^ Callaway, Ewen (2015). "Microbiomes raise privacy concerns". Nature. 521 (7551): 136. Bibcode:2015Natur.521..136C. doi:10.1038/521136a. PMID 25971486. S2CID 4393347.
- ^ Yong, Ed (2015-05-11). "Can The Microbes You Leave Behind Be Used to Identify You?". National Geographic. Retrieved 2015-05-17.