메틸전달효소
Methyltransferase메틸전달효소는 모두 기질을 메틸화하는 효소의 큰 그룹이지만 구조적인 특징에 따라 여러 개의 하위 클래스로 나눌 수 있다.메틸전달효소의 가장 일반적인 클래스는 클래스 I로, 모두 S-아데노실메티오닌(SAM) 결합을 위한 로스만 폴드를 포함한다. 클래스 II 메틸전달효소는 SET 도메인 히스톤 메틸전달효소에 의해 예시되는 SET 도메인과 막과 [1]관련된 클래스 III 메틸전달효소를 포함한다.메틸전달효소는 메틸전달반응에서 서로 다른 기질을 이용하는 다른 유형으로 분류될 수도 있다.이러한 유형에는 단백질 메틸전달효소, DNA/RNA 메틸전달효소, 천연물 메틸전달효소 및 비SAM 의존성 메틸전달효소 등이 포함됩니다.SAM은 메틸전달효소에 대한 전형적인 메틸 공여체이지만, 다른 메틸 공여체의 예는 자연에서 볼 수 있다.메틸 전달의 일반적인 메커니즘은 S2와N 같은 친핵성 공격이며, 메티오닌 황은 이탈기로서 작용하고, 메틸기는 효소 기질로 메틸기를 전달하는 전자 친핵기로서 작용한다.이 과정에서 SAM은 S-아데노실 호모시스테인(SAH)으로 변환된다.SAM-메틸 결합의 파괴와 기질-메틸 결합의 형성은 거의 동시에 일어난다.이러한 효소 반응은 많은 경로에서 발견되며 유전병, 암, 대사 질환과 관련이 있다.메틸 전달의 또 다른 유형은 1차 대사물, 단백질, 지질 및 RNA에서 불활성 탄소 원자의 메틸화인 라디칼 S-아데노실 메티오닌(SAM)이다.
기능.
유전학
다른 후생유전학적 변형뿐만 아니라 메틸화는 전사, 유전자 안정성, 부모의 [2]각인에 영향을 미친다.그것은 직접적으로 염색질 구조에 영향을 미치고 유전자 자체의 돌연변이 없이 유전자 전사를 조절하거나 심지어 유전자를 완전히 침묵시키거나 활성화시킬 수 있다.이러한 유전자 제어의 메커니즘은 복잡하지만, DNA의 저메틸화와 과메틸화는 많은 질병과 관련이 있다.
단백질 조절
단백질의 메틸화는 단백질-단백질 상호작용, 단백질-에서 조절 역할을 한다.DNA 상호작용과 단백질 활성화.
예: 중요한 유사분열 단백질인 RCC1은 염색체의 동원체와 상호작용할 수 있도록 메틸화된다.이는 메틸화가 히스톤 단백질 H2A 및 H2B에 대한 RCC1의 부착을 조절하기 때문에 단백질-단백질 상호작용 조절의 한 예이다.RCC1-크로마틴 상호작용은 또한 단백질-DNA 상호작용의 한 예이며, 이 단백질이 메틸화 될 때 RCC1의 또 다른 도메인이 DNA와 직접 상호작용하기 때문이다.RCC1이 메틸화되지 않은 경우 분할 셀은 여러 개의 스핀들 극을 가지며 일반적으로 생존할 수 없습니다.
p53은 DNA 손상 반응에서 리신의 활성화와 다른 단백질과의 상호작용을 조절하기 위해 리신에 메틸화된다.이것은 단백질-단백질 상호작용과 단백질 활성화의 조절의 예입니다. p53은 DNA 복구 경로를 활성화하고, 아포토시스를 시작하고, 세포 순환을 멈추는 것으로 알려진 종양 억제제입니다.전반적으로, DNA의 돌연변이에 반응하여 세포에 신호를 보내서 그것들을 고치거나 세포사멸을 시작함으로써 이러한 돌연변이가 암에 기여할 수 없도록 합니다.
NF-δB(염증에 관여하는 단백질)는 메틸전달효소 SETD6의 알려진 메틸화 타겟으로, 그 서브유닛 중 하나인 RelA를 억제함으로써 NF-δB 시그널링을 무효화한다.이것은 전사 활성화와 염증 반응을 감소시켜 NF-γB의 메틸화를 이 경로를 통한 세포 신호 전달을 [3]감소시키는 조절 과정으로 만든다.
천연물 메틸전달효소는 보조 인자, 신호 전달 분자 및 대사물의 가용성을 포함하여 대사 경로에 다양한 입력을 제공합니다.이것은 단백질의 활성을 조절함으로써 다양한 세포 경로를 조절한다.
종류들
히스톤메틸전달효소
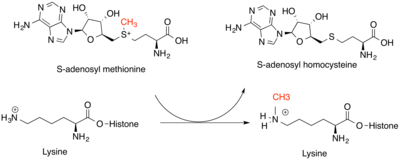
히스톤 메틸전달효소는 후생유전자 수준에서 유전자 조절에 매우 중요하다.이들은 주로 γ-질소의 리신과 히스톤 꼬리의 아르기닌 구아니디늄기를 수식한다.리신메틸전달효소 및 아르기닌메틸전달효소는 효소의 고유한 종류이지만, 둘 다 히스톤 기질에 대한 메틸 공여체로 SAM을 결합한다.리신 아미노산은 1기, 2기 또는 3기의 메틸기로 변형될 수 있으며, 아르기닌 아미노산은 1기 또는 2기의 메틸기로 변형될 수 있다.이것은 양전하와 잔류 소수성의 강도를 증가시켜 다른 단백질이 메틸 마크를 인식할 수 있게 합니다.이 수정의 효과는 히스톤 꼬리의 수정 위치 및 그 주변의 다른 히스톤 수정에 따라 달라집니다.변형 위치는 DNA 배열뿐만 아니라 작은 비코드 RNA 및 DNA 자체의 메틸화에 의해 부분적으로 결정될 수 있다.가장 일반적으로 척추동물에서 메틸화된 것은 히스톤 H3 또는 H4이다.변형 주변 유전자의 전사가 증가하거나 감소할 수 있다.전사의 증가는 염색질 응축의 감소에 의한 결과이며, 전사의 감소는 염색질 [4]응축의 증가로 인한 결과이다.히스톤의 메틸 표시는 염색질을 [5]추가로 수정할 수 있는 다른 단백질의 모집 부위의 역할을 함으로써 이러한 변화에 기여합니다.
N말단메틸전달효소
N-alpha 메틸전달효소는 단백질 표적에서 SAM에서 N 말단 질소로 메틸기를 전달한다.N 말단 메티오닌은 먼저 다른 효소에 의해 분해되며, X-프롤린-라이신 컨센서스 배열은 메틸전달효소에 의해 인식된다.알려진 모든 기질에 대해 X 아미노산은 알라닌, 세린 또는 프롤린이다.이 반응은 메틸화 단백질과 SAH를 생성한다. 사람에게 이러한 메틸전달효소의 알려진 표적은 RCC-1과 레티노블라스토마 단백질(과도한 세포 분열을 억제하는 종양 억제 단백질)을 포함한다.RCC-1 메틸화는 핵 포락선이 없을 때 일부 핵 단백질의 국부화를 조정하기 때문에 유사분열에서 특히 중요하다.RCC-1이 메틸화되지 않으면 여분의 스핀들 [6]극이 형성되어 세포분열이 비정상적으로 진행됩니다.망막아종 단백질 N-말단 메틸화의 기능은 알려져 있지 않다.
DNA/RNA메틸전달효소
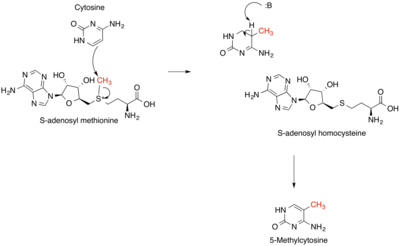
유전자 조절의 핵심 성분인 DNA 메틸화는 주로 5'메틸시토신을 형성하는 염기 시토신의 5-탄소에서 발생한다.[7]메틸화는 DNMT1, DNMT2, DNMT3를 포함한 DNA 메틸전달효소 효소에 의해 촉매되는 후생적 변형이다.이러한 효소들은 메틸 공여체로 S-아데노실메티오닌을 사용하며, 세 가지 형태 사이에 여러 고도로 보존된 구조적 특징을 포함한다. 이러한 효소들은 S-아데노실메티오닌 결합 부위, 반응 메커니즘에 중요한 티올라 음이온을 형성하는 바이시날 프롤린-시스테인 쌍, 시토신 기질 결합 포켓을 포함한다.DNA메틸전달효소의 많은 특징들은 박테리아에서 포유류에 이르기까지 많은 생물계급에 걸쳐 보존되어 있다.특정 유전자의 발현을 조절하는 것 외에도, 다양한 단백질 복합체가 있으며, 많은 것들이 인간의 건강에 영향을 미치며, 메틸화된 DNA 인식 부위에만 결합합니다.초기 DNA 메틸전달효소 중 많은 것들이 많은 [8]원시 RNA 종들을 보호하기 위해 RNA 세계에서 활성화 되어야 했던 RNA 메틸전달효소로부터 유래된 것으로 생각되어 왔다. RNA 메틸화는 다양한 종류의 RNA 종 viz.m에서 관찰되어 왔다.RNA, rRNA, tRNA, snRNA, snRNA, miRNA, tmRNA 및 바이러스 RNA 종.특정 RNA 메틸전달효소는 세포 주위에 널리 퍼져 있는 요구와 환경에 따라 RNA 종에 표시하기 위해 세포에 의해 사용된다. 2'-O-메틸화, m6A 메틸화, m1G 메틸화 및 m5C는 서로 다른 유형의 RNA에서 관찰되는 가장 일반적인 메틸화 표시이다.
6A는 다음과 [9]같이 화학 반응을 촉매하는 효소이다.
S-아데노실-L-메티오닌+DNA아데노실-L-호모시스테인+DNA6-메틸아미노푸린
m6A는 2015년까지 원핵생물에서 주로 발견되었으며, 일부 진핵생물에서도 확인되었습니다.m6A 메틸전달효소는 특히 숙주 [10]시스템이 제한효소를 통해 자신의 게놈을 소화하는 것을 방지하기 위해 C-6 위치에서 DNA의 아미노기를 메틸화하였습니다.
m5C는 유전자 전사를 조절하는 역할을 한다. m5C 전이효소는 시토신의 C-5 위치에 있는 DNA에서 C5-메틸시토신을 생성하는 효소이며 대부분의 식물과 일부 [11]진핵생물에서 발견된다.
천연물메틸전달효소
자연산 메틸전달효소(NPMT)는 자연적으로 생성된 작은 분자에 메틸기를 첨가하는 다양한 효소 그룹이다.많은 메틸전달효소처럼 SAM은 메틸 공여체로 이용되며 SAH가 생성된다.메틸기는 S, N, O 또는 C 원자에 첨가되며, 이들 원자 중 어느 원자가 수식되는지에 따라 분류되며, O-메틸전달효소는 가장 큰 종류를 나타낸다.이러한 반응의 메틸화 산물은 공동 인자, 색소, 신호 전달 화합물 및 대사물을 포함한 다양한 기능을 한다.NPMT는 이러한 화합물의 반응성과 가용성을 변경함으로써 규제 역할을 할 수 있습니다.이 효소들은 다른 종에 걸쳐 잘 보존되어 있지 않습니다. 왜냐하면 그것들은 종이나 더 작은 종의 특별한 경로를 위해 작은 분자를 제공하는 데 더 특별한 기능을 하기 때문입니다.이러한 다양성은 일반 산-염기 촉매, 금속 기반 촉매, 촉매 아미노산이 필요하지 않은 근접 및 용해 효과 등 다양한 촉매 전략을 반영합니다.NPMT는 기능적으로 가장 다양한 [12]종류의 메틸전달효소이다.
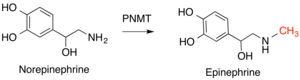
인간에서 이 효소 클래스의 중요한 예로는 노르에피네프린을 에피네프린으로 [14]변환하는 페닐레타놀라민 N-메틸전달효소(PNMT)와 히스타민 [15]대사 과정에서 히스타민을 메틸화하는 히스타민 N-메틸전달효소(HNMT)가 있다.카테콜-O-메틸전달효소(COMT)는 도파민, 에피네프린 및 노르에페린을 [16]포함하는 캐톨라민으로 알려진 분자의 클래스를 분해합니다.
비SAM 의존성 메틸전달효소
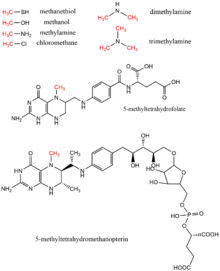
메탄올, 메틸테트라히드로폴레이트, 모노, 디, 트리메틸아민, 메탄티오르, 메틸테트라히드로메탄 및 클로로메탄은 모두 메틸기 공여체로 생물학에서 발견되는 메틸기 공여체이며, 일반적으로 보조인자 비타민 B12를 [17]사용하는 효소 반응에서 발견된다.이러한 기질들은 메티오닌 생합성, 메타노제네시스 및 아세트제네시스를 포함한 메틸전달경로에 기여한다.
래디칼 SAM메틸전달효소
서로 다른 단백질 구조와 촉매 작용 메커니즘을 기반으로, 래디칼 SAM(RS) 메틸화효소에는 세 가지 유형이 있습니다.클래스 A, B 및 C클래스 A RS 메틸화효소는 4가지 효소 중 가장 잘 특징지어지며 RlmN과 Cfr 모두와 관련이 있다.RlmN은 번역 충실도를 높이는 세균에 많이 존재하며, RlmN은 23S rRNA에서 아데노신 2503(A2503)의 C2와 아데노신(A37)의 C2의 메틸화를 촉매한다.한편 Cfr은 A2503의 C8의 메틸화도 촉매하고 C2메틸화도 [18]촉매한다.클래스 B는 현재 sp2-하이브리드 탄소 원자만을 촉매하는 클래스 A와 달리 서로 다른 기질 집합에서 sp2-하이브리드 탄소 원자와 sp3-하이브리드 탄소 원자를 모두 숙성시킬 수 있는 래디칼 SAM 메틸라아제 중 가장 큰 클래스이다.클래스 B를 다른 도메인과 구별하는 주요 차이점은 RS [19]도메인에 결합하는 추가 N 말단 코발라민 결합 도메인입니다.클래스 C 메틸화효소는 RS 효소인 코프로포르피리노겐 III 산화효소(HemN)와 상동배열을 가지며, 이는 또한 sp2-하이브리드 탄소 중심부의 메틸화를 촉매하지만 클래스 [18]A의 메커니즘에서 메틸화에 필요한 2개의 시스테인이 결여되어 있다.
임상적 의의
유전자 발현 및/또는 기능을 조절하는 생물학적 과정과 마찬가지로 비정상적인 DNA 메틸화는 ICF, 레트 증후군 및 연약성 [2]X 증후군과 같은 유전적 장애와 관련되어 있다.암세포는 일반적으로 DNA 메틸화 활성이 적지만, 정상세포에서 메틸화되지 않은 부위의 과메틸화는 종종 종양 억제 유전자를 비활성화하는 방법으로 기능한다.전체 DNA 메틸전달효소 활성의 억제가 치료 옵션으로 제안되었지만, 시토신 기질의 유사체인 DNMT 억제제는 시토신과의 유사성(오른쪽 참조)으로 인해 매우 독성이 높은 것으로 밝혀졌다. 이러한 뉴클레오티드와 유사성은 억제제를 DNA 번역에 포함시켜 비기능성을 유발한다.DNA를 합성하는 것.
항생제 라인졸리드의 리보솜 RNA 결합 부위를 변화시키는 메틸화효소는 리보솜 RNA에 작용하는 다른 항생제에 대한 교차저항을 일으킨다.이 유전자를 전달할 수 있는 플라스미드 벡터는 잠재적으로 위험한 교차저항의 [20]원인이다.
질병과 관련된 메틸전달효소 효소의 예:
- 티오푸린 메틸전달효소: 이 유전자의 결함은 티오푸린 화합물, 화학요법 및 면역억제제 치료에 사용되는 약물의 독성 축적을 일으킨다.
- 메티오닌합성효소: 비타민 B12 결핍에 의해 야기되는 유해성 빈혈은 메티오닌합성효소 보조인자의 결핍에 의해 야기된다.
약물 발견 및 개발 응용 프로그램
최근 연구는 메틸 대체 알킬기를 운반하는 S-아데노실 메티오닌(SAM) 유사체를 사용하기 위해 자연적으로 발생하는 항암제의 메틸화에 관여하는 메틸전달효소를 밝혀냈다.약물 발견 및 약물 개발의 맥락에서 차등적으로 알킬화된 SAM 유사체를 생성하고 이용하기 위한 촉진성 화학효소 플랫폼의 개발을 알킬란도미화라고 [21]한다.
암 치료에서의 응용
인간 세포에서는 m5C가 [22]암의 이상 종양 세포와 관련이 있는 것으로 밝혀졌다.m5C의 역할과 잠재적 적용은 암에서 손상된 DNA의 균형을 유지하는 것을 포함한다.암세포의 양쪽 타입(하이퍼메틸화/하이포메틸화)과 암의 환경 양쪽에서 m5C량을 변화시켜 종양세포를 [23]억제하는 등가점에 도달함으로써 DNA의 후생유전학적 수복을 적용할 수 있다.
예
예를 들어 다음과 같습니다.
레퍼런스
- ^ Katz, J. E.; Dlakic, M; Clarke S (18 July 2003). "Automated identification of putative methyltransferases from genomic open reading frames". Molecular & Cellular Proteomics. 2 (8): 525–40. doi:10.1074/mcp.M300037-MCP200. PMID 12872006.
- ^ a b Siedlecki, P; Zielenkiewicz, P (2006). "Mammalian DNA methyltransferases". Acta Biochimica Polonica. 53 (2): 245–56. doi:10.18388/abp.2006_3337. PMID 16582985.
- ^ Levy, Dan; et al. (5 December 2010). "Lysine methylation of the NF-κB subunit RelA by SETD6 couples activity of the histone methyltransferase GLP at chromatin to tonic repression of NF-κB signaling". Nature Immunology. 12 (1): 29–36. doi:10.1038/ni.1968. PMC 3074206. PMID 21131967.
- ^ Turner, Bryan M. (2001). Chromatin and gene regulation : mechanisms in epigenetics. Malden, MA: Blackwell Science. ISBN 978-0865427433.
- ^ Greer, Eric L.; Shi, Yang (3 April 2012). "Histone methylation: a dynamic mark in health, disease and inheritance". Nature Reviews Genetics. 13 (5): 343–357. doi:10.1038/nrg3173. PMC 4073795. PMID 22473383.
- ^ Clarke, Paul (May 2007). "Anchoring RCC1 by the tail". Nature Cell Biology. 9 (5): 485–487. doi:10.1038/ncb0507-485. PMID 17473856. S2CID 711645.
- ^ Lan, J; Hua, S; He, X; Zhang, Y (2010). "DNA methyltransferases and methyl-binding proteins of mammals". Acta Biochimica et Biophysica Sinica. 42 (4): 243–52. doi:10.1093/abbs/gmq015. PMID 20383462.
- ^ Rana, Ajay K.; Ankri, Serge (2016-01-01). "Reviving the RNA World: An Insight into the Appearance of RNA Methyltransferases". Frontiers in Genetics. 7: 99. doi:10.3389/fgene.2016.00099. PMC 4893491. PMID 27375676.
- ^ Kessler, Christoph; Manta, Vicentiu (1990-01-01). "Specificity of restriction endonucleases and DNA modification methyltransferases — a review (edition 3)". Gene. 92 (1): 1–240. doi:10.1016/0378-1119(90)90486-B. ISSN 0378-1119. PMID 2172084.
- ^ Narva, Kenneth E.; Van Etten, James L.; Slatko, Barton E.; Benner, Jack S. (1988-12-25). "The amino acid sequence of the eukaryotic DNA [N6-adenine]methyltransferase M·CviBIII, has regions of similarity with the prokaryotic isoschizomer M · TaqI and other DNA [N6-adenine] methyltransferases". Gene. 74 (1): 253–259. doi:10.1016/0378-1119(88)90298-3. ISSN 0378-1119. PMID 3248728.
- ^ Posfai, Janos; Bhagwat, Ashok S.; Roberts, Richard J. (1988-12-25). "Sequence motifs specific for cytosine methyltransferases". Gene. 74 (1): 261–265. doi:10.1016/0378-1119(88)90299-5. ISSN 0378-1119. PMID 3248729.
- ^ Liscombe, David K.; Louie, Gordon V.; Noel, Joseph P. (2012). "Architectures, mechanisms and molecular evolution of natural product methyltransferases". Natural Product Reports. 29 (10): 1238–50. doi:10.1039/c2np20029e. PMID 22850796.
- ^ Ashihara, Hiroshi; Yokota, Takao; Crozier, Alan (2013). Biosynthesis and catabolism of purine alkaloids. Advances in Botanical Research. Vol. 68. pp. 111–138. doi:10.1016/B978-0-12-408061-4.00004-3. ISBN 9780124080614.
- ^ "PNMT phenylethanolamine N-methyltransferase". NCBI Genetic Testing Registry. Retrieved 18 February 2014.
- ^ "HNMT histamine N-methyltransferase". NCBI Genetic Testing Registry. Retrieved 18 February 2014.
- ^ "COMT catechol-O-methyltransferase". NCBI Genetic Testing Registry. Retrieved 18 February 2014.
- ^ Ragsdale, S.W. "테트라히드로폴산염과 B12" 비타민과 호르몬을 포함한 메틸기 전달의 촉매 작용, 2008.
- ^ a b Bauerle, Matthew R.; Schwalm, Erica L.; Booker, Squire J. (2015-02-13). "Mechanistic Diversity of Radical S-Adenosylmethionine (SAM)-dependent Methylation". The Journal of Biological Chemistry. 290 (7): 3995–4002. doi:10.1074/jbc.R114.607044. ISSN 0021-9258. PMC 4326810. PMID 25477520.
- ^ Sofia, H. J.; Chen, G.; Hetzler, B. G.; Reyes-Spindola, J. F.; Miller, N. E. (2001-03-01). "Radical SAM, a novel protein superfamily linking unresolved steps in familiar biosynthetic pathways with radical mechanisms: functional characterization using new analysis and information visualization methods". Nucleic Acids Research. 29 (5): 1097–1106. doi:10.1093/nar/29.5.1097. ISSN 1362-4962. PMC 29726. PMID 11222759.
- ^ Morales G, Picazo JJ, Baos E, Candel FJ, Arribi A, Peláez B, Andrade R, de la Torre MA, Fereres J, Sánchez-García M (March 2010). "Resistance to linezolid is mediated by the cfr gene in the first report of an outbreak of linezolid-resistant Staphylococcus aureus". Clin. Infect. Dis. 50 (6): 821–5. doi:10.1086/650574. PMID 20144045.
- ^ Singh, S; Zhang, J; Huber, TD; Sunkara, M; Hurley, K; Goff, RD; Wang, G; Zhang, W; Liu, C; Rohr, J; Van Lanen, SG; Morris, AJ; Thorson, JS (7 April 2014). "Facile chemoenzymatic strategies for the synthesis and utilization of S-adenosyl-(L)-methionine analogues". Angewandte Chemie International Edition in English. 53 (15): 3965–9. doi:10.1002/anie.201308272. PMC 4076696. PMID 24616228.
- ^ Jones, Peter A. (1996-06-01). "DNA Methylation Errors and Cancer". Cancer Research. 56 (11): 2463–2467. ISSN 0008-5472. PMID 8653676.
- ^ D, Hanahan; Ra, Weinberg (2011-03-04). "Hallmarks of Cancer: The Next Generation". Cell. 144 (5): 646–74. doi:10.1016/j.cell.2011.02.013. PMID 21376230.