사카로미세스 게놈 데이터베이스
Saccharomyces Genome Database| 개발자 | J Michael Cherry, Gail Binkley, Stacia Engel, Rob Nash, Stuart Miyasato, Edith Wong, Shuai Weng |
|---|---|
| 운영 체제 | Unix, Mac, MS-Windows |
| 유형 | 생물정보학 도구, 모델 생물 데이터베이스 |
| 라이선스 | 공짜 |
| 웹 사이트 | http://www.yeastgenome.org |
사카로미세스 게놈 데이터베이스(SGD)는 일반적으로 빵집 또는 싹트기 [1]효모로 알려진 사카로미세스 세레비시아이 효모의 분자 생물학과 유전학에 대한 과학적 데이터베이스입니다.
사카로미세스 게놈 데이터베이스
SGD는 완전한 Saccharomyes cerevisiae 게놈 DNA 배열, 유전자와 그 생산물, 돌연변이의 표현형, 그리고 이러한 데이터를 뒷받침하는 문헌에 대한 인터넷 접속을 제공한다.피어 리뷰 문헌 리포트에서는 효모 유전자의 기능 및 상호작용에 관한 실험 결과를 고품질 수동 큐레이션으로 추출하여 잘 개발된 데이터베이스 내에 통합한다.데이터는 고품질의 높은 처리량과 결합되어 강력한 쿼리 엔진과 풍부한 게놈 브라우저인 Locus Summary 페이지에 게시됩니다.정보 수집의 복잡성에 기초하여, 정보를 통합하고 새로운 생물학적 [2]세부사항을 생산적으로 발견하기 위해 여러 생물 정보 도구를 사용한다.발아 효모의 기능 설명에 대한 골드 스탠다드는 SGD 자원에 의해 제공됩니다.SGD 자원은 또한 고등 유기체의 관련 유전자와 경로를 조사할 수 있는 플랫폼을 제공합니다.S. cerevisae genomic 배열이 공개된 후 SGD에 의해 제공되는 정보의 양과 특징의 수가 크게 증가했다.SGD는 기본 정보뿐만 아니라 유전자 특징과 유전자 간의 관계에 대한 자세한 정보를 제공하는 배열 유사성 검색과 같은 도구도 제공함으로써 연구자들에게 도움을 준다.SGD는 물리, 유전자 및 시퀀스 특징 맵을 나타내는 다양한 사용자 친화적이고 동적으로 생성된 그래픽 디스플레이를 사용하여 정보를 제공합니다.SGD의 모든 데이터는 [2]전 세계 연구자 및 교육자가 최적의 사용 편의성을 위해 설계된 웹 페이지를 통해 자유롭게 액세스할 수 있습니다.
정보 수집
바이오쿨레이터는 출판된 문헌 또는 데이터 세트의 검토를 포함하며, 주요 결과의 식별과 추상화를 이끈다.그 결과는 데이터베이스에 통합되고 적절한 유전자 또는 염색체 영역과 연관되기 위해 통제된 어휘를 사용한다.더 많은 데이터가 기록됨에 따라, 생물 의학 연구에 있어 생물 배치는 더욱 중요해지고 있다.
SGD는 발아 효모 S.cerevisiae에 대한 참조 게놈 배열을 유지합니다.SGD는 S. cerevisiae S288C 균주 배경의 게놈 배열의 원천이며, 유전자 카탈로그와 게놈의 염색체 특징을 포함한다.
SGD의 중요한 기능 중 하나는 효모 문헌의 생물화이다.SGD 바이오코쿠레이터는 S. cerevisiae와 관련된 모든 과학 문헌을 검색하고, 논문을 읽고,[2] 데이터베이스의 다양한 정의된 분야에서 주요 발견을 캡처합니다.
SGD의 바이오쿨레이터는 1차 문헌에서 기능을 식별하고 유전자 온톨로지의 [3]구조화된 지식 표현을 사용하여 용어와 연결함으로써 각 유전자에 주석을 다는 것을 목표로 한다.또한 높은 처리량 실험에서 식별된 함수와 계산적으로 예측된 함수 주석이 GO 주석 [4]프로젝트에서 포함됩니다.
생화학 경로는 SGD에 의해 수동으로 큐레이션되며 경로 도구 브라우저 버전 15.0(13)을 사용하여 제공됩니다.이용 가능한 모든 경로 도구 데이터 세트 중 가장 고도로 큐레이션된 데이터 세트 중 하나인 S. cerevisiae에 대한 SGD 생화학 경로 데이터 세트는 발아 효모의 황금 표준이다. SGD는 이러한 데이터를 업데이트하고 강화하기 위한 지속적인 노력을 지원한다.경로 도구 인터페이스는 분자 구조, 심전도 번호 및 전체 참조 목록과 함께 각 경로에 대한 완전한 설명을 제공합니다.업데이트된 경로 탐색기는 SGD에서 사용할 수 있는 다른 도구를 사용하여 추가 분석을 위해 경로에서 발견된 유전자 목록을 다운로드하는 등 몇 가지 향상된 기능을 제공한다.경로 브라우저는 Locus Summary 페이지의 'Pathways' 섹션을 통해 하이퍼링크됩니다.[ Path ]화면은, [2]http://pathway.yeastgenome.org 에서 입수할 수 있습니다.
명명법
SGD는 S. cerevisiae 게놈 명명법을 계속 유지합니다.그 일은 지역사회에서 정의된 명명기준을 촉진하고 합의된 가이드라인이 새로운 유전자를 명명하거나 이전에 식별된 유전자에 새로운 이름을 할당하는데 준수되도록 하는 것이다.커뮤니티 가이드라인은 유전자의 최초 공개명이 표준명이 된다고 명시하고 있다.단, 발표 전에 SGD에 유전자명을 등록하여 표시할 수 있다.의견 불일치나 명명 충돌이 있을 경우 커뮤니티 내 관련 연구자와 소통하고 가능하면 합의를 이끌어냅니다.문제의 유전자를 연구하는 사람들의 대다수는 그것이 SGD에 구현되기 전에 명명법 변경에 동의해야 한다.SGD는 유전적 이름을 유지하는 것 외에도 ORF, ARS 요소, tRNA 및 기타 염색체 특징의 이름도 합의된 형식에 부합하도록 보장한다.지난 2년 동안 154개의 새로운 유전자 이름이 할당되었고 21개의 지역 사회가 시작한 이름 변경이 [2]처리되었습니다.
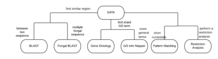
분석 방법
SGD에서는 몇 가지 다른 분석 도구가 제공됩니다.
BLAST, Basic Local Alignment Search Tool, 이 프로그램은 생물학적 시퀀스 간에 유사한 영역을 찾도록 설계되었습니다.SGD를 통해 사용자는 S. cerevisiae 시퀀스 데이터 세트의 BLAST 검색을 실행할 수 있다.
곰팡이 BLAST를 통해 여러 곰팡이 염기서열 검색 가능
GO(Gene Ontology) Term Finder는 유의한 공유 GO 용어 또는 그 부모를 검색하여 사용자가 유전자의 공통점을 발견하는 데 도움이 되는 유전자를 기술하는 데 사용됩니다.
GO 슬림 매퍼는 유전자 그룹의 주석을 보다 일반적인 용어에 매핑하거나 광범위한 범주로 분류합니다.
Pattern Matching은 사용자가 20개 미만의 짧은 뉴클레오티드 또는 펩타이드 배열을 검색할 수 있는 리소스입니다.
Restriction Analysis를 사용하면 시퀀스 이름 또는 임의의 DNA[5] 시퀀스를 입력하여 제한 분석을 수행할 수 있습니다.
레퍼런스
- ^ Cherry JM; Ball C; Weng S; Juvik G; Schmidt R; Adler C; Dunn B; Dwight S; Riles L; Mortimer RK; Botstein D (May 1997). "Genetic and physical maps of Saccharomyces cerevisiae". Nature. 387 (6632 Suppl): 67–73. doi:10.1038/387s067. PMC 3057085. PMID 9169866.
- ^ a b c d e Cherry, Michael; Hong, Eurie; amundsen, Craig; balakrishnan, rama; binkley, gail; chan, esther; christie, karen; costanzo, maria; dwight, selina; engel, stacia; fisk, dianna; hirschman, jodi; hitz, benjamin; karra, kalpana; krieger, cynthia; miyasato, stuart; nash, rob; park, julie; skrzypek, marek; simison, matt; weng, shuai; wong, edith (2011). "Saccharomyces Genome Database: the genomics resource of budding yeast". Nucleic Acids Research. 40 (2012): D700–D705. doi:10.1093/nar/gkr1029. PMC 3245034. PMID 22110037.
- ^ Dwight SS, Harris MA, Dolinski K, et al. (January 2002). "Saccharomyces Genome Database (SGD) provides secondary gene annotation using the Gene Ontology (GO)". Nucleic Acids Res. 30 (1): 69–72. doi:10.1093/nar/30.1.69. PMC 99086. PMID 11752257.
- ^ Hong EL, Balakrishnan R, Dong Q, et al. (January 2008). "Gene Ontology annotations at SGD: new data sources and annotation methods". Nucleic Acids Res. 36 (Database issue): D577–81. doi:10.1093/nar/gkm909. PMC 2238894. PMID 17982175.
- ^ "Saccharomyces Genome Database". Saccharomyces Genome Database. Stanford University. Retrieved 26 April 2018.
외부 링크
 사카로미세스 게놈 데이터베이스 ID(P3406) (용도 참조)
사카로미세스 게놈 데이터베이스 ID(P3406) (용도 참조)