게놈
Genome| 시리즈의 일부 |
| 유전학 |
|---|
 |

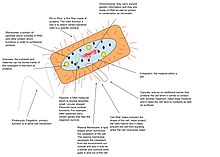
분자생물학과 유전학 분야에서 게놈은 모두 [1]유기체의 유전 정보이다.그것은 DNA(또는 RNA 바이러스의 RNA)의 뉴클레오티드 배열로 구성됩니다.핵 게놈은 단백질 코드화 유전자와 비코드화 유전자, 게놈의 다른 기능 영역(비코드화 DNA 참조), 그리고 존재하는 [2]경우 정크 DNA를 포함합니다.[3] 조류와 식물은 엽록체 게놈을 가진 엽록체를 포함하고 있으며 거의 모든 진핵생물은 미토콘드리아와 미토콘드리아 [2]게놈을 가지고 있다.
게놈 연구는 유전체학이라고 불린다.많은 유기체의 게놈 배열이 확인되었고 다양한 부위에 주석이 달렸습니다.국제 인간 게놈 프로젝트는 2004년에 호모 사피엔스의 게놈 배열을 보고했지만, 초기 "완료된" 배열은 대부분 반복 배열로 구성된 게놈의 8%가 누락되었다.
최초의 인간 게놈 프로젝트 연구에서 완전히 밝혀지지 않은 인간의 DNA에서 발견되는 많은 반복적인 배열의 염기서열을 처리할 수 있는 기술의 진보와 함께,[4] 과학자들은 2022년 3월에 최초의 인간 게놈 배열을 보고했습니다.
용어의 기원
게놈이라는 용어는 1920년 독일 함부르크 대학의 식물학 교수 Hans Winkler에 [5]의해 만들어졌다.옥스포드 사전은 이 이름이 유전자와 [6]염색체라는 단어의 혼합이라고 제안합니다.그러나 자세한 내용은 omics를 참조하십시오.바이오옴, 뿌리옴 등 관련 단어 몇 개가 이미 존재하여 게놈이 체계적으로 [7]들어맞는 어휘를 형성하고 있다.
게놈의 정의
"게놈"의 정확한 정의를 생각해 내는 것은 매우 어렵습니다.그것은 보통 유기체의 유전 정보를 운반하는 DNA (또는 때때로 RNA) 분자를 가리키지만, 때때로 정의에 포함할 분자를 결정하는 것은 어렵다; 예를 들어, 박테리아는 보통 필수적인 유전 물질을 모두 포함하지만 또한 sm을 포함하고 있는 하나 또는 두 개의 큰 DNA 분자 (염색체)를 가지고 있다.중요한 유전 정보를 가지고 있는 염색체 외 플라스미드 분자들.과학 문헌에서 흔히 사용되는 '게놈'의 정의는 [8]보통 박테리아에 있는 큰 염색체 DNA 분자로 제한된다.
진핵생물 게놈은 거의 모든 진핵생물 종이 핵염색체와 미토콘드리아에 여분의 DNA 분자를 포함하고 있기 때문에 더욱 정의하기 어렵다.게다가, 조류와 식물들은 엽록체 DNA를 가지고 있다.대부분의 교과서는 핵 게놈과 소기관(미토콘드리아와 엽록체) 게놈을 구별하기 때문에, 예를 들어 인간 게놈에 대해 언급할 때,[2][9] 그들은 핵에 있는 유전 물질만을 언급하고 있다.이것은 과학 문헌에서 '게놈'의 가장 일반적인 사용법이다.
대부분의 진핵생물은 이배체인데, 이것은 핵에 각 염색체의 두 개의 복사본이 있다는 것을 의미하지만, '게놈'은 각 염색체의 복사본 한 개만을 가리킨다.몇몇 진핵생물들은 포유류의 X와 Y 염색체와 같은 독특한 성염색체를 가지고 있기 때문에 게놈의 기술적 정의는 성염색체의 양쪽 모두를 포함해야 한다.예를 들어, 인간의 표준 기준 게놈을 언급할 때, 그것은 22개의 자동 염색체 각각에 하나의 X 염색체와 하나의 Y [10]염색체로 구성되어 있다.
순서 지정 및 매핑
게놈 배열은 개인이나 종의 모든 염색체를 구성하는 뉴클레오티드의 완전한 목록입니다.한 종 내에서, 대부분의 뉴클레오티드는 개인들 사이에서 동일하지만, 여러 개인들의 염기서열은 유전적 다양성을 이해하기 위해 필요하다.
1976년, 겐트 대학(벨기에)의 월터 피어스는 바이러스 RNA-게놈(박테리오파지 MS2)의 완전한 뉴클레오티드 서열을 최초로 확립했다.이듬해 프레드 생어는 첫 번째 DNA 유전자 염기서열을 완성했다.5386 염기쌍의 [11]파지 δ-X174.최초로 배열된 박테리아 게놈은 1995년 게놈 연구소의 팀에 의해 완성된 헤모필러스 인플루엔자이다.몇 달 후, 1980년대 중반에 시작된 유럽 주도의 노력의 결과로서 효모 사카로미세스 세레비시아의 16개 염색체의 염기서열이 발표되면서 최초의 진핵생물 게놈이 완성되었다.고고학자인 메타노코커스 잔나스키이의 첫 번째 게놈 배열은 1996년 게놈 연구소에 의해 다시 완성되었다.
새로운 기술의 개발로 게놈 배열이 매우 저렴하고 쉬워졌으며 완전한 게놈 배열의 수는 빠르게 증가하고 있다.미국 국립보건원은 게놈 정보의 [12]몇 가지 포괄적인 데이터베이스 중 하나를 관리하고 있습니다.완성된 수천 개의 게놈 배열 프로젝트 중에는 쌀, 쥐, 아라비도시스 탈리아나 식물, 복어, 그리고 박테리아 대장균이 포함된다.2013년 12월, 과학자들은 최초로 멸종된 인류 종인 네안데르탈인의 전체 게놈의 염기서열을 분석했습니다.이 게놈은 시베리아 [13][14]동굴에서 발견된 13만년 된 네안데르탈인의 발가락 뼈에서 추출됐다.
Manteia Predictive Medicine이 개척한 대규모 병렬 염기서열 분석과 같은 새로운 염기서열 분석 기술은 또한 개인 게놈 염기서열 분석의 가능성을 열어주었다.그 목표를 향한 중요한 단계는 2007년에 제임스 D의 게놈 전체를 완성한 것이다. 왓슨,[15] DNA 구조의 공동 발견자 중 한 명.
게놈 배열은 게놈의 모든 DNA 염기서열을 나열하는 반면, 게놈 지도는 랜드마크를 식별합니다.게놈 지도는 게놈 배열보다 덜 상세하고 게놈 주변을 탐색하는 데 도움이 된다.인간 게놈 프로젝트는 인간 게놈을 지도화하고 배열하기 위해 조직되었다.이 프로젝트의 기본 단계는 [16][17]파리의 제노스코프에서 Jean Weissenbach와 그의 팀이 만든 상세한 게놈 지도를 공개하는 것이었다.
참조 게놈 배열과 지도는 계속해서 업데이트되어 오류를 제거하고 높은 대립 유전자의 [18]복잡성 영역을 명확히 합니다.게놈 지도 제작 비용이 감소함에 따라 DNA와 같은 크라우드소싱된 과학적 노력에 자신의 게놈을 제출할 수 있을 정도로, 족보 사이트들이 [19]그것을 서비스로 제공할 수 있게 되었다.뉴욕 게놈 [20]센터의 LAND는 규모의 경제와 시민 [21]과학의 두 가지 사례입니다.
바이러스 게놈
바이러스 게놈은 RNA 또는 DNA로 구성될 수 있다.RNA 바이러스의 게놈은 단일 가닥 RNA 또는 이중 가닥 RNA일 수 있으며 하나 이상의 개별 RNA 분자(세그먼트: 모노파티트 또는 멀티파티트 게놈)를 포함할 수 있습니다.DNA 바이러스는 단가닥 또는 이중가닥의 게놈을 가질 수 있다.대부분의 DNA 바이러스 게놈은 단일 선형 DNA 분자로 구성되어 있지만, 일부는 원형 DNA [22]분자로 구성되어 있다.
원핵생물 게놈
원핵생물과 진핵생물은 DNA 게놈을 가지고 있다.고세균과 대부분의 박테리아는 단일 원형 [23]염색체를 가지고 있지만, 일부 박테리아 종은 선형 또는 다중 [24][25]염색체를 가지고 있다.DNA가 세균세포가 분열하는 속도보다 빨리 복제되면 단일 세포에 여러 개의 염색체 복제가 존재할 수 있고, DNA가 복제되는 속도보다 빨리 분열되면 분열이 일어나기 전에 염색체의 다중 복제가 시작돼 딸세포가 완전한 게놈을 물려받을 수 있고, 이미 부분적으로 유전체를 물려받을 수 있다.복제된 염색체.대부분의 원핵생물들은 그들의 [26]게놈에 거의 반복적인 DNA를 가지고 있지 않다.그러나 일부 공생 박테리아(예: Serratia Symbiotica)는 게놈을 감소시키고 의사유전자의 높은 비율을 가지고 있다: DNA의 40%만이 [27][28]단백질을 암호화한다.
어떤 박테리아들은 보조 유전 물질을 가지고 있고, 또한 그들의 게놈의 일부이기도 하며, 이것은 플라스미드로 운반된다.이를 위해 게놈이라는 단어를 염색체의 동의어로 사용해서는 안 된다.
진핵생물 게놈
진핵생물 게놈은 하나 이상의 선형 DNA 염색체로 구성되어 있다.염색체 수는 각각 한 쌍씩만 있는 잭 점퍼 개미와 무성 [29]선충에서부터 720쌍을 [30]가진 양치류까지 다양하다.진핵생물 게놈이 가진 DNA의 양은 다른 게놈에 비해 놀랍다.진핵생물의 게놈 크기가 무려 64,000배 이상 [31]변하기 때문에 DNA 단백질 코드와 비코드 유전자에 필요한 양보다 더 많은 양이다.그러나 이 특별한 특성은 반복 DNA와 Transposable Elements(TE; 트랜스포저블 요소)의 존재에 의해 발생합니다.
전형적인 인간 세포는 부모로부터 물려받은 22개의 자동 염색체 각각과 2개의 성 염색체를 가지고 있어 이중배체이다.난자, 정자, 포자, 꽃가루와 같은 배우자는 반수체인데, 이것은 그들이 각 염색체의 한 부만을 가지고 있다는 것을 의미한다.핵의 염색체 외에도 엽록체나 미토콘드리아와 같은 세포들은 그들만의 DNA를 가지고 있다.미토콘드리아는 종종 "미토콘드리아 게놈"이라고 불리는 그들만의 게놈을 가지고 있다고 한다.엽록체 내에서 발견되는 DNA는 "플라스톰"으로 언급될 수 있다.그들이 유래한 박테리아처럼, 미토콘드리아와 엽록체는 원형 염색체를 가지고 있다.
단백질 코딩 유전자의 엑손 인트론 구조가 존재하지만 다소 예외적인 원핵생물과는 달리, 진핵생물들은 일반적으로 그들의 유전자에 이러한 특징을 가지고 있고 그들의 게놈은 다양한 양의 반복적인 DNA를 가지고 있다.포유류와 식물에서, 게놈의 대부분은 반복적인 [32]DNA로 구성되어 있다.진핵 게놈의 유전자는 [33]FINDER를 사용하여 주석을 달 수 있습니다.
코드화 시퀀스
단백질을 만드는 명령을 전달하는 DNA 배열을 코드 배열이라고 합니다.코드 배열이 차지하는 게놈의 비율은 매우 다양하다.게놈이 클수록 반드시 더 많은 유전자를 포함할 필요는 없으며 복잡한 진핵생물에서 [32]게놈 크기가 증가함에 따라 비반복적 DNA의 비율은 감소한다.
비코딩 시퀀스
비부호화 배열에는 인트론, 비부호화 RNA의 배열, 조절 영역 및 반복 DNA가 포함됩니다.비코드 배열은 인간 게놈의 98%를 차지한다.게놈에는 두 가지 범주가 있다: 탠덤 반복과 삽입 반복.[34]
탠덤 반복
머리부터 꼬리까지 반복되는 짧은 비부호화 시퀀스를 탠덤 반복이라고 합니다.2-5개의 베이스페어 반복으로 구성된 마이크로위성은 30-35bp입니다.탠덤 반복은 인간 게놈의 약 4%, 초파리 [35]게놈의 9%를 차지한다.탠덤 반복은 기능할 수 있습니다.예를 들어, 텔로미어는 포유동물에서 TTAGG로 구성되어 있으며 염색체의 말단을 보호하는 데 중요한 역할을 한다.
다른 경우에는 엑손 또는 인트론에서 탠덤 반복 횟수 확대로 인해 [36]질병이 발생할 수 있습니다.예를 들어, 인간 유전자 헌팅틴(Htt)은 전형적으로 뉴클레오티드 CAG(폴리글루타민로 인코딩)의 6~29회 연속 반복을 포함한다.36회 이상 반복되면 신경변성 질환인 헌팅턴병이 발생한다.다양한 유전자의 유사한 연속 반복 팽창으로 인해 20개의 인간 질환이 발생하는 것으로 알려져 있다.확장된 폴리글라타민 작용이 뉴런의 죽음을 초래하는 메커니즘은 완전히 이해되지 않았다.한 가지 가능성은 단백질이 적절하게 접히지 않고 분해를 피하며, 대신 중요한 전사 인자를 격리하는 집합체에 축적되어 유전자 [36]발현을 변화시키는 것이다.
탠덤 반복은 일반적으로 복제 중 미끄러짐, 불균등한 교차 및 유전자 [37]변환에 의해 발생한다.
트랜스포저블 요소
Transposable Elements(TE; 트랜스포저블 요소)는 [35][26][38]게놈에서 위치를 바꿀 수 있는 정의된 구조를 가진 DNA의 배열입니다.TE는 복사 및 붙여넣기에 의해 복제되는 메커니즘 또는 게놈에서 제거되어 새로운 위치에 삽입될 수 있는 메커니즘으로 분류된다.인간 게놈에는 인간 DNA의 45% 이상을 구성하는 세 가지 중요한 종류의 TE가 있습니다; 이 세분류는 롱 인터스페드 핵 요소(LINEs), 인터스페드 핵 요소(SINEs), 그리고 내인성 레트로 바이러스입니다.이 요소들은 숙주 [31]유기체의 유전자 제어를 바꿀 수 있는 큰 잠재력을 가지고 있다.
TE의 이동은 진핵생물에서 게놈 진화의 원동력이다. 왜냐하면 TE의 삽입은 유전자 기능을 방해할 수 있고, TE들 사이의 상동 재조합은 복제를 만들 수 있으며, TE는 엑손과 조절 시퀀스를 새로운 [39]위치로 섞을 수 있기 때문이다.
레트로트랜스포존
역트랜스포존은[40] 주로 진핵생물에서 발견되지만 원핵생물에서는 발견되지 않으며 역트랜스포존은 많은 진핵생물들의 게놈에서 많은 부분을 형성한다.역트랜스포존은 RNA 중간체를 통해 전이되는 전이성 원소이다.역트랜스포존은[41] DNA로 구성되지만, 전이를 위해 RNA로 전사되고, 그 후 역트랜스포존이라고 불리는 특정 효소의 도움으로 RNA 전사가 다시 DNA 형성에 복사됩니다.유전자에 역전사 효소를 가지고 있는 역전사 효소는 스스로 전이를 일으킬 수 있지만 역전사 효소가 없는 유전자는 다른 역전사 효소에 의해 합성된 역전사 효소를 사용해야 한다.레트로트랜스포존은 RNA로 변환될 수 있으며, RNA는 다른 부위에서 [42]게놈으로 복제됩니다.레트로트랜스포존은 Long Terminal Repeat(LTR; 긴 단자 반복)과 Non-LTR(비 LTR;[39] 긴 단자 반복)로 나눌 수 있습니다.
장기말단반복(LTRs)은 고대 레트로바이러스 감염에서 유래한 것이므로 그들은 개그(바이러스의 구조단백질), 폴(역전사효소 및 인테그라아제), 프로(프로테아제), 그리고 경우에 따라서는 엔브(env) [38]유전자를 포함한 레트로바이러스 단백질과 관련된 단백질을 부호화한다.이 유전자들은 5'와 3' 양쪽 끝에 긴 반복이 있다.LTR은 대부분의 식물 게놈에서 가장 큰 부분으로 구성되어 있으며 게놈 [43]크기의 큰 변화를 설명할 수 있다고 보고되었다.
비장단말기 반복측정(Non-LTR)은 롱 인터스페드 핵요소(LINE), 쇼트 인터스페드 핵요소(SINE) 및 페넬로피 유사요소(PLE)로 분류된다.Dictyostelium discoideum에는 비LTR에 속하는 또 다른 DIRS 유사 요소가 있습니다.비 LTR은 진핵생물 [44]게놈에 널리 퍼져있다.
롱 인터스페드 요소(LINEs)는 역전사효소 및 엔도핵산가수분해효소에 대한 유전자를 인코딩하여 자율적인 트랜스포저블 요소로 만든다.인간 게놈은 약 50만 개의 LINE을 가지고 있으며 게놈의 [45]약 17%를 차지하고 있습니다.
짧은 산란 요소(SINE)는 보통 500개의 염기쌍보다 작고 비자율적이므로 전이를 [46]위해 LINE에 의해 인코딩된 단백질에 의존합니다.Alu 요소는 영장류에서 발견되는 가장 일반적인 SINE입니다.그것은 약 350개의 염기쌍이며 약 150,000개의 [39]복사본으로 인간 게놈의 약 11%를 차지한다.
DNA트랜스포존
DNA 트랜스포존은 역말단 반복 사이에 트랜스포사아제 효소를 부호화한다.발현될 때 트랜스포자아제는 트랜스포자 측면에 있는 말단 반전 반복을 인식하고 새로운 부위에 그 [35]절제 및 재삽입을 촉매한다.이 컷 앤 페이스트 메커니즘은 보통 원래 위치(100KB [39]이내) 근처에 트랜스포존을 재삽입합니다.DNA 트랜스포존은 박테리아에서 발견되며 인간 게놈의 3%와 회충 C. 엘레건스의 [39]게놈의 12%를 차지한다.
게놈 크기
게놈 크기는 반수체 게놈의 한 복사본에 있는 DNA 염기쌍의 총 수입니다.게놈 크기는 종에 따라 매우 다양하다.무척추동물은 작은 게놈을 가지고 있는데, 이것은 또한 소수의 전이성 요소들과 관련이 있다.어류와 양서류는 중간 크기의 게놈을 가지고 있고 새들은 상대적으로 게놈이 작지만 새들이 비행하는 과정에서 게놈의 상당 부분을 잃었다는 주장이 제기돼 왔다.이 손실 전에, DNA 메틸화는 [31]게놈의 적절한 확장을 가능하게 한다.
인간의 경우, 핵 게놈은 약 31억 [47]개의 DNA 뉴클레오티드로 구성되어 있으며, 각각 다른 염색체에 포함된 24개의 선형 분자, 가장 짧은 45,000,000개의 뉴클레오티드와 가장 긴 248,000개의 뉴클레오티드로 나뉜다.원핵생물이나 하위 [32][48]진핵생물에서 형태학적 복잡성과 게놈 크기 사이에는 명확하고 일관된 상관관계가 없다.게놈 크기는 주로 반복적인 DNA 요소의 확장과 수축의 함수이다.
게놈은 매우 복잡하기 때문에, 한 가지 연구 전략은 게놈의 유전자 수를 최소한으로 줄이고 문제의 유기체를 생존시키는 것이다.단세포 유기체에 대한 최소 게놈과 다세포 유기체에 대한 최소 게놈에 대한 실험 작업이 이루어지고 있다(발달 생물학 참조).그 작업은 생체내와 [49][50]실리코 둘 다이다.
전이성 요소로 인한 게놈 크기 차이
다세포 진핵생물 게놈에서 특히 이전에 언급되었던 게놈에는 엄청난 크기의 차이가 있다.이것의 대부분은 염색체에 [31]그들 자신의 새로운 복제를 만들어냄으로써 진화하는 전이성 원소의 다른 풍부함 때문이다.진핵생물 게놈은 종종 이러한 요소들의 수천 개의 복사본을 포함하고 있으며, 그들 대부분은 결함이 있는 돌연변이를 얻었다.
여기 유의하거나 대표적인 게놈의 표가 있습니다.배열된 게놈 목록은 #도 참조하십시오.
| 생물형 | 유기체 | 게놈 크기 (베이스 페어) | 유전자 수 | 메모 | |
|---|---|---|---|---|---|
| 바이러스 | 돼지 서코바이러스 타입 1 | 1,759 | 1.8kB | 진핵 [51]세포에서 자율적으로 복제되는 가장 작은 바이러스입니다. | |
| 바이러스 | 박테리오파지 MS2 | 3,569 | 3.5kB | 첫 번째 염기서열 RNA-게놈[52] | |
| 바이러스 | SV40 | 5,224 | 5.2kB | [53] | |
| 바이러스 | 페이지 δ-X174 | 5,386 | 5.4kB | 첫 번째 염기서열 DNA-게놈[54] | |
| 바이러스 | HIV | 9,749 | 9.7kB | [55] | |
| 바이러스 | 페이지 » | 48,502 | 48.5kB | 종종 재조합 DNA 복제를 위한 벡터로 사용됩니다. | |
| 바이러스 | 메가바이러스 | 1,259,197 | 1.3 MB | 2013년까지 가장 큰 바이러스 [59]게놈이었다. | |
| 바이러스 | 판도라바이러스살리누스 | 2,470,000 | 2.47 MB | 알려진 가장 큰 바이러스 [60]게놈입니다 | |
| 진핵생물 오가넬 | 인간 미토콘드리아 | 16,569 | 16.6 kB | [61] | |
| 세균 | 나스이아델토케팔린콜라(스트레인 NAS-ALF) | 112,091 | 112 kB | 137 | 가장 작은 비바이러스 게놈이야리프호퍼의 심비온트.[62] |
| 세균 | 카소넬라루디 | 159,662 | 160 kB | 실리스과 곤충의 내생충 | |
| 세균 | 부체네라 진딧물 | 600,000 | 600kB | 진딧물의[63] 내분비온트 | |
| 세균 | 위글스워시아 글로시니디아 | 700,000 | 700Kb | 체체파리의 내장에 있는 심비온트 | |
| 세균 – 시아노박테륨 | 프로클로로코커스 (1.7 Mb) | 1,700,000 | 1.7 MB | 1,884 | 알려진 가장 작은 시아노박테륨 게놈.지구상의 [64][65]주요 광합성자 중 하나지 |
| 세균 | 헤모필루스인플루엔자 | 1,830,000 | 1.8 MB | 살아있는 유기체의 첫 게놈 배열, 1995년 7월[66] | |
| 세균 | 대장균 | 4,600,000 | 4.6 MB | 4,288 | [67] |
| 세균 – 시아노박테륨 | 노스토크 시간형식 | 9,000,000 | 9 MB | 7,432 | 7432개의 열린[68] 판독 |
| 세균 | 솔리박터류(스트레인 Ellin 6076) | 9,970,000 | 10 MB | [69] | |
| 아메로이드 | 폴리카오스 두비움('아메바' 두비아) | 670,000,000,000 | 670 GB | 알려진 가장 큰 게놈([70]논란)[71] | |
| 식물. | 쯔베루사시 | 61,000,000 | 61 MB | 2014년에 [72]기록된 가장 작은 개화 식물 게놈입니다. | |
| 식물. | 아라비도시스탈리아나 | 135,000,000[73] | 135 MB | 27,655[74] | 2000년 [75]12월 첫 식물 게놈 배열 분석 |
| 식물. | 민달팽이속 | 480,000,000 | 480 MB | 73,013 | 첫 번째 트리 게놈 배열, 2006년 9월[76] |
| 식물. | 프리틸라리아 아시리아카 | 130,000,000,000 | 130 GB | ||
| 식물. | 파리자포니카(일본 토종, 옅은 꽃잎 | 150,000,000,000 | 150 GB | 알려진 가장 큰 식물 게놈[77] | |
| 식물 – 이끼 | 피지컬렐라 패튼스 | 480,000,000 | 480 MB | 2008년 [78]1월에 염기서열 분석된 브라이오피스의 첫 게놈. | |
| 곰팡이 – 효모 | 사카로미세스 세레비시아 | 12,100,000 | 12.1 MB | 6,294 | 최초의 진핵생물 게놈 배열, 1996[79] |
| 곰팡이 | 아스페르길루스니둘란스 | 30,000,000 | 30 MB | 9,541 | [80] |
| 네마토데 | 쯔바시카에 | 20,000,000 | 20 MB | [81] 알려진 가장 작은 동물 게놈[82] | |
| 네마토데 | 케노하브디시스엘레건스 | 100,300,000 | 100 MB | 19,000 | 최초의 다세포 동물 게놈 배열, 1998년 12월[83] |
| 곤충. | 드로소필라멜라노가스터(초파리) | 175,000,000 | 175 MB | 13,600 | 변형률에 따른 크기 변화(175~180Mb, 표준 y 와 변형률 175Mb)[84] |
| 곤충. | 꿀벌 | 236,000,000 | 236 MB | 10,157 | [85] |
| 곤충. | 봄빅스모리(실크나방) | 432,000,000 | 432 MB | 14,623 | 14[86],623개의 예측 유전자 |
| 곤충. | 솔레놉시스 인빅타(불개미) | 480,000,000 | 480 MB | 16,569 | [87] |
| 포유동물 | 근골근 | 2,700,000,000 | 2.7 GB | 20,210 | [88] |
| 포유동물 | 판파니스쿠스 | 3,286,640,000 | 3.3 GB | 20,000 | 보노보 - 추정 게놈 크기 32억9천만[89] bp |
| 포유동물 | 호모 사피엔스 | 3,117,000,000 | 3.1 GB | 20,000 | 2022년[47] 호모 사피엔스의 게놈 크기는 3.12Gbp로 추정된다. 인간[90] 게놈의 초기 배열 및 분석 |
| 새 | 담쟁이덩굴 | 1,043,000,000 | 1.0 GB | 20,000 | [91] |
| 물고기. | 복어의 일종인 테트라오돈 니그로비리디스 | 385,000,000 | 390 MB | 최소 척추동물 게놈은 340Mb[92][93] –[94] 385Mb로 추정됩니다. | |
| 물고기. | 폐어(마름모양 | 130,000,000,000 | 130 GB | 알려진 가장 큰 척추동물 게놈 | |
게놈의 변화
유기체의 모든 세포는 단일 세포에서 유래하기 때문에 동일한 게놈을 가지고 있을 것으로 예상되지만, 어떤 경우에는 차이가 생긴다.세포 분열 중에 DNA를 복제하는 과정과 환경 돌연변이에 노출되는 것 모두 체세포에 돌연변이를 일으킬 수 있다.어떤 경우에, 그러한 돌연변이는 세포들이 더 빨리 분열하고 주변 [95]조직을 침범하게 하기 때문에 암을 유발한다.인간 면역계의 특정 림프구에서 V(D)J 재조합은 각 세포가 고유한 항체 또는 T세포 수용체를 생성하도록 다른 게놈 배열을 생성한다.
감수분열 동안, 이배체 세포는 두 번 분열하여 반배체 생식세포를 생성한다.이 과정 동안, 재조합은 상동 염색체로부터 유전 물질의 재변형을 초래하여 각각의 생식체는 고유한 게놈을 가집니다.
게놈 전체의 재프로그래밍
쥐의 원시 생식세포에서 게놈 전체의 재프로그래밍은 전능성으로 이어지는 후생유전학적 각인 소거를 포함한다.재프로그래밍은 활성 DNA 탈메틸화에 의해 촉진되며, 활성 DNA 탈메틸화는 DNA 염기 제거 복구 [96]경로를 수반한다.이 경로는 원시 생식세포에서 CpG 메틸화(5mC) 소거에 사용된다.5mC의 소실은 TET1 및 TET2의 [97]높은 수치에 의해 구동되는 5-히드록시메틸사이토신(5hmC)으로의 변환을 통해 발생한다.
게놈 진화
게놈은 유기체의 유전자의 합보다 더 크고 특정 유전자와 그 생산물의 세부 사항을 고려하지 않고 측정되고 연구될 수 있는 특성을 가지고 있다.연구자들은 핵형(염색체 수), 게놈 크기, 유전자 순서, 코돈 사용 편향, GC 함량과 같은 특성을 비교하여 오늘날 존재하는 다양한 게놈을 생산할 수 있는 메커니즘이 무엇인지를 판단한다(최근 개요는 브라운 2002; 사코니와 페솔 2003; 벤페이와 프로토파파파파스 2004; 깁슨과 뮤즈 2004; 리즈; 2004; 2004).ory 2005).
복제는 게놈 형성에 중요한 역할을 한다.복제는 짧은 연속 반복의 연장에서부터 유전자 군집의 복제, 그리고 전체 염색체 또는 전체 게놈의 복제까지 다양할 수 있습니다.그러한 복제는 아마도 유전적인 참신성을 창조하는 데 있어 기초가 될 것이다.
수평 유전자 이동은 어떻게 종종 매우 멀리 떨어져 있는 두 유기체의 게놈의 작은 부분 사이에 극단적인 유사성이 있는지를 설명하기 위해 호출된다.수평적 유전자 이식은 많은 미생물들 사이에서 흔한 것으로 보인다.또한, 진핵 세포는 엽록체와 미토콘드리아 게놈에서 핵 염색체로 유전 물질이 전달되는 것을 경험한 것으로 보인다.최근의 경험적 데이터는 바이러스와 하위 바이러스 RNA 네트워크의 중요한 역할을 시사하며 유전자의 참신성과 자연스러운 게놈 편집을 창출하는 주요 추진 역할을 나타낸다.
픽션에서
공상과학소설 작품들은 게놈 배열의 가용성에 대한 우려를 보여준다.
마이클 크라이튼의 1990년 소설 '쥬라기 공원'과 후속작은 외딴 섬에 공룡 복제 테마파크를 만든 억만장자의 이야기를 담고 있다.한 유전학자가 고대 모기의 혈액에서 공룡 DNA를 추출해 그 틈새를 현생 종의 DNA로 메워 여러 종의 공룡을 만든다.혼돈 이론가는 공룡과 함께 생태계를 설계하는 것의 안전성에 대한 전문가의 의견을 구했고, 그는 프로젝트의 결과가 예측할 수 없고 궁극적으로 통제 불능이 될 것이라고 거듭 경고한다.게놈 정보 사용의 위험성에 대한 이러한 경고들이 이 책의 주요 주제이다.
1997년 영화 '가타카'는 미래주의 사회를 배경으로 아이의 게놈이 부모의 특성을 가장 이상적으로 조합하도록 설계돼 심장병 위험과 예측수명 등의 지표가 게놈을 기반으로 개개인에게 기록된다."In-Valids"로 알려진 우생학 프로그램 밖에서 임신한 사람들은 차별을 받고 하찮은 직업으로 좌천된다.이 영화의 주인공은 유전적인 역경을 극복하고 우주 항해사가 되고자 하는 꿈을 이루기 위해 일하는 불효자이다.이 영화는 유전자 정보가 유전공학 [98]아이들을 감당할 수 있는 사람들과 그렇지 못한 사람들 사이의 편견과 극심한 계급 차이를 부추기는 미래에 대해 경고한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Roth, Stephanie Clare (1 July 2019). "What is genomic medicine?". Journal of the Medical Library Association. University Library System, University of Pittsburgh. 107 (3): 442–448. doi:10.5195/jmla.2019.604. ISSN 1558-9439. PMC 6579593. PMID 31258451.
- ^ a b c Graur D (2016). Molecular Genome Evolution. Sunderland, MA, USA: Sinauer Associates, Inc.
- ^ Brosius, J (2009), "The Fragmented Gene", Annals of the New York Academy of Sciences, 1178 (1): 186–93, Bibcode:2009NYASA1178..186B, doi:10.1111/j.1749-6632.2009.05004.x, PMID 19845638, S2CID 8279434
- ^ Hartley, Gabrielle. "The Human Genome Project pieced together only 92% of the DNA – now scientists have finally filled in the remaining 8%". TheConversation.org. The Conversation US, Inc. Retrieved 4 April 2022.
- ^ Winkler HL (1920). Verbreitung und Ursache der Parthenogenesis im Pflanzen- und Tierreiche. Jena: Verlag Fischer.
- ^ "definition of Genome in Oxford dictionary". Retrieved 25 March 2014.
- ^ Lederberg J, McCray AT (2001). "'Ome Sweet 'Omics – A Genealogical Treasury of Words" (PDF). The Scientist. 15 (7). Archived from the original (PDF) on 29 September 2006.
- ^ Kirchberger PC, Schmidt ML, and Ochman H (2020). "The ingenuity of bacterial genomes". Annual Review of Microbiology. 74: 815–834. doi:10.1146/annurev-micro-020518-115822. PMID 32692614. S2CID 220699395.
- ^ Brown, TA (2018). Genomes 4. New York, NY, USA: Garland Science. ISBN 9780815345084.
- ^ "Ensembl Human Assembly and gene annotation (GRCh38)". Ensembl. Retrieved 30 May 2022.
- ^ "All about genes". www.beowulf.org.uk.
- ^ "Genome Home". 8 December 2010. Retrieved 27 January 2011.
- ^ Zimmer C (18 December 2013). "Toe Fossil Provides Complete Neanderthal Genome". The New York Times. Archived from the original on 2 January 2022. Retrieved 18 December 2013.
- ^ Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, et al. (January 2014). "The complete genome sequence of a Neanderthal from the Altai Mountains". Nature. 505 (7481): 43–49. Bibcode:2014Natur.505...43P. doi:10.1038/nature12886. PMC 4031459. PMID 24352235.
- ^ Wade N (31 May 2007). "Genome of DNA Pioneer Is Deciphered". The New York Times. Retrieved 2 April 2010.
- ^ "What's a Genome?". Genomenewsnetwork.org. 15 January 2003. Retrieved 27 January 2011.
- ^ "Mapping Factsheet". 29 March 2004. Archived from the original on 19 July 2010. Retrieved 27 January 2011.
- ^ Genome Reference Consortium. "Assembling the Genome". Retrieved 23 August 2016.
- ^ Kaplan, Sarah (17 April 2016). "How do your 20,000 genes determine so many wildly different traits? They multitask". The Washington Post. Retrieved 27 August 2016.
- ^ Check Hayden, Erika (2015). "Scientists hope to attract millions to 'DNA.LAND'". Nature. doi:10.1038/nature.2015.18514. S2CID 211729308.
- ^ Zimmer, Carl (25 July 2016). "Game of Genomes, Episode 13: Answers and Questions". STAT. Retrieved 27 August 2016.
- ^ Gelderblom, Hans R. (1996). "Structure and Classification of Viruses". Medical Microbiology (4th ed.). Galveston, TX: The University of Texas Medical Branch at Galveston. ISBN 9780963117212. PMID 21413309.
- ^ Samson RY, Bell SD (2014). "Archaeal chromosome biology". Journal of Molecular Microbiology and Biotechnology. 24 (5–6): 420–27. doi:10.1159/000368854. PMC 5175462. PMID 25732343.
- ^ Chaconas G, Chen CW (2005). "Replication of Linear Bacterial Chromosomes: No Longer Going Around in Circles". The Bacterial Chromosome: 525–540. doi:10.1128/9781555817640.ch29. ISBN 9781555812324.
- ^ "Bacterial Chromosomes". Microbial Genetics. 2002.
- ^ a b Koonin EV, Wolf YI (July 2010). "Constraints and plasticity in genome and molecular-phenome evolution". Nature Reviews. Genetics. 11 (7): 487–98. doi:10.1038/nrg2810. PMC 3273317. PMID 20548290.
- ^ McCutcheon JP, Moran NA (November 2011). "Extreme genome reduction in symbiotic bacteria". Nature Reviews. Microbiology. 10 (1): 13–26. doi:10.1038/nrmicro2670. PMID 22064560. S2CID 7175976.
- ^ Land M, Hauser L, Jun SR, Nookaew I, Leuze MR, Ahn TH, Karpinets T, Lund O, Kora G, Wassenaar T, Poudel S, Ussery DW (March 2015). "Insights from 20 years of bacterial genome sequencing". Functional & Integrative Genomics. 15 (2): 141–61. doi:10.1007/s10142-015-0433-4. PMC 4361730. PMID 25722247.
- ^ "Scientists sequence asexual tiny worm whose lineage stretches back 18 million years". ScienceDaily. Retrieved 7 November 2017.
- ^ Khandelwal S (March 1990). "Chromosome evolution in the genus Ophioglossum L.". Botanical Journal of the Linnean Society. 102 (3): 205–17. doi:10.1111/j.1095-8339.1990.tb01876.x.
- ^ a b c d Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 August 2020). "DNA methylation enables transposable element-driven genome expansion". Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. doi:10.1073/pnas.1921719117. ISSN 1091-6490. PMC 7431005. PMID 32719115.
- ^ a b c Lewin B (2004). Genes VIII (8th ed.). Upper Saddle River, NJ: Pearson/Prentice Hall. ISBN 978-0-13-143981-8.
- ^ Banerjee S, Bhandary P, Woodhouse M, Sen TZ, Wise RP, Andorf CM (April 2021). "FINDER: an automated software package to annotate eukaryotic genes from RNA-Seq data and associated protein sequences". BMC Bioinformatics. 44 (9): e89. doi:10.1186/s12859-021-04120-9. PMC 8056616. PMID 33879057.
- ^ Stojanovic N, ed. (2007). Computational genomics : current methods. Wymondham: Horizon Bioscience. ISBN 978-1-904933-30-4.
- ^ a b c Padeken J, Zeller P, Gasser SM (April 2015). "Repeat DNA in genome organization and stability". Current Opinion in Genetics & Development. 31: 12–19. doi:10.1016/j.gde.2015.03.009. PMID 25917896.
- ^ a b Usdin K (July 2008). "The biological effects of simple tandem repeats: lessons from the repeat expansion diseases". Genome Research. 18 (7): 1011–19. doi:10.1101/gr.070409.107. PMC 3960014. PMID 18593815.
- ^ Li YC, Korol AB, Fahima T, Beiles A, Nevo E (December 2002). "Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review". Molecular Ecology. 11 (12): 2453–65. doi:10.1046/j.1365-294X.2002.01643.x. PMID 12453231. S2CID 23606208.
- ^ a b Wessler SR (November 2006). "Transposable elements and the evolution of eukaryotic genomes". Proceedings of the National Academy of Sciences of the United States of America. 103 (47): 17600–01. Bibcode:2006PNAS..10317600W. doi:10.1073/pnas.0607612103. PMC 1693792. PMID 17101965.
- ^ a b c d e Kazazian HH (March 2004). "Mobile elements: drivers of genome evolution". Science. 303 (5664): 1626–32. Bibcode:2004Sci...303.1626K. doi:10.1126/science.1089670. PMID 15016989. S2CID 1956932.
- ^ "Transposon genetics". Encyclopedia Britannica. Retrieved 5 December 2020.
- ^ Sanders, Mark Frederick (2019). Genetic Analysis: an integrated approach third edition. New York: Pearson, always learning, and mastering. p. 425. ISBN 9780134605173.
- ^ Deininger PL, Moran JV, Batzer MA, Kazazian HH (December 2003). "Mobile elements and mammalian genome evolution". Current Opinion in Genetics & Development. 13 (6): 651–58. doi:10.1016/j.gde.2003.10.013. PMID 14638329.
- ^ Kidwell MG, Lisch DR (March 2000). "Transposable elements and host genome evolution". Trends in Ecology & Evolution. 15 (3): 95–99. doi:10.1016/S0169-5347(99)01817-0. PMID 10675923.
- ^ Richard GF, Kerrest A, Dujon B (December 2008). "Comparative genomics and molecular dynamics of DNA repeats in eukaryotes". Microbiology and Molecular Biology Reviews. 72 (4): 686–727. doi:10.1128/MMBR.00011-08. PMC 2593564. PMID 19052325.
- ^ Cordaux R, Batzer MA (October 2009). "The impact of retrotransposons on human genome evolution". Nature Reviews. Genetics. 10 (10): 691–703. doi:10.1038/nrg2640. PMC 2884099. PMID 19763152.
- ^ Han JS, Boeke JD (August 2005). "LINE-1 retrotransposons: modulators of quantity and quality of mammalian gene expression?". BioEssays. 27 (8): 775–84. doi:10.1002/bies.20257. PMID 16015595. S2CID 26424042.
- ^ a b Nurk, Sergey; et al. (31 March 2022). "The complete sequence of a human genome" (PDF). Science. 376 (6588): 44–53. Bibcode:2022Sci...376...44N. doi:10.1126/science.abj6987. PMID 35357919. S2CID 235233625.
- ^ Gregory TR, Nicol JA, Tamm H, Kullman B, Kullman K, Leitch IJ, Murray BG, Kapraun DF, Greilhuber J, Bennett MD (January 2007). "Eukaryotic genome size databases". Nucleic Acids Research. 35 (Database issue): D332–38. doi:10.1093/nar/gkl828. PMC 1669731. PMID 17090588.
- ^ Glass JI, Assad-Garcia N, Alperovich N, Yooseph S, Lewis MR, Maruf M, Hutchison CA, Smith HO, Venter JC (January 2006). "Essential genes of a minimal bacterium". Proceedings of the National Academy of Sciences of the United States of America. 103 (2): 425–30. Bibcode:2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
- ^ Forster AC, Church GM (2006). "Towards synthesis of a minimal cell". Molecular Systems Biology. 2 (1): 45. doi:10.1038/msb4100090. PMC 1681520. PMID 16924266.
- ^ Mankertz P (2008). "Molecular Biology of Porcine Circoviruses". Animal Viruses: Molecular Biology. Caister Academic Press. ISBN 978-1-904455-22-6.
- ^ Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, Min Jou W, Molemans F, Raeymaekers A, Van den Berghe A, Volckaert G, Ysebaert M (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature. 260 (5551): 500–07. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203. S2CID 4289674.
- ^ Fiers W, Contreras R, Haegemann G, Rogiers R, Van de Voorde A, Van Heuverswyn H, Van Herreweghe J, Volckaert G, Ysebaert M (May 1978). "Complete nucleotide sequence of SV40 DNA". Nature. 273 (5658): 113–20. Bibcode:1978Natur.273..113F. doi:10.1038/273113a0. PMID 205802. S2CID 1634424.
- ^ Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, Hutchison CA, Slocombe PM, Smith M (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ^ "Virology – Human Immunodeficiency Virus And Aids, Structure: The Genome And Proteins of HIV". Pathmicro.med.sc.edu. 1 July 2010. Retrieved 27 January 2011.
- ^ Thomason L, Court DL, Bubunenko M, Costantino N, Wilson H, Datta S, Oppenheim A (April 2007). "Recombineering: genetic engineering in bacteria using homologous recombination". Current Protocols in Molecular Biology. Chapter 1: Unit 1.16. doi:10.1002/0471142727.mb0116s78. ISBN 978-0-471-14272-0. PMID 18265390. S2CID 490362.
- ^ Court DL, Oppenheim AB, Adhya SL (January 2007). "A new look at bacteriophage lambda genetic networks". Journal of Bacteriology. 189 (2): 298–304. doi:10.1128/JB.01215-06. PMC 1797383. PMID 17085553.
- ^ Sanger F, Coulson AR, Hong GF, Hill DF, Petersen GB (December 1982). "Nucleotide sequence of bacteriophage lambda DNA". Journal of Molecular Biology. 162 (4): 729–73. doi:10.1016/0022-2836(82)90546-0. PMID 6221115.
- ^ Legendre M, Arslan D, Abergel C, Claverie JM (January 2012). "Genomics of Megavirus and the elusive fourth domain of Life". Communicative & Integrative Biology. 5 (1): 102–06. doi:10.4161/cib.18624. PMC 3291303. PMID 22482024.
- ^ Philippe N, Legendre M, Doutre G, Couté Y, Poirot O, Lescot M, Arslan D, Seltzer V, Bertaux L, Bruley C, Garin J, Claverie JM, Abergel C (July 2013). "Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes" (PDF). Science. 341 (6143): 281–86. Bibcode:2013Sci...341..281P. doi:10.1126/science.1239181. PMID 23869018. S2CID 16877147.
- ^ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). "Sequence and organization of the human mitochondrial genome". Nature. 290 (5806): 457–65. Bibcode:1981Natur.290..457A. doi:10.1038/290457a0. PMID 7219534. S2CID 4355527.
- ^ Bennett GM, Moran NA (5 August 2013). "Small, smaller, smallest: the origins and evolution of ancient dual symbioses in a Phloem-feeding insect". Genome Biology and Evolution. 5 (9): 1675–88. doi:10.1093/gbe/evt118. PMC 3787670. PMID 23918810.
- ^ Shigenobu S, Watanabe H, Hattori M, Sakaki Y, Ishikawa H (September 2000). "Genome sequence of the endocellular bacterial symbiont of aphids Buchnera sp. APS". Nature. 407 (6800): 81–86. Bibcode:2000Natur.407...81S. doi:10.1038/35024074. PMID 10993077.
- ^ Rocap G, Larimer FW, Lamerdin J, Malfatti S, Chain P, Ahlgren NA, et al. (August 2003). "Genome divergence in two Prochlorococcus ecotypes reflects oceanic niche differentiation". Nature. 424 (6952): 1042–47. Bibcode:2003Natur.424.1042R. doi:10.1038/nature01947. PMID 12917642. S2CID 4344597.
- ^ Dufresne A, Salanoubat M, Partensky F, Artiguenave F, Axmann IM, Barbe V, et al. (August 2003). "Genome sequence of the cyanobacterium Prochlorococcus marinus SS120, a nearly minimal oxyphototrophic genome". Proceedings of the National Academy of Sciences of the United States of America. 100 (17): 10020–25. Bibcode:2003PNAS..10010020D. doi:10.1073/pnas.1733211100. PMC 187748. PMID 12917486.
- ^ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, Bult CJ, Tomb JF, Dougherty BA, Merrick JM (July 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science. 269 (5223): 496–512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800. S2CID 10423613.
- ^ Blattner FR, Plunkett G, Bloch CA, Perna NT, Burland V, Riley M, et al. (September 1997). "The complete genome sequence of Escherichia coli K-12". Science. 277 (5331): 1453–62. doi:10.1126/science.277.5331.1453. PMID 9278503.
- ^ Meeks JC, Elhai J, Thiel T, Potts M, Larimer F, Lamerdin J, Predki P, Atlas R (2001). "An overview of the genome of Nostoc punctiforme, a multicellular, symbiotic cyanobacterium". Photosynthesis Research. 70 (1): 85–106. doi:10.1023/A:1013840025518. PMID 16228364. S2CID 8752382.
- ^ Challacombe JF, Eichorst SA, Hauser L, Land M, Xie G, Kuske CR (15 September 2011). Steinke D (ed.). "Biological consequences of ancient gene acquisition and duplication in the large genome of Candidatus Solibacter usitatus Ellin6076". PLOS ONE. 6 (9): e24882. Bibcode:2011PLoSO...624882C. doi:10.1371/journal.pone.0024882. PMC 3174227. PMID 21949776.
- ^ Parfrey LW, Lahr DJ, Katz LA (April 2008). "The dynamic nature of eukaryotic genomes". Molecular Biology and Evolution. 25 (4): 787–94. doi:10.1093/molbev/msn032. PMC 2933061. PMID 18258610.
- ^ ScienceShot: 2010년 10월 11일 웨이백 머신에 보관된 사상 최대 게놈: "아메바 두비아와 다른 원생동물의 측정은 1960년대에 매우 큰 게놈을 가지고 있는 것으로 보고된 바 있는 대략적인 생화학적 접근방식을 사용하여 이루어졌는데, 지금은 정확한 게놈 크기 결정을 위한 신뢰할 수 없는 방법으로 여겨지고 있습니다."
- ^ Fleischmann A, Michael TP, Rivadavia F, Sousa A, Wang W, Temsch EM, Greilhuber J, Müller KF, Heubl G (December 2014). "Evolution of genome size and chromosome number in the carnivorous plant genus Genlisea (Lentibulariaceae), with a new estimate of the minimum genome size in angiosperms". Annals of Botany. 114 (8): 1651–63. doi:10.1093/aob/mcu189. PMC 4649684. PMID 25274549.
- ^ "Genome Assembly". The Arabidopsis Information Resource (TAIR).
- ^ "Details - Arabidopsis thaliana - Ensembl Genomes 40". plants.ensembl.org.
- ^ Greilhuber J, Borsch T, Müller K, Worberg A, Porembski S, Barthlott W (November 2006). "Smallest angiosperm genomes found in lentibulariaceae, with chromosomes of bacterial size". Plant Biology. 8 (6): 770–77. doi:10.1055/s-2006-924101. PMID 17203433.
- ^ Tuskan GA, Difazio S, Jansson S, Bohlmann J, Grigoriev I, Hellsten U, et al. (September 2006). "The genome of black cottonwood, Populus trichocarpa (Torr. & Gray)" (PDF). Science. 313 (5793): 1596–604. Bibcode:2006Sci...313.1596T. doi:10.1126/science.1128691. PMID 16973872. S2CID 7717980.
- ^ Pellicer J, Fay MF, Leitch IJ (15 September 2010). "The largest eukaryotic genome of them all?". Botanical Journal of the Linnean Society. 164 (1): 10–15. doi:10.1111/j.1095-8339.2010.01072.x.
- ^ Lang D, Zimmer AD, Rensing SA, Reski R (October 2008). "Exploring plant biodiversity: the Physcomitrella genome and beyond". Trends in Plant Science. 13 (10): 542–49. doi:10.1016/j.tplants.2008.07.002. PMID 18762443.
- ^ "Saccharomyces Genome Database". Yeastgenome.org. Retrieved 27 January 2011.
- ^ Galagan JE, Calvo SE, Cuomo C, Ma LJ, Wortman JR, Batzoglou S, et al. (December 2005). "Sequencing of Aspergillus nidulans and comparative analysis with A. fumigatus and A. oryzae". Nature. 438 (7071): 1105–15. Bibcode:2005Natur.438.1105G. doi:10.1038/nature04341. PMID 16372000.
- ^ Leroy S, Bouamer S, Morand S, Fargette M (2007). "Genome size of plant-parasitic nematodes". Nematology. 9 (3): 449–50. doi:10.1163/156854107781352089.
- ^ Gregory TR (2005). "Animal Genome Size Database". Gregory, T.R. (2016). Animal Genome Size Database.
- ^ The C. elegans Sequencing Consortium (December 1998). "Genome sequence of the nematode C. elegans: a platform for investigating biology". Science. 282 (5396): 2012–18. Bibcode:1998Sci...282.2012.. doi:10.1126/science.282.5396.2012. PMID 9851916. S2CID 16873716.
- ^ Ellis LL, Huang W, Quinn AM, Ahuja A, Alfrejd B, Gomez FE, Hjelmen CE, Moore KL, Mackay TF, Johnston JS, Tarone AM (July 2014). "Intrapopulation genome size variation in D. melanogaster reflects life history variation and plasticity". PLOS Genetics. 10 (7): e1004522. doi:10.1371/journal.pgen.1004522. PMC 4109859. PMID 25057905.
- ^ Honeybee Genome Sequencing Consortium (October 2006). "Insights into social insects from the genome of the honeybee Apis mellifera". Nature. 443 (7114): 931–49. Bibcode:2006Natur.443..931T. doi:10.1038/nature05260. PMC 2048586. PMID 17073008.
- ^ The International Silkworm Genome (December 2008). "The genome of a lepidopteran model insect, the silkworm Bombyx mori". Insect Biochemistry and Molecular Biology. 38 (12): 1036–45. doi:10.1016/j.ibmb.2008.11.004. PMID 19121390.
- ^ Wurm Y, Wang J, Riba-Grognuz O, Corona M, Nygaard S, Hunt BG, et al. (April 2011). "The genome of the fire ant Solenopsis invicta". Proceedings of the National Academy of Sciences of the United States of America. 108 (14): 5679–84. Bibcode:2011PNAS..108.5679W. doi:10.1073/pnas.1009690108. PMC 3078418. PMID 21282665.
- ^ Church DM, Goodstadt L, Hillier LW, Zody MC, Goldstein S, She X, et al. (May 2009). Roberts RJ (ed.). "Lineage-specific biology revealed by a finished genome assembly of the mouse". PLOS Biology. 7 (5): e1000112. doi:10.1371/journal.pbio.1000112. PMC 2680341. PMID 19468303.
- ^ "Pan paniscus (pygmy chimpanzee)". nih.gov. Retrieved 30 June 2016.
- ^ Venter JC, Adams MD, Myers EW, Li PW, Mural RJ, Sutton GG, et al. (February 2001). "The sequence of the human genome". Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ^ International Chicken Genome Sequencing Consortium (December 2004). "Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution". Nature. 432 (7018): 695–716. Bibcode:2004Natur.432..695C. doi:10.1038/nature03154. ISSN 0028-0836. PMID 15592404.
- ^ Roest Crollius H, Jaillon O, Dasilva C, Ozouf-Costaz C, Fizames C, Fischer C, Bouneau L, Billault A, Quetier F, Saurin W, Bernot A, Weissenbach J (July 2000). "Characterization and repeat analysis of the compact genome of the freshwater pufferfish Tetraodon nigroviridis". Genome Research. 10 (7): 939–49. doi:10.1101/gr.10.7.939. PMC 310905. PMID 10899143.
- ^ Jaillon O, Aury JM, Brunet F, Petit JL, Stange-Thomann N, Mauceli E, et al. (October 2004). "Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype". Nature. 431 (7011): 946–57. Bibcode:2004Natur.431..946J. doi:10.1038/nature03025. PMID 15496914.
- ^ "Tetraodon Project Information". Archived from the original on 26 September 2012. Retrieved 17 October 2012.
- ^ Martincorena I, Campbell PJ (September 2015). "Somatic mutation in cancer and normal cells". Science. 349 (6255): 1483–89. Bibcode:2015Sci...349.1483M. doi:10.1126/science.aab4082. PMID 26404825. S2CID 13945473.
- ^ Hajkova P, Jeffries SJ, Lee C, Miller N, Jackson SP, Surani MA (July 2010). "Genome-wide reprogramming in the mouse germ line entails the base excision repair pathway". Science. 329 (5987): 78–82. Bibcode:2010Sci...329...78H. doi:10.1126/science.1187945. PMC 3863715. PMID 20595612.
- ^ Hackett JA, Sengupta R, Zylicz JJ, Murakami K, Lee C, Down TA, Surani MA (January 2013). "Germline DNA demethylation dynamics and imprint erasure through 5-hydroxymethylcytosine". Science. 339 (6118): 448–52. Bibcode:2013Sci...339..448H. doi:10.1126/science.1229277. PMC 3847602. PMID 23223451.
- ^ "Gattaca (movie)". Rotten Tomatoes.
추가 정보
- Benfey P, Protopapas AD (2004). Essentials of Genomics. Prentice Hall.
- Brown TA (2002). Genomes 2. Oxford: Bios Scientific Publishers. ISBN 978-1-85996-029-5.
- Gibson G, Muse SV (2004). A Primer of Genome Science (Second ed.). Sunderland, Mass: Sinauer Assoc. ISBN 978-0-87893-234-4.
- Gregory TR (2005). The Evolution of the Genome. Elsevier. ISBN 978-0-12-301463-4.
- Reece RJ (2004). Analysis of Genes and Genomes. Chichester: John Wiley & Sons. ISBN 978-0-470-84379-6.
- Saccone C, Pesole G (2003). Handbook of Comparative Genomics. Chichester: John Wiley & Sons. ISBN 978-0-471-39128-9.
- Werner E (December 2003). "In silico multicellular systems biology and minimal genomes". Drug Discovery Today. 8 (24): 1121–27. doi:10.1016/S1359-6446(03)02918-0. PMID 14678738.
외부 링크
- UCSC Genome Browser – 80개가 넘는 유기체의 게놈과 주석을 표시합니다.
- 게놈 센터howard.edu
- DNA 분자 생성
- 일부 비교 게놈 크기
- DNA 인터랙티브:DNA 과학의 역사
- 처음부터 DNA
- 인간 게놈 프로젝트의 모든 것 - Genome.gov
- 동물 게놈 크기 데이터베이스
- 식물 게놈 크기 데이터베이스
- GOLD: Genomes OnLine 데이터베이스
- 게놈 뉴스 네트워크
- NCBI Entrez 게놈 프로젝트 데이터베이스
- NCBI 게놈 프라이머
- GeneCards: 인간 유전자의 통합 데이터베이스
- BBC 뉴스 – 최종 게놈 '챕터' 공개
- IMG(Integrated Micro Genomes System)—DOE-JGI에 의한 게놈 분석용
- GeKnome Technologies 차세대 시퀀스 데이터 분석—Illumina 및 GeKnome Technologies의 454 서비스용 차세대 시퀀스 데이터 분석.