PMS2
PMS2| PMS2 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 별칭 | PMS2, HNPCC4, PMS2CL, PMSL2, MLH4, PMS1 호몰로고 2, 수리 시스템 구성 요소 불일치, MMRCS4 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 600259 MGI: 104288 HomoloGene: 133560 GeneCard: PMS2 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) |
| ||||||||||||||||||||||||
| RefSeq(단백질) |
|
| |||||||||||||||||||||||
| 위치(UCSC) | Cr 7: 5.97 – 6.01Mb | n/a | |||||||||||||||||||||||
| PubMed 검색 | [2] | [3] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
미스매치 리페어 엔도누클리스 PMS2는 인간에게 PMS2 유전자에 의해 암호화된 효소다.[4]
함수
이 유전자는 7번 염색체의 성단에서 발견되는 PMS2 유전자 구성원 중 하나이다. 인간 PMS2 관련 유전자는 7p12, 7p13, 7q11, 7q22 대역에 위치한다. 이러한 호몰로그램 1에서 5까지 인간 PMS2에 높은 수준의 정체성을 공유한다. 이 유전자의 산물은 DNA 불일치 수리에 관여한다. 단백질은 MLH1과 이단체를 이루며, 이 복합체는 일치하지 않는 베이스에 바인딩된 MSH2와 상호작용한다. 이 유전자의 결함은 유전성 비폴리포시스 대장암, 투르코트 증후군과 연관되어 있으며, 태곳적 원시 신경세포종양의 원인이다. 대안으로 분할된 대본 변형이 관찰되었다.[6]
수리 및 내시경 검사 작업 불일치
PMS2는 불일치 수리에 관여하며, MutL 호몰로그램에서 메타 바인딩 모티브의 무결성에 따라 달라지는 잠재 내분비 활성이 있는 것으로 알려져 있다. PMS2는 내분비증으로 불연속 DNA 가닥에 흠집을 주입한다.[7]
상호작용
PMS2는 헤테로디머 MutLα를 형성하여 MLH1과 상호작용하는 것으로 나타났다.[8][9][10][11][12][13] 잔류물 492-742에 위치한 MLH1의 상호 작용 영역을 위한 MLH3, PMS1, PMS2의 경쟁이 있다.[9]
PMS2의 상호작용 영역은 헵타드 반복을 가지고 있는데, 이는 헵타의 지퍼 단백질의 특징이다.[9] MLH1은 잔류물 506-756에서 PMS2와 상호작용한다.[10]
MutS 이성애자 MutSα 및 MutSβ는 결합 불일치 시 MutLα와 연관된다. MutLα는 새로운 DNA 가닥의 불일치 제거, 분해된 DNA의 재합성, DNA의 닉의 수리 등 다른 과정과 불일치 인식 단계에 합류하는 것으로 생각된다.[13] MutLα는 ATPase 활성도가 약한 것으로 보이며, 또한 DNA의 불연속 가닥에 빈틈을 도입하는 엔도누클레이저 활동을 가지고 있다. 이렇게 하면 EXO1에 의해 일치하지 않는 DNA 가닥이 5'에서 3'로 분해될 수 있다.[13] MutLα의 활성 부위는 PMS2 서브 유닛에 위치한다. PMS1과 PMS2는 MLH1과의 상호작용을 위해 경쟁한다.[13] PMS2의 상호작용에 있는 단백질은 탠덤 친화력 정화에 의해 확인되었다.[13][14]
인간 PMS2는 매우 낮은 수준으로 표현되며, 세포 주기가 강하게 조절되지 않는다고 믿어지지 않는다.[15]
p53 및 p73 관련 상호작용
PMS2는 또한 p53 및 p73과 상호작용하는 것으로 나타났다. P53이 없는 경우, PMS2-결핍 및 PMS2-효능세포는 여전히 시스플라틴으로 치료할 때 G2/M 체크포인트에서 세포 주기를 정지시킬 수 있다.[16] p53과 PMS2가 부족한 세포는 항암제에 대한 민감도가 높아진다. PMS2는 p53-결핍 세포의 세포 생존을 위한 보호 중재자로 p53과는 독립적으로 보호 DNA 손상 반응 경로를 변조한다.[16] PMS2와 MLH1은 불일치 수리에 의존하는 방식으로 p73 매개 세포사멸에 대항하여 세포사멸로부터 세포를 보호할 수 있다.[16]
PMS2는 p73과 상호 작용하여 p73을 안정화시켜 시스플라틴에 의한 사멸을 향상시킬 수 있다. 시스플라틴은 c-Abl에 의존하는 PMS2와 p73의 상호작용을 자극한다.[12] The MutLα complex may function as an adapter to bring p73 to the site of damaged DNA and also act as an activator of p73, due to the presence of PMS2.[12] It may also be possibly for overexpressed PMS2 to stimulate apoptosis in the absence of MLH1 and in the presence of p73 and cisplatin due to the stabilizing actions of PMS2 on p73.[12] DNA 손상 시 p53은 p21/WAF 경로를 통해 세포 주기 억제를 유도하고 MLH1과 PMS2의 표현으로 수리를 시작한다.[11] MSH1/PMS2 복합체는 DNA 손상 정도를 감지하는 역할을 하며, 손상이 수리할 수 없을 경우 p73을 안정화시켜 사멸을 개시한다.[11] PMS2의 손실이 항상 MLH1의 불안정성으로 이어지는 것은 아니다. MLH3와 PMS1로 콤플렉스를 형성할 수도 있기 때문이다.[17]
임상적 유의성
돌연변이
PMS2는 불일치 수리에 관여하는 DNA 수리 단백질을 암호화하는 유전자다. PMS2 유전자는 7p22 염색체에 위치하며 15개의 exon으로 구성되어 있다. PMS2 유전자의 엑손 11은 8개의 아데노신 코딩 반복을 가지고 있다.[18]
10만 건의 인간 암 샘플에 대한 포괄적인 유전학적 프로파일링 결과 PMS2의 촉진자 지역의 돌연변이는 특히 흑색종에서 높은 종양 돌연변이 부담(TMB)과 상당히 관련이 있는 것으로 나타났다.[19] TMB는 높은 TMB가 더 좋은 치료 결과와 연관되어 있는 암 면역 요법에 환자가 반응할 수 있는지 여부를 신뢰할 수 있는 예측 변수인 것으로 나타났다.[20]
PMS2와 같은 DNA 불일치에서 이질성 생식선 돌연변이는 자가 지배적인 린치 증후군으로 이어진다. 린치 증후군을 가진 가정의 2%만이 PMS2 유전자에 돌연변이를 가지고 있다.[21] PMS2 관련 린치 증후군을 처음 제시했을 때의 환자의 연령은 크게 달라지며, 보고된 연령은 23~77세까지 다양하다.[22]
드문 경우지만 균질성 결함은 이 증후군을 일으킬 수 있다. 그러한 경우 아이는 양부모로부터 유전자 변이를 물려받으며 그 상태를 투르코트 증후군 또는 체질 MMR 결핍증(CMMR-D)이라고 한다.[23] 2011년까지 이알릭 PMS2 세균라인 돌연변이로 인한 뇌종양 환자 36명이 보고됐다.[23] 투르코 증후군의 유전은 지배적이거나 열성적일 수 있다. 투르코 증후군의 열성 유전은 PMS2의 복합 이형 돌연변이에 의해 발생한다.[24] CMMR-D와 함께 보고된 57가족 중 31가족은 생식선 PMS2 돌연변이를 가지고 있다.[25] 60개의 PMS2 동형 돌연변이 또는 복합 이형 돌연변이 매개체 중 19가구는 CMR-D의 첫 발현으로 위장암이나 아데노마를 가지고 있었다.[25] PMS2에서 돌연변이를 확인할 때 유사유전체가 존재하면 혼동을 일으켜 돌연변이가 있는 PMS2의 존재에 대한 잘못된 긍정 결론을 초래할 수 있다.[18]
결핍과 과다압박
PMS2의 과다 압착은 과음성과 DNA 손상 내성을 초래한다.[26] PMS2의 결핍은 또한 MMR 기능이 감소하여 돌연변이가 번식할 수 있게 함으로써 유전적 불안정에 기여한다.[26] PMS2-/- 생쥐는 림프종과 사르코마가 발달한 것으로 나타났다. 또한 PMS2-/-인 수컷 생쥐는 멸균 상태임이 밝혀져 PMS2가 정조세포의 역할을 할 수 있음을 알 수 있었다.[7]
정상 결장에서의 역할
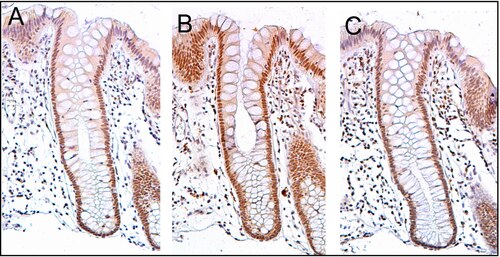
PMS2는 대장의 내부 표면에 줄지어 있는 대장암호 내에 있는 장세포(흡수세포)의 세포핵에서 높은 수준으로 표현된다(이미지, 패널 A 참조). PMS2, ERCC1 및 ERCC4(XPF) 단백질의 고표현을 수반하는 DNA 수리는 정상적이고 비가소성 대장상피에서 대장암호에서 매우 활발하게 나타난다. PMS2의 경우, 정상대장 상피에서 발현 수준이 암호의 77%~100%로 높다.[27]
세포는 암호기지에서 생성되어 암호기축을 따라 위쪽으로 이동하다가 며칠 후 대장내강으로 유출된다.[28] 그 지하에는 5~6개의 줄기세포가 있다.[28] 만약 암호화된 PMS2의 기저에 있는 줄기세포가 PMS2를 표현한다면, 일반적으로[29] 암호의 수천 개의 모든 세포도 PMS2를 표현하게 될 것이다. 이것은 이 절에 있는 이미지의 패널 A의 암호에 있는 대부분의 장세포에서 PMS2의 면역으로 보이는 갈색색으로 표시된다. ERCC4(XPF)와 ERCC1의 유사한 표현은 정상대장 상피의 각 대장암호에서 수천 개의 장세포에서 발생한다.
여기에 보이는 이미지의 조직 부분에도 헤마톡시린으로 역주사를 붙여 핵의 DNA를 청회색으로 얼룩지게 했다. 라미나 프로프리아 내 세포핵(상피암호 아래 및 주위에 있는 세포)은 헤마토실린 청회색을 주로 나타내며 PMS2, ERCC1 또는 ERCC4(XPF)의 발현이 거의 없다.
대장암
대장암에서 유래한 상피세포의 약 88%와 암에 인접한 10cm 이내의 상피세포의 약 50%(암이 발생할 가능성이 있는 현장결함)에 PMS2의 발현이 감소 또는 부재했다.[27]
대장 상피에 있는 PMS2의 결핍은 대부분 후생적 억압에 기인하는 것으로 보인다. 불일치 수리가 부족하고 부족하다고 분류되는 종양의 경우, 대부분의 PMS2 표현은 짝짓기 파트너인 MLH1이 부족하기 때문에 부족하다.[30] PMS2와 MLH1의 페어링이 안정화된다.[31] 산발성 암에서 MLH1이 손실된 것은 66건 중 65건의 촉진자 메틸화에 의한 후생적 소음 때문이다. 16개 암에서 Pms2는 MLH1 단백질 발현이 존재했음에도 부족했다. 이 16건 중 10건은 원인이 규명되지 않았으나 6건은 Pms2에서 이질균선 돌연변이를 보이는 것으로 나타났고, 그 뒤 종양에서 이질균이 상실될 가능성이 있는 것으로 나타났다. 따라서 Pms2에 대한 표현이 부족한 119종양 중 6개(5%)만이 PMS2의 돌연변이에 의한 것이었다.
ERCC1 및 ERCC4(XPF)와의 조정

현장 결함의 대장암호에서 PMS2가 감소하는 경우, DNA 수리 효소 ERCC1과 ERCC4(XPF)의 발현 감소와도 가장 많이 연관된다(이 절의 이미지 참조). ERCC1 및/또는 ERCC4(XPF)가 부족하면 DNA 손상이 누적될 수 있다. 그러한 과도한 DNA 손상은 종종 사멸을 초래한다.[32] 그러나 PMS2에 추가된 결함은 이러한 사멸을 억제할 수 있다.[33][34] 따라서 ERCC1 및/또는 ERCC4(XPF)가 부족할 때 DNA 손상이 증가하면 PMS2의 추가 결핍이 선택될 가능성이 높다. ERCC1 결핍된 중국 햄스터 난소 세포가 DNA 손상을 반복적으로 입었을 때, 생존 세포에서 추출된 5개의 복제 세포 중 3개가 Pms2에서 변이되었다.[35]
대장암으로의 진행
ERCC1, PMS2 이중 돌연변이 중국 햄스터 난소 세포는 자외선(DNA손상제)에 노출되었을 때 야생형 중국 햄스터 난소 세포보다 7,375배, ERCC1에서만 결함 세포보다 967배 높은 돌연변이 빈도를 보였다.[35] 따라서 ERCC1과 PMS2의 대장 세포 결핍은 게놈 불안정을 야기한다. PMS2와 ERCC4(XPF)에 대해 두 배의 결함이 있는 세포에 대해서도 유사한 유전학적 불안정한 상황이 예상된다. 이러한 불안정성은 돌연변이 표현형을 유발하여 대장암으로의 진행을 향상시킬 가능성이 높으며,[36] 대장암과 관련된 장 결함에서 PMS2와 ERCC1[또는 PMS2와 ERCC4 (XPF)]이 두 배로 부족한 세포의 존재를 설명할 수 있다. 하퍼와 에일드지에 의해 지적된 바와 같이,[37] DNA 손상에 적절하게 대응하고 치료하는 능력의 결함은 많은 형태의 암의 기초가 된다.
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000122512 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Nicolaides NC, Papadopoulos N, Liu B, Wei YF, Carter KC, Ruben SM, Rosen CA, Haseltine WA, Fleischmann RD, Fraser CM (Sep 1994). "Mutations of two PMS homologues in hereditary nonpolyposis colon cancer". Nature. 371 (6492): 75–80. doi:10.1038/371075a0. PMID 8072530. S2CID 4244907.
- ^ Nicolaides NC, Carter KC, Shell BK, Papadopoulos N, Vogelstein B, Kinzler KW (November 1995). "Genomic organization of the human PMS2 gene family". Genomics. 30 (2): 195–206. doi:10.1006/geno.1995.9885. PMID 8586419.
- ^ "Entrez Gene: PMS2 PMS2 postmeiotic segregation increased 2 (S. cerevisiae)".
- ^ a b van Oers JM, Roa S, Werling U, Liu Y, Genschel J, Hou H, Sellers RS, Modrich P, Scharff MD, Edelmann W (12 July 2010). "PMS2 endonuclease activity has distinct biological functions and is essential for genome maintenance". Proc. Natl. Acad. Sci. U.S.A. 107 (30): 13384–9. doi:10.1073/pnas.1008589107. PMC 2922181. PMID 20624957.
- ^ Mac Partlin M, Homer E, Robinson H, McCormick CJ, Crouch DH, Durant ST, Matheson EC, Hall AG, Gillespie DA, Brown R (February 2003). "Interactions of the DNA mismatch repair proteins MLH1 and MSH2 with c-MYC and MAX". Oncogene. 22 (6): 819–25. doi:10.1038/sj.onc.1206252. PMID 12584560.
- ^ a b c Kondo E, Horii A, Fukushige S (April 2001). "The interacting domains of three MutL heterodimers in man: hMLH1 interacts with 36 homologous amino acid residues within hMLH3, hPMS1 and hPMS2". Nucleic Acids Res. 29 (8): 1695–702. doi:10.1093/nar/29.8.1695. PMC 31313. PMID 11292842.
- ^ a b Guerrette S, Acharya S, Fishel R (March 1999). "The interaction of the human MutL homologues in hereditary nonpolyposis colon cancer". J. Biol. Chem. 274 (10): 6336–41. doi:10.1074/jbc.274.10.6336. PMID 10037723.
- ^ a b c Chen J, Sadowski I (March 2005). "Identification of the mismatch repair genes PMS2 and MLH1 as p53 target genes by using serial analysis of binding elements". Proc. Natl. Acad. Sci. U.S.A. 102 (13): 4813–8. doi:10.1073/pnas.0407069102. PMC 555698. PMID 15781865.
- ^ a b c d Shimodaira H, Yoshioka-Yamashita A, Kolodner RD, Wang JY (March 2003). "Interaction of mismatch repair protein PMS2 and the p53-related transcription factor p73 in apoptosis response to cisplatin". Proc. Natl. Acad. Sci. U.S.A. 100 (5): 2420–5. doi:10.1073/pnas.0438031100. PMC 151356. PMID 12601175.
- ^ "PMS2 Gene". The GeneCards Human Gene Database. Weizmann Institute of Science.
- ^ Meyers M, Theodosiou M, Acharya S, Odegaard E, Wilson T, Lewis JE, Davis TW, Wilson-Van Patten C, Fishel R, Boothman DA (January 1997). "Cell cycle regulation of the human DNA mismatch repair genes hMSH2, hMLH1, and hPMS2". Cancer Res. 57 (2): 206–8. PMID 9000555.
- ^ a b c Fedier A, Ruefenacht UB, Schwarz VA, Haller U, Fink D (October 2002). "Increased sensitivity of p53-deficient cells to anticancer agents due to loss of Pms2". Br. J. Cancer. 87 (9): 1027–33. doi:10.1038/sj.bjc.6600599. PMC 2364320. PMID 12434296.
- ^ Nakagawa H, Lockman JC, Frankel WL, Hampel H, Steenblock K, Burgart LJ, Thibodeau SN, de la Chapelle A (July 2004). "Mismatch repair gene PMS2: disease-causing germline mutations are frequent in patients whose tumors stain negative for PMS2 protein, but paralogous genes obscure mutation detection and interpretation". Cancer Res. 64 (14): 4721–7. doi:10.1158/0008-5472.CAN-03-2879. PMID 15256438.
- ^ a b Chadwick RB, Meek JE, Prior TW, Peltomaki P, de La Chapelle A (December 2000). "Polymorphisms in a pseudogene highly homologous to PMS2". Hum. Mutat. 16 (6): 530. doi:10.1002/1098-1004(200012)16:6<530::AID-HUMU15>3.0.CO;2-6. PMID 11102987.
- ^ 찰머스 ZR, 코넬리 CF, Fabrizio D, 게이 L, 알리, SM, 이들은 에니스 R, 슈록 A, 캠벨 B, Shlien A, Chmielecki J, 황이 F, 그는 Y, 태양 JTabori U, 케네디는 M, 리버 DS, Roels S화이트 J-오토 GA, 로스 JS, Garraway L, 밀러 VA스테판은 PJ, Frampton GM(4월 2017년)."10만 인간의 암 게놈들의 분석 종양 변화하는 부담의 풍경을 드러내".게놈 메드. 9(34):epub.doi:10.1186/s13073-017-0424-2.PMC5395719.PMID 28420421.>
- ^ Goodman AM, Kato S, Bazhenova L, Patel SP, Frampton GM, Miller V, Stephens PJ, Daniels GA, Kurzrock R (November 2017). "Tumor Mutational Burden as an Independent Predictor of Response to Immunotherapy in Diverse Cancers". Mol. Cancer Ther. 16 (11): 2598–2608. doi:10.1158/1535-7163.MCT-17-0386. PMC 5670009. PMID 28835386.>
- ^ "PMS2 - PMS2 postmeiotic segregation increased 2 (S. cerevisiae)". Genetics Home Reference. U.S. National Library of Medicine.
- ^ Senter L, Clendenning M, Sotamaa K, Hampel H, Green J, Potter JD, Lindblom A, Lagerstedt K, Thibodeau SN, Lindor NM, Young J, Winship I, Dowty JG, White DM, Hopper JL, Baglietto L, Jenkins MA, de la Chapelle A (August 2008). "The clinical phenotype of Lynch syndrome due to germ-line PMS2 mutations". Gastroenterology. 135 (2): 419–28. doi:10.1053/j.gastro.2008.04.026. PMC 2759321. PMID 18602922.
- ^ a b Johannesma PC, van der Klift HM, van Grieken NC, Troost D, Te Riele H, Jacobs MA, Postma TJ, Heideman DA, Tops CM, Wijnen JT, Menko FH (September 2011). "Childhood brain tumours due to germline bi-allelic mismatch repair gene mutations". Clin. Genet. 80 (3): 243–55. doi:10.1111/j.1399-0004.2011.01635.x. PMID 21261604. S2CID 23927730.
- ^ De Rosa M, Fasano C, Panariello L, Scarano MI, Belli G, Iannelli A, Ciciliano F, Izzo P (March 2000). "Evidence for a recessive inheritance of Turcot's syndrome caused by compound heterozygous mutations within the PMS2 gene". Oncogene. 19 (13): 1719–1723. doi:10.1038/sj.onc.1203447. PMID 10763829.
- ^ a b Herkert JC, Niessen RC, Olderode-Berends MJ, Veenstra-Knol HE, Vos YJ, van der Klift HM, Scheenstra R, Tops CM, Karrenbeld A, Peters FT, Hofstra RM, Kleibeuker JH, Sijmons RH (May 2011). "Paediatric intestinal cancer and polyposis due to bi-allelic PMS2 mutations: case series, review and follow-up guidelines". Eur. J. Cancer. 47 (7): 965–82. doi:10.1016/j.ejca.2011.01.013. PMID 21376568.
- ^ a b Gibson SL, Narayanan L, Hegan DC, Buermeyer AB, Liskay RM, Glazer PM (December 2006). "Overexpression of the DNA mismatch repair factor, PMS2, confers hypermutability and DNA damage tolerance". Cancer Lett. 244 (2): 195–202. doi:10.1016/j.canlet.2005.12.009. PMID 16426742.
- ^ a b c d e Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (2012). "Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer". Genome Integr. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ a b Baker AM, Cereser B, Melton S, Fletcher AG, Rodriguez-Justo M, Tadrous PJ, Humphries A, Elia G, McDonald SA, Wright NA, Simons BD, Jansen M, Graham TA (2014). "Quantification of crypt and stem cell evolution in the normal and neoplastic human colon". Cell Rep. 8 (4): 940–7. doi:10.1016/j.celrep.2014.07.019. PMC 4471679. PMID 25127143.
- ^ Nooteboom M, Johnson R, Taylor RW, Wright NA, Lightowlers RN, Kirkwood TB, Mathers JC, Turnbull DM, Greaves LC (2010). "Age-associated mitochondrial DNA mutations lead to small but significant changes in cell proliferation and apoptosis in human colonic crypts". Aging Cell. 9 (1): 96–9. doi:10.1111/j.1474-9726.2009.00531.x. PMC 2816353. PMID 19878146.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Chang DK, Ricciardiello L, Goel A, Chang CL, Boland CR (2000). "Steady-state regulation of the human DNA mismatch repair system". J. Biol. Chem. 275 (24): 18424–31. doi:10.1074/jbc.M001140200. PMID 10747992.
- ^ Norbury CJ, Zhivotovsky B (2004). "DNA damage-induced apoptosis". Oncogene. 23 (16): 2797–808. doi:10.1038/sj.onc.1207532. PMID 15077143.
- ^ Fukuhara S, Chang I, Mitsui Y, Chiyomaru T, Yamamura S, Majid S, Saini S, Deng G, Gill A, Wong DK, Shiina H, Nonomura N, Lau YF, Dahiya R, Tanaka Y (2015). "Functional role of DNA mismatch repair gene PMS2 in prostate cancer cells". Oncotarget. 6 (18): 16341–51. doi:10.18632/oncotarget.3854. PMC 4599273. PMID 26036629.
- ^ Marinovic-Terzic I, Yoshioka-Yamashita A, Shimodaira H, Avdievich E, Hunton IC, Kolodner RD, Edelmann W, Wang JY (2008). "Apoptotic function of human PMS2 compromised by the nonsynonymous single-nucleotide polymorphic variant R20Q". Proc. Natl. Acad. Sci. U.S.A. 105 (37): 13993–8. doi:10.1073/pnas.0806435105. PMC 2528866. PMID 18768816.
- ^ a b Nara K, Nagashima F, Yasui A (2001). "Highly elevated ultraviolet-induced mutation frequency in isolated Chinese hamster cell lines defective in nucleotide excision repair and mismatch repair proteins". Cancer Res. 61 (1): 50–2. PMID 11196196.
- ^ Loeb LA (2011). "Human cancers express mutator phenotypes: origin, consequences and targeting". Nat. Rev. Cancer. 11 (6): 450–7. doi:10.1038/nrc3063. PMC 4007007. PMID 21593786.
- ^ Harper JW, Elledge SJ (2007). "The DNA damage response: ten years after". Mol. Cell. 28 (5): 739–45. doi:10.1016/j.molcel.2007.11.015. PMID 18082599.