O-6-메틸과닌-DNA메틸전달효소
O-6-methylguanine-DNA methyltransferase| MGMT | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 별칭 | MGMT, Mgmt, AGT, AI267024, Agat, O-6-methylguanine-DNA 메틸전달효소 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 156569 MGI: 96977 HomoloGene: 31089 GeneCard: MGMT | ||||||||||||||||||||||||
| EC 번호 | 2.1.1.63 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) | |||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Chr 10: 129.47 – 129.77Mb | Cr 7: 136.89 – 137.13Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
O-alkylguanine6 DNA 알킬트랜스퍼레이즈(AGT, MGMT 또는 AGAT라고도 함)는 인간에서 O-메틸구아닌6 DNA 메틸트랜스퍼레이즈(MGMT) 유전자에 의해 인코딩되는 단백질이다.[5][6]O-메틸과닌6 DNA 메틸전달효소는 게놈 안정성을 위해 중요하다.자연적으로 발생하는 돌연변이 DNA 병변 O-메틸구아닌을6 구아닌으로 복구하고 DNA 복제와 전사 과정에서 불일치와 오류를 방지한다.따라서 MGMT의 상실은 알킬링제에 노출된 후 생쥐의 발암 위험을 증가시킨다.[7]두 박테리아 이소자미는 에이다와 오트다.null
기능 및 메커니즘
| '오알킬구아닌DNA알킬트랜스퍼레이제6' | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 2.1.1.63 | ||||||||
| CAS 번호. | 77271-19-3 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 입력 | ||||||||
| 엑스퍼시 | 나이스자이메 뷰 | ||||||||
| 케그 | KEG 입력 | ||||||||
| 메타사이크 | 대사통로 | ||||||||
| 프리암 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
알킬링 무타겐은 N7 위치에서 구아닌 기지를 우선적으로 수정하지만, 오알킬-과닌은6 DNA에서 발암성 주요 병변이다.이 DNA 인덕트는 S2N 메커니즘을 통해 수리 단백질 O-alkylguanine6 DNA 알킬트랜스퍼레이제에 의해 제거된다.이 단백질은 스토오치메트릭 반응에서 병변에서 알킬 그룹을 제거하고 활성 효소가 알킬화 후 재생되지 않기 때문에 진정한 효소가 아니다(자살 효소라고도 한다).단백질의 메틸 수용체 잔류물은 시스테인이다.[8]null
- 6-O-메틸과노신-과노신 디메틸화
임상적 유의성
뇌종양의 심각한 유형인 교모세포종 환자의 경우 암 치료제 테모졸로마이드가 유전자의 촉진제를 메틸화시킨 환자에게 더 효과적이다.[9]전체적으로 MGMT 메틸화는 임상 예측 모델에서 환자의 생존 장기화와 관련이 있다.[10]임상 환경에서 MGMT 프로모터 메틸레이션 상태 테스트의 경우 면역항생화학 또는 RNA 기반 검사보다 메틸화 특화 폴리머아제 연쇄반응(MS-PCR) 또는 파이로시퀀싱과 같은 DNA 기반 방법을 선호한다.[11]null
MGMT는 또한 유전자 치료 효율을 높이는 유용한 도구인 것으로 밝혀졌다.관심 트랜스젠과 MGMT로 구성된 두 가지 성분 벡터를 사용하여 체내 약물 선택을 활용하여 성공적인 변환 세포를 선택할 수 있다.[12]null
환경,[13] 담배 연기,[14] 식품,[15] 내생 대사 제품뿐만[16] 아니라 무타겐은 알킬레이트 또는 특정 메틸레이트 DNA를 생성하는 반응성 전기생성 종을 생성하여 6-O-메틸과닌(m6G)을 생성한다.null
1985년 야로쉬는 m6G를 가장 돌연변이 유발적이고 발암성이 강한 DNA의 알킬링 베이스로 확립한 초기 작업을 요약했다.[17]1994년 Rasouli-Nia 외 [18]연구진은 DNA에서 8개의 m6Gs가 손상되지 않은 것에 대해 약 1개의 돌연변이가 유도되었다는 것을 보여주었다.돌연변이는 자연선택의 과정에 의해 암으로 발전할 수 있다.[citation needed]null
암에서의 표현
| 암종류 | 암의 결핍 빈도 | 인접 필드 결함의 결여 빈도 |
|---|---|---|
| 자궁경부[19] | 61% | 39% |
| 콜로렉탈 | 40%-90%[20][21][22][23][24] | 11%-34%[20][21] |
| 미세 위성 불안정성을[25] 가진 대장 | 70% | 60% |
| 식도선상피종 | 71%-79%[26][27] | 89%[27] |
| 식도편평세포암 | 38%-96%[26][28][29] | 65%[29] |
| 촉진제 메틸화에 의한 교모세포종 | 44%-59%[30][31] | |
| 머리와 목의 편평한 세포암 | 54%[32] | |
| 간세포암(뇌염 C 바이러스 관련)[33] | 68% | 65% |
| 후두 | 54%-61%[34][35] | 38%[35] |
| 위 | 32%-88%[36][37] | 17%-78%[36][37] |
| 갑상선[38] | 87% |
후생적 억압
DNA 수리 결핍증을 가진 소수의 산발적인 암만이 DNA 수리 유전자에 돌연변이를 가지고 있다.그러나 DNA 수리 결핍증을 가진 산발적인 암의 대부분은 DNA 수리 유전자 발현을 줄이거나 침묵시키는 후생유전적 변화를 한 개 이상 가지고 있다.예를 들어 113개 순차 대장암에 대한 연구에서 DNA 수리 유전자 MGMT에서 오감 변이가 발생한 경우는 4명에 불과했고, MGMT 추진자 부위의 메틸화(후유전적 변화)로 인해 MGMT 발현이 감소된 경우는 대부분이었다.[39]null
MGMT는 여러 가지 방법으로 후생적으로 억제될 수 있다.[40]암에서 MGMT 표현을 억제할 때, 이것은 종종 그것의 촉진자 지역의 메틸화 때문이다.[40]그러나 히스톤 3의[41] 리신 9를 디메틸화하거나 miR-181d, miR-767-3p, miR-603을 포함한 다수의 마이크로RNA를 과다하게 표현하여 표현도 억제할 수 있다.[40][42][43]null
현장결함결함
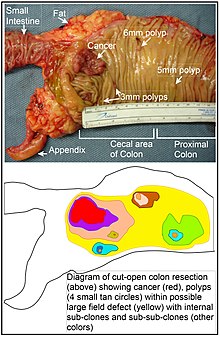
장 결함은 후생유전적 변화 및/또는 돌연변이에 의해 암의 발달을 지향하도록 전제된 상피의 영역 또는 "장"이다.대장암과 4개의 작은 용종이 같은 부위에 있는 대장 부분의 사진과 도표에도 필드 결함이 묘사되어 있다.루빈이 지적한 바와 같이, "암 연구에서의 대부분의 연구는 체내 종양이나 체외 이산 신소성 집중에 대해 잘 정의된 종양에 대해 행해져 왔다.[44]그러나 돌연변이 표현형 인간 대장종양에서 발견된 체 돌연변이의 80% 이상이 말단 종양 확장 이전에 발생한다는 증거가 있다."[45]마찬가지로 보겔슈타인 등은 종양에서 확인된 체세포 돌연변이의 절반 이상이 겉보기에는 정상세포가 성장하는 동안 (장 결함에서) 신소성 전 단계에서 발생했다고 지적한다.[46]null
위의 표에서 MGMT 결함은 대부분의 암을 둘러싼 장 결함(역사적으로 정상적인 조직)에서 확인되었다.만약 MGMT가 후생적으로 감소하거나 침묵한다면, 그것은 줄기세포에 선택적 우위를 부여하지 않을 것이다.그러나 MGMT의 발현이 감소하거나 없을 경우 돌연변이 발생률이 증가하며, 하나 이상의 돌연변이 유전자가 세포에 선택적 이점을 제공할 수 있다.그런 다음 돌연변이 줄기세포가 확장된 클론을 생성할 때 표현 결핍 MGMT 유전자는 선택적으로 중립적이거나 약간 유해한 승객(히치하이커) 유전자로 함께 운반될 수 있다.후생적으로 억압된 MGMT를 가진 클론의 지속적인 존재는 더 이상의 돌연변이를 계속 발생시킬 것이며, 그 중 일부는 종양을 발생시킬 수 있다.null
외생손상이 있는 결핍증
MGMT 결핍만으로는 암으로 진행하기에 충분하지 않을 수 있다.MGMT에서 균질 돌연변이를 가진 생쥐는 스트레스 없이 자랄 때 야생형 생쥐보다 암이 더 많이 발병하지 않았다.[47]그러나 아조시메탄과 덱스트란 황산염을 가진 생쥐에 대한 스트레스 치료는 MGMT 돌연변이 생쥐당 대장 종양을 4개 이상 유발했지만 야생형 생쥐당 종양은 1개 미만이었다.[48]null
다른 DNA 수리 유전자와 협조하는 억압
암에서는 다수의 DNA 수리 유전자가 동시에 억제되는 경우가 많다.[49]한 예로 MGMT를 포함하여, Jiang 외 [50]연구진은 40개의 아스트로사이토마에서 27개의 DNA 수리 유전자의 mRNA 발현을 비아스트로사이토마 개인으로부터 얻은 정상 뇌조직과 비교하여 평가한 연구를 수행했다.Among the 27 DNA repair genes evaluated, 13 DNA repair genes, MGMT, NTHL1, OGG1, SMUG1, ERCC1, ERCC2, ERCC3, ERCC4, MLH1, MLH3, RAD50, XRCC4 and XRCC5 were all significantly down-regulated in all three grades (II, III and IV) of astrocytomas.이 13개의 유전자를 저학년에서뿐만 아니라 고학년에서 억압한 것은 그것들이 초창기뿐만 아니라 후기 아스트로사이토마 단계에서도 중요할 수 있다는 것을 시사했다.또 다른 예에서 키타지마 [51]외 연구진은 위암 135개 검체에서 MGMT와 MLH1 발현에 대한 면역 활성도가 밀접하게 상관되어 있고 MGMT와 hMLH1의 상실이 종양 진행 중에 동시에 가속되는 것으로 보인다는 것을 발견했다.null
다수의 DNA 수리 유전자의 불충분한 발현이 암에서 종종 발견되며,[49] 암에서 주로 발견되는 수천 개의 돌연변이에 기여할 수 있다(암에서 돌연변이 빈도 참조).null
상호작용
O-메틸과닌-DNA 메틸전달효소는 에스트로겐 수용체 알파와 상호작용하는 것으로 나타났다.[52]null
참고 항목
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000170430 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000054612 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Tano K, Shiota S, Collier J, Foote RS, Mitra S (January 1990). "Isolation and structural characterization of a cDNA clone encoding the human DNA repair protein for O6-alkylguanine". Proc. Natl. Acad. Sci. U.S.A. 87 (2): 686–90. Bibcode:1990PNAS...87..686T. doi:10.1073/pnas.87.2.686. PMC 53330. PMID 2405387.
- ^ Natarajan AT, Vermeulen S, Darroudi F, Valentine MB, Brent TP, Mitra S, Tano K (January 1992). "Chromosomal localization of human O6-methylguanine-DNA methyltransferase (MGMT) gene by in situ hybridization". Mutagenesis. 7 (1): 83–5. doi:10.1093/mutage/7.1.83. PMID 1635460.
- ^ Shiraishi A, Sakumi K, Sekiguchi M (October 2000). "Increased susceptibility to chemotherapeutic alkylating agents of mice deficient in DNA repair methyltransferase". Carcinogenesis. 21 (10): 1879–83. doi:10.1093/carcin/21.10.1879. PMID 11023546.
- ^ Kaina B, Christmann M, Naumann S, Roos WP (August 2007). "MGMT: key node in the battle against genotoxicity, carcinogenicity and apoptosis induced by alkylating agents". DNA Repair (Amst.). 6 (8): 1079–99. doi:10.1016/j.dnarep.2007.03.008. PMID 17485253.
- ^ Hegi ME, Diserens AC, Gorlia T, Hamou MF, de Tribolet N, Weller M, Kros JM, Hainfellner JA, Mason W, Mariani L, Bromberg JE, Hau P, Mirimanoff RO, Cairncross JG, Janzer RC, Stupp R (2005). "MGMT gene silencing and benefit from temozolomide in glioblastoma". N. Engl. J. Med. 352 (10): 997–1003. doi:10.1056/NEJMoa043331. PMID 15758010.
- ^ Molenaar RJ, Verbaan D, Lamba S, Zanon C, Jeuken JW, Boots-Sprenger SH, Wesseling P, Hulsebos TJ, Troost D, van Tilborg AA, Leenstra S, Vandertop WP, Bardelli A, van Noorden CJ, Bleeker FE (2014). "The combination of IDH1 mutations and MGMT methylation status predicts survival in glioblastoma better than either IDH1 or MGMT alone". Neuro-Oncology. 16 (9): 1263–73. doi:10.1093/neuonc/nou005. PMC 4136888. PMID 24510240.
- ^ Preusser, M.; Janzer, Charles R.; Felsberg, J.; Reifenberger, G.; Hamou, M. F.; Diserens, A. C.; Stupp, R.; Gorlia, T.; Marosi, C.; Heinzl, H.; Hainfellner, J. A.; Hegi, M. (Oct 2008). "Anti-O6-methylguanine-methyltransferase (MGMT) immunohistochemistry in glioblastoma multiforme: observer variability and lack of association with patient survival impede its use as clinical biomarker". Brain Pathol. 18 (4): 520–532. doi:10.1111/j.1750-3639.2008.00153.x. PMC 8095504. PMID 18400046. S2CID 21167901.
- ^ Chang AH, Stephan MT, Lisowski L, Sadelain M (2008). "Erythroid-specific human factor IX delivery from in vivo selected hematopoietic stem cells following nonmyeloablative conditioning in hemophilia B mice". Mol. Ther. 16 (10): 1745–52. doi:10.1038/mt.2008.161. PMC 2658893. PMID 18682698.
- ^ Bartsch H, Montesano R (1984). "Relevance of nitrosamines to human cancer". Carcinogenesis. 5 (11): 1381–93. doi:10.1093/carcin/5.11.1381. PMID 6386215.
- ^ Christmann M, Kaina B (2012). "O(6)-methylguanine-DNA methyltransferase (MGMT): impact on cancer risk in response to tobacco smoke". Mutat. Res. 736 (1–2): 64–74. doi:10.1016/j.mrfmmm.2011.06.004. PMID 21708177.
- ^ Fahrer J, Kaina B (2013). "O6-methylguanine-DNA methyltransferase in the defense against N-nitroso compounds and colorectal cancer". Carcinogenesis. 34 (11): 2435–42. doi:10.1093/carcin/bgt275. PMID 23929436.
- ^ De Bont R, van Larebeke N (2004). "Endogenous DNA damage in humans: a review of quantitative data". Mutagenesis. 19 (3): 169–85. doi:10.1093/mutage/geh025. PMID 15123782.
- ^ Yarosh DB (1985). "The role of O6-methylguanine-DNA methyltransferase in cell survival, mutagenesis and carcinogenesis". Mutat. Res. 145 (1–2): 1–16. doi:10.1016/0167-8817(85)90034-3. PMID 3883145.
- ^ Rasouli-Nia A, Sibghat-Ullah, Mirzayans R, Paterson MC, Day RS (1994). "On the quantitative relationship between O6-methylguanine residues in genomic DNA and production of sister-chromatid exchanges, mutations and lethal events in a Mer- human tumor cell line". Mutat. Res. 314 (2): 99–113. doi:10.1016/0921-8777(94)90074-4. PMID 7510369.
- ^ Iliopoulos D, Oikonomou P, Messinis I, Tsezou A (2009). "Correlation of promoter hypermethylation in hTERT, DAPK and MGMT genes with cervical oncogenesis progression". Oncol. Rep. 22 (1): 199–204. doi:10.3892/or_00000425. PMID 19513524.
- ^ a b Shen L, Kondo Y, Rosner GL, Xiao L, Hernandez NS, Vilaythong J, Houlihan PS, Krouse RS, Prasad AR, Einspahr JG, Buckmeier J, Alberts DS, Hamilton SR, Issa JP (2005). "MGMT promoter methylation and field defect in sporadic colorectal cancer". J. Natl. Cancer Inst. 97 (18): 1330–8. doi:10.1093/jnci/dji275. PMID 16174854.
- ^ a b Lee KH, Lee JS, Nam JH, Choi C, Lee MC, Park CS, Juhng SW, Lee JH (2011). "Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence". Langenbecks Arch Surg. 396 (7): 1017–26. doi:10.1007/s00423-011-0812-9. PMID 21706233. S2CID 8069716.
- ^ Psofaki V, Kalogera C, Tzambouras N, Stephanou D, Tsianos E, Seferiadis K, Kolios G (2010). "Promoter methylation status of hMLH1, MGMT, and CDKN2A/p16 in colorectal adenomas". World J. Gastroenterol. 16 (28): 3553–60. doi:10.3748/wjg.v16.i28.3553. PMC 2909555. PMID 20653064.
- ^ Amatu A, Sartore-Bianchi A, Moutinho C, Belotti A, Bencardino K, Chirico G, Cassingena A, Rusconi F, Esposito A, Nichelatti M, Esteller M, Siena S (2013). "Promoter CpG island hypermethylation of the DNA repair enzyme MGMT predicts clinical response to dacarbazine in a phase II study for metastatic colorectal cancer". Clin. Cancer Res. 19 (8): 2265–72. doi:10.1158/1078-0432.CCR-12-3518. PMID 23422094.
- ^ Mokarram P, Zamani M, Kavousipour S, Naghibalhossaini F, Irajie C, Moradi Sarabi M, Hosseini SV (2013). "Different patterns of DNA methylation of the two distinct O6-methylguanine-DNA methyltransferase (O6-MGMT) promoter regions in colorectal cancer". Mol. Biol. Rep. 40 (5): 3851–7. doi:10.1007/s11033-012-2465-3. PMID 23271133. S2CID 18733871.
- ^ Svrcek M, Buhard O, Colas C, Coulet F, Dumont S, Massaoudi I, Lamri A, Hamelin R, Cosnes J, Oliveira C, Seruca R, Gaub MP, Legrain M, Collura A, Lascols O, Tiret E, Fléjou JF, Duval A (2010). "Methylation tolerance due to an O6-methylguanine DNA methyltransferase (MGMT) field defect in the colonic mucosa: an initiating step in the development of mismatch repair-deficient colorectal cancers". Gut. 59 (11): 1516–26. doi:10.1136/gut.2009.194787. PMID 20947886. S2CID 206950452.
- ^ a b Hasina R, Surati M, Kawada I, Arif Q, Carey GB, Kanteti R, Husain AN, Ferguson MK, Vokes EE, Villaflor VM, Salgia R (2013). "O-6-methylguanine-deoxyribonucleic acid methyltransferase methylation enhances response to temozolomide treatment in esophageal cancer". J Carcinog. 12: 20. doi:10.4103/1477-3163.120632. PMC 3853796. PMID 24319345.
- ^ a b Kuester D, El-Rifai W, Peng D, Ruemmele P, Kroeckel I, Peters B, Moskaluk CA, Stolte M, Mönkemüller K, Meyer F, Schulz HU, Hartmann A, Roessner A, Schneider-Stock R (2009). "Silencing of MGMT expression by promoter hypermethylation in the metaplasia-dysplasia-carcinoma sequence of Barrett's esophagus". Cancer Lett. 275 (1): 117–26. doi:10.1016/j.canlet.2008.10.009. PMC 4028828. PMID 19027227.
- ^ Ling ZQ, Li P, Ge MH, Hu FJ, Fang XH, Dong ZM, Mao WM (2011). "Aberrant methylation of different DNA repair genes demonstrates distinct prognostic value for esophageal cancer". Dig. Dis. Sci. 56 (10): 2992–3004. doi:10.1007/s10620-011-1774-z. PMID 21674174. S2CID 22913110.
- ^ a b Su Y, Yin L, Liu R, Sheng J, Yang M, Wang Y, Pan E, Guo W, Pu Y, Zhang J, Liang G (2014). "Promoter methylation status of MGMT, hMSH2, and hMLH1 and its relationship to corresponding protein expression and TP53 mutations in human esophageal squamous cell carcinoma". Med. Oncol. 31 (2): 784. doi:10.1007/s12032-013-0784-4. PMID 24366688. S2CID 22746140.
- ^ Morandi L, Franceschi E, de Biase D, Marucci G, Tosoni A, Ermani M, Pession A, Tallini G, Brandes A (2010). "Promoter methylation analysis of O6-methylguanine-DNA methyltransferase in glioblastoma: detection by locked nucleic acid based quantitative PCR using an imprinted gene (SNURF) as a reference". BMC Cancer. 10: 48. doi:10.1186/1471-2407-10-48. PMC 2843669. PMID 20167086.
- ^ Quillien V, Lavenu A, Karayan-Tapon L, Carpentier C, Labussière M, Lesimple T, Chinot O, Wager M, Honnorat J, Saikali S, Fina F, Sanson M, Figarella-Branger D (2012). "Comparative assessment of 5 methods (methylation-specific polymerase chain reaction, MethyLight, pyrosequencing, methylation-sensitive high-resolution melting, and immunohistochemistry) to analyze O6-methylguanine-DNA-methyltranferase in a series of 100 glioblastoma patients". Cancer. 118 (17): 4201–11. doi:10.1002/cncr.27392. PMID 22294349. S2CID 8145409.
- ^ Koutsimpelas D, Pongsapich W, Heinrich U, Mann S, Mann WJ, Brieger J (2012). "Promoter methylation of MGMT, MLH1 and RASSF1A tumor suppressor genes in head and neck squamous cell carcinoma: pharmacological genome demethylation reduces proliferation of head and neck squamous carcinoma cells". Oncol. Rep. 27 (4): 1135–41. doi:10.3892/or.2012.1624. PMC 3583513. PMID 22246327.
- ^ Zekri AR, Bahnasy AA, Shoeab FE, Mohamed WS, El-Dahshan DH, Ali FT, Sabry GM, Dasgupta N, Daoud SS (2014). "Methylation of multiple genes in hepatitis C virus associated hepatocellular carcinoma". J Adv Res. 5 (1): 27–40. doi:10.1016/j.jare.2012.11.002. PMC 4294722. PMID 25685469.
- ^ Pierini S, Jordanov SH, Mitkova AV, Chalakov IJ, Melnicharov MB, Kunev KV, Mitev VI, Kaneva RP, Goranova TE (2014). "Promoter hypermethylation of CDKN2A, MGMT, MLH1, and DAPK genes in laryngeal squamous cell carcinoma and their associations with clinical profiles of the patients". Head Neck. 36 (8): 1103–8. doi:10.1002/hed.23413. PMID 23804521. S2CID 11916790.
- ^ a b Paluszczak J, Misiak P, Wierzbicka M, Woźniak A, Baer-Dubowska W (2011). "Frequent hypermethylation of DAPK, RARbeta, MGMT, RASSF1A and FHIT in laryngeal squamous cell carcinomas and adjacent normal mucosa". Oral Oncol. 47 (2): 104–7. doi:10.1016/j.oraloncology.2010.11.006. PMID 21147548.
- ^ a b Jin J, Xie L, Xie CH, Zhou YF (2014). "Aberrant DNA methylation of MGMT and hMLH1 genes in prediction of gastric cancer". Genet. Mol. Res. 13 (2): 4140–5. doi:10.4238/2014.May.30.9. PMID 24938706.
- ^ a b Zou XP, Zhang B, Zhang XQ, Chen M, Cao J, Liu WJ (2009). "Promoter hypermethylation of multiple genes in early gastric adenocarcinoma and precancerous lesions". Hum. Pathol. 40 (11): 1534–42. doi:10.1016/j.humpath.2009.01.029. PMID 19695681.
- ^ Mokhtar M, Kondo K, Namura T, Ali AH, Fujita Y, Takai C, Takizawa H, Nakagawa Y, Toba H, Kajiura K, Yoshida M, Kawakami G, Sakiyama S, Tangoku A (2014). "Methylation and expression profiles of MGMT gene in thymic epithelial tumors". Lung Cancer. 83 (2): 279–87. doi:10.1016/j.lungcan.2013.12.004. PMID 24388682.
- ^ Halford S, Rowan A, Sawyer E, Talbot I, Tomlinson I (June 2005). "O(6)-methylguanine methyltransferase in colorectal cancers: detection of mutations, loss of expression, and weak association with G:C>A:T transitions". Gut. 54 (6): 797–802. doi:10.1136/gut.2004.059535. PMC 1774551. PMID 15888787.
- ^ a b c Cabrini G, Fabbri E, Lo Nigro C, Dechecchi MC, Gambari R (2015). "Regulation of expression of O6-methylguanine-DNA methyltransferase and the treatment of glioblastoma (Review)". Int. J. Oncol. 47 (2): 417–28. doi:10.3892/ijo.2015.3026. PMC 4501657. PMID 26035292.
- ^ Nakagawachi T, Soejima H, Urano T, Zhao W, Higashimoto K, Satoh Y, Matsukura S, Kudo S, Kitajima Y, Harada H, Furukawa K, Matsuzaki H, Emi M, Nakabeppu Y, Miyazaki K, Sekiguchi M, Mukai T (2003). "Silencing effect of CpG island hypermethylation and histone modifications on O6-methylguanine-DNA methyltransferase (MGMT) gene expression in human cancer". Oncogene. 22 (55): 8835–44. doi:10.1038/sj.onc.1207183. PMID 14647440.
- ^ Kushwaha D, Ramakrishnan V, Ng K, Steed T, Nguyen T, Futalan D, Akers JC, Sarkaria J, Jiang T, Chowdhury D, Carter BS, Chen CC (2014). "A genome-wide miRNA screen revealed miR-603 as a MGMT-regulating miRNA in glioblastomas". Oncotarget. 5 (12): 4026–39. doi:10.18632/oncotarget.1974. PMC 4147303. PMID 24994119.
- ^ Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC (2012). "miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression". Neuro-Oncology. 14 (6): 712–9. doi:10.1093/neuonc/nos089. PMC 3367855. PMID 22570426.
- ^ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays. 33 (3): 224–31. doi:10.1002/bies.201000067. PMID 21254148. S2CID 44981539.
- ^ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, Tavaré S, Shibata D (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proc. Natl. Acad. Sci. U.S.A. 97 (3): 1236–41. Bibcode:2000PNAS...97.1236T. doi:10.1073/pnas.97.3.1236. PMC 15581. PMID 10655514.
- ^ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science. 339 (6127): 1546–58. Bibcode:2013Sci...339.1546V. doi:10.1126/science.1235122. PMC 3749880. PMID 23539594.
- ^ Meira LB, Calvo JA, Shah D, Klapacz J, Moroski-Erkul CA, Bronson RT, Samson LD (2014). "Repair of endogenous DNA base lesions modulate lifespan in mice". DNA Repair (Amst.). 21: 78–86. doi:10.1016/j.dnarep.2014.05.012. PMC 4125484. PMID 24994062.
- ^ Wirtz S, Nagel G, Eshkind L, Neurath MF, Samson LD, Kaina B (2010). "Both base excision repair and O6-methylguanine-DNA methyltransferase protect against methylation-induced colon carcinogenesis". Carcinogenesis. 31 (12): 2111–7. doi:10.1093/carcin/bgq174. PMC 2994278. PMID 20732909.
- ^ a b Bernstein C, Bernstein H (2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World J Gastrointest Oncol. 7 (5): 30–46. doi:10.4251/wjgo.v7.i5.30. PMC 4434036. PMID 25987950.
- ^ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neurosci. Lett. 409 (2): 112–7. doi:10.1016/j.neulet.2006.09.038. PMID 17034947.
- ^ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer. 6 (2): 86–95. doi:10.1007/s10120-003-0213-z. PMID 12861399.
- ^ Teo AK, Oh HK, Ali RB, Li BF (October 2001). "The modified human DNA repair enzyme O(6)-methylguanine-DNA methyltransferase is a negative regulator of estrogen receptor-mediated transcription upon alkylation DNA damage". Mol. Cell. Biol. 21 (20): 7105–14. doi:10.1128/MCB.21.20.7105-7114.2001. PMC 99886. PMID 11564893.
추가 읽기
- Margison GP, Povey AC, Kaina B, Santibáñez Koref MF (2003). "Variability and regulation of O6-alkylguanine-DNA alkyltransferase". Carcinogenesis. 24 (4): 625–35. doi:10.1093/carcin/bgg005. PMID 12727789.