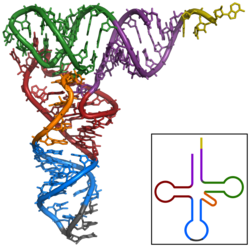
비부호화 RNA
Non-coding RNAncRNA(non-coding RNA)는 단백질로 변환되지 않은 RNA 분자입니다.기능성 비코드 RNA가 전사되는 DNA 배열은 종종 RNA 유전자라고 불립니다.풍부하고 기능적으로 중요한 비부호화 RNA의 종류에는 이동 RNA(tRNA)와 리보솜 RNA(RNA)뿐만 아니라 마이크로RNA, siRNA, piRNA, snoRNA, SNRNA, EXRNA, scaRNA 및 긴 NCNA와 같은 작은 RNA가 포함된다.
인간 게놈 내에 있는 코드화되지 않은 RNA의 수는 알려지지 않았지만, 최근의 전사체학 및 생물정보학 연구는 수천 개의 [1][2][3][4][5][6]RNA가 있다는 것을 시사한다.새롭게 식별된 많은 ncRNA는 [7]그 기능에 대해 검증되지 않았다.또한 많은 ncRNA가 기능하지 않고(때로는 정크 RNA라고도 함) 가짜 [8][9]전사의 산물일 가능성이 높다.
비암호화 RNA는 암과 알츠하이머를 포함한 질병에 기여하는 것으로 생각된다.
이력 및 검출
핵산은 1868년 Friedrich Miescher에[10] 의해 처음 발견되었고 1939년까지 RNA는 단백질 [11]합성에 관여했다.20년 후, 프란시스 크릭은 번역을 매개하는 기능성 RNA 성분을 예측했다; 그는 RNA가 순수한 폴리펩타이드보다 [12]mRNA 전사체를 가진 염기쌍에 더 적합하다고 추론했다.
특성화 된 최초의 비코드 RNA는 제빵 효모에서 발견된 알라닌 tRNA였으며, [13]그 구조는 1965년에 발표되었다.정제된 알라닌 tRNA 시료 Robert W. 할리 등시판용 빵집 효모 140kg을 사용해 정제 tRNA 1g만 [14]분석했습니다.80 뉴클레오티드 tRNA는 먼저 췌장 리보핵산가수분해효소(시토신 또는 우리딘으로 끝나는 단편 생성)와 타카디아스타아제 리보핵산가수분해효소 Tl(구아노신으로 끝나는 단편 생성)로 소화되어 배열되었다.그 후 크로마토그래피와 5'와 3' 말단의 식별은 RNA [14]염기서열을 확립하기 위해 단편들을 배열하는 데 도움을 주었다.원래 이 [13]tRNA에 대해 제안된 세 가지 구조 중 '클로버리프' 구조는 다음 여러 [15][16][17][18]출판물에서 독립적으로 제안되었다.클로버 리프 2차 구조는 1974년 [19][20]두 개의 독립적인 연구 그룹이 수행한 X선 결정학 분석에 따라 완성되었다.
리보솜 RNA가 다음으로 발견되었고 1980년대 초에 URNA가 그 뒤를 이었다.그 이후로, 새로운 비코드 RNA의 발견은 snoRNA, Xist, CRISPR 그리고 [21]더 많은 것들과 함께 계속되었다.최근 주목할 만한 추가 사항으로는 리보스위치 및 miRNA가 있다. 후자와 관련된 RNAi 메커니즘의 발견은 크레이그 C를 얻었다. 멜로와 앤드류는 2006년 노벨 [22]생리의학상을 수상했다.
최근 ncRNA의 발견은 실험적인 방법과 생체 정보적인 방법 모두를 통해 이루어졌다.
생물학적 역할
비코딩 RNA는 여러 그룹에 속하며 많은 세포 [23]과정에 관여합니다.이것들은 모든 또는 대부분의 세포 수명 동안 보존되는 중심적인 중요도의 ncRNA에서 하나 또는 소수의 밀접하게 관련된 종에 특정한 보다 일시적인 ncRNA에 이르기까지 다양하다.더 보존된 ncRNA는 마지막 공통 조상과 RNA 세계의 분자 화석이나 유물로 생각되며, 현재 그들의 역할은 대부분 DNA에서 [24][25][26]단백질로의 정보 흐름을 조절하는 데 있다.
번역중

보존되고 필수적이며 풍부한 ncRNA의 대부분은 번역과 관련되어 있습니다.리보솜이라고 불리는 리보핵단백질(RNP) 입자는 세포 내에서 번역이 일어나는 '요소'이다.리보솜은 60% 이상의 리보솜 RNA로 구성되어 있다; 이것들은 원핵생물에서 3개의 ncRNA와 진핵생물에서 4개의 ncRNA로 구성되어 있다.리보솜 RNA는 뉴클레오티드 배열을 단백질로 변환하는 것을 촉매한다.또 다른 ncRNA 세트인 Transfer RNA는 mRNA와 단백질 사이에서 '어댑터 분자'를 형성한다.H/ACA 박스와 C/D 박스 snoRNA는 고세균과 진핵생물에서 발견되는 ncRNA이다.RNAase MRP는 진핵생물로 제한된다.두 그룹의 ncRNA는 모두 rRNA 성숙에 관여한다.snoRNA는 rRNA, tRNA 및 snRNA의 공유변형을 유도합니다.RNase MRP는 내부 전사 스페이서 1을 18S와 5.8S의 rRNA 사이에서 절단합니다.유비쿼터스 ncRNA, RNase P는 RNase [28]MRP의 진화적 친척이다. RNase P는 전구체 tRNA의 5'-리더 요소를 분해하여 tRNA의 성숙한 5'-ends를 생성함으로써 tRNA 서열을 성숙시킨다.SRP라고 불리는 또 다른 유비쿼터스 RNP는 특정 초기 단백질을 인식하고 진핵생물의 소포체와 원핵생물의 혈장막으로 운반한다.박테리아에서 Transfer-messenger RNA(tmRNA)는 정지된 리보솜을 구하고, 불완전한 폴리펩타이드에 태그를 붙이고, 이상 [citation needed]mRNA의 분해를 촉진하는 데 관여하는 RNP이다.
RNA 스플라이싱 내
진핵생물에서 스플라이싱은 인트론 배열을 제거하기 위해 필수적인 스플라이싱 반응을 수행하며, 이 과정은 성숙한 mRNA 형성에 필요하다.스플라이소좀은 snRNP 또는 tri-snRNP라고도 알려진 또 다른 RNP입니다.스플라이소좀에는 큰 형태와 작은 형태 두 가지가 있다.주요 스플라이소좀의 ncRNA 성분은 U1, U2, U4, U5, U6입니다.마이너 스플라이소좀의 ncRNA 구성 요소는 U11, U12, U5, U4atac [citation needed]및 U6atac입니다.
또 다른 인트론 그룹은 숙주 전사물로부터 그들 자신의 제거를 촉매할 수 있습니다; 이것들은 자기 분열 RNA라고 불립니다.자가 스플라이싱 RNA에는 그룹 I 촉매 인트론과 그룹 II 촉매 인트론의 두 가지 주요 그룹이 있습니다.이러한 ncRNA는 광범위한 [citation needed]유기체의 mRNA, tRNA 및 rRNA 전구체로부터 자체 절제를 촉매한다.
포유동물에서 snoRNA는 mRNA의 대체 스플라이싱을 조절할 수 있는 것으로 밝혀졌다. 예를 들어 snoRNA HBII-52는 세로토닌 수용체 2C의 [29]스플라이싱을 조절한다.
선충류에서 SmY ncRNA는 mRNA 트랜스스플라이싱에 [citation needed]관여하는 것으로 보인다.
DNA 리플리케이션 중
Y RNA는 염색질 및 시작 단백질과의 상호작용을 통해 DNA 복제를 위해 필요한 줄기 고리입니다(원산지 인식 [31][32]복합체 포함).또한 Ro60 리보핵단백질[33] 입자의 성분으로 전신성 홍반성 [34]루푸스 환자의 자가면역항체의 표적이 된다.
유전자 조절 중
수천 개의 유전자의 발현은 ncRNA에 의해 조절된다.이 규정은 트랜스 또는 cis에서 발생할 수 있습니다.유전자의 인핸서 영역에서 전사된 인핸서 RNA라고 불리는 특별한 유형의 ncRNA가 유전자 [citation needed]발현을 촉진하는 작용을 한다는 증거가 증가하고 있다.
트랜스액션
고등 진핵생물에서 마이크로 RNA는 유전자 발현을 조절한다.단일 miRNA는 수백 개의 유전자의 발현 수준을 낮출 수 있다.성숙한 miRNA 분자가 작용하는 메커니즘은 하나 이상의 메신저 RNA(mRNA) 분자, 일반적으로 3' UTR에 부분 상보적인 것이다.miRNA의 주요 기능은 유전자 발현을 하향 조절하는 것이다.
ncRNA RNase P도 유전자 발현에 영향을 미치는 것으로 나타났다.인간핵에서 RNase P는 RNA 중합효소 III에 의해 전사된 다양한 ncRNA의 정상적이고 효율적인 전사에 필요하다.여기에는 tRNA, 5S rRNA, SRP RNA 및 U6 snRNA 유전자가 포함됩니다.RNase P는 활성 tRNA [35]및 5S rRNA 유전자의 Pol III 및 크로마틴과의 연관성을 통해 전사에 그 역할을 한다.
메타조안 ncRNA인 7SK RNA는 RNA 중합효소 II 신장인자 P-TEFb의 음의 조절인자로 작용하며, 이 활성은 스트레스 반응 [citation needed]경로의 영향을 받는 것으로 나타났다.
박테리아 ncRNA, 6S RNA는 특히 시그마70 특이성 인자를 포함하는 RNA 중합효소 홀로엔자임과 관련이 있습니다.이 상호작용은 정지상 [citation needed]동안 시그마70 의존 프로모터로부터의 발현을 억제한다.
또 다른 세균 ncRNA인 옥시S RNA는 샤인-달가노 배열에 결합함으로써 리보솜 결합을 흡수함으로써 번역을 억제한다.OxyS RNA는 [citation needed]대장균의 산화 스트레스에 반응하여 유도된다.
B2 RNA는 쥐 세포의 열 충격에 반응하여 mRNA 전사를 억제하는 작은 비코드 RNA 중합효소 III 전사체입니다.B2 RNA는 코어 Pol II에 결합함으로써 전사를 억제한다.이러한 상호작용을 통해 B2 RNA는 프로모터에서 사전 개시 복합체로 모여 RNA [36]합성을 차단한다.
최근의 연구는 단지 ncRNA 배열의 전사 작용이 유전자 발현에 영향을 미칠 수 있다는 것을 보여주었다.ncRNA의 RNA 중합효소 II 전사는 Chrisoscaromyces pombe의 염색질 재모델링을 위해 필요하다.여러 종의 ncRNA가 [37]전사됨에 따라 크로마틴은 점차 개방배치로 변환된다.
시스 작용
다수의 ncRNA가 단백질 코딩 유전자의 5' UTR(Untranslated Regions)에 내장되어 다양한 방식으로 발현에 영향을 준다.예를 들어, 리보스위치는 작은 표적 분자와 직접 결합할 수 있다; 표적 결합은 유전자의 [citation needed]활동에 영향을 미친다.
RNA 리더 배열은 아미노산 생합성 오퍼론의 첫 번째 유전자의 상류에서 발견된다.이러한 RNA 원소는 오퍼론의 최종 생성물 아미노산이 풍부한 매우 짧은 펩타이드 배열을 코드하는 영역에서 두 가지 가능한 구조 중 하나를 형성합니다.조절아미노산이 과잉되어 리더 전사체 상에서의 리보솜 이동이 저해되지 않을 때 터미네이터 구조가 형성된다.조절아미노산의 하전 tRNA가 결핍되면 리더펩타이드를 번역하는 리보솜이 정지하고 항거미자 구조를 형성한다.이것은 RNA 중합효소가 오퍼론을 전사할 수 있게 한다.알려진 RNA 리더는 히스티딘 오퍼론 리더, 류신 오퍼론 리더, 트레오닌 오퍼론 리더 및 트립토판 오퍼론 [citation needed]리더입니다.
철 응답 요소(IRE)는 철 응답 단백질(IRP)에 의해 결합됩니다.IRE는 철 대사에 관여하는 생성물을 가진 다양한 mRNA의 UTR에서 발견된다.철분 농도가 낮으면 IRP에 의해 페리틴 mRNA IRE가 결합되어 변환 [citation needed]억제가 이루어집니다.
내부 리보솜 진입부위(IRES)는 단백질 [citation needed]합성 과정의 일부로서 mRNA 배열 중간에 번역 개시를 가능하게 하는 RNA 구조이다.
게놈 방어에 있어서
포유류의 고환과 체세포에서 발현되는 Piwi-상호작용 RNA(piRNA)는 Piwi 단백질과 RNA-단백질 복합체를 형성한다.이러한 piRNA 복합체(piRCs)는 특히 정자 형성에 있는 역트랜스포존 및 다른 유전 요소의 전사 유전자 소음과 관련이 있다.
CRISPR(Clustered Regularly Interspace Short Palindromic Repeats)은 많은 박테리아와 고세균의 DNA에서 발견되는 반복입니다.반복은 유사한 길이의 스페이서로 구분됩니다.이러한 스페이서는 파지에서 파생되어 세포를 감염으로부터 보호하는 데 도움을 줄 수 있는 것으로 입증되었습니다.
염색체 구조
텔로머라아제(Telomerase)는 진핵생물 염색체의 말단에서 발견되는 텔로머 영역에 특정 DNA 염기서열 반복(척추동물에서 TTAGGG)을 추가하는 RNP 효소이다.텔로미어는 응축된 DNA 물질을 포함하고 있어 염색체에 안정감을 준다.이 효소는 Telomerase RNA를 운반하는 역전사효소이며, Telomerase RNA는 복제 주기 후에 짧아지는 텔로미어를 연장할 때 템플릿으로 사용됩니다.
Xist(X-비활성 특이적 전사체)는 태반 포유류의 X염색체 위에 있는 긴 ncRNA 유전자로 Bar body를 형성하는 X염색체 불활성화 과정의 주요 이펙터 역할을 한다.안티센스 RNA인 Tsix는 Xist의 음의 조절제이다.Tsix 발현이 결여된 X염색체(따라서 Xist 전사가 높은 수준)는 일반 염색체보다 더 자주 비활성화된다.XY 성결정 시스템을 사용하는 드로소필리드에서도 roX(X상의 RNA) RNA는 용량 [38]보상에 관여한다.Xist와 roX는 모두 히스톤 수식 효소의 신병을 통해 전사의 후생적 조절에 의해 작동한다.
이관능성 RNA
이중 기능성 RNA 또는 이중 기능성 RNA는 두 가지 다른 기능을 [39][40]가진 RNA입니다.알려진 2관능성 RNA의 대부분은 단백질과 ncRNA를 모두 코드하는 mRNA이다.그러나 점점 더 많은 ncRNA가 두 가지 다른 ncRNA 범주(예: H/ACA box snoRNA와 miRNA)[41][42]로 분류된다.
2관능성 RNA의 잘 알려진 두 가지 예는 SgrS RNA와 RNAIII이다.그러나 소수의 다른 2관능성 RNA가 존재하는 것으로 알려져 있다(예: 스테로이드 수용체 활성화제/SRA,[43] VegT RNA,[44][45] Oskar RNA,[46] ENOD40,[47] p53[48] RNA 및 SR1 RNA).[49] 최근 2관능성 RNA는 Biocimie의 [50]특별호 주제이다.
호르몬으로서
특정 비코드 RNA와 호르몬 조절 경로의 제어 사이에는 중요한 연관성이 있다.드로소필라에서는 엑디손과 청소년 호르몬과 같은 호르몬이 특정 miRNA의 발현을 촉진할 수 있다.또한, 이 조절은 케노하브디티스 엘레강스 [51]발생 내의 뚜렷한 시간적 지점에서 발생한다.포유류에서 miR-206은 에스트로겐 수용체 [52]알파의 중요한 조절제이다.
비코드 RNA는 당뇨병과 [53]같은 내분비 질환뿐만 아니라 여러 내분비 기관의 발달에 중요하다.특히 MCF-7 세포주에서는 17β-에스트라디올을 첨가하면 에스트로겐 활성화 코딩 [54]유전자 근처에서 lncRNA라고 불리는 비코딩 RNA의 전사가 증가했다.
병원성 회피
C. elegans는 박테리아 [55][56]병원체의 단일 비코드 RNA에 노출된 후 병원성 회피를 배우고 유전하는 것으로 나타났다.
질병에서의 역할
단백질과 마찬가지로 체내 ncRNA 레퍼토리의 돌연변이나 불균형은 다양한 질병을 일으킬 수 있다.
암
많은 ncRNA가 암 [5]조직에서 비정상적인 발현 패턴을 보인다.여기에는 miRNA, 긴 mRNA 유사 ncRNA,[57][58] GAS5,[59] SNORD50,[60][61] 텔로머라아제 RNA 및 Y RNA가 포함된다.miRNA는 많은 단백질 코딩 [62][63]유전자의 대규모 조절에 관여하며, Y RNA는 DNA [31]복제의 시작, 텔로머라아제의 프라이머 역할을 하는 텔로머라아제 RNA, 염색체 말단에서 텔로머 영역을 확장하는 RNP(자세한 정보는 텔로미어 및 질병 참조)에 중요하다.긴 mRNA 유사 ncRNA의 직접 기능은 명확하지 않습니다.
miR-16-1과 miR-15 1차 전구체의 생식선 돌연변이는 통제 [64][65]집단에 비해 만성 림프구 백혈병 환자에서 훨씬 더 빈번한 것으로 나타났다.
has-mir-196a2와 겹치는 희귀 SNP(rs11614913)가 비소세포 [66]폐암과 관련이 있는 것으로 밝혀졌다.마찬가지로, 다수의 유방암 관련 유전자를 조절할 것으로 예측된 17개의 miRNA의 화면에서는 환자의 miR-17과 miR-30c-1에서 변이를 발견했다. 이 환자들은 BRCA1 또는 BRCA2 돌연변이의 비보균자였으며,[67] 이러한 miRNA의 변이에 의해 가족성 유방암이 유발될 가능성을 제시하였다.p53 종양 억제제는 종양의 형성과 진행을 막는데 가장 중요한 약제이다.p53 단백질은 세포 스트레스 반응을 조정하는 데 중요한 역할을 하는 전사 인자로 기능합니다.p53은 암에서 중요한 역할 외에도 당뇨병, 허혈 후 세포사망, 헌팅턴, 파킨슨, 알츠하이머와 같은 다양한 신경변성 질환과 같은 다른 질병들과 관련이 있다.연구에 따르면 p53 발현은 비부호화 [4]RNA에 의해 조절되는 것으로 나타났다.
암세포에서 조절되는 비부호화 RNA 장애의 또 다른 예는 긴 비부호화 RNA Link00707이다.Linc00707고 해면 갯솜 뼈에 miRNAs 간 세포의 carcinoma,[69]위 cancer[70]이나 유방 cancer,[71][72]에 중간엽 줄기 cells,[68]marrow-derived 것이며, 따라서 간 세포 암 진행되는 것인 기여를 확산과 전이고, 또는 간접적으로 단백질의 표현을 규제하고 골 형성을 촉진한다 upregulated. 암에 관련된각각 공격성을 나타냅니다.
프라더윌리 증후군
C/D 박스 snoRNA SNORD116의 48개 복사본이 삭제된 것이 Prader-Wili [73][74][75][76]증후군의 주요 원인인 것으로 나타났다.프레이더 윌리는 과식과 학습 장애와 관련된 발달 장애입니다.SNORD116은 다수의 단백질 코드 유전자 내에 잠재적 표적 부위를 가지며, 대체 [77]스플라이싱을 조절하는 역할을 할 수 있다.
자폐증
작은 핵소체 RNA SNORD115 유전자 클러스터를 포함하는 염색체 궤적은 자폐적 [78][79]특성을 가진 약 5%의 개인에서 복제되었다.SNORD115 클러스터를 복제하도록 설계된 마우스 모델은 자폐증과 같은 [80]행동을 나타낸다.최근 사후의 뇌조직에 대한 작은 연구는 [81]대조군과 비교하여 자폐성 뇌의 전전두피질과 소뇌에서 긴 비코드 RNA의 발현 변화를 보여주었다.
연골모발저형성증
RNAase MRP 내의 돌연변이는 작은 키, [82][83][84]희박한 머리카락, 골격 이상, 억제된 면역 체계와 같은 여러 증상과 관련된 질병인 연골-모발 저형성을 유발하는 것으로 나타났다.가장 잘 특징지어진 변이는 보존된 의사 노트의 루프 영역 2개 염기 5'에 있는 뉴클레오티드 70에서 A-G 전이이다.그러나 RNase MRP 내의 다른 많은 돌연변이들도 CHH를 유발한다.
알츠하이머병
안티센스 RNA, BACE1-AS는 반대편 가닥에서 BACE1로 전사되어 알츠하이머병 [85]환자에서 상향 조절된다.BACE1-AS는 BACE1 mRNA 안정성을 높이고 전사 후 피드포워드 메커니즘을 통해 추가 BACE1을 생성함으로써 BACE1의 발현을 조절한다.또한 동일한 메커니즘으로 노인성 플라크의 주요 성분인 베타 아밀로이드의 농도를 증가시킨다.BACE1-AS 농도는 알츠하이머 환자 및 아밀로이드 전구체 단백질 트랜스제닉 마우스에서 상승한다.
miR-96과 난청
성숙한 miR-96의 종자 영역 내 변화는 인간과 생쥐에서 상염색체 우성 진행성 난청과 관련이 있다.호모 접합 돌연변이 생쥐들은 달팽이관 반응을 보이지 않는 심각한 청각장애였다.헤테로 접합 생쥐와 인간은 점차적으로 [86][87][88]청력을 잃는다.
미토콘드리아 전이 RNA
미토콘드리아 tRNA 내의 많은 돌연변이는 MELAS 증후군, MERF 증후군, 만성 진행성 외안근염과 [89][90][91][92]같은 질병과 관련이 있다.
기능성 RNA(fRNA)와 ncRNA의 구별
과학자들은 독립형 RNA [93][94][95]전사일 수도 있고 아닐 수도 있는 RNA 수준에서 기능하는 영역을 설명하기 위해 기능성 RNA와 ncRNA를 구별하기 시작했다.이는 fRNA(리보스위치, SECIS 요소 및 기타 cis 규제 영역 등)가 ncRNA가 아님을 의미합니다.그러나 fRNA는 mRNA를 포함할 수 있는데, 이는 단백질을 코드하는 RNA이기 때문에 기능적이기 때문이다.추가로 인공적으로 진화한 RNA도 fRNA 포괄적 용어에 속한다.일부 출판물은[21] ncRNA와 fRNA가 거의 동의어에 가깝다고 기술하고 있지만, 다른 출판물은 주석이 달린 ncRNA의 상당 부분이 기능이 [8][9]없을 가능성이 높다고 지적했다.또한 단백질 코드 RNA(메신저 RNA)와의 구별은 이미 한정자 mRNA에 [96]의해 주어지기 때문에 RNA라는 용어를 사용하는 것이 제안되었다.이는 "비코딩" RNA를 코드화할 때 유전자의 애매함을 제거한다. 게다가 출판된 문헌과 [97][98][99]데이터 세트에 잘못 표기된 다수의 ncRNA가 있을 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S, Patel S, et al. (May 2005). "Transcriptional maps of 10 human chromosomes at 5-nucleotide resolution". Science. 308 (5725): 1149–54. Bibcode:2005Sci...308.1149C. doi:10.1126/science.1108625. PMID 15790807. S2CID 13047538.
- ^ ENCODE Project Consortium, Birney E, Stamatoyannopoulos JA, Dutta A, Guigó R, Gingeras TR, et al. (June 2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature. 447 (7146): 799–816. Bibcode:2007Natur.447..799B. doi:10.1038/nature05874. PMC 2212820. PMID 17571346.
- ^ Washietl S, Pedersen JS, Korbel JO, Stocsits C, Gruber AR, Hackermüller J, et al. (June 2007). "Structured RNAs in the ENCODE selected regions of the human genome". Genome Research. 17 (6): 852–64. doi:10.1101/gr.5650707. PMC 1891344. PMID 17568003.
- ^ a b Morris KV, ed. (2012). Non-coding RNAs and Epigenetic Regulation of Gene Expression: Drivers of Natural Selection. Caister Academic Press. ISBN 978-1-904455-94-3.
- ^ a b Shahrouki P, Larsson E (2012). "The non-coding oncogene: a case of missing DNA evidence?". Frontiers in Genetics. 3: 170. doi:10.3389/fgene.2012.00170. PMC 3439828. PMID 22988449.
- ^ van Bakel H, Nislow C, Blencowe BJ, Hughes TR (May 2010). Eddy SR (ed.). "Most "dark matter" transcripts are associated with known genes". PLOS Biology. 8 (5): e1000371. doi:10.1371/journal.pbio.1000371. PMC 2872640. PMID 20502517.
- ^ Hüttenhofer A, Schattner P, Polacek N (May 2005). "Non-coding RNAs: hope or hype?". Trends in Genetics. 21 (5): 289–97. doi:10.1016/j.tig.2005.03.007. PMID 15851066.
- ^ a b Brosius J (May 2005). "Waste not, want not--transcript excess in multicellular eukaryotes". Trends in Genetics. 21 (5): 287–8. doi:10.1016/j.tig.2005.02.014. PMID 15851065.
- ^ a b Palazzo AF, Lee ES (2015). "Non-coding RNA: what is functional and what is junk?". Frontiers in Genetics. 6: 2. doi:10.3389/fgene.2015.00002. PMC 4306305. PMID 25674102.
- ^ Dahm R (February 2005). "Friedrich Miescher and the discovery of DNA". Developmental Biology. 278 (2): 274–88. doi:10.1016/j.ydbio.2004.11.028. PMID 15680349.
- ^ Caspersson T, Schultz J (1939). "Pentose nucleotides in the cytoplasm of growing tissues". Nature. 143 (3623): 602–3. Bibcode:1939Natur.143..602C. doi:10.1038/143602c0. S2CID 4140563.
- ^ Crick FH (1958). "On protein synthesis". Symposia of the Society for Experimental Biology. 12: 138–63. PMID 13580867.
- ^ a b Holley RW, et al. (March 1965). "Structure of a Ribonucleic Acid". Science. 147 (3664): 1462–5. Bibcode:1965Sci...147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ a b "The Nobel Prize in Physiology or Medicine 1968". Nobel Foundation. Retrieved 2007-07-28.
- ^ Madison JT, Everett GA, Kung H (July 1966). "Nucleotide sequence of a yeast tyrosine transfer RNA". Science. 153 (3735): 531–4. Bibcode:1966Sci...153..531M. CiteSeerX 10.1.1.1001.2662. doi:10.1126/science.153.3735.531. PMID 5938777. S2CID 9265016.
- ^ Zachau HG, Dütting D, Feldmann H, Melchers F, Karau W (1966). "Serine specific transfer ribonucleic acids. XIV. Comparison of nucleotide sequences and secondary structure models". Cold Spring Harbor Symposia on Quantitative Biology. 31: 417–24. doi:10.1101/SQB.1966.031.01.054. PMID 5237198.
- ^ Dudock BS, Katz G, Taylor EK, Holley RW (March 1969). "Primary structure of wheat germ phenylalanine transfer RNA". Proceedings of the National Academy of Sciences of the United States of America. 62 (3): 941–5. Bibcode:1969PNAS...62..941D. doi:10.1073/pnas.62.3.941. PMC 223689. PMID 5257014.
- ^ Cramer F, Doepner H, Haar F VD, Schlimme E, Seidel H (December 1968). "On the conformation of transfer RNA". Proceedings of the National Academy of Sciences of the United States of America. 61 (4): 1384–91. Bibcode:1968PNAS...61.1384C. doi:10.1073/pnas.61.4.1384. PMC 225267. PMID 4884685.
- ^ Ladner JE, et al. (November 1975). "Structure of yeast phenylalanine transfer RNA at 2.5 A resolution". Proceedings of the National Academy of Sciences of the United States of America. 72 (11): 4414–8. Bibcode:1975PNAS...72.4414L. doi:10.1073/pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, et al. (January 1973). "Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain". Science. 179 (4070): 285–8. Bibcode:1973Sci...179..285K. doi:10.1126/science.179.4070.285. PMID 4566654. S2CID 28916938.
- ^ a b Eddy SR (December 2001). "Non-coding RNA genes and the modern RNA world". Nature Reviews. Genetics. 2 (12): 919–29. doi:10.1038/35103511. PMID 11733745. S2CID 18347629.
- ^ Daneholt, Bertil. "Advanced Information: RNA interference". The Nobel Prize in Physiology or Medicine 2006. Archived from the original on 2007-01-20. Retrieved 2007-01-25.
- ^ Monga I, Banerjee I (November 2019). "Computational Identification of piRNAs Using Features Based on RNA Sequence, Structure, Thermodynamic and Physicochemical Properties". Current Genomics. 20 (7): 508–518. doi:10.2174/1389202920666191129112705. PMC 7327968. PMID 32655289.
- ^ Jeffares DC, Poole AM, Penny D (January 1998). "Relics from the RNA world". Journal of Molecular Evolution. 46 (1): 18–36. Bibcode:1998JMolE..46...18J. doi:10.1007/PL00006280. PMID 9419222. S2CID 2029318.
- ^ Poole AM, Jeffares DC, Penny D (January 1998). "The path from the RNA world". Journal of Molecular Evolution. 46 (1): 1–17. Bibcode:1998JMolE..46....1P. doi:10.1007/PL00006275. PMID 9419221. S2CID 17968659.
- ^ Poole A, Jeffares D, Penny D (October 1999). "Early evolution: prokaryotes, the new kids on the block". BioEssays. 21 (10): 880–9. doi:10.1002/(SICI)1521-1878(199910)21:10<880::AID-BIES11>3.0.CO;2-P. PMID 10497339.
- ^ Ban N, Nissen P, Hansen J, Moore PB, Steitz TA (August 2000). "The complete atomic structure of the large ribosomal subunit at 2.4 A resolution". Science. 289 (5481): 905–20. Bibcode:2000Sci...289..905B. CiteSeerX 10.1.1.58.2271. doi:10.1126/science.289.5481.905. PMID 10937989.
- ^ Zhu Y, Stribinskis V, Ramos KS, Li Y (May 2006). "Sequence analysis of RNase MRP RNA reveals its origination from eukaryotic RNase P RNA". RNA. 12 (5): 699–706. doi:10.1261/rna.2284906. PMC 1440897. PMID 16540690.
- ^ Kishore S, Stamm S (January 2006). "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C". Science. 311 (5758): 230–2. Bibcode:2006Sci...311..230K. doi:10.1126/science.1118265. PMID 16357227. S2CID 44527461.
- ^ Stein AJ, Fuchs G, Fu C, Wolin SL, Reinisch KM (May 2005). "Structural insights into RNA quality control: the Ro autoantigen binds misfolded RNAs via its central cavity". Cell. 121 (4): 529–39. doi:10.1016/j.cell.2005.03.009. PMC 1769319. PMID 15907467.
- ^ a b Christov CP, Gardiner TJ, Szüts D, Krude T (September 2006). "Functional requirement of noncoding Y RNAs for human chromosomal DNA replication". Molecular and Cellular Biology. 26 (18): 6993–7004. doi:10.1128/MCB.01060-06. PMC 1592862. PMID 16943439.
- ^ Zhang AT, Langley AR, Christov CP, Kheir E, Shafee T, Gardiner TJ, Krude T (June 2011). "Dynamic interaction of Y RNAs with chromatin and initiation proteins during human DNA replication". Journal of Cell Science. 124 (Pt 12): 2058–69. doi:10.1242/jcs.086561. PMC 3104036. PMID 21610089.
- ^ Hall AE, Turnbull C, Dalmay T (April 2013). "Y RNAs: recent developments". Biomolecular Concepts. 4 (2): 103–10. doi:10.1515/bmc-2012-0050. PMID 25436569. S2CID 12575326.
- ^ Lerner MR, Boyle JA, Hardin JA, Steitz JA (January 1981). "Two novel classes of small ribonucleoproteins detected by antibodies associated with lupus erythematosus". Science. 211 (4480): 400–2. Bibcode:1981Sci...211..400L. doi:10.1126/science.6164096. PMID 6164096.
- ^ Reiner R, Ben-Asouli Y, Krilovetzky I, Jarrous N (June 2006). "A role for the catalytic ribonucleoprotein RNase P in RNA polymerase III transcription". Genes & Development. 20 (12): 1621–35. doi:10.1101/gad.386706. PMC 1482482. PMID 16778078.
- ^ Espinoza CA, Allen TA, Hieb AR, Kugel JF, Goodrich JA (September 2004). "B2 RNA binds directly to RNA polymerase II to repress transcript synthesis". Nature Structural & Molecular Biology. 11 (9): 822–9. doi:10.1038/nsmb812. PMID 15300239. S2CID 22199826.
- ^ Hirota K, Miyoshi T, Kugou K, Hoffman CS, Shibata T, Ohta K (November 2008). "Stepwise chromatin remodelling by a cascade of transcription initiation of non-coding RNAs". Nature. 456 (7218): 130–4. Bibcode:2008Natur.456..130H. doi:10.1038/nature07348. PMID 18820678. S2CID 4416402.
- ^ Park Y, Kelley RL, Oh H, Kuroda MI, Meller VH (November 2002). "Extent of chromatin spreading determined by roX RNA recruitment of MSL proteins". Science. 298 (5598): 1620–3. Bibcode:2002Sci...298.1620P. doi:10.1126/science.1076686. PMID 12446910. S2CID 27167367.
- ^ Wadler CS, Vanderpool CK (December 2007). "A dual function for a bacterial small RNA: SgrS performs base pairing-dependent regulation and encodes a functional polypeptide". Proceedings of the National Academy of Sciences of the United States of America. 104 (51): 20454–9. Bibcode:2007PNAS..10420454W. doi:10.1073/pnas.0708102104. PMC 2154452. PMID 18042713.
- ^ Dinger ME, Pang KC, Mercer TR, Mattick JS (November 2008). McEntyre J (ed.). "Differentiating protein-coding and noncoding RNA: challenges and ambiguities". PLOS Computational Biology. 4 (11): e1000176. Bibcode:2008PLSCB...4E0176D. doi:10.1371/journal.pcbi.1000176. PMC 2518207. PMID 19043537.
- ^ Saraiya AA, Wang CC (November 2008). Goldberg DE (ed.). "snoRNA, a novel precursor of microRNA in Giardia lamblia". PLOS Pathogens. 4 (11): e1000224. doi:10.1371/journal.ppat.1000224. PMC 2583053. PMID 19043559.
- ^ Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N, Meister G (November 2008). "A human snoRNA with microRNA-like functions". Molecular Cell. 32 (4): 519–28. doi:10.1016/j.molcel.2008.10.017. PMID 19026782.
- ^ Leygue E (August 2007). "Steroid receptor RNA activator (SRA1): unusual bifaceted gene products with suspected relevance to breast cancer". Nuclear Receptor Signaling. 5: e006. doi:10.1621/nrs.05006. PMC 1948073. PMID 17710122.
- ^ Zhang J, King ML (December 1996). "Xenopus VegT RNA is localized to the vegetal cortex during oogenesis and encodes a novel T-box transcription factor involved in mesodermal patterning". Development. 122 (12): 4119–29. doi:10.1242/dev.122.12.4119. PMID 9012531.
- ^ Kloc M, Wilk K, Vargas D, Shirato Y, Bilinski S, Etkin LD (August 2005). "Potential structural role of non-coding and coding RNAs in the organization of the cytoskeleton at the vegetal cortex of Xenopus oocytes". Development. 132 (15): 3445–57. doi:10.1242/dev.01919. PMID 16000384.
- ^ Jenny A, Hachet O, Závorszky P, Cyrklaff A, Weston MD, Johnston DS, Erdélyi M, Ephrussi A (August 2006). "A translation-independent role of oskar RNA in early Drosophila oogenesis". Development. 133 (15): 2827–33. doi:10.1242/dev.02456. PMID 16835436.
- ^ Gultyaev AP, Roussis A (2007). "Identification of conserved secondary structures and expansion segments in enod40 RNAs reveals new enod40 homologues in plants". Nucleic Acids Research. 35 (9): 3144–52. doi:10.1093/nar/gkm173. PMC 1888808. PMID 17452360.
- ^ Candeias MM, Malbert-Colas L, Powell DJ, Daskalogianni C, Maslon MM, Naski N, Bourougaa K, Calvo F, Fåhraeus R (September 2008). "P53 mRNA controls p53 activity by managing Mdm2 functions". Nature Cell Biology. 10 (9): 1098–105. doi:10.1038/ncb1770. PMID 19160491. S2CID 5122088.
- ^ Gimpel M, Preis H, Barth E, Gramzow L, Brantl S (December 2012). "SR1--a small RNA with two remarkably conserved functions". Nucleic Acids Research. 40 (22): 11659–72. doi:10.1093/nar/gks895. PMC 3526287. PMID 23034808.
- ^ Francastel C, Hubé F (November 2011). "Coding or non-coding: Need they be exclusive?". Biochimie. 93 (11): vi–vii. doi:10.1016/S0300-9084(11)00322-1. PMID 21963143.
- ^ Sempere LF, Sokol NS, Dubrovsky EB, Berger EM, Ambros V (July 2003). "Temporal regulation of microRNA expression in Drosophila melanogaster mediated by hormonal signals and broad-Complex gene activity". Developmental Biology. 259 (1): 9–18. doi:10.1016/S0012-1606(03)00208-2. PMID 12812784.
- ^ Adams BD, Furneaux H, White BA (May 2007). "The micro-ribonucleic acid (miRNA) miR-206 targets the human estrogen receptor-alpha (ERalpha) and represses ERalpha messenger RNA and protein expression in breast cancer cell lines". Molecular Endocrinology. 21 (5): 1132–47. doi:10.1210/me.2007-0022. PMID 17312270.
- ^ Knoll M, Lodish HF, Sun L (March 2015). "Long non-coding RNAs as regulators of the endocrine system". Nature Reviews. Endocrinology. 11 (3): 151–60. doi:10.1038/nrendo.2014.229. hdl:1721.1/116703. PMC 4376378. PMID 25560704.
- ^ Li W, Notani D, Ma Q, Tanasa B, Nunez E, Chen AY, Merkurjev D, Zhang J, Ohgi K, Song X, Oh S, Kim HS, Glass CK, Rosenfeld MG (June 2013). "Functional roles of enhancer RNAs for oestrogen-dependent transcriptional activation". Nature. 498 (7455): 516–20. Bibcode:2013Natur.498..516L. doi:10.1038/nature12210. PMC 3718886. PMID 23728302.
- ^ "Researchers discover how worms pass knowledge of a pathogen to offspring". phys.org. Retrieved 11 October 2020.
- ^ Kaletsky, Rachel; Moore, Rebecca S.; Vrla, Geoffrey D.; Parsons, Lance R.; Gitai, Zemer; Murphy, Coleen T. (9 September 2020). "C. elegans interprets bacterial non-coding RNAs to learn pathogenic avoidance". Nature. 586 (7829): 445–451. Bibcode:2020Natur.586..445K. doi:10.1038/s41586-020-2699-5. ISSN 1476-4687. PMC 8547118. PMID 32908307. S2CID 221626129.
- ^ Pibouin L, Villaudy J, Ferbus D, Muleris M, Prospéri MT, Remvikos Y, Goubin G (February 2002). "Cloning of the mRNA of overexpression in colon carcinoma-1: a sequence overexpressed in a subset of colon carcinomas". Cancer Genetics and Cytogenetics. 133 (1): 55–60. doi:10.1016/S0165-4608(01)00634-3. PMID 11890990.
- ^ Fu X, Ravindranath L, Tran N, Petrovics G, Srivastava S (March 2006). "Regulation of apoptosis by a prostate-specific and prostate cancer-associated noncoding gene, PCGEM1". DNA and Cell Biology. 25 (3): 135–41. doi:10.1089/dna.2006.25.135. PMID 16569192.
- ^ Mourtada-Maarabouni M, Pickard MR, Hedge VL, Farzaneh F, Williams GT (January 2009). "GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer". Oncogene. 28 (2): 195–208. doi:10.1038/onc.2008.373. PMID 18836484.
- ^ Dong XY, Guo P, Boyd J, Sun X, Li Q, Zhou W, Dong JT (August 2009). "Implication of snoRNA U50 in human breast cancer". Journal of Genetics and Genomics = Yi Chuan Xue Bao. 36 (8): 447–54. doi:10.1016/S1673-8527(08)60134-4. PMC 2854654. PMID 19683667.
- ^ Christov CP, Trivier E, Krude T (March 2008). "Noncoding human Y RNAs are overexpressed in tumours and required for cell proliferation". British Journal of Cancer. 98 (5): 981–8. doi:10.1038/sj.bjc.6604254. PMC 2266855. PMID 18283318.
- ^ Farh KK, Grimson A, Jan C, Lewis BP, Johnston WK, Lim LP, Burge CB, Bartel DP (December 2005). "The widespread impact of mammalian MicroRNAs on mRNA repression and evolution". Science. 310 (5755): 1817–21. Bibcode:2005Sci...310.1817F. doi:10.1126/science.1121158. PMID 16308420. S2CID 1849875.
- ^ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs". Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Calin GA, Ferracin M, Cimmino A, Di Leva G, Shimizu M, Wojcik SE, Iorio MV, Visone R, Sever NI, Fabbri M, Iuliano R, Palumbo T, Pichiorri F, Roldo C, Garzon R, Sevignani C, Rassenti L, Alder H, Volinia S, Liu CG, Kipps TJ, Negrini M, Croce CM (October 2005). "A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia". The New England Journal of Medicine. 353 (17): 1793–801. doi:10.1056/NEJMoa050995. PMID 16251535.
- ^ Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K, Rassenti L, Kipps T, Negrini M, Bullrich F, Croce CM (November 2002). "Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia". Proceedings of the National Academy of Sciences of the United States of America. 99 (24): 15524–9. Bibcode:2002PNAS...9915524C. doi:10.1073/pnas.242606799. PMC 137750. PMID 12434020.
- ^ Hu Z, Chen J, Tian T, Zhou X, Gu H, Xu L, Zeng Y, Miao R, Jin G, Ma H, Chen Y, Shen H (July 2008). "Genetic variants of miRNA sequences and non-small cell lung cancer survival". The Journal of Clinical Investigation. 118 (7): 2600–8. doi:10.1172/JCI34934. PMC 2402113. PMID 18521189.
- ^ Shen J, Ambrosone CB, Zhao H (March 2009). "Novel genetic variants in microRNA genes and familial breast cancer". International Journal of Cancer. 124 (5): 1178–82. doi:10.1002/ijc.24008. PMID 19048628. S2CID 20960029.
- ^ Jia, Bo; Wang, Zhiping; Sun, Xiang; Chen, Jun; Zhao, Jianjiang; Qiu, Xiaoling (December 2019). "Long noncoding RNA LINC00707 sponges miR-370-3p to promote osteogenesis of human bone marrow-derived mesenchymal stem cells through upregulating WNT2B". Stem Cell Research & Therapy. 10 (1): 67. doi:10.1186/s13287-019-1161-9. ISSN 1757-6512. PMC 6387535. PMID 30795799.
- ^ Tu, Jianfei; Zhao, Zhongwei; Xu, Min; Chen, Minjiang; Weng, Qiaoyou; Wang, Jiangmei; Ji, Jiansong (July 2019). "LINC00707 contributes to hepatocellular carcinoma progression via sponging miR‐206 to increase CDK14". Journal of Cellular Physiology. 234 (7): 10615–10624. doi:10.1002/jcp.27737. ISSN 0021-9541. PMID 30488589. S2CID 54119752.
- ^ Xie, Min; Ma, Tianshi; Xue, Jiangyang; Ma, Hongwei; Sun, Ming; Zhang, Zhihong; Liu, Minjuan; Liu, Yinghua; Ju, Songwen; Wang, Zhaoxia; De, Wei (February 2019). "The long intergenic non-protein coding RNA 707 promotes proliferation and metastasis of gastric cancer by interacting with mRNA stabilizing protein HuR". Cancer Letters. 443: 67–79. doi:10.1016/j.canlet.2018.11.032. PMID 30502359. S2CID 54611497.
- ^ Li, Tong; Li, Yunpeng; Sun, Hongyan (2019-06-06). "MicroRNA-876 is sponged by long noncoding RNA LINC00707 and directly targets metadherin to inhibit breast cancer malignancy". Cancer Management and Research. 11: 5255–5269. doi:10.2147/cmar.s210845. PMC 6559252. PMID 31239777.
- ^ Yuan, R.-X.; Bao, D.; Zhang, Y. (May 2020). "Linc00707 promotes cell proliferation, invasion, and migration via the miR-30c/CTHRC1 regulatory loop in breast cancer". European Review for Medical and Pharmacological Sciences. 24 (9): 4863–4872. doi:10.26355/eurrev_202005_21175. ISSN 1128-3602. PMID 32432749.
- ^ Sahoo T, del Gaudio D, German JR, Shinawi M, Peters SU, Person RE, Garnica A, Cheung SW, Beaudet AL (June 2008). "Prader-Willi phenotype caused by paternal deficiency for the HBII-85 C/D box small nucleolar RNA cluster". Nature Genetics. 40 (6): 719–21. doi:10.1038/ng.158. PMC 2705197. PMID 18500341.
- ^ Skryabin BV, Gubar LV, Seeger B, Pfeiffer J, Handel S, Robeck T, Karpova E, Rozhdestvensky TS, Brosius J (December 2007). "Deletion of the MBII-85 snoRNA gene cluster in mice results in postnatal growth retardation". PLOS Genetics. 3 (12): e235. doi:10.1371/journal.pgen.0030235. PMC 2323313. PMID 18166085.
- ^ Ding F, Li HH, Zhang S, Solomon NM, Camper SA, Cohen P, Francke U (March 2008). Akbarian S (ed.). "SnoRNA Snord116 (Pwcr1/MBII-85) deletion causes growth deficiency and hyperphagia in mice". PLOS ONE. 3 (3): e1709. Bibcode:2008PLoSO...3.1709D. doi:10.1371/journal.pone.0001709. PMC 2248623. PMID 18320030.
- ^ Ding F, Prints Y, Dhar MS, Johnson DK, Garnacho-Montero C, Nicholls RD, Francke U (June 2005). "Lack of Pwcr1/MBII-85 snoRNA is critical for neonatal lethality in Prader-Willi syndrome mouse models". Mammalian Genome. 16 (6): 424–31. doi:10.1007/s00335-005-2460-2. PMID 16075369. S2CID 12256515.
- ^ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A (January 2008). "snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions". Gene. 408 (1–2): 172–9. doi:10.1016/j.gene.2007.10.037. PMC 6800007. PMID 18160232.
- ^ Bolton PF, Veltman MW, Weisblatt E, Holmes JR, Thomas NS, Youings SA, Thompson RJ, Roberts SE, Dennis NR, Browne CE, Goodson S, Moore V, Brown J (September 2004). "Chromosome 15q11-13 abnormalities and other medical conditions in individuals with autism spectrum disorders". Psychiatric Genetics. 14 (3): 131–7. doi:10.1097/00041444-200409000-00002. PMID 15318025. S2CID 37344935.
- ^ Cook EH, Scherer SW (October 2008). "Copy-number variations associated with neuropsychiatric conditions". Nature. 455 (7215): 919–23. Bibcode:2008Natur.455..919C. doi:10.1038/nature07458. PMID 18923514. S2CID 4377899.
- ^ Nakatani J, Tamada K, Hatanaka F, Ise S, Ohta H, Inoue K, Tomonaga S, Watanabe Y, Chung YJ, Banerjee R, Iwamoto K, Kato T, Okazawa M, Yamauchi K, Tanda K, Takao K, Miyakawa T, Bradley A, Takumi T (June 2009). "Abnormal behavior in a chromosome-engineered mouse model for human 15q11-13 duplication seen in autism". Cell. 137 (7): 1235–46. doi:10.1016/j.cell.2009.04.024. PMC 3710970. PMID 19563756.
- ^ Ziats MN, Rennert OM (March 2013). "Aberrant expression of long noncoding RNAs in autistic brain". Journal of Molecular Neuroscience. 49 (3): 589–93. doi:10.1007/s12031-012-9880-8. PMC 3566384. PMID 22949041.
- ^ Ridanpää M, van Eenennaam H, Pelin K, Chadwick R, Johnson C, Yuan B, vanVenrooij W, Pruijn G, Salmela R, Rockas S, Mäkitie O, Kaitila I, de la Chapelle A (January 2001). "Mutations in the RNA component of RNase MRP cause a pleiotropic human disease, cartilage-hair hypoplasia". Cell. 104 (2): 195–203. doi:10.1016/S0092-8674(01)00205-7. PMID 11207361. S2CID 13977736.
- ^ Martin AN, Li Y (March 2007). "RNase MRP RNA and human genetic diseases". Cell Research. 17 (3): 219–26. doi:10.1038/sj.cr.7310120. PMID 17189938.
- ^ Kavadas FD, Giliani S, Gu Y, Mazzolari E, Bates A, Pegoiani E, Roifman CM, Notarangelo LD (December 2008). "Variability of clinical and laboratory features among patients with ribonuclease mitochondrial RNA processing endoribonuclease gene mutations". The Journal of Allergy and Clinical Immunology. 122 (6): 1178–84. doi:10.1016/j.jaci.2008.07.036. PMID 18804272.
- ^ Faghihi MA, Modarresi F, Khalil AM, Wood DE, Sahagan BG, Morgan TE, Finch CE, St Laurent G, Kenny PJ, Wahlestedt C (July 2008). "Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase". Nature Medicine. 14 (7): 723–30. doi:10.1038/nm1784. PMC 2826895. PMID 18587408.
- ^ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (May 2009). "Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss". Nature Genetics. 41 (5): 609–13. doi:10.1038/ng.355. PMID 19363479. S2CID 11113852.
- ^ Lewis MA, Quint E, Glazier AM, Fuchs H, De Angelis MH, Langford C, van Dongen S, Abreu-Goodger C, Piipari M, Redshaw N, Dalmay T, Moreno-Pelayo MA, Enright AJ, Steel KP (May 2009). "An ENU-induced mutation of miR-96 associated with progressive hearing loss in mice". Nature Genetics. 41 (5): 614–8. doi:10.1038/ng.369. PMC 2705913. PMID 19363478.
- ^ Soukup GA (June 2009). "Little but loud: small RNAs have a resounding affect on ear development". Brain Research. 1277: 104–14. doi:10.1016/j.brainres.2009.02.027. PMC 2700218. PMID 19245798.
- ^ Taylor, RW; Turnbull, DM (May 2005). "Mitochondrial DNA mutations in human disease". Nature Reviews. Genetics. 6 (5): 389–402. doi:10.1038/nrg1606. PMC 1762815. PMID 15861210.
- ^ Yarham, JW; Elson, JL; Blakely, EL; McFarland, R; Taylor, RW (September 2010). "Mitochondrial tRNA mutations and disease". Wiley Interdisciplinary Reviews: RNA. 1 (2): 304–24. doi:10.1002/wrna.27. PMID 21935892. S2CID 43123827.
- ^ Zifa, E; Giannouli, S; Theotokis, P; Stamatis, C; Mamuris, Z; Stathopoulos, C (January 2007). "Mitochondrial tRNA mutations: clinical and functional perturbations". RNA Biology. 4 (1): 38–66. doi:10.4161/rna.4.1.4548. PMID 17617745. S2CID 11965790.
- ^ Abbott, JA; Francklyn, CS; Robey-Bond, SM (2014). "Transfer RNA and human disease". Frontiers in Genetics. 5: 158. doi:10.3389/fgene.2014.00158. PMC 4042891. PMID 24917879.
- ^ Carter RJ, Dubchak I, Holbrook SR (October 2001). "A computational approach to identify genes for functional RNAs in genomic sequences". Nucleic Acids Research. 29 (19): 3928–38. doi:10.1093/nar/29.19.3928. PMC 60242. PMID 11574674.
- ^ Pedersen JS, Bejerano G, Siepel A, Rosenbloom K, Lindblad-Toh K, Lander ES, Kent J, Miller W, Haussler D (April 2006). "Identification and classification of conserved RNA secondary structures in the human genome". PLOS Computational Biology. 2 (4): e33. Bibcode:2006PLSCB...2...33P. doi:10.1371/journal.pcbi.0020033. PMC 1440920. PMID 16628248.
- ^ Thomas JM, Horspool D, Brown G, Tcherepanov V, Upton C (January 2007). "GraphDNA: a Java program for graphical display of DNA composition analyses". BMC Bioinformatics. 8: 21. doi:10.1186/1471-2105-8-21. PMC 1783863. PMID 17244370.
- ^ Brosius J, Raabe CA (February 2015). "What is an RNA? A top layer for RNA classification". RNA Biology. 13 (2): 140–4. doi:10.1080/15476286.2015.1128064. PMC 4829331. PMID 26818079.
- ^ Ji, Zhe; Song, Ruisheng; Regev, Aviv; Struhl, Kevin (2015-12-19). "Many lncRNAs, 5'UTRs, and pseudogenes are translated and some are likely to express functional proteins". eLife. 4: e08890. doi:10.7554/eLife.08890. ISSN 2050-084X. PMC 4739776. PMID 26687005.
- ^ Tosar, Juan Pablo; Rovira, Carlos; Cayota, Alfonso (2018-01-22). "Non-coding RNA fragments account for the majority of annotated piRNAs expressed in somatic non-gonadal tissues". Communications Biology. 1 (1): 2. doi:10.1038/s42003-017-0001-7. ISSN 2399-3642. PMC 6052916. PMID 30271890.
- ^ Housman, Gali; Ulitsky, Igor (January 2016). "Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs". Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. 1859 (1): 31–40. doi:10.1016/j.bbagrm.2015.07.017. ISSN 0006-3002. PMID 26265145.
외부 링크
(웨이백 머신 복사)
- Rfam Database - 수백 개의 관련 ncRNA 패밀리의 큐레이티드
- NONCODE.org : 모든 종류의 비코딩 RNA 무료 데이터베이스(tRNA 및 rRNA 제외)
- ncRNA BMC Genomics 2014 RNAcon 예측 및 분류, 15:127
- ENCODE 스레드 탐색기 비코딩 RNA 특성화.자연 (저널)
- Non-Coding RNA Databases Resource (NRDR) - 인터넷을 통해 이용 가능한 비코딩 RNA 데이터베이스와 관련된 데이터의 큐레이티드 소스
- DASHR - 코드화되지 않은 소형 RNA의 데이터베이스 2018