X-비활성화
X-inactivationX-불활성화(영국의 유전학자 메리 라이온의 이름을 따서 라이온화라고도 함)는 X 염색체의 복사본 중 하나가 암컷 포유동물에서 불활성화되는 과정이다.불활성 X염색체는 헤테로크로마틴이라고 불리는 전사 불활성 구조로 포장됨으로써 침묵된다.거의 모든 암컷 포유동물이 두 개의 X염색체를 가지고 있기 때문에, X불활성화는 X염색체의 단일 복사본만을 가지고 있는 수컷보다 X염색체 유전자 생산물을 두 배나 더 많이 가지고 있는 것을 막는다.
특정 배아 세포에서 어떤 X염색체가 불활성화 될지는 사람과 같은 태반 포유동물에서 무작위로 선택되지만, 일단 X염색체가 불활성화되면 그것은 세포와 유기체 내 그 후손들의 평생 동안 불활성화 될 것이다.그 결과 유기체의 모든 세포에서 불활성화 X염색체의 선택은 무작위 분포이며, 종종 약 절반의 세포는 부성 X염색체를 가지고 있고 절반은 불활성화 모성 X염색체를 가지고 있다. 그러나 일반적으로 X불활성화는 한 유기체 내에서 세포주 전체에 불균등하게 분포되어 있다.X-비활성화).
태반 포유동물에서의 무작위 X-불활성화와 달리 유대류에서의 불활성화는 부계유래 X염색체에만 적용된다.
메커니즘
설치류 X염색체 활성화 주기
아래 단락은 설치류에만 관련이 있으며 대부분의 포유동물에서 XI를 반영하지 않습니다.X-불활성화는 여성 생활 전반에 걸쳐 X 염색체의 활성화 주기의 일부이다.난자와 수정 접합자는 처음에는 모체 전사를 사용하며 접합 유전자가 활성화될 때까지 전체 배아 게놈은 침묵한다.그 후, 모든 마우스 세포는 4~8개의 세포 단계 [3][4][5][6]배아에서 부계 유래 X염색체의 초기 각인 불활성화를 겪는다.태아외 조직(태반과 배아를 지탱하는 다른 조직을 발생시키는)은 이러한 조기 각인 불활성화를 유지하며, 따라서 모성 X 염색체만이 이러한 조직들에서 활동합니다.
초기 배반포에서, 이 초기, 각인된 X-불활성화는 내부 세포 덩어리의 세포에서 역전되고, 이러한 세포에서 두 X 염색체는 다시 활성화된다.각각의 세포들은 독립적으로 그리고 무작위로 X [5]염색체의 한 복사본을 비활성화한다.이 불활성화 사건은 생식선을 제외하고 개인의 수명 동안 돌이킬 수 없다.감수분열 전 여성배아라인에서는 X-불활성화가 역전되어 감수분열 후 모든 단수성 난모세포가 단일 활성 X염색체를 포함한다.
개요
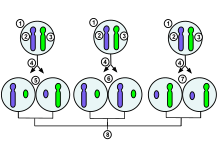
Xi는 비활성, Xa는 활성 X 염색체입니다.X는PM 부계, X to는 모계 X염색체를 나타낸다.X를 운반하는M 난자가 Y나 X를 운반하는P 정자에 의해 수정되면 이배체 접합자가 형성된다.접합자부터 성인 단계, 그리고 다음 세대의 난자에 이르기까지, X 염색체는 다음과 같은 변화를 겪습니다.
- XiPM Xi 접합자 → 접합자 게놈 활성화 진행 중, 다음과 같은 결과를 초래합니다.
- XaP XaM → 임프린트(임프린트) X 불활성화가 진행되어 다음과 같은 결과를 초래합니다.
- XiP XaM → 초기 배반포 단계에서 X-활성화가 진행되어 다음과 같은 결과를 초래합니다.
- XaP XaM → 배반포 단계에서 배아 혈통(세포 질량)에서 무작위 X-불활성화 과정을 거치면서 다음과 같은 결과를 초래한다.
- XiPM Xa ORP XaM Xi → 감수분열 전 원시 생식세포에서 X-재활성화가 진행되어 다음과 같은 결과를 초래한다.
- XaM XaP 이배체 생식세포가 감수생식 정지 상태야감수분열 I은 배란으로 끝나기 때문에 인간의 생식세포는 발달 첫 주부터 사춘기까지 이 단계에서 존재한다.감수 분열의 완성은 다음과 같이 이어진다.
- XaM AND XaP 반배체배아세포(란).
X 활성화 주기는 생쥐에게 가장 잘 연구되었지만, 인간에게는 여러 가지 연구가 있다.대부분의 증거가 생쥐에서 나오기 때문에 위의 계획은 생쥐의 사건을 나타낸다.감수분열의 완성은 명확성을 위해 여기서 단순화된다.1~4단계는 시험관내 수정 배아 및 줄기세포 분화에서 연구할 수 있다. X-활성화는 발달 중인 배아에서 발생하고 이후 (6~7)단계는 암컷 몸 안에서 이루어지기 때문에 연구하기가 훨씬 어렵다.
타이밍.
각 과정의 시기는 종에 따라 다르며, 많은 경우 정확한 시간은 활발하게 논의됩니다.[이 표의 X-활성화 인간 타이밍의 전체 부분은 매우 의심스러우며 경험적 데이터에 의해 적절히 입증될 때까지 제거되어야 한다]
| 과정 | 마우스 | 인간 | |
| 1 | 접합체유전체활성화 | 2[7] ~ 4 셀 스테이지 | 2[7]~8셀 스테이지 |
| 2 | 임프린트(모성) X-비활성화 | 4~8셀[6][8] 스테이지 | 사람에게서[9] 발생하는지는 불명확하다. |
| 3 | X활성화 | 배반포 초기 단계 | 배반포 초기 단계 |
| 4 | 태아 계통의 랜덤 X 불활성화(내세포 질량) | 후기 배반포 | 착상[9] 후 배반포 말기 |
| 5 | 감수분열 전 원시배아세포의 X-재활성화 | 개발 4주차부터 14주차까지[10][11] |
셀 세대 간 비활성화 상태 상속
특정 X염색체를 불활성화한 각 세포의 후손들도 같은 염색체를 불활성화시킬 것이다.암컷이 X-연결 유전자에 대해 헤테로 접합되어 있을 때 거북이의 색칠에서 관찰될 수 있는 이 현상은 모자이즘과 혼동되어서는 안 된다. 모자이즘은 같은 개체에서 다양한 세포 집단의 유전자형 차이를 구체적으로 언급하는 용어이다; X-불활성화는 후생유전학적 변화이다.다른 표현형의 궤양은 유전자형 수준의 변화가 아니다.따라서 개별 세포나 혈통의 경우 불활성화는 치우치거나 무작위적이지 않으며, 이는 X-연계 유전 [12]장애의 여성 '캐리어'에서 가벼운 증상을 일으킬 수 있다.
활성 X 염색체 1개 선택
전형적인 암컷은 두 개의 X 염색체를 가지고 있으며, 주어진 세포에서 한 개의 염색체는 활성(Xa로 지정)이고 한 개의 염색체는 비활성(Xi)일 것이다.하지만, X 염색체를 추가로 복제한 개인들에 대한 연구는 두 개 이상의 X 염색체를 가진 세포에는 여전히 Xa가 하나 있고, 나머지 X 염색체들은 모두 불활성화 되어 있다는 것을 보여준다.이것은 여성의 X 염색체의 기본 상태가 불활성화이지만, 항상 하나의 X 염색체가 활성 상태로 유지되도록 선택된다는 것을 나타냅니다.
X염색체 불활성화는 상피세포(배아를 낳는 세포)에서 위경화가 일어날 때 일어나는 무작위 과정으로 이해된다.모체 X염색체와 부성 X염색체는 불활성화 확률이 동일하다.이것은 여성이 X염색체를 두 개 가지고 있는 반면 남성은 하나만 가지고 있기 때문에 X염색체를 가지고 있는 경우 남성보다 약 50% 더 자주 X연계 장애를 겪을 것으로 예상되지만, 실제로 여성에게서 이러한 장애의 발생은 그보다 훨씬 더 낮다.이러한 불균형에 대한 한 가지 설명은 불활성화 X 염색체 상의 12-20%의 유전자가 발현된 상태로 유지되어 X염색체에 의해 코드된 결함 유전자에 대한 추가적인 보호를 제공한다는 것이다.일부에서는[who?] 이러한 차이가 우선적(랜덤하지 않은) 불활성화의 증거가 되어야 한다고 제안합니다.부계 X염색체의 우선 불활성화는 유대류 및 [14]배아를 둘러싼 막을 형성하는 세포 계통 모두에서 일어나는 반면 태반 포유류에서는 모계 또는 모계 유래 X염색체가 다른 세포 [15]계통에서 불활성화될 수 있다.
X염색체 불활성화 기간이 이 차이를 설명한다.불활성화는 위배 중에 상피세포에서 일어나 [16]배아를 일으킨다.불활성화는 세포 수준에서 일어나 모자이크 발현을 초래하며, 세포 조각은 비활성 모성 X염색체를 가지며, 다른 조각은 비활성 부성 X염색체를 가진다.예를 들어, 혈우병에 대한 헤테로 접합 여성(X-연계 질환)은 간세포의 약 절반이 정상적으로 기능할 것이며, 이는 일반적으로 정상적인 혈액 [17][18]응고를 보장하기에 충분하다.우연은 훨씬 더 많은 기능 장애가 있는 세포를 야기할 수 있지만, 그러한 통계적 극단성은 거의 없다.염색체의 유전적 차이는 또한 하나의 X염색체가 불활성화를 겪을 가능성을 더 높인다.또한 X염색체가 그 성장을 방해하거나 그것을 생존할 수 없게 만드는 돌연변이를 가지고 있다면, X염색체가 무작위로 불활성화된 세포는 정상 대립 유전자를 무작위로 불활성화한 세포보다 선택적으로 유리할 것이다.따라서, 불활성화는 처음에는 무작위이지만, 정상 대립 유전자를 불활성화하는 세포는 결국 과잉 성장하여 거의 모든 세포가 동일한 X-염색체를 [17]활성화하는 기능적으로 정상적인 세포로 대체될 것이다.
X염색체에 결합해 불활성화를 막는 자가암호화 차단인자가 있다는 가설이[19] 있다.모델은 제한 차단 인자가 있다고 가정하고, 따라서 사용 가능한 차단 인자 분자가 하나의 X 염색체에 결합하면 나머지 X 염색체는 불활성화로부터 보호되지 않는다.이 모델은 많은 X염색체를 가진 세포에서 단일 Xa의 존재와 정상 수의 두 배의 자동 [20]염색체를 가진 세포 선에서 두 개의 활성 X염색체의 존재로 뒷받침된다.
X 염색체에 존재하는 X 불활성화 중심(XIC)의 시퀀스는 X 염색체의 침묵을 제어합니다.가상의 차단 인자는 XIC 내의 시퀀스에 결합할 것으로 예측됩니다.
헤테로 접합 여성의 X-연계 장애 발현
암컷 X 헤테로 접합의 효과는 칼리코 고양이의 독특한 털 패턴과 같은 일부 국소적 특성에서 뚜렷하게 나타납니다.그러나 질병의 발현과 같이 이러한 여성들에게서 국소화되지 않은 특성의 발현을 완전히 이해하는 것은 더 어려울 수 있다.
남성은 X염색체의 복사본 하나만 가지고 있기 때문에, 발현된 모든 X염색체 유전자(또는 모집단의 특정 유전자에 대한 여러 변종 형태의 경우 대립 유전자)는 그 염색체의 복사본에 위치한다.그러나 암컷은 주로 활성 상태로 유지되는 X염색체 복사본에 위치한 유전자나 대립 유전자를 발현합니다.특정 표현형에 개인 차이를 일으키는 한 유전자 또는 여러 유전자의 상황을 고려한다면(즉, 그 표현형에 대한 모집단에서 관찰된 변화를 유발한다), 동종 접합 여성의 경우 두 가지 복제의 대립 유전자가 동일하기 때문에 염색체의 어떤 복사가 활성화되지 않은지는 특별히 중요하지 않다.그러나 원인 유전자에 헤테로 접합된 여성의 경우 염색체의 한 복사본이 다른 복사본에 비해 비활성화되는 것은 표현형 가치에 직접적인 영향을 미칠 수 있다.이러한 현상으로 인해 해당 유전자 또는 해당 [21]유전자에 동종 접합을 하는 암컷보다 관련 유전자에 이형 접합을 하는 암컷에서 표현형 변이의 증가가 관찰된다.표현형 변이가 발현될 수 있는 많은 다른 방법들이 있다.많은 경우에, 헤테로 접합 여성은 증상이 없거나 X-연결 아드레날레우키오디스트로피와 [22]같은 특정 장애의 경미한 증상만 보일 수 있다.
헤테로 접합 암컷에서 표현형의 분화는 X-불활성 스큐잉의 존재에 의해 더욱 심해진다.일반적으로 각 X염색체는 세포의 절반에서 무음화되지만 염색체의 선택적 불활성화가 발생하면 이 과정이 왜곡된다.변형이 일어나는 것은 우연히 또는 바람직하지 않은 [23][24]돌연변이와 같이 염색체를 다소 자주 침묵시키는 염색체의 물리적 특성에 의해 일어나는 것으로 생각된다.
평균적으로, 각 X 염색체는 세포의 절반에서 불활성화되지만, "분명히 정상적인" 여성의 5-20%는 [23]X 불활성화 왜곡을 보인다.스큐잉이 존재하는 경우 광범위한 증상 표현이 발생할 수 있으며, 그 결과 스큐잉 비율에 따라 경미함에서 중대함까지 표현이 달라집니다.일란성 여성 쌍둥이가 멩크스병(X-연계 질환)의 발현에 있어 극단적 편차를 보여 한쪽 쌍둥이는 사망했고 다른 한쪽은 무증상으로 [25]남아 있었다.
X 불활성화 왜곡은 불활성화를 일으키는 메커니즘의 문제 또는 염색체 자체의 [23][24]문제로 인해 발생할 수 있다고 생각된다.그러나 표현형과 스큐잉 사이의 연관성은 여전히 의문시되고 있으며 사례별로 검토해야 한다.Duchenne과 Becker 근이영양증(DMD)에 대해 헤테로 접합된 증상 및 무증상 여성 모두를 조사한 연구는 전사 발현과 왜곡된 X-불활성화 사이에 뚜렷한 연관성을 발견하지 못했다.이 연구는 두 메커니즘이 독립적으로 조절되며,[26] 다른 알려지지 않은 요소들이 작용하고 있다는 것을 알 수 있습니다.
염색체 성분
X 염색체의 X 불활성화 중심(또는 단순히 XIC)은 X 불활성화를 일으키기에 필요하고 충분합니다.XIC를 자동 염색체상에 배치하는 염색체 전위는 자동 염색체의 불활성화를 가져오며, XIC가 결여된 X염색체는 [27][28]불활성화되지 않는다.
XIC는 X-불활성화에 관여하는 4개의 비번역 RNA 유전자 Xist, Tsix, Jpx 및 Ftx를 포함한다.XIC는 또한 알려진 조절 [29]단백질과 알려지지 않은 조절 단백질 모두에 대한 결합 부위를 포함합니다.
Xist 및 Tsix RNA
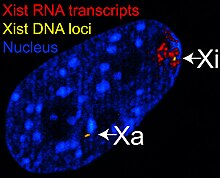
X-비활성 특이 전사체(Xist) 유전자는 그것이 [30]전사되는 X염색체의 특이적 사일런싱을 매개하는 역할을 하는 큰 비코드 RNA를 암호화한다.비활성 X 염색체는 Xist [31]RNA에 의해 코팅되지만 Xa는 코팅되지 않습니다(오른쪽 그림 참조).Xist 유전자가 없는 X염색체는 [32]불활성화될 수 없다.Xist 유전자를 다른 염색체에 인위적으로 배치하고 발현시키는 것은 [33][27]그 염색체의 침묵으로 이어진다.
불활성화되기 전에, 두 X 염색체는 Xist 유전자의 Xist RNA를 약하게 발현시켰다.불활성화 과정 동안 미래의 Xa는 Xist 발현을 멈추는 반면, 미래의 Xi는 Xist RNA 생산을 극적으로 증가시킨다.미래의 Xi에서는 Xist RNA가 XIC에서 [33]확산되어 염색체를 점진적으로 코팅합니다. Xist RNA는 Xa에 국소화되지 않습니다.Xi를 따라 유전자의 침묵은 Xist RNA에 의해 코팅된 직후에 일어난다.
Xist와 같이, Tsix 유전자는 단백질을 암호화하는 것으로 믿어지지 않는 큰 RNA를 암호화한다.Tsix RNA는 Xist로 전사되는데, 이는 Tsix 유전자가 Xist 유전자와 겹치고 Xist [28]유전자의 반대쪽 DNA 가닥에 전사된다는 것을 의미합니다.Tsix는 Xist의 음의 조절제이다; Tsix 발현이 결여된 X염색체는 일반 염색체보다 훨씬 더 자주 비활성화된다.
Xist와 같이, 불활성화되기 전에, 두 X염색체는 Tsix 유전자의 Tsix RNA를 약하게 발현한다.X-불활성화가 시작되면 미래의 Xi는 Tsix RNA 발현을 멈추는 반면 Xa는 며칠 동안 Tsix 발현을 계속한다.
Rep A는 X 불활성화를 위해 또 다른 긴 비코딩 RNA인 Xist와 함께 작동하는 긴 비코딩 RNA입니다.A의원은 Xite의 발현 제거와 함께 Xist의 안티센스인 Tsix의 기능을 억제한다.PRC2를 끌어들여 X염색체 [29]중 하나를 불활성화시킴으로써 Tsix 영역의 메틸화를 촉진한다.
사일런트
비활성 X염색체는 활성 X염색체와 달리 대부분의 유전자를 발현하지 않는다.이는 Xi DNA를 압축하고 대부분의 유전자의 발현을 막는 억제성 헤테로크로마틴에 의해 Xi가 침묵하기 때문이다.
Xa에 비해 Xi는 높은 수준의 DNA 메틸화, 낮은 수준의 히스톤 아세틸화, 낮은 수준의 히스톤 H3 리신-4 메틸화 및 높은 수준의 히스톤 H3 리신-9 메틸화 및 H3 리신-27 메틸화 마크를 가지며, 이들은 모두 [34]Xist에 의해 포섭된 PRC2 착체에 의해 배치된다.PRC2는 DNA 손상 [35]반응을 포함한 여러 과정에서 염색질 압축 및 염색질 리모델링을 조절한다.또한, 매크로H2A(H2AFY)라고 불리는 히스톤 변이체는 [36][37]Xi를 따라 있는 뉴클레오솜에서만 발견된다.
변호사단
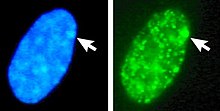
Xi와 같은 헤테로크로마틴으로 포장된 DNA는 Xa와 같은 에크로마틴으로 포장된 DNA보다 더 응축되어 있다.비활성 X는 Bar [38]body라고 불리는 핵 내에서 이산적인 몸을 형성합니다.Bar body는 일반적으로 핵의 주변에 위치하고 세포 주기 내에서 늦게 복제되며, Xi를 포함하므로 헤테로크로마틴 수정과 Xist RNA를 포함합니다.
비활성 X염색체 발현 유전자
X염색체를 따라 있는 유전자의 일부가 Xi에서 불활성화에서 탈출한다.Xist 유전자는 Xi에서 높은 수준으로 발현되며 Xa에서는 [39]발현되지 않습니다.다른 많은 유전자들은 불활성화에서 탈출한다; 어떤 유전자들은 Xa와 Xi에서 동등하게 발현되고, 다른 유전자들은 양쪽 염색체에서 발현되는 동안 여전히 Xa에서 [40][41][42]주로 발현된다.인간 Xi의 유전자의 4분의 1이 [40]탈출할 수 있다.쥐에 대한 연구는 주어진 세포 유형에서 유전자의 3%에서 15%가 불활성화에서 탈출하고 탈출하는 유전자의 정체성은 [41][42]조직마다 다르다고 시사한다.
불활성화에서 벗어나는 많은 유전자들은 X 염색체의 대다수와 달리 Y 염색체에 존재하는 유전자를 포함하는 X 염색체의 영역을 따라 존재한다.이 지역들은 성 염색체를 따라 있는 대다수의 유전자들과 달리, 어느 한 성별의 개인이 이 지역의 모든 유전자의 두 개의 복사본을 받게 되기 때문에 의사 자동 염색체 영역이라고 불립니다.어느 한 성(性)의 개인이 의사 자동 염색체 영역의 모든 유전자의 두 개의 복사본을 받을 것이기 때문에, 여성에 대한 용량 보상은 필요하지 않기 때문에, DNA의 이러한 영역은 X 불활성화를 피하기 위한 메커니즘을 진화시켰다고 가정한다.Xi의 유사 자가 염색체 영역의 유전자는 Xi의 전형적인 변형이 없고 Xist RNA 결합이 거의 없다.
불활성 X를 따라 침묵하지 않는 유전자의 존재는 터너 증후군(X0)이나 클라인펠터 증후군(XXY)과 같은 X 염색체의 비정상적인 숫자를 가진 인간의 결함을 설명한다.이론적으로 X-불활성화는 영향을 받는 개인과 정상적인 염색체 보체를 가진 개인 사이의 유전자 용량 차이를 제거해야 한다.그러나 영향을 받는 개인에서는 X-불활성화가 불완전하며, 이러한 비침묵 유전자의 용량은 상염색체 부배수와 유사하게 X-불활성화를 벗어나기 때문에 다를 것이다.
X 불활성화로부터의 탈출을 제어하는 정확한 메커니즘은 알려져 있지 않지만, 무음 영역과 탈출 영역은 뚜렷한 크로마틴 [41][43]마크를 가지고 있는 것으로 나타났다.X-불활성화로부터의 탈출은 탈출 염색체 [2]도메인 내에서 긴 비부호화 RNA(lncRNA)의 발현에 의해 매개될 수 있다고 제안되었다.
실험 생물학에서 사용
스탠리 마이클 가틀러는 X-크롬 불활성화를 이용하여 암의 클론 기원을 증명했다.성별 결합 G6PD 유전자의 동질효소에 대해 헤테로 접합된 여성의 정상 조직과 종양을 검사한 결과, 그러한 개인의 종양 세포는 G6PD의 한 형태만을 발현하는 반면, 정상 조직은 두 개의 다른 표현형을 발현하는 세포의 거의 동등한 혼합물로 구성되어 있는 것으로 나타났다.이 패턴은 집단이 아닌 단세포가 [44]암으로 성장한다는 것을 암시한다.하지만, 이 패턴은 많은 암 유형에서 잘못된 것으로 입증되었고, 일부 암은 [45]기원이 다복제일 수 있다는 것을 암시한다.
또한 X염색체에 위치한 다형 인간 안드로겐 수용체(HUMARA)의 메틸화(불활성화) 상태를 측정하는 것은 여성 암 생체 [46]검사의 클로날리티를 평가하는 가장 정확한 방법으로 간주된다.이 방법에 의해 다양한 종양이 테스트되었으며, 신장 세포암과 [47]같은 일부 종양이 모노클로널로 발견된 반면 다른 종양(예: 중피종[48])은 다세포로 보고되었다.
연구자들은 또한 X염색체 불활성화를 이용하여 상염색체의 활동을 억제하는 것을 연구해 왔다.예를 들어, 장 외 연구진은 Xist 유전자의 복사본을 트리소미 21(다운 증후군)[49]을 가진 개인으로부터 파생된 줄기세포의 21번 염색체의 한 복사본에 삽입했다.삽입된 Xist 유전자는 Bar body 형성을 유도하고, 안정적인 헤테로크로마틴 변형을 유발하며, 염색체 21의 여분의 복사본에서 대부분의 유전자를 침묵시킨다.이러한 변형된 줄기세포에서 Xist 매개 유전자 침묵은 다운 증후군과 관련된 결함의 일부를 되돌리는 것으로 보인다.
역사
1959년 Susumu Ohno는 포유류의 두 X 염색체가 다르다는 것을 보여주었다. 하나는 자가 염색체와 비슷하고 다른 하나는 응축되어 이색체이다.[50]이 연구결과는 두 그룹의 조사자와 독립적으로 X 염색체 중 하나가 불활성화되었음을 시사했다.
1961년, Mary Lyon은 털색 유전자에 [51]헤테로 접합된 암컷 생쥐의 얼룩덜룩한 표현형을 설명하기 위해 하나의 암컷 X 염색체의 무작위 불활성화를 제안했다.리옹 가설은 또한 여성 세포에서 X염색체 한 개가 고도로 응축되어 있고 X염색체 한 개만 가진 쥐가 불임 여성으로 발달했다는 연구 결과도 설명했습니다.어니스트 보이틀러에 이 suggested[52], 포도당-6-인산 탈수소 효소(글루코스-6-인산 탈수소 효소)결핍증에 이형의 여성들 공부하다는 적혈구의 두개의 붉은 세포군을 그런 heterozygotes에:부족한 세포와 정상 cells,[53]은 불활화 X염색체( 빨간 세포의 전구 세포의 핵에서)포함하고 있는지에 따라 다양했다.그 n 있습니다.또는 결함이 있는 G6PD 대립 유전자.
「 」를 참조해 주세요.
레퍼런스
- ^ Gartler SM, Varadarajan KR, Luo P, Canfield TK, Traynor J, Francke U, Hansen RS (September 2004). "Normal histone modifications on the inactive X chromosome in ICF and Rett syndrome cells: implications for methyl-CpG binding proteins". BMC Biology. 2: 21. doi:10.1186/1741-7007-2-21. PMC 521681. PMID 15377381.
- ^ a b Reinius B, Shi C, Hengshuo L, Sandhu KS, Radomska KJ, Rosen GD, Lu L, Kullander K, Williams RW, Jazin E (November 2010). "Female-biased expression of long non-coding RNAs in domains that escape X-inactivation in mouse". BMC Genomics. 11: 614. doi:10.1186/1471-2164-11-614. PMC 3091755. PMID 21047393.
- ^ Takagi N, Sasaki M (August 1975). "Preferential inactivation of the paternally derived X chromosome in the extra embryonic membranes of the mouse". Nature. 256 (5519): 640–2. Bibcode:1975Natur.256..640T. doi:10.1038/256640a0. PMID 1152998. S2CID 4190616.
- ^ Cheng MK, Disteche CM (August 2004). "Silence of the fathers: early X inactivation". BioEssays. 26 (8): 821–4. doi:10.1002/bies.20082. PMID 15273983.[데드링크]
- ^ a b Okamoto I, Otte AP, Allis CD, Reinberg D, Heard E (January 2004). "Epigenetic dynamics of imprinted X inactivation during early mouse development". Science. 303 (5658): 644–9. Bibcode:2004Sci...303..644O. doi:10.1126/science.1092727. PMID 14671313. S2CID 26326026.
- ^ a b Deng Q, Ramsköld D, Reinius B, Sandberg R (January 2014). "Single-cell RNA-seq reveals dynamic, random monoallelic gene expression in mammalian cells". Science. 343 (6167): 193–6. Bibcode:2014Sci...343..193D. doi:10.1126/science.1245316. PMID 24408435. S2CID 206552108.
- ^ a b Xue Z, Huang K, Cai C, Cai L, Jiang CY, Feng Y, Liu Z, Zeng Q, Cheng L, Sun YE, Liu JY, Horvath S, Fan G (August 2013). "Genetic programs in human and mouse early embryos revealed by single-cell RNA sequencing". Nature. 500 (7464): 593–7. Bibcode:2013Natur.500..593X. doi:10.1038/nature12364. PMC 4950944. PMID 23892778.
- ^ Borensztein M, Syx L, Ancelin K, Diabangouaya P, Picard C, Liu T, Liang JB, Vassilev I, Galupa R, Servant N, Barillot E, Surani A, Chen CJ, Heard E (March 2017). "Xist-dependent imprinted X inactivation and the early developmental consequences of its failure". Nature Structural & Molecular Biology. 24 (3): 226–233. doi:10.1038/nsmb.3365. PMC 5337400. PMID 28134930.
- ^ a b Deng X, Berletch JB, Nguyen DK, Disteche CM (June 2014). "X chromosome regulation: diverse patterns in development, tissues and disease". Nature Reviews. Genetics. 15 (6): 367–78. doi:10.1038/nrg3687. PMC 4117651. PMID 24733023.
- ^ Vértesy Á, Arindrarto W, Roost MS, Reinius B, Torrens-Juaneda V, Bialecka M, et al. (May 2018). "Parental haplotype-specific single-cell transcriptomics reveal incomplete epigenetic reprogramming in human female germ cells". Nature Communications. 9 (1): 1873. Bibcode:2018NatCo...9.1873V. doi:10.1038/s41467-018-04215-7. PMC 5951918. PMID 29760424.
- ^ Guo F, Yan L, Guo H, Li L, Hu B, Zhao Y, et al. (June 2015). "The Transcriptome and DNA Methylome Landscapes of Human Primordial Germ Cells". Cell. 161 (6): 1437–52. doi:10.1016/j.cell.2015.05.015. PMID 26046443.
- ^ Puck JM, Willard HF (January 1998). "X inactivation in females with X-linked disease". The New England Journal of Medicine. 338 (5): 325–8. doi:10.1056/NEJM199801293380611. PMID 9445416.
- ^ Balaton BP, Cotton AM, Brown CJ (30 December 2015). "Derivation of consensus inactivation status for X-linked genes from genome-wide studies". Biology of Sex Differences. 6 (35): 35. doi:10.1186/s13293-015-0053-7. PMC 4696107. PMID 26719789.
- ^ Graves JA (1996). "Mammals that break the rules: genetics of marsupials and monotremes". Annual Review of Genetics. 30: 233–60. doi:10.1146/annurev.genet.30.1.233. PMID 8982455.
- ^ Lyon MF (January 1972). "X-chromosome inactivation and developmental patterns in mammals". Biological Reviews of the Cambridge Philosophical Society. 47 (1): 1–35. doi:10.1111/j.1469-185X.1972.tb00969.x. PMID 4554151. S2CID 39402646.
- ^ Migeon, B (2010). "X chromosome inactivation in human cells". The Biomedical & Life Sciences Collection. Henry Stewart Talks, Ltd: 1–54. Retrieved 15 December 2013.
- ^ a b Gartler SM, Goldman MA (2001). "X-Chromosome Inactivation" (PDF). Encyclopedia of Life Sciences. Nature Publishing Group: 1–2.
- ^ Connallon T, Clark AG (April 2013). "Sex-differential selection and the evolution of X inactivation strategies". PLOS Genetics. 9 (4): e1003440. doi:10.1371/journal.pgen.1003440. PMC 3630082. PMID 23637618.
- ^ Avner, Philip; Heard, Edith (January 2001). "X-chromosome inactivation: counting, choice and initiation". Nature Reviews Genetics. 2 (1): 59–67. doi:10.1038/35047580. ISSN 1471-0064. PMID 11253071. S2CID 5234164.
- ^ Barakat TS, Gribnau J (2010). "X Chromosome Inactivation and Embryonic Stem Cells". In Meshorer E, Plath K (eds.). The Cell Biology of Stem Cells. Landes Bioscience and Springer Science+Business Media.
- ^ Ma L, Hoffman G, Keinan A (March 2015). "X-inactivation informs variance-based testing for X-linked association of a quantitative trait". BMC Genomics. 16: 241. doi:10.1186/s12864-015-1463-y. PMC 4381508. PMID 25880738.
- ^ Habekost CT, Pereira FS, Vargas CR, Coelho DM, Torrez V, Oses JP, Portela LV, Schestatsky P, Felix VT, Matte U, Torman VL, Jardim LB (October 2015). "Progression rate of myelopathy in X-linked adrenoleukodystrophy heterozygotes". Metabolic Brain Disease. 30 (5): 1279–84. doi:10.1007/s11011-015-9672-2. PMID 25920484. S2CID 11375978.
- ^ a b c Belmont JW (June 1996). "Genetic control of X inactivation and processes leading to X-inactivation skewing". American Journal of Human Genetics. 58 (6): 1101–8. PMC 1915050. PMID 8651285.
- ^ a b Holle JR, Marsh RA, Holdcroft AM, Davies SM, Wang L, Zhang K, Jordan MB (July 2015). "Hemophagocytic lymphohistiocytosis in a female patient due to a heterozygous XIAP mutation and skewed X chromosome inactivation". Pediatric Blood & Cancer. 62 (7): 1288–90. doi:10.1002/pbc.25483. PMID 25801017. S2CID 5516967.
- ^ Burgemeister AL, Zirn B, Oeffner F, Kaler SG, Lemm G, Rossier E, Büttel HM (November 2015). "Menkes disease with discordant phenotype in female monozygotic twins". American Journal of Medical Genetics. Part A. 167A (11): 2826–9. doi:10.1002/ajmg.a.37276. PMC 6475897. PMID 26239182.
- ^ Brioschi S, Gualandi F, Scotton C, Armaroli A, Bovolenta M, Falzarano MS, Sabatelli P, Selvatici R, D'Amico A, Pane M, Ricci G, Siciliano G, Tedeschi S, Pini A, Vercelli L, De Grandis D, Mercuri E, Bertini E, Merlini L, Mongini T, Ferlini A (August 2012). "Genetic characterization in symptomatic female DMD carriers: lack of relationship between X-inactivation, transcriptional DMD allele balancing and phenotype". BMC Medical Genetics. 13: 73. doi:10.1186/1471-2350-13-73. PMC 3459813. PMID 22894145.
- ^ a b Lee JT, Jaenisch R (March 1997). "Long-range cis effects of ectopic X-inactivation centres on a mouse autosome". Nature. 386 (6622): 275–9. Bibcode:1997Natur.386..275L. doi:10.1038/386275a0. PMID 9069285. S2CID 10899129.
- ^ a b Lee JT, Davidow LS, Warshawsky D (April 1999). "Tsix, a gene antisense to Xist at the X-inactivation centre". Nature Genetics. 21 (4): 400–4. doi:10.1038/7734. PMID 10192391. S2CID 30636065.
- ^ a b Mercer, T.R., Dinger, M.E., Mattick, J.S., (2009)긴 비코드 RNA: 기능에 대한 통찰력.Nature Reviews Genetics. (10) 155~159.
- ^ Hoki Y, Kimura N, Kanbayashi M, Amakawa Y, Ohhata T, Sasaki H, Sado T (January 2009). "A proximal conserved repeat in the Xist gene is essential as a genomic element for X-inactivation in mouse". Development. 136 (1): 139–46. doi:10.1242/dev.026427. PMID 19036803.
- ^ Ng K, Pullirsch D, Leeb M, Wutz A (January 2007). "Xist and the order of silencing" (Review Article). EMBO Reports. 8 (1): 34–9. doi:10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100.
Figure 1 Xist RNA encompasses the X from which it is transcribed.
{{cite journal}}:외부 링크quote= - ^ Penny GD, Kay GF, Sheardown SA, Rastan S, Brockdorff N (1996). "Requirement for Xist in X chromosome inactivation". Nature. 379 (6561): 131–7. Bibcode:1996Natur.379..131P. doi:10.1038/379131a0. PMID 8538762. S2CID 4329368.
- ^ a b Herzing LB, Romer JT, Horn JM, Ashworth A (March 1997). "Xist has properties of the X-chromosome inactivation centre". Nature. 386 (6622): 272–5. Bibcode:1997Natur.386..272H. doi:10.1038/386272a0. PMID 9069284. S2CID 4371247.
- ^ Ng K, Pullirsch D, Leeb M, Wutz A (January 2007). "Xist and the order of silencing" (Review Article). EMBO Reports. 8 (1): 34–9. doi:10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100.
Table 1 Features of the inactive X territory
{{cite journal}}: 외부 링크 (도움말)– 발신원
Chow JC, Yen Z, Ziesche SM, Brown CJ (2005). "Silencing of the mammalian X chromosome". Annual Review of Genomics and Human Genetics. 6: 69–92. doi:10.1146/annurev.genom.6.080604.162350. PMID 16124854.
Lucchesi JC, Kelly WG, Panning B (2005). "Chromatin remodeling in dosage compensation". Annual Review of Genetics. 39: 615–51. CiteSeerX 10.1.1.328.2992. doi:10.1146/annurev.genet.39.073003.094210. PMID 16285873. - ^ Veneti Z, Gkouskou KK, Eliopoulos AG (July 2017). "Polycomb Repressor Complex 2 in Genomic Instability and Cancer". Int J Mol Sci. 18 (8): 1657. doi:10.3390/ijms18081657. PMC 5578047. PMID 28758948.
- ^ Costanzi C, Pehrson JR (June 1998). "Histone macroH2A1 is concentrated in the inactive X chromosome of female mammals". Nature. 393 (6685): 599–601. Bibcode:1998Natur.393..599C. doi:10.1038/31275. PMID 9634239. S2CID 205001095.
- ^ Costanzi C, Stein P, Worrad DM, Schultz RM, Pehrson JR (June 2000). "Histone macroH2A1 is concentrated in the inactive X chromosome of female preimplantation mouse embryos" (PDF). Development. 127 (11): 2283–9. doi:10.1242/dev.127.11.2283. PMID 10804171.
- ^ Barr ML, Bertram EG (April 1949). "A morphological distinction between neurones of the male and female, and the behaviour of the nucleolar satellite during accelerated nucleoprotein synthesis". Nature. 163 (4148): 676–677. Bibcode:1949Natur.163..676B. doi:10.1038/163676a0. PMID 18120749. S2CID 4093883.
- ^ Plath K, Mlynarczyk-Evans S, Nusinow DA, Panning B (2002). "Xist RNA and the mechanism of X chromosome inactivation". Annual Review of Genetics. 36: 233–78. doi:10.1146/annurev.genet.36.042902.092433. PMID 12429693.
- ^ a b Carrel L, Willard HF (March 2005). "X-inactivation profile reveals extensive variability in X-linked gene expression in females". Nature. 434 (7031): 400–4. Bibcode:2005Natur.434..400C. doi:10.1038/nature03479. PMID 15772666. S2CID 4358447.
- ^ a b c Calabrese JM, Sun W, Song L, Mugford JW, Williams L, Yee D, Starmer J, Mieczkowski P, Crawford GE, Magnuson T (November 2012). "Site-specific silencing of regulatory elements as a mechanism of X inactivation". Cell. 151 (5): 951–63. doi:10.1016/j.cell.2012.10.037. PMC 3511858. PMID 23178118.
- ^ a b Yang F, Babak T, Shendure J, Disteche CM (May 2010). "Global survey of escape from X inactivation by RNA-sequencing in mouse". Genome Research. 20 (5): 614–22. doi:10.1101/gr.103200.109. PMC 2860163. PMID 20363980.
- ^ Berletch JB, Yang F, Disteche CM (June 2010). "Escape from X inactivation in mice and humans". Genome Biology. 11 (6): 213. doi:10.1186/gb-2010-11-6-213. PMC 2911101. PMID 20573260.
- ^ Linder D, Gartler SM (October 1965). "Glucose-6-phosphate dehydrogenase mosaicism: utilization as a cell marker in the study of leiomyomas". Science. 150 (3692): 67–9. Bibcode:1965Sci...150...67L. doi:10.1126/science.150.3692.67. PMID 5833538. S2CID 33941451.
- ^ Parsons BL (2008). "Many different tumor types have polyclonal tumor origin: evidence and implications". Mutation Research. 659 (3): 232–47. doi:10.1016/j.mrrev.2008.05.004. PMID 18614394.
- ^ Chen GL, Prchal JT (September 2007). "X-linked clonality testing: interpretation and limitations". Blood. 110 (5): 1411–9. doi:10.1182/blood-2006-09-018655. PMC 1975831. PMID 17435115.
- ^ Petersson F, Branzovsky J, Martinek P, Korabecna M, Kruslin B, Hora M, et al. (July 2014). "The leiomyomatous stroma in renal cell carcinomas is polyclonal and not part of the neoplastic process". Virchows Archiv. 465 (1): 89–96. doi:10.1007/s00428-014-1591-9. PMID 24838683. S2CID 24870232.
- ^ Comertpay S, Pastorino S, Tanji M, Mezzapelle R, Strianese O, Napolitano A, Baumann F, Weigel T, Friedberg J, Sugarbaker P, Krausz T, Wang E, Powers A, Gaudino G, Kanodia S, Pass HI, Parsons BL, Yang H, Carbone M (December 2014). "Evaluation of clonal origin of malignant mesothelioma". Journal of Translational Medicine. 12: 301. doi:10.1186/s12967-014-0301-3. PMC 4255423. PMID 25471750.
- ^ Jiang J, Jing Y, Cost GJ, Chiang JC, Kolpa HJ, Cotton AM, et al. (August 2013). "Translating dosage compensation to trisomy 21". Nature. 500 (7462): 296–300. Bibcode:2013Natur.500..296J. doi:10.1038/nature12394. PMC 3848249. PMID 23863942.
- ^ Ohno S, Kaplan WD, Kinosita R (October 1959). "Formation of the sex chromatin by a single X-chromosome in liver cells of Rattus norvegicus". Experimental Cell Research. 18 (2): 415–8. doi:10.1016/0014-4827(59)90031-X. PMID 14428474.
- ^ Lyon MF (April 1961). "Gene action in the X-chromosome of the mouse (Mus musculus L.)". Nature. 190 (4773): 372–3. Bibcode:1961Natur.190..372L. doi:10.1038/190372a0. PMID 13764598. S2CID 4146768.
- ^ Beutler E (January 2008). "Glucose-6-phosphate dehydrogenase deficiency: a historical perspective". Blood. 111 (1): 16–24. doi:10.1182/blood-2007-04-077412. PMID 18156501.
- ^ Beutler E, Yeh M, Fairbanks VF (January 1962). "The normal human female as a mosaic of X-chromosome activity: studies using the gene for C-6-PD-deficiency as a marker". Proceedings of the National Academy of Sciences of the United States of America. 48 (1): 9–16. Bibcode:1962PNAS...48....9B. doi:10.1073/pnas.48.1.9. PMC 285481. PMID 13868717.
추가 정보
- Huynh KD, Lee JT (May 2005). "X-chromosome inactivation: a hypothesis linking ontogeny and phylogeny". Nature Reviews. Genetics. 6 (5): 410–8. doi:10.1038/nrg1604. PMID 15818384. S2CID 19245310.
- Goto T, Monk M (June 1998). "Regulation of X-chromosome inactivation in development in mice and humans" (Review Article). Microbiology and Molecular Biology Reviews. 62 (2): 362–78. doi:10.1128/MMBR.62.2.362-378.1998. PMC 98919. PMID 9618446.
- Lyon MF (December 2003). "The Lyon and the LINE hypothesis". Seminars in Cell & Developmental Biology (Review Article). 14 (6): 313–8. doi:10.1016/j.semcdb.2003.09.015. PMID 15015738.
- Ng K, Pullirsch D, Leeb M, Wutz A (January 2007). "Xist and the order of silencing" (Review Article). EMBO Reports. 8 (1): 34–9. doi:10.1038/sj.embor.7400871. PMC 1796754. PMID 17203100.
- Cerase A, Pintacuda G, Tattermusch A, Avner P (August 2015). "Xist localization and function: new insights from multiple levels". Genome Biology. 16: 166. doi:10.1186/s13059-015-0733-y. PMC 4539689. PMID 26282267.
외부 링크
 Wikimedia Commons X 염색체 불활성화 관련 매체
Wikimedia Commons X 염색체 불활성화 관련 매체- Kruszelnicki K (2004-02-11). "Hybrid Auto-Immune Women 3". ABC Science.