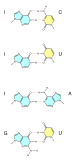
베이스 페어
Base pair
염기쌍(bp)은 수소결합에 의해 서로 결합되는 2개의 핵염기로 이루어진 2가닥 핵산의 기본단위이다.그들은 DNA 이중나선의 구성 요소를 형성하고 DNA와 RNA 모두의 접힌 구조에 기여한다. 특정한 수소 결합 패턴에 의해 지시되는 "왓슨-크릭"(또는 "왓슨-크릭-프랭클린") 염기쌍(과닌-시토신 및 아데닌-티민)[1]은 DNA 나선이 그것의 미묘하게 의존하는 규칙적인 나선 구조를 유지하도록 한다.뉴클레오티드 [2]배열이 기반 쌍 구조의 상호 보완적인 성질은 DNA의 각 가닥 안에 인코딩된 유전자 정보의 중복된 복사본을 제공합니다.DNA 이중나선에 의해 제공되는 규칙적인 구조와 데이터 중복은 DNA를 유전 정보의 저장에 잘 적합하게 만드는 반면, DNA와 유입되는 뉴클레오티드 사이의 염기쌍은 DNA 중합효소가 DNA를 복제하고 RNA 중합효소가 RNA로 DNA를 전사하는 메커니즘을 제공한다. 많은 DNA 결합 단백질은 특이성을 인식할 수 있다.유전자의 특정 조절 영역을 식별하는 ic 염기서열 패턴.
분자내 염기쌍은 단일가닥 핵산 내에서 발생할 수 있다.이는 특히 RNA 분자(예: 전달 RNA)에서 중요하다. 여기서 왓슨-크릭 염기쌍(과닌-시토신 및 아데닌-우라실)은 짧은 이중 가닥 나선 형성을 허용하고, 광범위한 비-왓슨-크릭 상호작용(예: G-U 또는 A-S)은 RNA로 접히는 것을 허용한다.또한 전달 RNA(tRNA)와 메신저 RNA(mRNA) 간의 염기쌍은 mRNA의 뉴클레오티드 배열을 유전자 코드를 통해 단백질의 아미노산 배열로 변환시키는 분자 인식 이벤트의 기초를 형성한다.
DNA는 보통 이중 가닥이기 때문에 개별 유전자나 유기체의 전체 게놈의 크기는 종종 염기쌍으로 측정된다.따라서 총 염기쌍의 수는 텔로미어의 단일 가닥 영역을 제외하고 가닥 중 하나에 있는 뉴클레오티드의 수와 같다.반수체 인간 게놈(23개 염색체)은 길이가 약 32억 염기로 추정되며 20,000-25,000개의 단백질 코드화 [3][4][5]유전자를 포함하고 있다.킬로베이스(kb)는 분자생물학에서 1000개의 DNA 또는 [6]RNA 염기쌍과 같은 측정 단위입니다.지구상의 총 DNA 염기쌍 수는 5.0×10으로37 추정되며 무게는 500억 [7]톤이다.이에 비해 생물권의 총 질량은 4TtC(탄소 [8]1조 톤)에 달하는 것으로 추정되고 있다.
수소 결합 및 안정성
수소 결합은 위에서 설명한 염기쌍 구성 규칙의 기초가 되는 화학적 상호작용입니다.수소 결합 공여체와 수용체의 적절한 기하학적 대응은 "오른쪽" 쌍만 안정적으로 형성할 수 있게 한다.GC 함량이 높은 DNA는 GC 함량이 낮은 DNA보다 안정적이다.하지만, 일반적인 믿음과는 달리, 수소 결합은 DNA를 크게 안정화시키지 않는다; 안정화는 주로 쌓이는 [9]상호작용에 기인한다.
더 큰 핵염기인 아데닌과 구아닌은 퓨린이라고 불리는 이중 고리 화학 구조의 구성원이고, 더 작은 핵염기인 시토신과 티민은 피리미딘이라고 불리는 단일 고리 화학 구조의 구성원입니다.퓨린은 피리미딘과 상보적이다.피리미딘-피리미딘 쌍은 수소 결합을 확립하기에는 분자가 너무 멀기 때문에 에너지적으로 바람직하지 않다.푸린-푸린 쌍은 분자가 너무 가깝기 때문에 에너지적으로 바람직하지 않다.AT, GC 또는 UA(RNA 내)의 퓨린피리미딘 염기쌍은 적절한 이중구조를 나타낸다.다른 퓨린-피리미딘 쌍은 AC, GT 및 UG(RNA 내)뿐이며, 수소 공여체와 수용체의 패턴이 일치하지 않기 때문에 이러한 쌍은 일치하지 않습니다.두 개의 수소 결합을 가진 GU 쌍은 RNA에서 꽤 자주 발생합니다(흔들 염기 쌍 참조).
쌍을 이룬 DNA와 RNA 분자는 상온에서 비교적 안정적이지만, 두 개의 뉴클레오티드 가닥은 분자의 길이, 잘못된 짝의 정도(있는 경우), 그리고 GC 함량에 의해 결정되는 녹는점 위에서 분리될 것입니다.GC 함량이 높을수록 녹는 온도가 높아지므로 Thermus thermophilus와 같은 극호성 생물의 게놈이 특히 GC가 풍부한 것은 놀랄 일이 아니다.반대로 자주 분리해야 하는 게놈의 영역(예를 들어, 자주 전사되는 유전자의 프로모터 영역)은 상대적으로 GC가 낮다(TATA 상자 참조).PCR 반응을 위한 프라이머를 설계할 때 GC 함량과 용해 온도도 고려해야 합니다.
예
다음 DNA 염기서열은 쌍쌍의 이중가닥 패턴을 보여줍니다.관례상 윗줄은 5µ 끝에서 3µ 끝까지 쓰기 때문에 아랫줄은 3µ에서 5µ로 쓴다.
- 염기쌍 DNA 배열:
ATCGATTGAGCTCTAGCGTAGCTAACTCGAGATCGC
- RNA 가닥에서 유라실이 티민 대신 치환되는 해당 RNA 배열:
AUCGAUUGAGCUCUAGCGUAGCUAACUCGAGAUCGC
기본 아날로그 및 인터캘레이터
뉴클레오티드의 화학적 유사체는 적절한 뉴클레오티드를 대체할 수 있고 비표준 염기쌍을 확립하여 DNA 복제와 DNA 전사에서의 오류(대부분 점 돌연변이)를 초래할 수 있다.이것은 그들의 이성 화학 작용 때문이다.일반적인 변이원성 염기 유사체는 티민과 유사하지만 에놀 형태로 구아닌과 염기쌍을 이룰 수 있는 5-브로모우라실이다.
DNA 인터칼레이터로 알려진 다른 화학물질들은 단일 가닥의 인접한 염기들 사이의 틈새에 들어맞고 염기서로서 "위장"함으로써 프레임시프트 돌연변이를 유도하여, DNA 복제 기구가 인터칼레이터된 부위에 추가적인 뉴클레오티드를 건너뛰거나 삽입하도록 한다.대부분의 인터칼레이터는 큰 다방향족 화합물이며 발암물질로 알려져 있거나 의심되는 물질입니다.예를 들어 브롬화 에티듐과 아크리딘이 있다.
불일치 복구
일치하지 않는 염기쌍은 DNA 복제 오류 및 상동 재조합 중 중간체로서 생성될 수 있다.불일치 복구 프로세스는 일반적으로 정상적인 DNA 염기쌍의 긴 시퀀스 내에서 소수의 염기쌍을 인식하고 올바르게 복구해야 합니다.DNA 복제 중에 형성된 불일치를 복구하기 위해, 템플릿 스트랜드와 새로 형성된 스트랜드를 구별하도록 몇 가지 독특한 복구 과정이 진화하여 새롭게 삽입된 잘못된 뉴클레오티드만 제거된다([10]변이를 일으키지 않기 위해).DNA 복제 중 불일치 복구에 사용된 단백질과 이 과정에서 결함의 임상적 의미는 DNA 불일치 복구 기사에 설명되어 있다.재조합 중 오쌍보정의 과정은 유전자 변환에 기술되어 있다.
부자연스러운 베이스 페어(UBP)
부자연 염기쌍(UBP)은 실험실에서 생성되고 자연에서는 발생하지 않는 DNA의 설계 서브유닛(또는 핵염기)이다.자연에서 발견된 두 개의 염기쌍인 A-T(아데닌 – 티민)와 G-C(구아닌 – 시토신) 외에, 세 번째 염기쌍을 형성하기 위해 새롭게 생성된 핵염기를 사용하는 DNA 배열이 설명되었다.스티븐 A가 이끄는 팀을 포함한 몇몇 연구 단체들은 DNA를 위한 세 번째 염기쌍을 찾고 있다. 베너, 필리프 마를리에, 플로이드 E 로메스버그와 히라오 [11]이치로.대체 수소 결합, 소수성 상호작용 및 금속 배위에 기초한 일부 새로운 염기쌍이 [12][13][14][15]보고되었다.
1989년 스티븐 베너(당시 취리히에 있는 스위스 연방 공과대학에서 근무)와 그의 팀은 [16]체외 DNA 분자로 변형된 형태의 시토신과 구아닌을 이끌었다.RNA와 단백질을 코드한 뉴클레오티드는 체외에서 성공적으로 복제되었다.그 이후 베너 교수팀은 [17]공급원료의 필요성을 없애기 위해 처음부터 외국 기지를 만들 수 있는 세포를 개발하려고 노력해 왔다.
2002년 일본의 히라오 이치로( h amino一郞) 그룹은 비표준 아미노산을 단백질에 [18]결합시키기 위해 전사 및 번역에 기능하는 2-아미노-8-(2-티에닐) 푸린(s)과 피리딘-2-온(y)의 부자연스러운 염기쌍을 개발했다.2006년에는 복제 및 [19]전사를 위한 세 번째 염기쌍으로 7-(2-티에닐)이미다조[4,5-b]피리딘(Ds)과 피롤-2-카르발알데히드(Pa)를 생성했다.그 후 PCR [20][21]증폭에서 Ds와 4-[3-(6-아미노헥사나미도)-1-프로피닐]-2-니트로피롤(Px)을 고충실성 쌍으로 발견했다.2013년, 그들은 Ds-Px 쌍을 시험관내 선택(SELEX)에 의한 DNA 압타머 생성에 적용하고 유전자 알파벳 확장이 표적 [22]단백질에 대한 DNA 압타머 친화력을 유의하게 증가시킨다는 것을 입증했다.
2012년, 캘리포니아 샌디에고에 있는 스크립스 연구소의 화학 생물학자인 플로이드 롬스버그가 이끄는 미국 과학자 그룹은 그의 팀이 부자연스러운 [14]염기쌍을 설계했다고 발표했다.두 개의 새로운 인공 뉴클레오티드 또는 부자연스러운 염기쌍(UBP)은 d5SICS와 dNaM으로 명명되었다.좀 더 기술적으로, 소수성 핵산염기를 가진 이러한 인공 뉴클레오티드는 DNA에서 [17][23](d5SICS–dNaM) 복합체 또는 염기쌍을 형성하는 두 개의 융합 방향족 고리를 특징으로 한다.그의 팀은 부자연스러운 염기쌍을 포함하는 다양한 시험관 또는 "시험관" 템플릿을 설계했으며, 그들은 DNA와 PCR 기반 애플리케이션의 [14]PCR 증폭이라는 최신 표준 시험관 기술을 사용하여 거의 모든 시퀀스 컨텍스트에서 높은 충실도로 효율적으로 복제되었음을 확인했다.그 결과에 따르면 PCR 및 PCR 기반 애플리케이션의 경우 d5SICS-dNaM 부자연 염기쌍은 기능적으로 자연 염기쌍과 동등하며, 모든 유기체가 사용하는 다른 두 개의 자연 염기쌍인 A-T와 G-C와 결합하면 완전히 기능하고 확장된 6글자의 "유전자 알파벳"[23]을 제공한다.
2014년 스크립스 연구소의 같은 팀은 자연 T-A와 C-G 염기쌍을 포함하는 플라스미드로 알려진 원형 DNA를 UBP Romesberg의 실험실이 설계하여 자연 B를 성공적으로 복제한 일반적인 박테리아 대장균의 세포에 삽입했다고 보고했다.여러 [11]세대에 걸쳐 짝을 이루다.이 전염은 대장균 세포의 성장을 방해하지 않았고 자연적인 DNA 복구 메커니즘으로 인해 비정상적인 염기쌍을 잃을 기미를 보이지 않았다.이것은 살아있는 유기체가 확장된 유전자 코드를 따라 다음 [23][24]세대에 전달되는 최초의 알려진 사례이다.롬스버그는 그와 그의 동료들이 충분히 안정적이고 세포가 분열할 때 자연 그대로 쉽게 복제될 수 있는 뉴클레오티드의 디자인을 다듬기 위해 300개의 변종을 만들었다고 말했다.이는 부분적으로 d5SICSTP와 dNaMTP의 트리포스페이트를 대장균에 [23]효율적으로 수입하는 뉴클레오티드 삼인산 트랜스포터를 발현하는 지지 조류 유전자를 추가함으로써 달성되었다.그런 다음 자연 세균 복제 경로는 d5SICS–dNaM을 포함하는 플라스미드를 정확하게 복제하기 위해 이들을 사용한다.다른 연구원들은 이 박테리아가 인간이 만든 DNA 서브유닛을 [25]복제한 것에 놀랐다.
세 번째 염기쌍의 성공적인 통합은 DNA에 의해 암호화될 수 있는 아미노산의 수를 기존의 20개의 아미노산에서 이론적으로 가능한 172개로 크게 확장함으로써 생물들이 새로운 [11]단백질을 생산할 수 있는 가능성을 확장하는 목표를 향한 중요한 돌파구이다.DNA의 인공 문자열은 아직 아무 의미도 없지만, 과학자들은 그것들이 산업적 또는 [26]의약적으로 사용될 수 있는 새로운 단백질을 생산하도록 설계될 수 있다고 추측한다.전문가들은 이 부자연스러운 염기쌍을 포함한 합성 DNA가 다른 DNA [25][26]코드에 기초한 생명체의 가능성을 높여준다고 말했다.
비표준 베이스 페어링
표준 쌍 외에, 일부 조건은 대체 염기 배향, 수소 결합의 수 및 기하학으로 염기 쌍을 선호할 수 있다.이러한 쌍은 로컬백본 형상의 변경을 수반합니다.
이들[28] 중 가장 일반적인 것은 전사와 일부 tRNA 합성효소에 [29]의해 tRNA가 충전되는 동안 많은 코돈의 세 번째 염기 위치에서 tRNA와 mRNA 사이에 발생하는 워블 염기쌍이다.그것들은 또한 일부 RNA [30]배열의 2차 구조에서 관찰되었다.
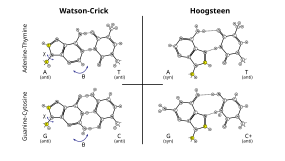
또한 Hoogsteen 염기쌍(일반적으로 A•U/T 및 G•C로 표기)은 표준 Watson-Crick [27]쌍과 동적 평형 상태에서 일부 DNA 배열(예: CA 및 TA 디뉴클레오티드)에 존재할 수 있다.그것들은 또한 일부 단백질에서도 관찰되었다.DNA [31]콤플렉스
이들 대체 염기쌍 외에 RNA 2차 및 3차 [32]구조에서 광범위한 염기 수소 결합이 관찰된다.이러한 결합은 종종 RNA의 정확하고 복잡한 모양과 상호작용 [32]파트너와의 결합에 필요합니다.
길이 측정
D/RNA 분자의 길이를 설명하기 위해 일반적으로 사용되는 약어는 다음과 같습니다.
- bp = 염기쌍—1bp는 가닥을 따라 약 3.4Ω(14ppm)[33]의 길이에 해당하며, DNA와 RNA의 경우 각각 약 618 또는 643 달톤에 해당한다.
- kb (= kbp) = 킬로 베이스 주파수 = 1,000 bp
- Mb(= Mbp) = 메가 베이스 주파수 = 1,000,000 bp
- Gb = 기가 베이스 주파수 = 1,000,000 bp.
단일 가닥 DNA/RNA의 경우, 뉴클레오티드의 단위는 쌍이 아니기 때문에 nt(또는 knt, Mnt, Gnt)로 단축된다.컴퓨터 스토리지 유닛과 베이스를 구별하기 위해 기본 쌍으로 kbp, Mbp, Gbp 등을 사용할 수 있습니다.
센티모건은 염색체를 따라 거리를 암시하는 데 자주 사용되지만, 이에 대응하는 염기쌍의 수는 매우 다양하다.인간 게놈에서 센티모건은 약 100만쌍의 [34][35]염기쌍이다.
「 」를 참조해 주세요.
레퍼런스
- ^ Spencer, M. (10 January 1959). "The stereochemistry of deoxyribonucleic acid. II. Hydrogen-bonded pairs of bases". Acta Crystallographica. 12 (1): 66–71. doi:10.1107/S0365110X59000160. ISSN 0365-110X.
- ^ Zhurkin VB, Tolstorukov MY, Xu F, Colasanti AV, Olson WK (2005). "Sequence-Dependent Variability of B-DNA". DNA Conformation and Transcription. pp. 18–34. doi:10.1007/0-387-29148-2_2. ISBN 978-0-387-25579-8.
{{cite book}}:누락 또는 비어 있음title=(도움말) - ^ Moran LA (2011-03-24). "The total size of the human genome is very likely to be ~3,200 Mb". Sandwalk.blogspot.com. Retrieved 2012-07-16.
- ^ "The finished length of the human genome is 2.86 Gb". Strategicgenomics.com. 2006-06-12. Retrieved 2012-07-16.
- ^ International Human Genome Sequencing Consortium (October 2004). "Finishing the euchromatic sequence of the human genome". Nature. 431 (7011): 931–45. Bibcode:2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
- ^ Cockburn AF, Newkirk MJ, Firtel RA (December 1976). "Organization of the ribosomal RNA genes of Dictyostelium discoideum: mapping of the nontranscribed spacer regions". Cell. 9 (4 Pt 1): 605–13. doi:10.1016/0092-8674(76)90043-X. PMID 1034500. S2CID 31624366.
- ^ Nuwer R (18 July 2015). "Counting All the DNA on Earth". The New York Times. New York. ISSN 0362-4331. Archived from the original on 2022-01-01. Retrieved 2015-07-18.
- ^ "The Biosphere: Diversity of Life". Aspen Global Change Institute. Basalt, CO. Retrieved 2015-07-19.
- ^ Yakovchuk P, Protozanova E, Frank-Kamenetskii MD (2006-01-30). "Base-stacking and base-pairing contributions into thermal stability of the DNA double helix". Nucleic Acids Research. 34 (2): 564–74. doi:10.1093/nar/gkj454. PMC 1360284. PMID 16449200.
- ^ Putnam, Christopher D. (September 2021). "Strand discrimination in DNA mismatch repair". DNA Repair. 105: 103161. doi:10.1016/j.dnarep.2021.103161. PMC 8785607. PMID 34171627.
- ^ a b c Fikes BJ (May 8, 2014). "Life engineered with expanded genetic code". San Diego Union Tribune. Archived from the original on 9 May 2014. Retrieved 8 May 2014.
- ^ Yang Z, Chen F, Alvarado JB, Benner SA (September 2011). "Amplification, mutation, and sequencing of a six-letter synthetic genetic system". Journal of the American Chemical Society. 133 (38): 15105–12. doi:10.1021/ja204910n. PMC 3427765. PMID 21842904.
- ^ Yamashige R, Kimoto M, Takezawa Y, Sato A, Mitsui T, Yokoyama S, Hirao I (March 2012). "Highly specific unnatural base pair systems as a third base pair for PCR amplification". Nucleic Acids Research. 40 (6): 2793–806. doi:10.1093/nar/gkr1068. PMC 3315302. PMID 22121213.
- ^ a b c Malyshev DA, Dhami K, Quach HT, Lavergne T, Ordoukhanian P, Torkamani A, Romesberg FE (July 2012). "Efficient and sequence-independent replication of DNA containing a third base pair establishes a functional six-letter genetic alphabet". Proceedings of the National Academy of Sciences of the United States of America. 109 (30): 12005–10. Bibcode:2012PNAS..10912005M. doi:10.1073/pnas.1205176109. PMC 3409741. PMID 22773812.
- ^ Takezawa, Yusuke; Müller, Jens; Shionoya, Mitsuhiko (2017-05-05). "Artificial DNA Base Pairing Mediated by Diverse Metal Ions". Chemistry Letters. 46 (5): 622–633. doi:10.1246/cl.160985. ISSN 0366-7022.
- ^ Switzer C, Moroney SE, Benner SA (1989). "Enzymatic incorporation of a new base pair into DNA and RNA". J. Am. Chem. Soc. 111 (21): 8322–8323. doi:10.1021/ja00203a067.
- ^ a b Callaway E (May 7, 2014). "Scientists Create First Living Organism With 'Artificial' DNA". Nature News. Huffington Post. Retrieved 8 May 2014.
- ^ Hirao I, Ohtsuki T, Fujiwara T, Mitsui T, Yokogawa T, Okuni T, Nakayama H, Takio K, Yabuki T, Kigawa T, Kodama K, Yokogawa T, Nishikawa K, Yokoyama S (February 2002). "An unnatural base pair for incorporating amino acid analogs into proteins". Nature Biotechnology. 20 (2): 177–82. doi:10.1038/nbt0202-177. PMID 11821864. S2CID 22055476.
- ^ Hirao I, Kimoto M, Mitsui T, Fujiwara T, Kawai R, Sato A, Harada Y, Yokoyama S (September 2006). "An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA". Nature Methods. 3 (9): 729–35. doi:10.1038/nmeth915. PMID 16929319. S2CID 6494156.
- ^ 키모토 M. 등(2009) DNA 분자의 효율적인 PCR 증폭 및 기능화를 위한 부자연스러운 염기쌍 시스템.핵산 제37호, e14호
- ^ Yamashige R, Kimoto M, Takezawa Y, Sato A, Mitsui T, Yokoyama S, Hirao I (March 2012). "Highly specific unnatural base pair systems as a third base pair for PCR amplification". Nucleic Acids Research. 40 (6): 2793–806. doi:10.1093/nar/gkr1068. PMC 3315302. PMID 22121213.
- ^ Kimoto M, Yamashige R, Matsunaga K, Yokoyama S, Hirao I (May 2013). "Generation of high-affinity DNA aptamers using an expanded genetic alphabet". Nature Biotechnology. 31 (5): 453–7. doi:10.1038/nbt.2556. PMID 23563318. S2CID 23329867.
- ^ a b c d Malyshev DA, Dhami K, Lavergne T, Chen T, Dai N, Foster JM, Corrêa IR, Romesberg FE (May 2014). "A semi-synthetic organism with an expanded genetic alphabet". Nature. 509 (7500): 385–8. Bibcode:2014Natur.509..385M. doi:10.1038/nature13314. PMC 4058825. PMID 24805238.
- ^ Sample I (May 7, 2014). "First life forms to pass on artificial DNA engineered by US scientists". The Guardian. Retrieved 8 May 2014.
- ^ a b "Scientists create first living organism containing artificial DNA". The Wall Street Journal. Fox News. May 8, 2014. Retrieved 8 May 2014.
- ^ a b Pollack A (May 7, 2014). "Scientists Add Letters to DNA's Alphabet, Raising Hope and Fear". New York Times. Retrieved 8 May 2014.
- ^ a b Evgenia N. Nikolova; Eunae Kim; Abigail A. Wise; Patrick J. O'Brien; Ioan Andricioaei; Hashim M. Al-Hashimi (2011). "Transient Hoogsteen base pairs in canonical duplex DNA". Nature. 470 (7335): 498–502. Bibcode:2011Natur.470..498N. doi:10.1038/nature09775. PMC 3074620. PMID 21270796.
- ^ Murphy, Frank V; Ramakrishnan, V (2004-11-21). "Structure of a purine-purine wobble base pair in the decoding center of the ribosome". Nature Structural & Molecular Biology. 11 (12): 1251–1252. doi:10.1038/nsmb866. ISSN 1545-9993. PMID 15558050. S2CID 27022506.
- ^ Vargas-Rodriguez, Oscar; Musier-Forsyth, Karin (June 2014). "Wobble puts RNA on target". Nature. 510 (7506): 480–481. doi:10.1038/nature13502. ISSN 1476-4687. PMID 24919145. S2CID 205239383.
- ^ Garg, Ankur; Heinemann, Udo (2017-11-09). "A novel form of RNA double helix based on G·U and C·A+wobble base pairing". RNA. 24 (2): 209–218. doi:10.1261/rna.064048.117. ISSN 1355-8382. PMC 5769748. PMID 29122970.
- ^ Jun Aishima, Rossitza K. Gitti, Joyce E. Noah, Hin Hark Gan, Tamar Schlick, Cynthia Wolberger (2002). "A Hoogsteen base pair embedded in undistorted B‐DNA". Nucleic Acids Res. 30 (23): 5244–5252. doi:10.1093/nar/gkf661. PMC 137974. PMID 12466549.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ a b Leontis, Neocles B; Westhof, Eric (2003). "Analysis of RNA motifs". Current Opinion in Structural Biology. 13 (3): 300–308. doi:10.1016/S0959-440X(03)00076-9. PMID 12831880.
- ^ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (December 2014). Molecular Biology of the Cell (6th ed.). New York/Abingdon: Garland Science, Taylor & Francis Group. p. 177. ISBN 978-0-8153-4432-2.
- ^ "NIH ORDR – Glossary – C". Rarediseases.info.nih.gov. Retrieved 2012-07-16.
- ^ Scott MP, Matsudaira P, Lodish H, Darnell J, Zipursky L, Kaiser CA, Berk A, Krieger M (2004). Molecular Cell Biology (Fifth ed.). San Francisco: W. H. Freeman. p. 396. ISBN 978-0-7167-4366-8.
...in humans 1 centimorgan on average represents a distance of about 7.5x105 base pairs.
추가 정보
- Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). Molecular Biology of the Gene (5th ed.). Pearson Benjamin Cummings: CSHL Press. (특히 6장 및 9장 참조)
- Sigel A, Sigel H, Sigel RK, eds. (2012). Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. Vol. 10. Springer. doi:10.1007/978-94-007-2172-2. ISBN 978-9-4007-2171-5. S2CID 92951134.
- Clever GH, Shionoya M (2012). "Chapter 10. Alternative DNA Base-Pairing through Metal Coordination". Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. Vol. 10. pp. 269–294. doi:10.1007/978-94-007-2172-2_10. ISBN 978-94-007-2171-5. PMID 22210343.
- Megger DA, Megger N, Mueller J (2012). "Chapter 11. Metal-Mediated Base Pairs in Nucleic Acids with Purine and Pyrimidine-Derived Neucleosides". Interplay between Metal Ions and Nucleic Acids. Metal Ions in Life Sciences. Vol. 10. pp. 295–317. doi:10.1007/978-94-007-2172-2_11. ISBN 978-94-007-2171-5. PMID 22210344.