RecBCD
RecBCD| 엑소데옥시리보핵산가수분해효소V | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 식별자 | |||||||||
| EC 번호 | 3.1.11.5 | ||||||||
| CAS 번호 | 37350-26-8 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| |||||||||
RecBCD(EC 3.1.11.5, Exonuclease V, E.coli exonuclease V, E.coli exonuclease V, 유전자 RecBC 엔도엔자임, RecBC 디옥시리보핵산가수분해효소, 유전자 RecBC DNase, 유전자 RECBCD 효소)는 대장균 재조합을 개시하는 효소이다.재생 오류, 엔도핵산 분해효소, 산화 손상 및 기타 여러 가지 요인.[2][3]RecBCD 효소는 DNA의 가닥을 풀거나 분리하는 헬리케이스와 DNA에 [1]단가닥 흠집을 내는 핵산가수분해효소이다.
구조.
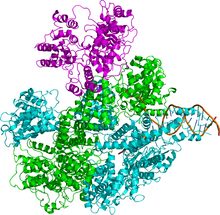
효소 복합체는 RecB, RecC, RecD라는 세 개의 서로 다른 서브유닛으로 구성되어 있으므로 복합체는 RecBCD로 명명됩니다(그림 1).recD [4]유전자가 발견되기 전에, 이 효소는 "RecBC"로 알려져 있었다.각 서브유닛은 별도의 유전자에 의해 부호화된다.
| 유전자 | 쇠사슬 | 단백질 | 기능. |
|---|---|---|---|
| 레시비 | 베타. | P08394 | 3'-5' 헬리케이스, 핵산가수분해효소 |
| 인식 | 감마 | P07648 | Chi(크로스오버 핫스팟 설치 프로그램)를 인식합니다. |
| 인식 | 알파 | P04993 | 5'-3' 헬리케이스 |
기능.
RecD 및 RecB 서브유닛은 모두 헬리케이스, 즉 DNA(또는 다른 단백질의 경우 RNA)를 풀어주는 에너지 의존 분자 모터이다.또한 RecB 서브유닛은 핵산가수분해효소 [5]기능을 가지고 있다.마지막으로 RecBCD 효소(아마 RecC 서브유닛)는 Chi(그리스 문자 χ로 지정되기도 함)로 알려진 DNA의 특정 배열인 5'-GCTGG-3'을 인식한다.
RecBCD는 서로 다른[6] 속도로 이동하는 두 개의 헬리케이스가 있고 Chi DNA [7][8]염기서열을 인식하고 변형될 수 있기 때문에 헬리케이스 중에서 특이하다.RecBCD는 선형 이중사슬(ds) DNA의 끝을 열렬히 결합시킨다.RecD 헬리케이스는 효소가 풀림을 시작하는 5' 끝의 가닥에서 이동하며, RecB는 3' 끝의 가닥에서 이동한다.RecB는 RecD보다 느리기 때문에 단일 가닥(ss) DNA 루프가 RecB보다 먼저 축적됩니다(그림 2).이것은 전자 현미경으로 [9]관찰된 2개의 ss 꼬리 (짧은 3' 끝 꼬리, 긴 5' 끝 꼬리)와 1개의 ss 루프 (3' 끝 가닥)를 가진 DNA 구조를 생성한다.ss 꼬리는 첫 번째 ss 루프를 보완하는 두 번째 ss 루프를 생성하기 위해 아닐할 수 있다. 그러한 트윈 루프 구조는 처음에는 "토끼 귀"라고 불렸다.
작용 메커니즘
RecB에서 핵산가수분해효소를 푸는 동안 반응 조건, 특히 Mg 이온과 ATP의 농도2+ 비율에 따라 다른 방식으로 작용할 수 있다. (1) ATP가 초과되면 효소는 단순히 Chi(초기 3' 끝을 가진 가닥)로 가닥을 끊는다(그림 2).[10][11]풀림이 계속되어 종점 부근에서 Chi와 함께 3's의 꼬리를 생성합니다.이 꼬리는 RecA 단백질에 의해 결합될 수 있으며, 이것은 온전한 상동 DNA [12]이중체와의 가닥 교환을 촉진합니다.RecBCD가 DNA의 말단에 도달하면 세 개의 서브유닛이 모두 분해되고 효소는 1시간 [13]이상 비활성 상태로 유지된다. Chi에서 작용한 RecBCD 분자는 다른 DNA 분자를 공격하지 않는다. (2) Mg 이온이 초과하면2+ RecBCD는 양쪽 DNA 가닥을 단핵적으로 절단하지만 (흔히 5'[14] 꼬리는 절단된다.)RecBCD가 3' 끝 가닥에서 Chi 부위와 마주치면, 풀림이 일시 중지되고 3' 꼬리의 소화가 [15]감소한다.RecBCD가 다시 풀리면 반대쪽 가닥(즉, 5' 꼬리)[16][17]을 잘라내고 RecA 단백질을 3' 끝 [12]가닥에 적재합니다.하나의 DNA 분자에 대한 반응을 완료한 후, 효소는 빠르게 두 번째 DNA를 공격하고, 첫 번째 DNA에서와 같은 반응이 일어난다.
비록 어떤 반응도 세포 내 DNA의 분석에 의해 검증되지 않았지만, 반응 중간체의 일시적인 특성 때문에, 유전적 증거는 첫 번째 반응이 [2]세포에서 그것을 더 거의 모방한다는 것을 보여준다.예를 들어, Chi의 활성은 세포 및 ATP와의 반응 모두에서 3' 측에 대한 뉴클레오티드의 영향을 받지만 Mg가 초과되는2+ 것은 아니다 [PMIDs 27401752, 27330137].검출 가능한 핵산가수분해효소 활성이 결여된 RecBCD 돌연변이는 세포 내에서 높은 Chi 핫스팟 활성을 유지하고 외부 [18]세포에서 Chi를 찌른다.세포 내 한 DNA 분자의 Chi 부위는 다른 DNA의 Chi 활동을 감소시키거나 제거하며, 아마도 과도한 ATP 조건 하에서 체외에서 관찰된 RecBCD의 Chi 의존적 분해와 [19][20]Chi에서의 DNA 조각이 반영될 것이다.
두 반응 조건 모두에서 3' 가닥은 Chi의 하류에 그대로 남아 있다.그런 다음 RecA 단백질은 RecBCD에 [12]의해 3' 꼬리에 능동적으로 부하된다.RecBCD는 떨어지지 않고 최소 60kb의 DNA를 풀 수 있지만, 어떤 미결정 지점에서 RecBCD는 DNA로부터 분리된다.RecA는 온전한 DNA 이중에서 동일하거나 거의 동일한 가닥과 결합된 DNA 가닥의 교환을 시작합니다. 이 가닥 교환은 D-루프와 같은 접합 DNA 분자를 생성합니다(그림 2).관절 DNA 분자는 침입한 3' 끝 가닥에 의해 프라이밍된 복제 또는 D-루프의 절단 및 홀리데이 접합의 형성에 의해 분해되는 것으로 생각된다.홀리데이 접합부는 RuvABC 복합체에 의해 선형 DNA로 분해되거나 RecG 단백질에 의해 분리될 수 있다.이러한 각각의 사건들은 부모의 DNA가 다를 수 있는 유전자 표지의 새로운 조합으로 온전한 DNA를 생성할 수 있다.이 과정, 상동 재조합은 이중가닥 DNA 파괴의 복구를 완료한다.
적용들
RecBCD는 단백질-DNA [21]상호작용의 기능을 더 잘 이해하기 위해 사용되는 실험 기술로서 단일 분자 형광을 사용하기 위한 모델 효소이다.효소는 또한 활동을 위해 DNA 말단이 필요하기 때문에 원형 이중 가닥 DNA의 준비에서 단일 가닥 또는 이중 가닥의 선형 DNA를 제거하는 데 유용하다.
레퍼런스
- ^ a b Singleton MR, Dillingham MS, Gaudier M, Kowalczykowski SC, Wigley DB (November 2004). "Crystal structure of RecBCD enzyme reveals a machine for processing DNA breaks". Nature. 432 (7014): 187–93. Bibcode:2004Natur.432..187S. doi:10.1038/nature02988. PMID 15538360. S2CID 2916995.
- ^ a b Smith, GR (June 2012). "How RecBCD Enzyme and Chi Promote DNA Break Repair and Recombination: a Molecular Biologist's View". Microbiol Mol Biol Rev. 76 (2): 217–28. doi:10.1128/MMBR.05026-11. PMC 3372252. PMID 22688812.
- ^ Spies M, Kowalczykowski SC (2003). "Homologous recombination by RecBCD and RecF pathways". In Higgins P (ed.). Bacterial Chromosomes. Washington, D.C: ASM Press. pp. 389–403. ISBN 1-55581-232-5.
- ^ Amundsen SK, Taylor AF, Chaudhury AM, Smith GR (August 1986). "recD: the gene for an essential third subunit of exonuclease V." Proc Natl Acad Sci U S A. 83 (15): 5558–62. Bibcode:1986PNAS...83.5558A. doi:10.1073/pnas.83.15.5558. PMC 386327. PMID 3526335.
- ^ Yu M, Souaya J, Julin DA (February 1998). "The 30-kDa C-terminal domain of the RecB protein is critical for the nuclease activity, but not the helicase activity, of the RecBCD enzyme from Escherichia coli". Proc Natl Acad Sci U S A. 95 (3): 981–6. doi:10.1073/pnas.95.3.981. PMC 18645. PMID 9448271.
- ^ Taylor AF, Smith GR (June 2003). "RecBCD enzyme is a DNA helicase with fast and slow motors of opposite polarity". Nature. 423 (6942): 889–93. Bibcode:2003Natur.423..889T. doi:10.1038/nature01674. PMID 12815437. S2CID 4302346.
- ^ Taylor AF, Smith GR (June 1992). "RecBCD enzyme is altered upon cutting DNA at a Chi recombination hotspot". Proc Natl Acad Sci U S A. 89 (12): 5226–30. Bibcode:1992PNAS...89.5226T. doi:10.1073/pnas.89.12.5226. PMC 49264. PMID 1535156.
- ^ Amundsen SK, Taylor AF, Reddy M, Smith GR (December 2007). "Intersubunit signaling in RecBCD enzyme, a complex protein machine regulated by Chi hot spots". Genes Dev. 21 (24): 3296–307. doi:10.1101/gad.1605807. PMC 2113030. PMID 18079176.
- ^ Taylor A, Smith GR (November 1980). "Unwinding and rewinding of DNA by the RecBC enzyme". Cell. 22 (2 Pt 2): 447–57. doi:10.1016/0092-8674(80)90355-4. PMID 6256081. S2CID 22123298.
- ^ Ponticelli, A. S.; Schultz, D. W.; Taylor, A. F.; Smith, G. R. (May 1985). "Chi-dependent DNA strand cleavage by RecBC enzyme". Cell. 41 (1): 145–151. doi:10.1016/0092-8674(85)90069-8. PMID 3888404. S2CID 38361329.
- ^ Taylor AF, Schultz DW, Ponticelli AS, Smith GR (May 1985). "RecBC enzyme nicking at Chi sites during DNA unwinding: location and orientation-dependence of the cutting". Cell. 41 (1): 153–63. doi:10.1016/0092-8674(85)90070-4. PMID 3888405. S2CID 8551482.
- ^ a b c Anderson DG, Kowalczykowski SC (July 1997). "The translocating RecBCD enzyme stimulates recombination by directing RecA protein onto ssDNA in a Chi-regulated manner". Cell. 90 (1): 77–86. doi:10.1016/S0092-8674(00)80315-3. PMID 9230304.
- ^ Taylor AF, Smith GR (April 1999). "Regulation of homologous recombination: Chi inactivates RecBCD enzyme by disassembly of the three subunits". Genes Dev. 13 (7): 890–900. doi:10.1101/gad.13.7.890. PMC 316601. PMID 10197988.
- ^ Dixon DA, Kowalczykowski SC (April 1993). "The recombination hotspot Chi is a regulatory sequence that acts by attenuating the nuclease activity of the E. coli RecBCD enzyme". Cell. 73 (1): 87–96. doi:10.1016/0092-8674(93)90162-J. PMID 8384931. S2CID 19718817.
- ^ Spies M, Amitani I, Baskin RJ, Kowalczykowski SC (November 2007). "RecBCD enzyme switches lead motor subunits in response to Chi recognition". Cell. 131 (4): 694–705. doi:10.1016/j.cell.2007.09.023. PMC 2151923. PMID 18022364.
- ^ Taylor AF, Smith GR (October 1995). "Strand specificity of nicking of DNA at Chi sites by RecBCD enzyme. Modulation by ATP and magnesium levels". J Biol Chem. 270 (41): 24459–67. doi:10.1074/jbc.270.41.24459. PMID 7592661.
- ^ Anderson DG, Kowalczykowski SC (March 1997). "The recombination hot spot chi is a regulatory element that switches the polarity of DNA degradation by the RecBCD enzyme". Genes Dev. 11 (5): 571–81. doi:10.1101/gad.11.5.571. PMID 9119222.
- ^ Amundsen SK, Smith GR (January 2007). "Chi hotspot activity in Escherichia coli without RecBCD exonuclease activity: implications for the mechanism of recombination". Genetics. 175 (1): 41–54. doi:10.1534/genetics.106.065524. PMC 1774988. PMID 17110484.
- ^ Köppen A, Krobitsch S, Thoms B, Wackernagel W (July 1995). "Interaction with the recombination hot spot Chi in vivo converts the RecBCD enzyme of Escherichia coli into a Chi-independent recombinase by inactivation of the RecD subunit". Proc Natl Acad Sci U S A. 92 (14): 6249–53. Bibcode:1995PNAS...92.6249K. doi:10.1073/pnas.92.14.6249. PMC 41495. PMID 7541534.
- ^ Myers RS, Kuzminov A, Stahl FW (July 1995). "The recombination hot spot Chi activates RecBCD recombination by converting Escherichia coli to a recD mutant phenocopy". Proc Natl Acad Sci U S A. 92 (14): 6244–8. Bibcode:1995PNAS...92.6244M. doi:10.1073/pnas.92.14.6244. PMC 41494. PMID 7603978.
- ^ Bianco PR, Brewer LR, Corzett M, Balhorn R, Yeh Y, Kowalczykowski SC, Baskin RJ (January 2001). "Processive translocation and DNA unwinding by individual RecBCD enzyme molecules". Nature. 409 (6818): 374–8. Bibcode:2001Natur.409..374B. doi:10.1038/35053131. PMID 11201750. S2CID 4399125.
외부 링크
- 미국 국립 의학 도서관(MeSH)의 엑소데옥시리보핵산가수분해효소+V
- 미국 국립 의학 도서관(MeSH)의 Exodexyribonuclease+V,+E+coli
- EC 3.1.11.5