RAD52
RAD52| RAD52 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 식별자 | |||||||||||||||||||||||||
| 별칭 | RAD52, RAD52 호몰로고, DNA 수리 단백질 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 600392 MGI: 101949 호몰로진: 31118 GeneCard: RAD52 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 | |||||||||||||||||||||||||
| 앙상블 | |||||||||||||||||||||||||
| 유니프로트 | |||||||||||||||||||||||||
| RefSeq(mRNA) |
| ||||||||||||||||||||||||
| RefSeq(단백질) | |||||||||||||||||||||||||
| 위치(UCSC) | Chr 12: 0.91 – 0.99Mb | Chr 6: 119.88 – 119.9Mb | |||||||||||||||||||||||
| PubMed 검색 | [3] | [4] | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
RAD52 호몰로컬(S. cerebisiae)은 인간에서 RAD52 유전자에 의해 인코딩되는 단백질이다.[5][6]
함수
이 유전자에 의해 암호화된 단백질은 DNA 이중 가닥 파괴 수리와 동음이의 재조합에 중요한 단백질인 사카로마이오스 세레비시아에 Rad52와 유사성을 공유한다.이 유전자 생산물은 단일 가닥의 DNA 끝을 결합하고 보완 DNA 가닥의 분리에 필요한 DNA-DNA 상호작용을 중재하는 것으로 나타났다.또한 RAD51 관련 DNA 재조합 및 보수에 대한 역할을 제시한 DNA 재조합 단백질 RAD51과 상호작용하는 것으로 밝혀졌다.[6]
DNA 재조합 수리의 역할
RAD52는 RAD51 기능을 효모 사카로마이오스 세레비시아와 생쥐와 인간의 포유류 세포 모두에서 동음이의 재조합 수리(HRRR)로 매개한다.그러나 RAD52 단백질은 효모와 인간의 HRR에서 뚜렷하게 다른 기능을 가지고 있다.S. 세레비사에서는 Rad52 단백질이 단독으로 작용하여 Rad51 단백질을 재조합 전 단계에서 복제 단백질 A로 미리 코팅된 단일 가닥 DNA에 쉽게 로딩한다.[7][8]
그러나 생쥐와 인간의 경우 BRCA2는 주로 동음이의 쌍과 가닥 침투를 위해 활동하는 형태인 ssDNA에 대한 RAD51의 질서 있는 조립을 중재한다.[9]또한 BRCA2는 RAD51을 dsDNA에서 리디렉션하고 ssDNA와의 분리를 방지한다.[9]또한 RAD51B(RAD51L1) RAD51C(RAD51L2) RAD51D(RAD51L3) XRCC2의 4개의 파라로그는 BCDX2 콤플렉스라고 불리는 콤플렉스를 형성한다.이 단지는 RAD51 채용이나 피해현장 안정화에 참여한다.[10]BCDX2 복합체는 RAD51 뉴클레오프로틴 필라멘트의 조립이나 안정성을 촉진하여 작용하는 것으로 보인다.단, BRCA2 돌연변이가 있는 경우 인간 RAD52는 RAD51 조립체를 ssDNA에 중재할 수 있으며,[11] BRCA2보다 효율은 낮지만 동음이의 재조합 DNA 복구에서 BRCA2를 대체할 수 있다.
또한 인간 RAD52는 ERCC1과 결합하여 단일 가닥의 오류 발생 가능성이 있는 동질 DNA 수리 경로를 촉진한다.[12]오류가 발생하기 쉬우나 달리 복구할 수 없는 DNA 손상이 있는 세포의 생존을 위해 이 수리 경로가 필요할 수 있다.
인간 RAD52는 또한 세포 주기의 G0/G1 단계 동안 활성 전사 현장에서 DNA 이중 스트랜드 파손의 복구에 중요한 역할을 한다.이러한 이중 가닥 파손의 수리는 RAD52에 의존하는 RNA 템플릿 기반 재결합 메커니즘을 사용하는 것으로 보인다.[13]코카인 신드롬 B 단백질(ERCC6에 의해 코드화됨)은 활성 전사 현장의 이중 스트랜드 브레이크에서 국소화되며, RAD51C, RAD52에 이어 RAD51C, RAD52가 새로 합성된 RNA를 템플릿으로 사용하여 동종 재조합 수리를 수행한다.[13]
마이크로RNA와 암 위험
전령 RNA(mRNA)의 세 가지 주요 비번역 영역(3'UTR)은 종종 전승 후 RNA 음소화를 유발할 수 있는 규제 시퀀스를 포함하고 있다.그러한 3'-UTR은 종종 miRNA(microRNA)를 위한 결합 사이트를 포함한다.miRNA는 3'-UTR 내의 특정 사이트에 결합함으로써 번역을 억제하거나 성적서의 저하를 직접 유발하여 다양한 mRNA의 유전자 발현을 감소시킬 수 있다.
마이크로RNA(miRNAs)는 인간 게놈의 단백질 코딩 유전자의 60% 이상의 발현을 조절하는 것으로 보인다.[14]마이크로RNA 1대 miR-210은 RAD52를 누른다.[15]데블린 외 연구진에서 언급한 바와 같이 miR-210은 대부분의 고형 종양에서 상향 조절되며 임상 결과에 부정적인 영향을 미친다.[16]
또한 RAD52의 3'-UTR에는 마이크로RNA let-7의 바인딩 사이트가 있다.let-7의 결합을 감소시키는 let-7(rs7963551)의 결합 부위에서 단일 뉴클레오티드 다형성(SNP)을 가진 여성은 RAD52(간에서[17] 이 SNP에서 나타난 바와 같이)의 발현이 증가했을 가능성이 있다.3인방의 이 SNP를 가진 여성들.RAD52의 UTR는 승산비 0.84, 95% 신뢰구간 0.75-0.95로 유방암 위험이 감소하는 것으로 나타났다.[18]
한족 인구에서, let-7 (rs7963551)에 대한 RAD52 결합 사이트의 3'-UTR에서 위와 같은 SNP는 교모종의 위험을 줄였다.RAD52 rs7963551 유전자형과 관련된 교모종의 위험은 41세 이상에서는 오즈비율이 0.44(SNP가 없는 것과 비교), 41세 이하에서는 오즈비율이 0.58이었다.[19]
Li 외 [17]연구진은 RAD52 rs7963551 CC 유전자형(위의 동일한 SNP)을 가진 개인들 사이에서 간세포암 위험이 중국 인구의 AA 유전자형에 비해 현저히 감소했음을 발견했다.그들은 또한 44개의 정상적인 인간 간 조직 샘플에서 rs7963551 SNP의 존재는 RAD52 mRNA 발현의 상당한 증가와 관련이 있다는 것을 발견했다.
따라서 증가된 RAD52 표현은 다양한 암에 대해 보호된다.
RAD52에서 변경된 마이크로RNA 결합 부위와 암 발생성에 미치는 영향에 대한 또 다른 연구는 Naccarati 등이 수행했다.[20]그들은 자주 변형되어 대장암 위험에 영향을 미친 RAD52 마이크로RNA 결합 사이트 두 곳을 발견했다.rs1051669에서 동질성 또는 이질성 SNP를 가진 개인은 대장암에 걸릴 위험이 증가했다(또는 동질성의 경우 OR 1.78, 95% CI 1.13–2.80, p = 0.01, 그리고 이성질의 경우 OR 1.72, 95% CI 1.10–2.692, p = 0.02).다른 RAD52 SNP(rs11571475)의 이질성 운반체는 대장암 위험 감소에 있었다(OR 0.76, 95% CI 0.58–1.00, p = 0.05).검사한 동질 재조합 수리 경로의 21개 유전자와 비동질 종단 접합 경로의 7개 유전자 중 RAD52의 2개 유전자와 MRE11A의 1개 유전자는 모두 평가할 수 있을 만큼 높은 빈도에 있고 대장암의 위험에 영향을 미친 마이크로RNA 결합 영역에서 발견된 유일한 SNP였다.
DNA 손상은 암의 근본적인 원인인 것으로 보이며,[21] DNA 수리의 결핍은 많은 형태의 암의 기초가 되는 것으로 보인다.[22]DNA 수리가 부족하면 DNA 손상이 누적되는 경향이 있다.그러한 과도한 DNA 손상은 오류가 발생하기 쉬운 전이 합성 때문에 DNA 복제 중 돌연변이 오류를 증가시킬 수 있다.과도한 DNA 손상은 또한 DNA 수리 중 오류로 인한 후생유전적 변화를 증가시킬 수 있다.[23][24]그러한 돌연변이와 후생적 변화는 암을 유발할 수 있다.마이크로RNA 결합변형으로 인한 RAD52 매개 DNA 수리의 빈번한 증가나 결핍은 유방암, 뇌암, 간암 또는 대장암의 예방이나 진행에 기여할 가능성이 있다.
상호작용
RAD52는 RAD51과 상호작용하는 것으로 나타났다.[25]Rad52는 RPA 단백질을 간섭함으로써 ssDNA에 대한 Rad51의 하중을 완화할 것이다.
내적 보완
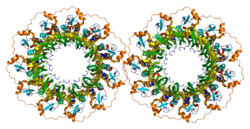
유전자에 의해 인코딩된 폴리펩타이드의 여러 복사본이 골재를 형성할 때, 이 단백질 구조를 멀티머라고 한다.멀티머가 특정 유전자의 서로 다른 두 돌연변이 알레르기에 의해 생성된 폴리펩타이드로 형성되는 경우, 혼합 멀티머는 각각의 돌연변이만으로 형성된 비혼합 멀티머보다 더 큰 기능적 활성을 보일 수 있다.이 경우 그 현상을 '내부 보완'이라고 한다.C단자 잘린 단백질을 나타내는 사카로마이오스 세레비시아래 RAD52 돌연변이 알레르기가 다른 RAD52 돌연변이 미스 알레르기를 보완하는 것으로 밝혀졌다.[26]이러한 유전자 내 보완의 발견은 RAD52 단백질이 구성 단량체 사이의 상호작용을 가능하게 하는 복합체 구조를 가지고 있음을 시사한다.
참조
- ^ a b c GRCh38: 앙상블 출시 89: ENSG00000002016 - 앙상블, 2017년 5월
- ^ a b c GRCm38: 앙상블 릴리스 89: ENSMUSG000030166 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Mouse PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ Shen Z, Denison K, Lobb R, Gatewood JM, Chen DJ (Jan 1995). "The human and mouse homologs of the yeast RAD52 gene: cDNA cloning, sequence analysis, assignment to human chromosome 12p12.2-p13, and mRNA expression in mouse tissues". Genomics. 25 (1): 199–206. doi:10.1016/0888-7543(95)80126-7. PMID 7774919.
- ^ a b "Entrez Gene: RAD52 RAD52 homolog (S. cerevisiae)".
- ^ Shinohara A, Ogawa T (1998). "Stimulation by Rad52 of yeast Rad51-mediated recombination". Nature. 391 (6665): 404–7. doi:10.1038/34943. PMID 9450759. S2CID 4304549.
- ^ New JH, Sugiyama T, Zaitseva E, Kowalczykowski SC (1998). "Rad52 protein stimulates DNA strand exchange by Rad51 and replication protein A". Nature. 391 (6665): 407–10. doi:10.1038/34950. PMID 9450760. S2CID 4408959.
- ^ a b Holloman WK (2011). "Unraveling the mechanism of BRCA2 in homologous recombination". Nat. Struct. Mol. Biol. 18 (7): 748–54. doi:10.1038/nsmb.2096. PMC 3647347. PMID 21731065.
- ^ Chun J, Buechelmaier ES, Powell SN (2013). "Rad51 paralog complexes BCDX2 and CX3 act at different stages in the BRCA1-BRCA2-dependent homologous recombination pathway". Mol. Cell. Biol. 33 (2): 387–95. doi:10.1128/MCB.00465-12. PMC 3554112. PMID 23149936.
- ^ Feng Z, Scott SP, Bussen W, Sharma GG, Guo G, Pandita TK, Powell SN (2011). "Rad52 inactivation is synthetically lethal with BRCA2 deficiency". Proc. Natl. Acad. Sci. U.S.A. 108 (2): 686–91. doi:10.1073/pnas.1010959107. PMC 3021033. PMID 21148102.
- ^ Stark JM, Pierce AJ, Oh J, Pastink A, Jasin M (2004). "Genetic steps of mammalian homologous repair with distinct mutagenic consequences". Mol. Cell. Biol. 24 (21): 9305–16. doi:10.1128/MCB.24.21.9305-9316.2004. PMC 522275. PMID 15485900.
- ^ a b Wei L, Nakajima S, Böhm S, Bernstein KA, Shen Z, Tsang M, Levine AS, Lan L (2015). "DNA damage during the G0/G1 phase triggers RNA-templated, Cockayne syndrome B-dependent homologous recombination". Proc. Natl. Acad. Sci. U.S.A. 112 (27): E3495–504. doi:10.1073/pnas.1507105112. PMC 4500203. PMID 26100862.
- ^ Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Crosby ME, Kulshreshtha R, Ivan M, Glazer PM (2009). "MicroRNA regulation of DNA repair gene expression in hypoxic stress". Cancer Res. 69 (3): 1221–9. doi:10.1158/0008-5472.CAN-08-2516. PMC 2997438. PMID 19141645.
- ^ Devlin C, Greco S, Martelli F, Ivan M (2011). "miR-210: More than a silent player in hypoxia". IUBMB Life. 63 (2): 94–100. doi:10.1002/iub.427. PMC 4497508. PMID 21360638.
- ^ a b Li Z, Guo Y, Zhou L, Ge Y, Wei L, Li L, Zhou C, Wei J, Yuan Q, Li J, Yang M (2015). "Association of a functional RAD52 genetic variant locating in a miRNA binding site with risk of HBV-related hepatocellular carcinoma". Mol. Carcinog. 54 (9): 853–8. doi:10.1002/mc.22156. PMID 24729511. S2CID 25174260.
- ^ Jiang Y, Qin Z, Hu Z, Guan X, Wang Y, He Y, Xue J, Liu X, Chen J, Dai J, Jin G, Ma H, Wang S, Shen H (2013). "Genetic variation in a hsa-let-7 binding site in RAD52 is associated with breast cancer susceptibility". Carcinogenesis. 34 (3): 689–93. doi:10.1093/carcin/bgs373. PMID 23188672.
- ^ Lu C, Chen YD, Han S, Wei J, Ge Y, Pan W, Jiang T, Qiu XG, Yang M (2014). "A RAD52 genetic variant located in a miRNA binding site is associated with glioma risk in Han Chinese". J. Neurooncol. 120 (1): 11–7. doi:10.1007/s11060-014-1527-x. PMID 25012956. S2CID 1082923.
- ^ Naccarati A, Rosa F, Vymetalkova V, Barone E, Jiraskova K, Di Gaetano C, Novotny J, Levy M, Vodickova L, Gemignani F, Buchler T, Landi S, Vodicka P, Pardini B (2015). "Double-strand break repair and colorectal cancer: gene variants within 3' UTRs and microRNAs binding as modulators of cancer risk and clinical outcome". Oncotarget. 7 (17): 23156–69. doi:10.18632/oncotarget.6804. PMC 5029617. PMID 26735576.
- ^ Kastan MB (2008). "DNA damage responses: mechanisms and roles in human disease: 2007 G.H.A. Clowes Memorial Award Lecture". Mol. Cancer Res. 6 (4): 517–24. doi:10.1158/1541-7786.MCR-08-0020. PMID 18403632.
- ^ Harper JW, Elledge SJ (2007). "The DNA damage response: ten years after". Mol. Cell. 28 (5): 739–45. doi:10.1016/j.molcel.2007.11.015. PMID 18082599.
- ^ O'Hagan HM, Mohammad HP, Baylin SB (2008). "Double strand breaks can initiate gene silencing and SIRT1-dependent onset of DNA methylation in an exogenous promoter CpG island". PLOS Genetics. 4 (8): e1000155. doi:10.1371/journal.pgen.1000155. PMC 2491723. PMID 18704159.
- ^ Cuozzo C, Porcellini A, Angrisano T, Morano A, Lee B, Di Pardo A, Messina S, Iuliano R, Fusco A, Santillo MR, Muller MT, Chiariotti L, Gottesman ME, Avvedimento EV (Jul 2007). "DNA damage, homology-directed repair, and DNA methylation". PLOS Genetics. 3 (7): e110. doi:10.1371/journal.pgen.0030110. PMC 1913100. PMID 17616978.
- ^ Chen G, Yuan SS, Liu W, Xu Y, Trujillo K, Song B, Cong F, Goff SP, Wu Y, Arlinghaus R, Baltimore D, Gasser PJ, Park MS, Sung P, Lee EY (Apr 1999). "Radiation-induced assembly of Rad51 and Rad52 recombination complex requires ATM and c-Abl" (PDF). The Journal of Biological Chemistry. 274 (18): 12748–52. doi:10.1074/jbc.274.18.12748. PMID 10212258. S2CID 2587580.
- ^ Livingston DM, Boundy-Mills KL. Saccharomyces serebisiae RAD52 allele은 C-단자 절단 단백질: 활동과 오감 변이의 유전적 보완을 표현한다.유전학.1993;133(1):39-49.
추가 읽기
- Muris DF, Bezzubova O, Buerstedde JM, Vreeken K, Balajee AS, Osgood CJ, Troelstra C, Hoeijmakers JH, Ostermann K, Schmidt H (Nov 1994). "Cloning of human and mouse genes homologous to RAD52, a yeast gene involved in DNA repair and recombination". Mutation Research. 315 (3): 295–305. doi:10.1016/0921-8777(94)90040-x. hdl:1765/3072. PMID 7526206.
- Shen Z, Denison K, Lobb R, Gatewood JM, Chen DJ (Jan 1995). "The human and mouse homologs of the yeast RAD52 gene: cDNA cloning, sequence analysis, assignment to human chromosome 12p12.2-p13, and mRNA expression in mouse tissues". Genomics. 25 (1): 199–206. doi:10.1016/0888-7543(95)80126-7. PMID 7774919.
- Park MS (Jun 1995). "Expression of human RAD52 confers resistance to ionizing radiation in mammalian cells". The Journal of Biological Chemistry. 270 (26): 15467–70. doi:10.1074/jbc.270.26.15467. PMID 7797537.
- Shen Z, Pardington-Purtymun PE, Comeaux JC, Moyzis RK, Chen DJ (Sep 1996). "UBL1, a human ubiquitin-like protein associating with human RAD51/RAD52 proteins". Genomics. 36 (2): 271–9. doi:10.1006/geno.1996.0462. PMID 8812453.
- Shen Z, Pardington-Purtymun PE, Comeaux JC, Moyzis RK, Chen DJ (Oct 1996). "Associations of UBE2I with RAD52, UBL1, p53, and RAD51 proteins in a yeast two-hybrid system". Genomics. 37 (2): 183–6. doi:10.1006/geno.1996.0540. PMID 8921390.
- Chen G, Yuan SS, Liu W, Xu Y, Trujillo K, Song B, Cong F, Goff SP, Wu Y, Arlinghaus R, Baltimore D, Gasser PJ, Park MS, Sung P, Lee EY (Apr 1999). "Radiation-induced assembly of Rad51 and Rad52 recombination complex requires ATM and c-Abl" (PDF). The Journal of Biological Chemistry. 274 (18): 12748–52. doi:10.1074/jbc.274.18.12748. PMID 10212258. S2CID 2587580.
- Kito K, Wada H, Yeh ET, Kamitani T (Dec 1999). "Identification of novel isoforms of human RAD52". Biochimica et Biophysica Acta (BBA) - Gene Structure and Expression. 1489 (2–3): 303–14. doi:10.1016/s0167-4781(99)00214-6. PMID 10673031.
- Stasiak AZ, Larquet E, Stasiak A, Müller S, Engel A, Van Dyck E, West SC, Egelman EH (Mar 2000). "The human Rad52 protein exists as a heptameric ring". Current Biology. 10 (6): 337–40. doi:10.1016/S0960-9822(00)00385-7. PMID 10744977. S2CID 16087838.
- Parsons CA, Baumann P, Van Dyck E, West SC (Aug 2000). "Precise binding of single-stranded DNA termini by human RAD52 protein". The EMBO Journal. 19 (15): 4175–81. doi:10.1093/emboj/19.15.4175. PMC 306603. PMID 10921897.
- Mer G, Bochkarev A, Gupta R, Bochkareva E, Frappier L, Ingles CJ, Edwards AM, Chazin WJ (Oct 2000). "Structural basis for the recognition of DNA repair proteins UNG2, XPA, and RAD52 by replication factor RPA". Cell. 103 (3): 449–56. doi:10.1016/S0092-8674(00)00136-7. PMID 11081631. S2CID 16640087.
- Ranatunga W, Jackson D, Flowers II RA, Borgstahl GE (Jul 2001). "Human RAD52 protein has extreme thermal stability". Biochemistry. 40 (29): 8557–62. doi:10.1021/bi0155089. PMID 11456495.
- Van Dyck E, Stasiak AZ, Stasiak A, West SC (Oct 2001). "Visualization of recombination intermediates produced by RAD52-mediated single-strand annealing". EMBO Reports. 2 (10): 905–9. doi:10.1093/embo-reports/kve201. PMC 1084079. PMID 11571269.
- Kim PM, Allen C, Wagener BM, Shen Z, Nickoloff JA (Nov 2001). "Overexpression of human RAD51 and RAD52 reduces double-strand break-induced homologous recombination in mammalian cells". Nucleic Acids Research. 29 (21): 4352–60. doi:10.1093/nar/29.21.4352. PMC 60192. PMID 11691922.
- Yáñez RJ, Porter AC (Feb 2002). "Differential effects of Rad52p overexpression on gene targeting and extrachromosomal homologous recombination in a human cell line". Nucleic Acids Research. 30 (3): 740–8. doi:10.1093/nar/30.3.740. PMC 100286. PMID 11809887.
- Jackson D, Dhar K, Wahl JK, Wold MS, Borgstahl GE (Aug 2002). "Analysis of the human replication protein A:Rad52 complex: evidence for crosstalk between RPA32, RPA70, Rad52 and DNA". Journal of Molecular Biology. 321 (1): 133–48. doi:10.1016/S0022-2836(02)00541-7. PMID 12139939.
- Kagawa W, Kurumizaka H, Ishitani R, Fukai S, Nureki O, Shibata T, Yokoyama S (Aug 2002). "Crystal structure of the homologous-pairing domain from the human Rad52 recombinase in the undecameric form". Molecular Cell. 10 (2): 359–71. doi:10.1016/S1097-2765(02)00587-7. PMID 12191481.
- Singleton MR, Wentzell LM, Liu Y, West SC, Wigley DB (Oct 2002). "Structure of the single-strand annealing domain of human RAD52 protein". Proceedings of the National Academy of Sciences of the United States of America. 99 (21): 13492–7. doi:10.1073/pnas.212449899. PMC 129701. PMID 12370410.
- Liu J, Meng X, Shen Z (Oct 2002). "Association of human RAD52 protein with transcription factors". Biochemical and Biophysical Research Communications. 297 (5): 1191–6. doi:10.1016/S0006-291X(02)02353-7. PMID 12372413.
- Han J, Hankinson SE, De Vivo I, Colditz GA, Hunter DJ (Oct 2002). "No association between a stop codon polymorphism in RAD52 and breast cancer risk". Cancer Epidemiology, Biomarkers & Prevention. 11 (10 Pt 1): 1138–9. PMID 12376524.
- Kitao H, Yuan ZM (Dec 2002). "Regulation of ionizing radiation-induced Rad52 nuclear foci formation by c-Abl-mediated phosphorylation". The Journal of Biological Chemistry. 277 (50): 48944–8. doi:10.1074/jbc.M208151200. PMID 12379650.