에세리키아 바이러스 T4
Escherichia virus T4
| 에세리키아 바이러스 T4 | |
|---|---|
 | |
| Escherichia 바이러스 T4 (비리온의 EM of virion) | |
| 바이러스 분류 | |
| (순위 없음): | 바이러스 |
| 영역: | 듀플로드나비리아 |
| 킹덤: | 흥공비래 |
| 문: | 우로비코타주 |
| 클래스: | 카우도비리케테스 |
| 순서: | 카우도비랄레스속 |
| 가족: | 묘비루스과 |
| 속: | 테콰트로바이러스 |
| 종: | 에세리키아 바이러스 T4 |
| 스트레인스[2] | |
| 동의어[3] | |
| 장내세균 T4 | |
대장균 바이러스 T4는 대장균 박테리아를 감염시키는 박테리오파지의 한 종류입니다.이것은 묘비루스과의 테벤비루스아과에 속하는 이중가닥 DNA 바이러스입니다.T4는 용해성 수명 주기가 아닌 분해성 수명 주기만을 거칠 수 있습니다.이 종은 이전에 T-even bacteriophage로 명명되었는데, 이 이름은 다른 균주들 중에서도 (또는 분리된), 장박테리아 T2, 장박테리아 T4 및 장박테리아 T6를 포함합니다.
연구에 사용
1940년대로 거슬러 올라가 오늘날에도 계속되고 있는 T-even 파지는 가장 잘 연구된 모형 생물로 여겨집니다.모델 생물체는 보통 5개 이하의 유전자로 단순해야 합니다.그러나 T-even 파지는 사실 이 파지의 유전 정보가 약 300개의 유전자로 구성되어 있는 가장 크고 복잡한 바이러스 중 하나입니다.T-even 바이러스는 그 복잡성과 동시에 핵산 염기인 사이토신 대신 특이한 염기인 하이드록시메틸사이토신(HMC)을 가지고 있는 것으로 밝혀졌습니다.[citation needed]
게놈과 구조
T4 바이러스의 이중 가닥 DNA 게놈은 약 169kbp 길이이며[4] 289개의 단백질을 암호화합니다.T4 유전체는 말기적으로 중복되어 있습니다.DNA 복제 시, 긴 다중 게놈 길이의 컨캐터가 형성되며, 아마도 복제의 롤링 서클 메커니즘에 의해 형성됩니다.[5]포장 시, 콘캐터는 동일한 길이의 불특정 위치에서 절단되어 원본의 원형 순열을 나타내는 여러 게놈으로 이어집니다.[6]T4 유전체는 진핵세포와 유사한 인트론 서열을 지니고 있습니다.
번역.
샤인-달가노 서열 GAGGG는 바이러스 T4 초기 유전자에서 지배적인 반면, 서열 GGAG는 초기 mRNA 분해를 개시하는 T4 엔도뉴클레아제 RegB의 표적입니다.[7]
바이러스입자구조

T4는 폭이 약 90nm이고 길이가 약 200nm인 비교적 큰 바이러스입니다(대부분 바이러스의 길이는 25~200nm입니다).DNA 게놈은 캡시드라고도 알려진 정이십면체 머리에 담겨 있습니다.[8]T4의 꼬리는 속이 비어 있어서 부착 후에 감염된 세포로 핵산을 전달할 수 있습니다.T4와 같은 Myoviridae 파지는 꼬리의 집합체와 기능에 관여하는 많은 수의 단백질을 가진 복잡한 수축 꼬리 구조를 가지고 있습니다.[9]꼬리 섬유는 숙주 세포 표면 수용체를 인식하는데도 중요해서, 박테리아가 바이러스의 숙주 범위 안에 있는지를 결정합니다.[10]
13개의 상이한 단백질(유전자 산물 5, 5.4, 6, 7, 8, 9, 10, 11, 12, 25, 27, 48 및 53)의 127개의 폴리펩티드 사슬을 포함하는 6메가달톤 T4 염기판의 구조가 최근 원자적으로 상세하게 기술되었다.gp54 및 주관 단백질 gp19에 의해 형성된 꼬리관의 근위 영역의 원자 모델 또한 생성되었습니다.테이프 측정 단백질 gp29는 베이스 플레이트-테일 튜브 복합체에 존재하지만 모델링할 수 없었습니다.[11]
박테리오파지 (파지) T4 비리온의 조립 동안, 파지 유전자에 의해 암호화된 형태유전학적 단백질은 특징적인 서열로 서로 상호작용합니다.바이러스 감염 동안 생성된 이러한 단백질 각각의 양에서 적절한 균형을 유지하는 것은 정상적인 파지 T4 형태 생성에 매우 중요한 것으로 보입니다.[12]비리온 구조를 결정하는 파지 T4 암호화된 단백질은 주요 구조적 성분, 작은 구조적 성분 및 형태생성 서열의 특정 단계를 촉매하는 비구조적 단백질을 포함합니다.[13]페이지 T4 형태형성은 Yap과 Rossman에 의해 상세히 설명된 것처럼 머리, 꼬리 그리고 긴 꼬리 섬유의 세 가지 독립적인 경로로 나뉩니다.[14]
감염과정
T4 바이러스는 대장균 세포 표면에 OmpC 포린 단백질과 리포다당류(LPS)를 긴 꼬리 섬유(LTF)로 결합시켜 대장균 감염을 시작합니다.[15][16]인식 신호는 LTF를 통해 베이스 플레이트로 전송됩니다.이것은 대장균 세포 표면에 비가역적으로 결합하는 짧은 꼬리 섬유(STF)를 풀어줍니다.베이스 플레이트는 입체구조를 변화시키고 테일 시스는 수축하여 테일 튜브의 끝에 있는 GP5가 세포의 외막에 구멍을 내게 합니다.[17]GP5의 리소자임 도메인은 활성화되고 주변 펩티도글리칸 층을 분해합니다.막의 남은 부분이 분해되고 바이러스의 머리에서 나온 DNA가 꼬리관을 통해 이동하여 대장균 세포로 들어갈 수 있습니다.[citation needed]
1952년 허쉬와 체이스는[18] 파지 DNA가 단백질과 구별되는 숙주 세균 세포로 들어가 감염시 파지의 유전적 물질이라는 주요 증거를 제시했습니다.이 발견은 일반적으로 DNA가 다른 유기체의 유전 물질이라는 것을 암시했습니다.[citation needed]
생식
세균에 들어갈 때부터 파괴될 때까지 용해 수명 주기는 약 30분(37°C)이 걸립니다.독이 있는 박테리오파지는 들어온 직후 세균 숙주에서 증식합니다.자손의 수가 일정량에 도달하면 숙주가 용해되거나 분해되기 때문에 새로운 숙주 세포가 방출되고 감염됩니다.[19]숙주가 용해되고 방출되는 과정을 용해 주기라고 합니다.라이틱 사이클(Lytic cycle)은 감염된 세포와 세포막의 파괴를 수반하는 바이러스 번식의 사이클입니다.이 주기는 숙주세포와 그 기계를 제치고 번식하는 바이러스를 포함합니다.따라서 숙주세포를 번식시키고 감염시키기 위해서는 바이러스가 5단계를 거쳐야 합니다.[citation needed]
라이프 사이클이 완료된 후 호스트 셀은 터져서 새로 구축된 바이러스를 환경으로 방출하여 호스트 셀을 파괴합니다.T4는 감염된 숙주 당 약 100-150개의 바이러스 입자의 폭발 크기를 가지고 있습니다.[citation needed]
Benzer (1955–1959)는 rIIA 및 rIIB 유전자에 결함이 있는 박테리오파지 T4 돌연변이를 이용하여 유전자의 미세 구조를 연구하기 위한 시스템을 개발했습니다.[20][21][22]재조합, 특히 결실 돌연변이 간의 재조합을 검출하기 위해 사용된 기술은 보완 테스트와 교차였습니다.이러한 유전자 실험은 유전자 안에서 돌연변이 부위의 독특한 선형 순서를 발견하게 했습니다.이 결과는 유전자가 독립적으로 돌연변이를 일으킬 수 있는 많은 부위를 가진 DNA의 길이와 동일한 선형 구조를 가지고 있다는 핵심 아이디어에 대한 강력한 증거를 제공했습니다.[citation needed]
흡착 및 침투
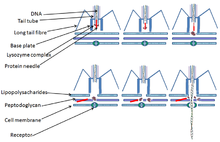
다른 모든 바이러스와 마찬가지로, T-even 파지는 숙주의 표면에 무작위로 붙지 않고, 대신 숙주의 표면에서 발견되는 특정한 단백질 구조인 수용체에 "검색"되어 결합합니다.이 수용체들은 파지에 따라 다양한데, 테이초산, 세포벽 단백질과 지방다당류, 편모, 그리고 필라 모두 파지가 결합하는 수용체 역할을 할 수 있습니다.T-even 파지가 숙주를 감염시키고 수명 주기를 시작하기 위해서는 첫 번째 감염 과정인 세균 세포에 파지를 흡착하는 과정에 들어가야 합니다.흡착은 파지-호스트 쌍(phage-host pair)의 값 특성이며, 숙주 세포 표면에서의 파지의 흡착은 2단계 과정, 즉 가역적(reversible) 및 비가역적(reversible)으로 설명됩니다.그것은 파지 꼬리 섬유가 파지를 숙주의 적절한 수용체에 결합하는 것을 도울 때 시작되는 파지 꼬리 구조를 포함합니다.이 과정은 가역적입니다.베이스 플레이트의 구성요소들 중 하나 이상은 파지를 박테리아에 결합시키는 비가역적 과정을 매개합니다.[citation needed]
침투는 또한 파지 유전 물질을 박테리아 내부에 주입하는 것을 수반하는 파지 숙주 감염의 가치 특징입니다.핵산의 침투는 비가역적 흡착 단계 이후에 일어납니다.파지 핵산의 침투와 관련된 메커니즘은 각 파지마다 구체적입니다.이 침투 메커니즘은 전기화학적 막 전위, ATP 분자, 펩티도글리칸 층의 효소적 분열을 포함할 수 있고, 또는 이 세 가지 요소 모두가 박테리아 세포 내부의 핵산 침투에 필수적일 수 있습니다.T2 박테리오파지(T4 유사 파지) 침투 메커니즘에 대한 연구가 이루어졌으며 파지의 꼬리는 박테리아 세포벽 내부로 침투하지 않으며 이 파지의 침투는 내부 막에 전기화학적 막 전위를 포함한다는 것을 보여주었습니다.[23][24]
복제 및 패키징
바이러스 T4 게놈은 롤링 서클 복제를 사용하여 숙주 세포 내에서 합성됩니다.[5]살아있는 세포에서 DNA 복제에 걸리는 시간은 바이러스에 감염된 대장균의 바이러스 T4 DNA 신장률로 측정되었습니다.[25]37°C에서 기하급수적으로 DNA가 증가하는 기간 동안, 그 비율은 초당 749개의 뉴클레오티드였습니다.바이러스 T4 DNA 합성 중 복제당 염기쌍당 변이율은 10개당 1.7개로 매우−8 정확한 DNA 복사 메커니즘이며 [26]300개 사본에서 1개의 오류만 발생합니다.그 바이러스는 또한 독특한 DNA 복구 메커니즘을 코드화합니다.[27]T4 파지 헤드는 비계 단백질 주위에 빈 채로 조립되어 나중에 분해됩니다.따라서 DNA는 작은 구멍을 통해 프로헤드로 들어갈 필요가 있으며, 이는 gp17의 헥사머가 먼저 DNA와 상호작용함으로써 얻어지는데, 이는 모터와 뉴클레아제의 역할도 합니다.T4 DNA 패키징 모터는 초당 최대 2000개의 염기쌍의 속도로 DNA를 바이러스 캡시드에 로딩하는 것으로 밝혀졌습니다.관련 출력은, 크기가 커지면, 보통의 자동차 엔진과 맞먹게 됩니다.[28]
풀어주다
바이러스 번식과 증식의 마지막 단계는 숙주 세포로부터의 비리온의 방출에 의해 결정됩니다.비리온의 방출은 세균의 혈장막이 파괴된 후에 일어납니다.바이러스 단백질이 펩티도글리칸이나 세포막을 공격하는 것을 특징으로 하는 숙주세포를 비포접 바이러스가 용해합니다.박테리아의 용해는 세포 안에 있는 캡시드가 세포벽을 분해하는 효소인 라이소자임을 방출할 때 발생합니다.방출된 박테리오파지는 다른 세포들을 감염시키고, 바이러스 증식 주기는 그 세포들 안에서 반복됩니다.[citation needed]
다중도 재활성화
다중 재활성화(multipity reactivation, MR)는 비활성화된 유전체 손상을 포함하는 두 개 이상의 바이러스 유전체가 감염된 세포 내에서 상호작용하여 실행 가능한 바이러스 유전체를 형성하는 과정입니다.살바도르 루리아(Salvador Luria)는 1946년 UV 조사 바이러스 T4를 연구하던 중 MR을 발견하고 재조합 메커니즘에 의해 손상된 바이러스의 관찰된 재활성화가 일어난다고 제안했습니다.(참조 참조).[29][30][31]이것은 1952년 허쉬-체이스 실험에 의해 관련 바이러스 T2에서 DNA가 유전 물질로 확인되기 전에 일어난 일입니다.[18]
Luria(1984,[32] pg. 97)에 의해 기억되듯이, 조사된 바이러스의 재활성화("다중성 재활성화"라고 함)의 발견은 즉시 초기 파지 그룹 내의 방사선 손상 복구 연구에서 많은[27] 활동을 시작했습니다(1981년 Bernstein에 의해 검토됨).나중에 알고 보니 루리아가 발견한 상호간의 도움에 의한 손상된 바이러스의 복구는 DNA 복구의 특별한 경우 중 하나에 불과했습니다.박테리아와 그 바이러스뿐만 아니라 인간을 포함한 모든 종류의 세포는 이제 DNA 손상을 복구하기 위한 복잡한 생화학적 과정을 가지고 있는 것으로 알려져 있습니다(DNA 복구 참조).DNA 복구 과정은 노화, 암, 불임 등을 예방하는 데 중요한 역할을 하는 것으로도 알려져 있습니다.[citation needed]
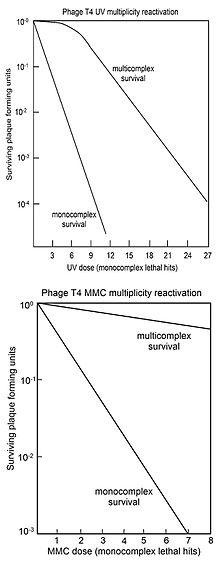
MR은 일반적으로 유전체 손상제의 투여량에 대해 다중 감염 세포(다중 복합체)의 플라크 형성 능력의 생존을 플라크로 표시하는 "생존 곡선"으로 표시됩니다.비교를 위해, 단일 감염된 세포(단일 복합체)의 바이러스 플라크 형성 능력의 생존은 또한 유전체 손상제의 투여량에 대해 플롯됩니다.위 그림은 바이러스 T4 다중 복합체와 단일 복합체에 대한 생존 곡선으로 자외선의 선량이 증가하는 것을 보여줍니다.생존은 로그 척도로 표시되므로 다중 복합체의 생존이 단일 복합체의 생존보다 매우 큰 요인(선량에 따라 다름)에 의해 초과된다는 것을 알 수 있습니다.다중 복합체의 UV 비활성화 곡선은 초기 어깨를 갖습니다.다중 복잡한 생존 곡선에 어깨를 가진 다른 바이러스 T4 DNA 손상제는 X선과[33][34] EMS(ethyl methane sulfonate)입니다.[27]어깨가 있다는 것은 두 가지 재조합 과정을 사용한다는 것을 의미하는 것으로 해석되어 왔습니다.[35]첫 번째 경로는 ("어깨"에 있는) 높은 효율로 DNA를 복구하지만 손상이 증가함에 따라 그 능력이 포화됩니다; 두 번째 경로는 모든 손상 수준에서 기능합니다.다중 복합체에서 방출된 생존 T4 바이러스는 돌연변이의 증가를 보이지 않으며, 이는 UV 조사 바이러스의 MR이 정확한 과정임을 나타냅니다.[35]
아래 그림은 DNA 손상제인 mitomycin C (MMC)에 의한 바이러스 T4의 비활성화에 대한 생존 곡선을 보여줍니다.이 경우 다중 복합체에 대한 생존 곡선은 초기 숄더가 없으므로 위에서 설명한 두 번째 재조합 수리 과정만 활성화되었음을 나타냅니다.이 프로세스에 의한 수리의 효율성은 1,000개의 단일 복합체 중 1개의 복합체만 생존할 수 있는 MMC 용량이 다중 복합체의 약 70%의 생존을 가능하게 한다는 관찰에 의해 나타납니다.DNA 손상제 P32 붕괴, 프소랄렌 플러스 근자외선조사(PUVA), N-메틸-N'-니트로-N-니트로소구아니딘(MNNG), 메틸메탄설포네이트(MMS)[27] 및 아질산에 대해서도 유사한 다중 복합 생존 곡선(어깨 없음)을 얻었습니다.
바이러스 T4에서 MR에 필요한 것으로 밝혀진 몇몇 유전자들은 원핵생물, 진핵생물 그리고 고세균에서 재조합에 필수적인 유전자들을 위한 오르톨로그임이 증명되었습니다.이것은 예를 들어, 대장균으로부터 RecA에 대한 3차원 구조 상동성을 갖는 단백질을 특정하는 T4 유전자 uvSX[36] 및 진핵생물에서 상동성 단백질 RAD51 및 고세균에서 RadA를 포함합니다.MR 동안 DNA 손상의 효율적이고 정확한 재조합 복구는 진핵생물의 감수분열 동안 발생하는 재조합 복구 과정과 유사할 수 있다고 제안되었습니다.[37]
역사
박테리오파지는 1915년 영국 과학자 프레드릭 트워트와 펠릭스에 의해 처음 발견되었습니다.1917년의 헤렐.1930년대 후반, T. L. 라키텐은 생하수도의 혼합물 또는 생하수도에 감염된 대장균의 용해물을 Milislav Demrec와 Ugo Fano 두 연구자에게 제안했습니다.이 두 연구원들은 대장균으로부터 T3, T4, T5, 그리고 T6를 분리했습니다.또한 1932년에 J. 브론펜브레너 연구원은 T2 파지를 연구하고 연구했는데, T2 파지는 바이러스로부터 분리되었습니다.[38]이 격리는 하수도보다는 분변 물질로 이루어졌습니다.어쨌든 막스 델브뤼크는 열 개의 페이지를 발견하는 데 관여했습니다.그의 역할은 박테리오파지를 1형(T1), 2형(T2), 3형(T3) 등으로 명명하는 것이었습니다.[citation needed]
T4 바이러스가 분리된 구체적인 시간과 장소는 불분명하지만 하수나 분변 물질에서 발견되었을 가능성이 있습니다.T4와 유사한 바이러스들은 Thomas F에 의해 논문에서 설명되었습니다. 1944년 11월 앤더슨, 막스 델브뤼크, 밀리슬라프 데메렉.[39]1943년 살바도르 루리아와 델브뤼크는 파지 저항성을 위한 박테리아 돌연변이가 선택에 대한 반응이라기 보다는 선택의 부재에서 발생한다는 것을 보여주었습니다.[32]1943년 이전 세균학자들의 전통적인 지혜는 박테리아는 염색체와 유전자가 없다는 것이었습니다.루리아-델브룩 실험은 박테리아가 다른 확립된 모델 유전자 유기체처럼 유전자를 가지고 있고, 이것들이 자발적으로 돌연변이를 일으켜 돌연변이를 생성할 수 있으며, 이 돌연변이는 복제되어 클론 혈통을 형성할 수 있음을 보여주었습니다.그 해, 그들은 또 다른 파지 실험자인 알프레드 허시와도 협력하기 시작했습니다.[40] (세 사람은 1969년에 "바이러스의 복제 메커니즘과 유전학에 대한 연구"로 노벨 생리학·의학상을 공동 수상했습니다.)
파지 그룹은 막스 델브룩을 중심으로 한 생물학자들의 비공식 네트워크로 주로 박테리오파지 T4에 대한 기초 연구를 수행했으며 20세기 중반에 미생물 유전학과 분자 생물학의 기원에 많은 중요한 기여를 했습니다.1961년, 파지 그룹의 초기 멤버인 시드니 브레너(Sydney Brenner)는 케임브리지 캐번디시 연구소(Cavendish Laboratory)에서 프랜시스 크릭(Francis Crick), 레슬리 바넷(Leslie Barnett), 리처드 왓츠토빈(Richard Watts-Tobin)과 협력하여 단백질의 유전 코드의 기본적인 성격을 입증하는 유전자 실험을 수행했습니다.[41]T4 페이지의 rIIB 유전자 돌연변이로 수행된 이 실험들은 단백질을 암호화하는 유전자에 대해, 그 유전자의 DNA의 세 개의 순차적인 염기가 그 단백질의 각각의 연속적인 아미노산을 특정한다는 것을 보여주었습니다.따라서 유전자 코드는 삼중항 코드이며, 각 삼중항(코돈이라고 함)은 특정 아미노산을 지정합니다.그들은 또한 단백질을 암호화하는 DNA 서열에서 코돈이 서로 겹치지 않는다는 증거를 얻었고, 그러한 서열은 고정된 시작점에서 읽혀집니다.[citation needed]
1962-1964년 동안 T4 연구원들은 실험실 조건에서 파지의 성장에 필수적인 거의 모든 유전자의 기능을 연구할 기회를 제공했습니다.[42][43]이러한 연구는 조건부 치명적 돌연변이의 두 부류의 발견으로 촉진되었습니다.이러한 돌연변이의 한 종류는 호박 돌연변이로 알려져 있습니다.[44]조건부 치명적인 돌연변이의 또 다른 종류는 온도에 민감한 돌연변이라고[45] 불립니다. 이 두 종류의 돌연변이에 대한 연구는 수많은 근본적인 생물학적 문제에 대한 상당한 통찰력을 이끌어냈습니다.따라서 DNA 복제, 수리 및 재조합의 기계에 사용되는 단백질의 기능과 상호 작용, 그리고 바이러스가 단백질 및 핵산 성분(분자 형태 생성)으로부터 어떻게 조립되는지에 대한 이해를 얻었습니다.또한, 코돈을 종결시키는 사슬의 역할을 설명했습니다.한 주목할 만한 연구는 T4 파지의 주요 머리 단백질을 암호화하는 유전자에 결함이 있는 호박 돌연변이를 사용했습니다.[46]이 실험은 널리 알려진 증거를 제공했지만, 1964년 이전에는 아직 증명되지 않은 "서열 가설"에 의해 단백질의 아미노산 서열이 단백질을 결정하는 유전자의 뉴클레오티드 서열에 의해 특정된다는 것입니다.따라서, 이 연구는 암호화된 단백질과 유전자의 공선성을 증명했습니다.
막스 델브뤼크, 살바도르 루리아, 알프레드 허쉬, 제임스 D를 포함한 많은 노벨상 수상자들이 T4 혹은 T4와 유사한 바이러스를 연구했습니다. 왓슨, 프란시스 크릭.T4 바이러스를 연구한 다른 중요한 과학자들로는 마이클 로스만, 시모어 벤저, 브루스 알버트, 지젤라 모식,[47] 리차드 렌스키, 그리고 제임스 불이 있습니다.
참고 항목
참고문헌
- ^ Padilla-Sanchez, Victor (2021). "Structural Model of Bacteriophage T4". WikiJournal of Science. 4 (1): 5. doi:10.15347/WJS/2021.005.
- ^ "ICTV 9th Report (2011) Myoviridae". International Committee on Taxonomy of Viruses (ICTV). Archived from the original on 26 December 2018. Retrieved 26 December 2018.
- ^ "ICTV Taxonomy history: Escherichia virus T4". International Committee on Taxonomy of Viruses (ICTV). Retrieved 26 December 2018.
Caudovirales > Myoviridae > Tevenvirinae > T4virus > Escherichia virus T4
- ^ Miller ES, Kutter E, Mosig G, Arisaka F, Kunisawa T, Rüger W (March 2003). "Bacteriophage T4 genome". Microbiology and Molecular Biology Reviews. 67 (1): 86–156, table of contents. doi:10.1128/mmbr.67.1.86-156.2003. PMC 150520. PMID 12626685.
- ^ a b Bernstein H, Bernstein C (July 1973). "Circular and branched circular concatenates as possible intermediates in bacteriophage T4 DNA replication". Journal of Molecular Biology. 77 (3): 355–61. doi:10.1016/0022-2836(73)90443-9. PMID 4580243.
- ^ Madigan M, Martinko J, eds. (2006). Brock Biology of Microorganisms (11th ed.). Prentice Hall. ISBN 978-0-13-144329-7.
- ^ Malys N (January 2012). "Shine-Dalgarno sequence of bacteriophage T4: GAGG prevails in early genes". Molecular Biology Reports. 39 (1): 33–9. doi:10.1007/s11033-011-0707-4. PMID 21533668. S2CID 17854788.
- ^ Prescott LM, Harley JP, Klein DA (2008). Microbiology (seventh ed.). McGraw-Hill. ISBN 978-007-126727-4.
- ^ Leiman PG, Arisaka F, van Raaij MJ, Kostyuchenko VA, Aksyuk AA, Kanamaru S, Rossmann MG (December 2010). "Morphogenesis of the T4 tail and tail fibers". Virology Journal. 7: 355. doi:10.1186/1743-422X-7-355. PMC 3004832. PMID 21129200.
- ^ Ackermann HW, Krisch HM (1997). "A catalogue of T4-type bacteriophages". Archives of Virology. 142 (12): 2329–45. doi:10.1007/s007050050246. PMID 9672598. S2CID 39369249.
- ^ Taylor NM, Prokhorov NS, Guerrero-Ferreira RC, Shneider MM, Browning C, Goldie KN, Stahlberg H, Leiman PG (May 2016). "Structure of the T4 baseplate and its function in triggering sheath contraction". Nature. 533 (7603): 346–52. Bibcode:2016Natur.533..346T. doi:10.1038/nature17971. PMID 27193680. S2CID 4399265.
- ^ Floor E (February 1970). "Interaction of morphogenetic genes of bacteriophage T4". Journal of Molecular Biology. 47 (3): 293–306. doi:10.1016/0022-2836(70)90303-7. PMID 4907266.
- ^ Snustad DP (August 1968). "Dominance interactions in Escherichia coli cells mixedly infected with bacteriophage T4D wild-type and amber mutants and their possible implications as to type of gene-product function: catalytic vs. stoichiometric". Virology. 35 (4): 550–63. doi:10.1016/0042-6822(68)90285-7. PMID 4878023.
- ^ Yap ML, Rossmann MG (2014). "Structure and function of bacteriophage T4". Future Microbiology. 9 (12): 1319–27. doi:10.2217/fmb.14.91. PMC 4275845. PMID 25517898.
- ^ Yu F, Mizushima S (August 1982). "Roles of lipopolysaccharide and outer membrane protein OmpC of Escherichia coli K-12 in the receptor function for bacteriophage T4". Journal of Bacteriology. 151 (2): 718–22. doi:10.1128/JB.151.2.718-722.1982. PMC 220313. PMID 7047495.
- ^ Furukawa H, Mizushima S (May 1982). "Roles of cell surface components of Escherichia coli K-12 in bacteriophage T4 infection: interaction of tail core with phospholipids". Journal of Bacteriology. 150 (2): 916–24. doi:10.1128/JB.150.2.916-924.1982. PMC 216445. PMID 7040345.
- ^ Maghsoodi A, Chatterjee A, Andricioaei I, Perkins NC (December 2019). "How the phage T4 injection machinery works including energetics, forces, and dynamic pathway". Proceedings of the National Academy of Sciences of the United States of America. 116 (50): 25097–25105. Bibcode:2019PNAS..11625097M. doi:10.1073/pnas.1909298116. PMC 6911207. PMID 31767752.
- ^ a b HERSHEY AD, CHASE M (May 1952). "Independent functions of viral protein and nucleic acid in growth of bacteriophage". The Journal of General Physiology. 36 (1): 39–56. doi:10.1085/jgp.36.1.39. PMC 2147348. PMID 12981234.
- ^ Sherwood, Linda (2011). Prescott's Microbiology (eighth ed.). McGraw-Hill.
- ^ Benzer S. "rII 지역에서의 모험" 페이지와 분자생물학의 기원(2007) John Cairns 편집, Gunther S.스텐트, 제임스 D.Watson, Cold Spring Harbor 정량 생물학 연구소, Long Island, New York ISBN 978-0879698003
- ^ Benzer S (June 1955). "Fine structure of a genetic region in bacteriophage". Proceedings of the National Academy of Sciences of the United States of America. 41 (6): 344–54. Bibcode:1955PNAS...41..344B. doi:10.1073/pnas.41.6.344. PMC 528093. PMID 16589677.
- ^ Benzer S (November 1959). "On the topology of genetic fine structure". Proceedings of the National Academy of Sciences of the United States of America. 45 (11): 1607–20. Bibcode:1959PNAS...45.1607B. doi:10.1073/pnas.45.11.1607. PMC 222769. PMID 16590553.
- ^ Norkin, Leonard C. (2010). Virology, Molecular Biology and Pathogenesis. Washington: American Society for Microbiology. p. 31. ISBN 978-1-55581-453-3.
- ^ Prescott LM, Harley JP, Klein DA (2008). Microbiology (seventh ed.). McGraw Hill. p. 427. ISBN 978-007-126727-4.
- ^ McCarthy D, Minner C, Bernstein H, Bernstein C (1976). "DNA elongation rates and growing point distributions of wild-type phage T4 and a DNA-delay amber mutant". J Mol Biol. 106 (4): 963–81. doi:10.1016/0022-2836(76)90346-6. PMID 789903.
- ^ Drake JW (1970) 돌연변이의 분자적 기초.홀든데이, 샌프란시스코 ISBN 0816224501 ISBN 978-0816224500
- ^ a b c d 번스타인 C. "박테리오파지의 디옥시리보핵산 수복"Microbiol Rev. 1981 3월;45(1):72-98검토.PMID6261109
- ^ Rao VB, Black LW (December 2010). "Structure and assembly of bacteriophage T4 head". Virology Journal. 7: 356. doi:10.1186/1743-422X-7-356. PMC 3012670. PMID 21129201.
- ^ Luria SE (1947). "Reactivation of Irradiated Bacteriophage by Transfer of Self-Reproducing Units". Proc. Natl. Acad. Sci. U.S.A. 33 (9): 253–64. Bibcode:1947PNAS...33..253L. doi:10.1073/pnas.33.9.253. PMC 1079044. PMID 16588748.
- ^ LURIA SE, DULBECCO R (1948). "Lethal mutations, and inactivation of individual genetic determinants in bacteriophage". Genetics. 33 (6): 618. PMID 18100306.
- ^ Luria SE, Dulbecco R (1949). "Genetic Recombinations Leading to Production of Active Bacteriophage from Ultraviolet Inactivated Bacteriophage Particles". Genetics. 34 (2): 93–125. doi:10.1093/genetics/34.2.93. PMC 1209443. PMID 17247312.
- ^ a b 살바도르 E.루리아.슬롯머신, 고장난 시험관: 자서전하퍼 & 로우, 뉴욕: 1984.228쪽.ISBN 0-06-015260-5 (미국 및 캐나다)
- ^ WATSON JD (1952). "The properties of x-ray inactivated bacteriophage". J. Bacteriol. 63 (4): 473–85. doi:10.1128/JB.63.4.473-485.1952. PMC 169298. PMID 14938320.
- ^ HARM W (1958). "Multiplicity reactivation, marker rescue, and genetic recombination in phage T4 following x-ray inactivation". Virology. 5 (2): 337–61. doi:10.1016/0042-6822(58)90027-8. PMID 13544109.
- ^ a b Yarosh DB (1978). "UV-induced mutation in bacteriophage T4". J. Virol. 26 (2): 265–71. doi:10.1128/JVI.26.2.265-271.1978. PMC 354064. PMID 660716.
- ^ Story RM, Bishop DK, Kleckner N, Steitz TA (1993). "Structural relationship of bacterial RecA proteins to recombination proteins from bacteriophage T4 and yeast". Science. 259 (5103): 1892–6. Bibcode:1993Sci...259.1892S. doi:10.1126/science.8456313. PMID 8456313.
- ^ Bernstein C (1979). "Why are babies young? Meiosis may prevent aging of the germ line". Perspect. Biol. Med. 22 (4): 539–44. doi:10.1353/pbm.1979.0041. PMID 573881. S2CID 38550472.
- ^ Willey, Joanne. Prescott's Microbiology (seventh ed.). McGraw-Hill.
- ^ Abedon ST (June 2000). "The murky origin of Snow White and her T-even dwarfs". Genetics. 155 (2): 481–6. doi:10.1093/genetics/155.2.481. PMC 1461100. PMID 10835374.
- ^ Morange, 분자생물학사, pp 43-44
- ^ CRICK FH, BARNETT L, BRENNER S, WATTS-TOBIN RJ (December 1961). "General nature of the genetic code for proteins". Nature. 192 (4809): 1227–32. Bibcode:1961Natur.192.1227C. doi:10.1038/1921227a0. PMID 13882203. S2CID 4276146.
- ^ Edgar RS 조건부 치사: 파지와 분자생물학의 기원 (2007) John Cairns 편집, Gunther S.스텐트, 제임스 D.Watson, Cold Spring Harbor 정량 생물학 연구소, Long Island, New York ISBN 978-0879698003
- ^ Edgar B (October 2004). "The genome of bacteriophage T4: an archeological dig". Genetics. 168 (2): 575–82. doi:10.1093/genetics/168.2.575. PMC 1448817. PMID 15514035.
- ^ Epstein RH, Bolle A, Steinberg CM, Kellenberger E, Boy de la Tour E, Chevalley R, Edgar RS, Susman M, Denhardt GH, Lielausis A (1963). "Physiological Studies of Conditional Lethal Mutants of Bacteriophage T4D". Cold Spring Harbor Symposia on Quantitative Biology. 28: 375–394. doi:10.1101/SQB.1963.028.01.053. ISSN 0091-7451.
- ^ Edgar RS, Lielausis I (April 1964). "Temperature-sensitive mutants of bacteriophage T4D: Their isolation and Characterization". Genetics. 49 (4): 649–62. doi:10.1093/genetics/49.4.649. PMC 1210603. PMID 14156925.
- ^ Sarabhai AS, Stretton AO, Brenner S, Bolle A (January 1964). "Co-linearity of the gene with the polypeptide chain". Nature. 201 (4914): 13–7. Bibcode:1964Natur.201...13S. doi:10.1038/201013a0. PMID 14085558. S2CID 10179456.
- ^ Nossal NG, Franklin JL, Kutter E, Drake JW (November 2004). "Anecdotal, historical and critical commentaries on genetics. Gisela Mosig". Genetics. 168 (3): 1097–104. doi:10.1093/genetics/168.3.1097. PMC 1448779. PMID 15579671.
추가열람
- Leiman P.G.; Kanamaru S; Mesyanzhinov V.V.; Arisaka F.; Rossmann M.G. (2003). "Structure and morphogenesis of bacteriophage T4". Cellular and Molecular Life Sciences. 60 (11): 2356–2370. doi:10.1007/s00018-003-3072-1. PMID 14625682. S2CID 2228357.
- Karam, J., Petrov, V., Nolan, J., Chin, D., Shatley, C., Krisch, H., 그리고 Letarov, A.T4와 유사한 파지 유전체 프로젝트.https://web.archive.org/web/20070523215704/http ://phage.bioc.tulane.edu/ . (T4와 같은 파지 전체 유전자 염기서열 보관소)
- 모식, G, F.아이저링. 2006년.T4 및 관련 페이지: 구조 및 개발, R. Calendar 및 ST. 아베돈 (eds.), 박테리오파지스.옥스포드 대학교 출판부, 옥스포드. (T4페이지 생물학 리뷰) ISBN 0-19-514850-9
- Filee J. Tetart F.; Suttle C.A.; Krisch H.M. (2005). "Marine T4-type bacteriophages, a ubiquitous component of the dark matter of the biosphere". Proc. Natl. Acad. Sci. USA. 102 (35): 12471–6. Bibcode:2005PNAS..10212471F. doi:10.1073/pnas.0503404102. PMC 1194919. PMID 16116082. (야생에서의 유병률 및 T4 유사 파지의 표시)
- Chibani-Chennoufi S.; Canchaya C.; Bruttin A.; Brussow H. (2004). "Comparative genomics of the T4-Like Escherichia coli phage JS98: implications for the evolution of T4 phages". J. Bacteriol. 186 (24): 8276–86. doi:10.1128/JB.186.24.8276-8286.2004. PMC 532421. PMID 15576776. (T4 유사 파지의 특성화)
- Desplats C, Krisch HM (May 2003). "The diversity and evolution of the T4-type bacteriophages". Res. Microbiol. 154 (4): 259–67. doi:10.1016/S0923-2508(03)00069-X. PMID 12798230.
- Miller, E.S.; Kutter E.; Mosig G.; Arisaka F.; Kunisawa T.; Ruger W. (2003). "Bacteriophage T4 genome". Microbiol. Mol. Biol. Rev. 67 (1): 86–156. doi:10.1128/MMBR.67.1.86-156.2003. PMC 150520. PMID 12626685. (유전체 관점에서 T4호기 검토)
- Desplats C.; Dez C.; Tetart F.; Eleaume H.; Krisch H.M. (2002). "Snapshot of the genome of the pseudo-T-even bacteriophage RB49". J. Bacteriol. 184 (10): 2789–2804. doi:10.1128/JB.184.10.2789-2804.2002. PMC 135041. PMID 11976309. (T4 유사 파지인 RB49 유전체 개요)
- Malys N, Chang DY, Baumann RG, Xie D, Black LW (2002). "A bipartite bacteriophage T4 SOC and HOC randomized peptide display library: detection and analysis of phage T4 terminase (gp17) and late sigma factor (gp55) interaction". J Mol Biol. 319 (2): 289–304. doi:10.1016/S0022-2836(02)00298-X. PMID 12051907. (단백질 상호작용 연구를 위한 생명공학에서의 T4 파지 응용)
- Tétart F.; Desplats C.; Kutateladze M.; Monod C.; Ackermann H.-W.; Krisch H.M. (2001). "Phylogeny of the major head and tail genes of the wide-ranging T4-type bacteriophages". J. Bacteriol. 183 (1): 358–366. doi:10.1128/JB.183.1.358-366.2001. PMC 94885. PMID 11114936. (야생에서의 T4형 염기서열 유병률 표시)
- Abedon S.T. (2000). "The murky origin of Snow White and her T-even dwarfs". Genetics. 155 (2): 481–6. doi:10.1093/genetics/155.2.481. PMC 1461100. PMID 10835374. (T4 유사파지 T2, T4, T6의 분리에 대한 역사적 기술)
- Ackermann HW, Krisch HM (1997). "A catalogue of T4-type bacteriophages". Arch. Virol. 142 (12): 2329–45. doi:10.1007/s007050050246. PMID 9672598. S2CID 39369249. Archived from the original on 1 November 2001. (당시 알려진 T4 유사 파지의 거의 완전한 목록)
- Monod C, Repoila F, Kutateladze M, Tétart F, Krisch HM (March 1997). "The genome of the pseudo T-even bacteriophages, a diverse group that resembles T4". J. Mol. Biol. 267 (2): 237–49. doi:10.1006/jmbi.1996.0867. PMID 9096222. (다양한 T4 유사 파지의 유전체 관점에서의 개요)
- Kutter E.; Gachechiladze K.; Poglazov A.; Marusich E.; Shneider M.; Aronsson P.; Napuli A.; Porter D.; Mesyanzhinov V. (1995). "Evolution of T4-related phages". Virus Genes. 11 (2–3): 285–297. doi:10.1007/BF01728666. PMID 8828153. S2CID 20529415. (다양한 T4 유사 파지의 게놈 비교)
- 카람, J.D. 외 1994.박테리오파지 T4의 분자생물학ASM Press, Washington, DC. (제2 T4 성경, Mosig and Eiserling, 2006, 생물학에 대해 배우기 시작함) ISBN 1-55581-064-0
- 에디, 1992년 S. R.티이븐 박테리오파지 속의 인트론.박사 논문.볼더에 있는 콜로라도 대학교.(3장에서는 다양한 T4 유사 파지의 개요 및 당시 새로운 T4 유사 파지의 분리에 대해 설명합니다.
- Surdis, T.J. et al UC Santa Cruz, 1978년 11월, "T4에 특이적인 박테리오파지 부착 방법", 분석, 개요.
- 매튜스, C.K., E.M.커터, G.모식, P.B.버젯.1983. 박테리오파지 T4.American Society for Microbiology, Washington, DC. (최초의 T4 성경; 여기의 모든 정보가 Karam et al., 1994에서 복제된 것은 아닙니다; T4와 유사한 페이지에 대한 역사적 개요는 Doermann의 서론 장을 특히 참조하십시오) ISBN 0-914826-56-5
- 러셀, R.L. 1967.티이븐 박테리오파지 중의 종실.박사 논문.캘리포니아 공과대학.(T4 유사 파지의 RB 계열 분리)
- Malys N, Nivinskas R (2009). "Non-canonical RNA arrangement in T4-even phages: accommodated ribosome binding site at the gene 26-25 intercistronic junction". Mol Microbiol. 73 (6): 1115–1127. doi:10.1111/j.1365-2958.2009.06840.x. PMID 19708923. (T4로 특징지어지는 희귀한 번역 규정 유형)
- Kay D.; Fildes P. (1962). "Hydroxymethylcytosine-containing and tryptophan-dependent bacteriophages isolated from city effluents". J. Gen. Microbiol. 27: 143–6. doi:10.1099/00221287-27-1-143. PMID 14454648. (T4 유사 파지 격리, Ox2 파지 격리 포함)


