코로나바이러스 3' 줄기루프 II 유사 모티프(s2m)
Coronavirus 3′ stem-loop II-like motif (s2m)| 코로나바이러스 3' 줄기루프 II 유사 모티프(s2m) | |
|---|---|
 | |
| 식별자 | |
| 기호. | s2m |
| Rfam | RF00164 |
| 기타자료 | |
| RNA형 | 시스레그 |
| 도메인 | 진핵생물; 바이러스 |
| 그렇게 | SO:0000233 |
| PDB 구조물 | PDBe |
코로나바이러스 3' 줄기 루프 II 유사 모티프(s2m이라고도 함)는 아스트로바이러스, 코로나바이러스 및 말 라이노바이러스 유전체의 3' 비번역 영역(3'UTR)에서 확인된 2차 구조 모티프입니다.[1][2] 그 기능은 알려져 있지 않지만 다양한 바이러스 3' UTR 영역이 바이러스 복제 및 패키징에 역할을 하는 것으로 밝혀졌습니다.
이 모티프는 뉴클레오티드 서열과 2차 구조 접힘 모두에서 보존되는 것으로 보이며, 이는 보존을 위한 강력한 진화적 선택을 나타냅니다. 서로 다른 바이러스 계열에서 이 보존된 모티프의 존재는 적어도 두 개의 개별 재조합 사건의 결과로 제안됩니다.[2] 현재까지 s2m은 Astroviridae, Caliciviridae, Picornaviridae 및 Coronaviridae의 4가지 양성 감각 단일 가닥 RNA 바이러스 계열에서 설명되었습니다. s2m를 포함한 바이러스는 새, 박쥐, 말, 개, 인간을 포함한 광범위한 고등 척추동물을 감염시키고 다양한 조직 친화성을 나타낼 수 있습니다.[3][4] 오직 멀리 떨어져 있는 곤충 종에만 s2m의 이종상동체가 있는 것 같습니다.[5]
코로나바이러스에서 확인된 다른 RNA 계열에는 코로나바이러스 프레임 이동 자극 요소, 코로나바이러스 패키징 신호 및 코로나바이러스 3' UTR 의사노트가 포함됩니다.

생물학적 의의
기능적으로 바이러스 RNA에 의한 숙주 침입 동안, s2m은 먼저 바이러스 RNA가 숙주 단백질 합성을 대체하는 메커니즘으로서 하나 이상의 단백질에 결합하는 것으로 보입니다. 이것은 리보솜 RNA 접힘의 s2m RNA 거대분자 치환에서도 관찰되었습니다. s2m RNA 요소는 항바이러스제 및 안티센스 올리고뉴클레오티드 설계의 효과적인 표적이기도 합니다.[6][2]
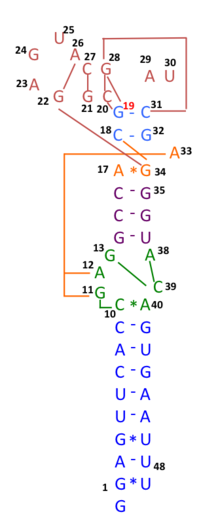
인플루엔자 A 바이러스 H1N1과 유사성을 공유하는 SARS-CoV-2의 잠재적 상호작용 인간 마이크로RNA 표적이 치료 표적으로 확인되었습니다.[7]
SARS-CoV-1과 SARS-CoV-2 s2m의 다른 구조
s2m SARS-CoV-1 RNA의 전체 X선(2.7-Å) 결정 구조는 NMR에 의해 결정되는 SARS-CoV-2 s2m 2차 구조와 다릅니다.[8] SARS-CoV-2 유전체 RNA에서 5' UTR과 s2m 사이의 장거리 상호작용이 제안되었습니다.[9]
SARS-CoV-2 s2m의 돌연변이 : 보존된 RNA 모티프가 아닌 재조합 핫스팟
2020년 COVID-19 대유행 기간 동안 호주 SARS ‐CoV ‐2 분리주의 많은 유전체 서열은 s2m에 결손 또는 돌연변이(29742G>A 또는 29742G>U; "G19A" 또는 "G19U")를 가지며, 이는 RNA 요소에서 RNA 재조합이 발생했을 수 있음을 시사합니다. 29742G("G19"), 29744G("G21"), 29751G("G28")가 재조합 핫스팟으로 예측되었습니다.[10] 29742G>U 변이는 이란에서 호주와 뉴질랜드로 돌아오는 여행객들과도 연관이 있었습니다.[11] 다이아몬드 프린세스 크루즈의 3명의 환자에서 29736G > T와 29751G > T ("G13" 및 "G28")의 두 가지 돌연변이가 SARS-CoV-2의 s2m에서 발견되었습니다. 이 결과는 SARS-CoV-2의 s2m이 다른 코로나바이러스에서 발견되는 보존된 RNA 모티프가 아닌 RNA 재조합/돌연변이 핫스팟임을 시사합니다.[12]
분자동역학 시뮬레이션에 따르면 SARS-CoV-2의 S2M 변이체 29734G>C(G11C)와 29742G>U(G19U) 모두 RNA 구조 안정성을 변화시켜 SARS-CoV-2 복제에서 s2m의 기능적 관련성에 대한 의문을 제기합니다.[13]
참고 항목
참고문헌
- ^ Jonassen CM, Jonassen TO, Grinde B (April 1998). "A common RNA motif in the 3' end of the genomes of astroviruses, avian infectious bronchitis virus and an equine rhinovirus". The Journal of General Virology. 79 ( Pt 4) (4): 715–8. doi:10.1099/0022-1317-79-4-715. PMID 9568965.
- ^ a b c d e Robertson MP, Igel H, Baertsch R, Haussler D, Ares M, Scott WG (January 2005). "The structure of a rigorously conserved RNA element within the SARS virus genome". PLOS Biology. 3 (1): e5. doi:10.1371/journal.pbio.0030005. PMC 539059. PMID 15630477.
- ^ Tengs T, Kristoffersen AB, Bachvaroff TR, Jonassen CM (April 2013). "A mobile genetic element with unknown function found in distantly related viruses". Virology Journal. 10 (1): 132. doi:10.1186/1743-422X-10-132. PMC 3653767. PMID 23618040.
- ^ Rangan R, Zheludev IN, Hagey RJ, et al. (August 2020). "RNA genome conservation and secondary structure in SARS-CoV-2 and SARS-related viruses: a first look". RNA. 26 (8): 937–959. doi:10.1261/rna.076141.120. PMC 7373990. PMID 32398273.
- ^ Tengs T, Delwiche CF, Monceyron Jonassen C (March 2021). "A genetic element in the SARS-CoV-2 genome is shared with multiple insect species". The Journal of General Virology. 102 (3). doi:10.1099/jgv.0.001551. PMC 8515862. PMID 33427605.
- ^ Lulla V, Wandel MP, Bandyra KJ, et al. (June 2021). "Targeting the Conserved Stem Loop 2 Motif in the SARS-CoV-2 Genome". J Virol. 95 (14): e0066321. doi:10.1128/JVI.00663-21. PMC 8223950. PMID 33963053.
- ^ Chan AP, Choi Y, Schork NJ (November 2020). "Conserved genomic terminals OF SARS-COV-2 as co-evolving functional elements and potential therapeutic targets". mSphere. 5 (6): e00754-20. doi:10.1128/mSphere.00754-20. PMC 7690956. PMID 33239366.
- ^ Rangan R, Watkins AM, Chacon J, et al. (March 2021). "De novo 3D models of SARS-CoV-2 RNA elements from consensus experimental secondary structures". Nucleic Acids Res. 49 (6): 3092–3108. doi:10.1093/nar/gkab119. PMC 8034642. PMID 33693814.
- ^ Ziv O, Price J, Shalamova L, et al. (December 2020). "The Short- and Long-Range RNA-RNA Interactome of SARS-CoV-2". Molecular Cell. 80 (6): 1067–1077. doi:10.1016/j.molcel.2020.11.004. PMC 7643667. PMID 33259809.
- ^ Yeh TY, Contreras GP (July 2020). "Emerging viral mutants in Australia suggest RNA recombination event in the SARS-CoV-2 genome". The Medical Journal of Australia. 213 (1): 44–44.e1. doi:10.5694/mja2.50657. PMC 7300921. PMID 32506536.
- ^ Eden JS, Rockett R, Carter I, et al. (April 2020). "An emergent clade of SARS-CoV-2 linked to returned travellers from Iran". Virus Evolution. 6 (1): veaa027. doi:10.1093/ve/veaa027. PMC 7147362. PMID 32296544.
- ^ Yeh TY, Contreras GP (1 July 2021). "Viral transmission and evolution dynamics of SARS-CoV-2 in shipboard quarantine". Bull. World Health Organ. 99 (7): 486–495. doi:10.2471/BLT.20.255752. PMC 8243027. PMID 34248221.
- ^ Ryder SP, Morgan BR, Coskun P, et al. (May 2021). "Analysis of Emerging Variants in Structured Regions of the SARS-CoV-2 Genome". Evol Bioinform Online. 17: 11769343211014167. doi:10.1177/11769343211014167. PMC 8114311. PMID 34017166.
외부 링크
- Rfam의 코로나바이러스 3' 줄기 루프 II 유사 모티프(s2m) 페이지
- S2M 모티프



