ATP합성효소
ATP synthase| ATP 합성효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 X선 결정학에 의해 결정되는 ATP 합성효소의 분자 모델.스테이터는 여기에 표시되어 있지 않습니다. | |||||||||
| 식별자 | |||||||||
| EC 번호 | 7.1.2.2 | ||||||||
| CAS 번호 | 9000-83-3 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
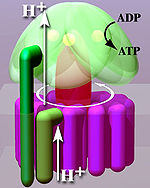
ATP 합성효소는 아데노신2인산(ADP)과 무기인산(Pi)을 사용하여 에너지 저장 분자 아데노신3인산(ATP)의 형성을 촉매하는 단백질이다.P-O 결합(포스포디에스테르 결합)의 형성에 의해 ADP가 변화하므로 연결효소(ligase)로 분류된다.ATP 합성효소는 분자 기계이다.ATP 합성효소에 의해 촉매되는 전반적인 반응은 다음과 같다.
- ADP + Pi + 2H+out atp ATP + HO2 + 2H+in
ADP와i P에서 ATP의 형성은 에너지적으로 불리하며 일반적으로 반대 방향으로 진행됩니다.이 반응을 진전시키기 위해, ATP 합성효소는 세포 호흡 중의 ATP 합성을 진핵 생물의 내부 미토콘드리아 막 또는 박테리아 내 혈장 막에 걸친 양성자(H+) 농도 차이에 의해 만들어진 전기 화학적 구배에 결합시킨다.식물에서 광합성하는 동안, ATP는 틸라코이드 막을 통해 틸라코이드 내강에 생성된 양성자 구배를 사용하여 엽록체 스트로마로 합성됩니다.
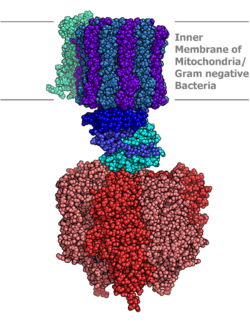
진핵생물 ATP 합성효소는 F-ATPase이며, ATP 효소에 대해 "역방향"으로 실행됩니다.이 기사는 주로 이런 유형을 다루고 있다.F-ATPase는 ATP [1][2]생성을 허용하는 회전 운동 메커니즘을 가진 F와1 F라는 두O 개의 주요 소단위들로 구성됩니다.
명명법
F1 분율은 "Fraction 1"과O F(첨자 문자 "0"이 아닌 "o"로 표기됨)라는 용어에서 유래했으며, 이름은 ATP 합성효소의 [3][4]F 단위를O 억제할 수 있는 자연 유래 항생물질의 일종인 올리고마이신의 결합 분율에서 유래했다.이들 기능영역은 다른 단백질 서브유닛으로 구성됩니다.표 참조.이 효소는 유산소 호흡을 통한 ATP 합성에 사용된다.
구조 및 기능
틸라코이드막과 미토콘드리아막 안에 위치한 ATP 합성효소는 두 개의 영역O F와1 F로 구성됩니다.F는O F의1 회전을 일으키며 c-링으로 구성되며 a, 2 b, F6의 서브유닛으로 구성된다.F는1 α, β, δ 및 δ 서브유닛으로 이루어진다.F는1 ATP를 가수분해할 수 있는 수용성 부분을 가지고 있다.반면O F는 주로 소수성 영역을 가지고 있다.FO1 F는 [7]막을 가로지르는 양성자 이동 경로를 만듭니다.
F1 영역
ATP1 합성효소의 F 부분은 친수성이며 ATP를 가수분해하는 역할을 한다.F단위는1 미토콘드리아 매트릭스 공간으로 돌출되어 있다.서브유닛α 및 β는 6개의 결합부위를 가진 헥사머를 형성한다.이들 중 3개는 촉매적으로 비활성화되어 ADP와 결합합니다.
다른 세 개의 서브유닛은 ATP 합성을 촉매한다.다른1 F 서브유닛 θ, θ, θ는 회전모터기구(로터/액슬)의 일부이다.β 서브유닛은 β가 합성된 후 ATP가 결합 및 방출될 수 있는 구조 변화(즉, 폐쇄, 반개방 및 개방 상태)를 거치게 한다.F1 입자는 크고 [8]음염색으로 투과 전자 현미경으로 볼 수 있습니다.이것들은 미토콘드리아 내부의 막을 후추로 만드는 지름 9 nm의 입자들이다.
| 서브유닛 | 인간 유전자 | 메모 |
|---|---|---|
| 알파 | ATP5A1, ATPAF2 | |
| 베타. | ATP5B, ATPAF1 | |
| 감마 | ATP5C1 | |
| 델타 | ATP5D | 미토콘드리아 "델타"는 박테리아/염소 플라스틱 엡실론이다. |
| 엡실론 | ATP5E | 미토콘드리아 특유의 것 |
| OSCP | ATP5O | 박테리아와 엽록체 버전에서는 "델타"라고 불립니다. |
FO 영역

F는O 8개의 서브유닛과 트랜스막 고리를 가진 수용성 단백질이다.고리는 양성자화 및 탈양성자화 시 구조변화를 거치는 나선 루프나선 단백질과 함께 사량체 형태를 가지며, 인접한 서브유닛을 회전시켜 F의O 회전도 유발하며, F의 구성에도1 영향을 미쳐 알파 및 베타 서브유닛의 상태 전환을 초래한다.ATPO 합성효소의 F 영역은 미토콘드리아 막에 내장된 양성자 모공이다.이 서브유닛은 a, b, c의 3개의 주요 서브유닛으로 구성되어 있으며, 6개의 c 서브유닛은 로터링을 구성하고 서브유닛 b는 αβ 헥사머의 회전을 방해하는 F OSCP에 접속하는1 레버를 구성하고 있습니다.서브유닛 a는 b를 c링에 [11]접속한다.인간은 6개의 추가 서브 유닛, d, e, f, g, F6, 8(또는 A6L)을 가지고 있다.효소의 이 부분은 미토콘드리아 내막에 위치하며 F 영역에서1 ATP 합성을 유발하는 회전에 양성자 전이를 결합한다.
진핵생물에서 미토콘드리아O F는 막-굴곡 이합체를 형성한다.이 이합체들은 크리스테의 끝에서 긴 열로 스스로 배열되는데, 아마도 크리스테 [12]형성의 첫 단계일 것이다.이합체 효모O F영역의 원자모델은 cryo-EM에 의해 전체 분해능 3.[13]6Ω으로 측정되었다.
| 서브유닛 | 인간 유전자 |
|---|---|
| a | MT-ATP6 |
| b | ATP5F1 |
| c | ATP5G1, ATP5G2, ATP5G3 |
바인딩 모델

1960년대부터 1970년대까지 UCLA 교수인 Paul Boyer는 결합 변화, 즉 플립 플랍 메커니즘 이론을 개발했는데, 이것은 ATP 합성이 감마 서브 유닛의 회전에 의해 생성된 ATP 합성효소의 구조 변화에 의존한다고 가정했다.존 E의 연구 그룹입니다. 당시 캠브리지의 MRC 분자생물학 연구소에서 워커는 ATP 합성효소의 F 촉매 도메인을1 결정화했다.당시 알려진 가장 큰 비대칭 단백질 구조였던 이 구조는 Boyer의 회전-카탈리시스 모델이 본질적으로 정확하다는 것을 보여주었다.이를 설명한 공로로 보이어와 워커는 1997년 노벨 화학상의 절반을 공동 수상했다.
F의1 결정 구조는 회전하는 비대칭 감마 서브 유닛 주위에 주황색 세그먼트처럼 배열된 알파 서브 유닛과 베타 서브 유닛(각 3개)을 번갈아 나타냈다.현재 ATP 합성 모델(대체 촉매 모델이라고 알려져 있음)에 따르면, 전자전달계에 의해 공급되는 (H+) 양성자 양이온에 의해 생성된 트랜스막 전위는 ATP 합성효소의 F 영역을O 통해 막간 공간에서 막으로 (H+) 양성자 양이온을 구동한다.F(c-subunits의 고리)의O 일부는 양성자가 막을 통과할 때 회전합니다.c-링은 비대칭 중앙 줄기(주로 감마 서브유닛으로 구성됨)에 단단히 부착되어 F의 알파벳331 내에서 회전하여 3개의 촉매 뉴클레오티드 결합 부위가 ATP 합성으로 이어지는 일련의 구조 변화를 겪도록 한다.주요1 F 서브유닛은 알파벳을33 F의O 비회전 부분에 결합하는 주변 레버에 의해 중앙 레버 로터에 동조하여 회전하는 것을 방지합니다.온전한 ATP 합성효소의 구조는 현재 복합체의 전자 저온 현미경(cryo-EM) 연구에서 낮은 분해능으로 알려져 있다.ATP 합성효소의 저온 전자파 모델은 말초 줄기가 F에서O F로 결합하면서1 복합체를 감싸는 유연한 구조임을 시사합니다.적절한 조건 하에서 효소 반응은 또한 역방향으로 수행될 수 있으며, ATP 가수분해는 막을 가로질러 양성자를 펌핑합니다.
결합 변화 메커니즘은 β 서브유닛의 활성 부위가 세 가지 [14]상태 사이를 순환하는 것을 포함한다."느슨한" 상태에서는 ADP와 인산염이 활성 부위로 들어갑니다. 인접한 다이어그램에서는 이것이 분홍색으로 표시됩니다.그 후 효소는 형태 변화를 겪고 이러한 분자들을 함께 밀어 붙이고, 결과적인 "긴축" 상태의 활성 부위는 매우 높은 친화력으로 새로 생성된 ATP 분자와 결합합니다.마지막으로 활성 부위는 개방 상태(주황색)로 돌아가 ATP를 방출하고 더 많은 ADP와 인산염과 결합하여 다음 ATP [15]생산 사이클에 대비합니다.
생리학적 역할
다른 효소들과 마찬가지로 FF ATP 합성효소의 활성은1O 가역적이다.많은 양의 ATP는 그것이 막 투과 양성자 구배를 만들도록 합니다. 이것은 전자 전달 사슬을 가지고 있지 않은 박테리아를 발효시켜 오히려 ATP를 가수 분해하여 양성자 구배를 만드는데 사용됩니다. 그들은 편모세포와 영양소의 세포로의 운반을 촉진하기 위해 사용합니다.
생리학적 조건에서 박테리아를 호흡시킬 때, 일반적으로 ATP 합성효소는 반대 방향으로 움직이며, 전자전달계에 의해 만들어진 양성자 원동력을 에너지원으로 사용하면서 ATP를 생성한다.이러한 방식으로 에너지를 생성하는 전체적인 과정을 산화적 인산화라고 한다.같은 과정이 미토콘드리아에서 일어나는데, 여기서 ATP 합성효소는 미토콘드리아 내부의 막에 위치하고1 F 부분은 미토콘드리아 매트릭스로 돌출된다.ATP-합성효소에 의한 ATP의 소비는 양성자 양이온을 매트릭스로 펌프한다.
진화
ATP 합성효소의 진화는 기능적으로 독립적인 두 개의 서브유닛이 결합되어 새로운 [16][17]기능을 획득하는 모듈식이었던 것으로 생각된다.이 연관성은 진화 역사 초기에 일어난 것으로 보이는데, 왜냐하면 본질적으로 모든 생명체 [16]왕국에 동일한 구조와 활동이 존재하기 때문입니다.F-ATP 합성효소는 V-ATPase와 [18]높은 기능적 및 기계적 유사성을 보인다.그러나 F-ATP 합성효소는 양성자 구배를 이용하여 ATP를 생성하는 반면, V-ATPase는 ATP를 희생하여 양성자 구배를 생성하며, pH 값은 [19]1만큼 낮습니다.
F 영역은 또한1 헥사머릭 DNA 헬리케이스(특히 Rho 인자)와 유의미한 유사성을 보이며, 전체 효소 영역은 H-powered+
T3SS 또는 편모 운동 [18][20][21]복합체와 어느 정도 유사성을 보인다.F 영역의1 αβ33 헥사머는 헥사머 DNA 헬리케이스와 구조적으로 유의미한 유사성을 보이며, 둘 다 중앙 기공과 3배 회전 대칭을 가진 고리를 형성한다.두 가지 모두 모공 내 고분자의 상대적인 회전에 의존적인 역할을 한다. DNA 헬리케이스는 DNA 분자를 따라 그들의 움직임을 구동하고 초코일을 감지하기 위해 DNA의 나선 형태를 사용하는 반면, αβ33 육각형은 효소 반응을 [22]촉진하기 위해 β 서브유닛의 회전을 통해 구조 변화를 사용한다.
F+
입자의O H 모터는 편모를 [18]구동하는 H 모터와+
기능적으로 매우 유사합니다.둘 다 에너지원으로서 H 전위 구배를 사용하여+
근처의 정지 단백질에 대해 상대적으로 회전하는 많은 작은 알파 나선 단백질의 고리를 특징으로 한다.그러나 편모 모터의 전체적인 구조는 F 입자보다O 훨씬 복잡하고 약 30개의 회전 단백질을 가진 고리가 F 복합체의O 10, 11 또는 14개의 나선 단백질보다 훨씬 크기 때문에 이 연결은 미약하다.그러나 보다 최근의 구조 데이터는 고리와 줄기가 구조적으로 F [21]입자와1 유사하다는 것을 보여준다.
ATP 합성효소의 기원에 대한 모듈식 진화 이론은 독립적인 기능을 가진 두 개의 서브유닛, 즉 ATPase 활성을 가진 DNA 헬리케이스와 H+
모터가 결합할 수 있었고, 모터의 회전은 헬리케이스의 ATP 효소 활성을 [16][22]역방향으로 몰았다.그 후 이 복합체는 더 큰 효율성을 발전시켰고 결국 오늘날의 복잡한 ATP 합성 효소로 발전했다.또는 DNA 헬리케이스/H+
모터 복합체는 [16]H 모터를 반대로+
구동하는 헬리케이스의 ATPase 활성과 함께 H 펌프 활성을 가질+
수 있다.이것은 역반응을 수행하고 ATP [17][23][24]합성효소로 작용하도록 진화했을 수 있다.
억제제
ATP 합성효소의 다양한 자연 및 합성 억제제가 발견되었다.[25]이것들은 ATP 합성효소의 구조와 메커니즘을 조사하는데 사용되어 왔다.일부는 치료에 도움이 될 수 있다.ATP 합성효소 억제제에는 펩타이드 억제제, 폴리페놀성 피토케티드, 유기소 화합물, 폴리엔성 α-피론 유도체, 양이온 억제제, 기질 유사체, 아미노산 수식제 및 기타 다양한 [25]화학물질이 포함되어 있습니다.가장 일반적으로 사용되는 ATP 합성효소 억제제 중 일부는 올리고마이신과 DCCD이다.
다른 유기체에
박테리아
대장균 ATP 합성효소는 알려진 가장 단순한 형태의 ATP 합성효소로 8개의 다른 서브유닛 유형을 가지고 [11]있다.
세균성 F-ATPases는 때때로 반대로 작용하여 ATP 효소로 [26]변할 수 있습니다.일부 박테리아는 A/V형 ATP 효소를 [9]양방향으로 사용하여 F-ATPase를 가지고 있지 않습니다.
효모균
효모 ATP 합성효소는 가장 잘 연구된 진핵생물 ATP 합성효소 중 하나이며, 5개의1 F, 8개의O F 서브유닛, 7개의 관련 단백질이 확인되었다.[7]이 단백질의 대부분은 다른 진핵생물에서 [27][28][29][30]상동성을 가지고 있다.
식물.
식물에서, ATP 합성효소는 엽록체에도 존재한다.이 효소는 틸라코이드 막에 통합되어 CF-부분이1 스트로마로마에 달라붙어 광합성과 ATP 합성의 어두운 반응이 일어납니다.엽록체 ATP 합성효소의 전체적인 구조와 촉매 메커니즘은 세균 효소의 구조와 거의 동일하다.그러나 엽록체에서 양성자 구동력은 호흡 전자전달계가 아니라 1차 광합성 단백질에 의해 발생한다.합성효소는 감마 서브유닛에 40-aa 삽입물이 있어 [31]어두울 때 낭비 활동을 억제한다.
포유동물
소(Bos taurus) 심장 미토콘드리아에서 분리된 ATP 합성효소는 생화학 및 구조 면에서 가장 잘 특징지어지는 ATP 합성효소이다.소의 심장은 심장 근육에 미토콘드리아의 농도가 높기 때문에 효소의 공급원으로 사용된다.그들의 유전자는 인간의 ATP 합성효소와 [32][33][34]밀접한 상동성을 가지고 있다.
ATP 합성효소의 성분을 코드하는 인간 유전자:
- ATP5A1
- ATP5B
- ATP5C1, ATP5D, ATP5E, ATP5F1, ATP5G1, ATP5G2, ATP5G3, ATP5H, ATP5I, ATP5J2, ATP5L, ATP5O
- MT-ATP6, MT-ATP8
기타 진핵생물
몇몇 다른 계통들에 속하는 진핵 생물들은 ATP 합성효소의 매우 특별한 조직을 가지고 있다.유글레노조아 ATP 합성효소는 다른 미토콘드리아 ATP 합성효소처럼 부메랑 모양의1 F 헤드를 가진 이합체를 형성하지만, F 서브콤플렉스는 많은O 독특한 서브유닛을 가지고 있다.그것은 카르디올리핀을 사용한다.억제성1 IF는 또한 트리파노소마티다와 [35]공유되는 방식으로 서로 다르게 결합한다.
고세균
고고학자는 일반적으로 F-ATPase를 가지고 있지 않다.대신, 그들은 구조적으로 V-ATPase와 유사하지만 주로 ATP 합성 [26]효소로 기능하는 회전 기계인 A-ATPase/합성 효소를 사용하여 ATP를 합성한다.박테리아 F-ATPase와 같이, 그것은 또한 ATP [9]효소로 기능하는 것으로 여겨진다.
LUCA 이전
F-ATPase 유전자 연결과 유전자 질서는 고대 원핵생물 계통에 걸쳐 광범위하게 보존되어 있으며, 이는 이 시스템이 마지막 보편적인 공통 조상인 [36]LUCA 이전에 이미 존재했음을 암시한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Okuno D, Iino R, Noji H (June 2011). "Rotation and structure of FoF1-ATP synthase". Journal of Biochemistry. 149 (6): 655–664. doi:10.1093/jb/mvr049. PMID 21524994.
- ^ Junge W, Nelson N (June 2015). "ATP synthase". Annual Review of Biochemistry. 84: 631–657. doi:10.1146/annurev-biochem-060614-034124. PMID 25839341.
- ^ Kagawa Y, Racker E (May 1966). "Partial resolution of the enzymes catalyzing oxidative phosphorylation. 8. Properties of a factor conferring oligomycin sensitivity on mitochondrial adenosine triphosphatase". The Journal of Biological Chemistry. 241 (10): 2461–2466. doi:10.1016/S0021-9258(18)96640-8. PMID 4223640.
- ^ Mccarty RE (November 1992). "A PLANT BIOCHEMIST'S VIEW OF H+-ATPases AND ATP SYNTHASES". The Journal of Experimental Biology. 172 (Pt 1): 431–441. doi:10.1242/jeb.172.1.431. PMID 9874753.
- ^ PDB: 5ARA; Zhou A, Rohou A, Schep DG, Bason JV, Montgomery MG, Walker JE, et al. (October 2015). "Structure and conformational states of the bovine mitochondrial ATP synthase by cryo-EM". eLife. 4: e10180. doi:10.7554/eLife.10180. PMC 4718723. PMID 26439008.
- ^ Goodsell D (December 2005). "ATP Synthase". Molecule of the Month. doi:10.2210/rcsb_pdb/mom_2005_12.
- ^ a b Velours J, Paumard P, Soubannier V, Spannagel C, Vaillier J, Arselin G, Graves PV (May 2000). "Organisation of the yeast ATP synthase F(0):a study based on cysteine mutants, thiol modification and cross-linking reagents". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1458 (2–3): 443–456. doi:10.1016/S0005-2728(00)00093-1. PMID 10838057.
- ^ Fernandez Moran H, Oda T, Blair PV, Green DE (July 1964). "A macromolecular repeating unit of mitochondrial structure and function. Correlated electron microscopic and biochemical studies of isolated mitochondria and submitochondrial particles of beef heart muscle". The Journal of Cell Biology. 22 (1): 63–100. doi:10.1083/jcb.22.1.63. PMC 2106494. PMID 14195622.
- ^ a b c Stewart AG, Laming EM, Sobti M, Stock D (April 2014). "Rotary ATPases--dynamic molecular machines". Current Opinion in Structural Biology. 25: 40–48. doi:10.1016/j.sbi.2013.11.013. PMID 24878343.
- ^ PDB: 1VZS; Carbajo RJ, Silvester JA, Runswick MJ, Walker JE, Neuhaus D (September 2004). "Solution structure of subunit F(6) from the peripheral stalk region of ATP synthase from bovine heart mitochondria". Journal of Molecular Biology. 342 (2): 593–603. doi:10.1016/j.jmb.2004.07.013. PMID 15327958.
- ^ a b Ahmad Z, Okafor F, Laughlin TF (2011). "Role of Charged Residues in the Catalytic Sites of Escherichia coli ATP Synthase". Journal of Amino Acids. 2011: 785741. doi:10.4061/2011/785741. PMC 3268026. PMID 22312470.
- ^ Blum TB, Hahn A, Meier T, Davies KM, Kühlbrandt W (March 2019). "Dimers of mitochondrial ATP synthase induce membrane curvature and self-assemble into rows". Proceedings of the National Academy of Sciences of the United States of America. 116 (10): 4250–4255. doi:10.1073/pnas.1816556116. PMC 6410833. PMID 30760595.
- ^ Guo H, Bueler SA, Rubinstein JL (November 2017). "Atomic model for the dimeric FO region of mitochondrial ATP synthase". Science. 358 (6365): 936–940. Bibcode:2017Sci...358..936G. doi:10.1126/science.aao4815. PMC 6402782. PMID 29074581.
- ^ Gresser MJ, Myers JA, Boyer PD (October 1982). "Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model". The Journal of Biological Chemistry. 257 (20): 12030–12038. doi:10.1016/S0021-9258(18)33672-X. PMID 6214554.
- ^ Nakamoto RK, Baylis Scanlon JA, Al-Shawi MK (August 2008). "The rotary mechanism of the ATP synthase". Archives of Biochemistry and Biophysics. 476 (1): 43–50. doi:10.1016/j.abb.2008.05.004. PMC 2581510. PMID 18515057.
- ^ a b c d Doering C, Ermentrout B, Oster G (December 1995). "Rotary DNA motors". Biophysical Journal. 69 (6): 2256–2267. Bibcode:1995BpJ....69.2256D. doi:10.1016/S0006-3495(95)80096-2. PMC 1236464. PMID 8599633.
- ^ a b Crofts A. "Lecture 10:ATP synthase". Life Sciences at the University of Illinois at Urbana–Champaign.
- ^ a b c "ATP Synthase". InterPro Database.
- ^ Beyenbach KW, Wieczorek H (February 2006). "The V-type H+ ATPase: molecular structure and function, physiological roles and regulation". The Journal of Experimental Biology. 209 (Pt 4): 577–589. doi:10.1242/jeb.02014. PMID 16449553.
- ^ Skordalakes E, Berger JM (July 2003). "Structure of the Rho transcription terminator: mechanism of mRNA recognition and helicase loading". Cell. 114 (1): 135–146. doi:10.1016/S0092-8674(03)00512-9. PMID 12859904. S2CID 5765103.
- ^ a b Imada K, Minamino T, Uchida Y, Kinoshita M, Namba K (March 2016). "Insight into the flagella type III export revealed by the complex structure of the type III ATPase and its regulator". Proceedings of the National Academy of Sciences of the United States of America. 113 (13): 3633–3638. Bibcode:2016PNAS..113.3633I. doi:10.1073/pnas.1524025113. PMC 4822572. PMID 26984495.
- ^ a b Martinez LO, Jacquet S, Esteve JP, Rolland C, Cabezón E, Champagne E, et al. (January 2003). "Ectopic beta-chain of ATP synthase is an apolipoprotein A-I receptor in hepatic HDL endocytosis". Nature. 421 (6918): 75–79. Bibcode:2003Natur.421...75M. doi:10.1038/nature01250. PMID 12511957. S2CID 4333137.
- ^ Cross RL, Taiz L (January 1990). "Gene duplication as a means for altering H+/ATP ratios during the evolution of FOF1 ATPases and synthases". FEBS Letters. 259 (2): 227–229. doi:10.1016/0014-5793(90)80014-a. PMID 2136729. S2CID 32559858.
- ^ Cross RL, Müller V (October 2004). "The evolution of A-, F-, and V-type ATP synthases and ATPases: reversals in function and changes in the H+/ATP coupling ratio". FEBS Letters. 576 (1–2): 1–4. doi:10.1016/j.febslet.2004.08.065. PMID 15473999. S2CID 25800744.
- ^ a b Hong S, Pedersen PL (December 2008). "ATP synthase and the actions of inhibitors utilized to study its roles in human health, disease, and other scientific areas". Microbiology and Molecular Biology Reviews. 72 (4): 590–641, Table of Contents. doi:10.1128/MMBR.00016-08. PMC 2593570. PMID 19052322.
- ^ a b Kühlbrandt W, Davies KM (January 2016). "Rotary ATPases: A New Twist to an Ancient Machine". Trends in Biochemical Sciences. 41 (1): 106–116. doi:10.1016/j.tibs.2015.10.006. PMID 26671611.
- ^ Devenish RJ, Prescott M, Roucou X, Nagley P (May 2000). "Insights into ATP synthase assembly and function through the molecular genetic manipulation of subunits of the yeast mitochondrial enzyme complex". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1458 (2–3): 428–442. doi:10.1016/S0005-2728(00)00092-X. PMID 10838056.
- ^ Kabaleeswaran V, Puri N, Walker JE, Leslie AG, Mueller DM (November 2006). "Novel features of the rotary catalytic mechanism revealed in the structure of yeast F1 ATPase". The EMBO Journal. 25 (22): 5433–5442. doi:10.1038/sj.emboj.7601410. PMC 1636620. PMID 17082766.
- ^ Stock D, Leslie AG, Walker JE (November 1999). "Molecular architecture of the rotary motor in ATP synthase". Science. 286 (5445): 1700–1705. doi:10.1126/science.286.5445.1700. PMID 10576729.
- ^ Liu S, Charlesworth TJ, Bason JV, Montgomery MG, Harbour ME, Fearnley IM, Walker JE (May 2015). "The purification and characterization of ATP synthase complexes from the mitochondria of four fungal species". The Biochemical Journal. 468 (1): 167–175. doi:10.1042/BJ20150197. PMC 4422255. PMID 25759169.
- ^ Hahn A, Vonck J, Mills DJ, Meier T, Kühlbrandt W (May 2018). "Structure, mechanism, and regulation of the chloroplast ATP synthase". Science. 360 (6389): eaat4318. doi:10.1126/science.aat4318. PMC 7116070. PMID 29748256.
- ^ Abrahams JP, Leslie AG, Lutter R, Walker JE (August 1994). "Structure at 2.8 A resolution of F1-ATPase from bovine heart mitochondria". Nature. 370 (6491): 621–628. Bibcode:1994Natur.370..621A. doi:10.1038/370621a0. PMID 8065448. S2CID 4275221.
- ^ Gibbons C, Montgomery MG, Leslie AG, Walker JE (November 2000). "The structure of the central stalk in bovine F(1)-ATPase at 2.4 A resolution". Nature Structural Biology. 7 (11): 1055–1061. doi:10.1038/80981. PMID 11062563. S2CID 23229994.
- ^ Menz RI, Walker JE, Leslie AG (August 2001). "Structure of bovine mitochondrial F(1)-ATPase with nucleotide bound to all three catalytic sites: implications for the mechanism of rotary catalysis". Cell. 106 (3): 331–341. doi:10.1016/s0092-8674(01)00452-4. PMID 11509182. S2CID 1266814.
- ^ Mühleip A, McComas SE, Amunts A (November 2019). "Structure of a mitochondrial ATP synthase with bound native cardiolipin". eLife. 8: e51179. doi:10.7554/eLife.51179. PMC 6930080. PMID 31738165.
- "Different from the rest". eLife. December 24, 2019.
- ^ Matzke NJ, Lin A, Stone M, Baker MA (July 2021). "Flagellar export apparatus and ATP synthetase: Homology evidenced by synteny predating the Last Universal Common Ancestor". BioEssays. 43 (7): e2100004. doi:10.1002/bies.202100004. PMID 33998015. S2CID 234747849.
추가 정보
- 닉 레인:중요한 질문: 에너지, 진화, 그리고 복잡한 삶의 기원, Ww Norton, 2015-07-20, ISBN 978-0393088816 (ATP 합성효소의 모델을 보여주는 그림 10과의 링크 포인트)
외부 링크
- 보리스 A.Feniouk: "ATP 합성효소 - 훌륭한 분자 기계"
- Urbana-Champaign에 있는 일리노이 대학의 Antony Crofts의 ATP 합성효소 강의가 잘 설명되었습니다.
- OPM 데이터베이스의 F형, V형 및 A형 ATPase 전이 양성자와 나트륨
- 1997년 노벨 화학상 수상자 폴 D.보이어와 존 E.보행기는 ATP 합성의 효소적 메커니즘을 위한 것이다; 그리고 옌스 C.Skou, 이온수송효소 Na+
, K-ATPase의 +
발견을 위해. - 하버드 멀티미디어 제작 사이트 - 비디오– ATP 합성 애니메이션
- David Goodsell: "ATP Synthase - 이달의 분자"