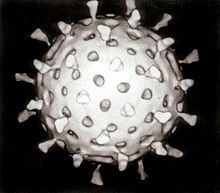
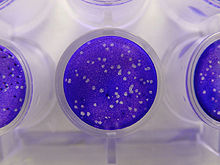
바이러스학
Virology바이러스학은 생물학적 바이러스에 대한 과학적 연구이다.미생물의 검출, 구조, 분류, 진화, 생식을 위한 숙주 세포의 감염과 이용, 숙주 생물 생리와 면역과의 상호작용, 그들이 일으키는 질병, 그들을 고립시키고 배양하는 기술, 그리고 연구와 테라에서의 사용에 초점을 맞춘 미생물학의 하위 분야이다.py.
마르티누스 베이제린크(1898)에 의한 담배 모자이크병의 원인물질(TMV)의 새로운 병원체로서의 식별은 세균학과 구별되는 분야로서 바이러스학 분야의 공식적인 시작으로서 현재 인정되고 있다.그는 그 원인이 세균이나 곰팡이 감염이 아니라 전혀 다른 것이라는 것을 깨달았다.베이제린크는 그의 'contagium vivum fluidum'에서 신비한 인물을 묘사하기 위해 "바이러스"라는 단어를 사용했다.로잘린드 프랭클린은 1955년에 담배 모자이크 바이러스의 전체 구조를 제안했다.
바이러스학은 바이러스를 전파하거나 시각화하는 방법이나 바이러스 감염에 대한 특정 실험실 테스트가 없을 때 시작되었다.현재 바이러스학의 주축이 되고 있는 바이러스 핵산(RNA와 DNA)과 단백질을 분리하는 방법은 존재하지 않았다.현재 바이러스와 그 구성 요소의 구조와 기능을 관찰하는 많은 방법이 있다.수천 개의 다른 바이러스들이 현재 알려져 있고 바이러스학자들은 종종 식물이나 박테리아, 다른 미생물, 또는 동물을 감염시키는 바이러스들 중 하나를 전문으로 한다.인간을 감염시키는 바이러스는 현재 의학 바이러스학자들에 의해 연구되고 있다.
바이러스학은 생물학, 건강, 동물 복지, 농업, 생태학을 망라하는 광범위한 과목이다.바이러스를 연구하는데 사용되는 방법은 여러 가지가 있는데, 이 글에서는 바이러스의 주요 방법과 역사적 중요성을 개략적으로 설명합니다.개별 바이러스 및 바이러스 질환에 대한 설명은 별도의 위키피디아 문서와 바이러스 포털에 나와 있습니다.
역사
루이 파스퇴르는 광견병의 원인물질을 찾을 수 없었고 현미경으로 [1]검출하기에는 너무 작은 병원체에 대해 추측했다.1884년, 프랑스의 미생물학자 찰스 체임벌랜드는 체임벌랜드 필터(또는 파스퇴르-체임벌랜드 필터)를 발명했는데,[2] 체임벌랜드 필터는 체임벌랜드를 통과한 용액에서 모든 박테리아를 제거할 수 있을 만큼 작은 모공을 가지고 있습니다.1892년, 러시아의 생물학자 드미트리 이바노프스키가 이 필터를 사용하여 현재 담배 모자이크 바이러스로 알려진 것을 연구했습니다: 감염된 담배 식물의 찌그러진 잎 추출물은 박테리아를 제거하기 위해 여과한 후에도 전염성을 유지했습니다.이바노프스키는 이 감염이 박테리아에 의해 생성된 독소에 의한 것일 수도 있다고 주장했지만,[3] 그는 그 생각을 추구하지 않았다.당시에는 모든 감염물질이 필터에 의해 유지되고 영양 배지에서 배양될 수 있다고 생각되었다. 이것은 [4]질병의 세균 이론의 일부였다.
1898년, 네덜란드의 미생물학자 마르티누스 베이제린크는 실험을 반복했고 여과된 용액이 새로운 형태의 감염 [5]물질을 포함하고 있다는 것을 확신하게 되었다.그는 이 물질이 분열하는 세포에서만 증식하는 것을 관찰했지만 실험 결과 입자로 만들어지지 않자 이를 페트로슘 비붐 플루이넘(용해성 살아있는 배아)이라고 부르며 바이러스라는 단어를 다시 도입했다.베이제린크는 바이러스는 본질적으로 액체라고 주장했는데, 이 이론은 나중에 [3]미립자라는 것을 증명한 웬델 스탠리에 의해 신빙성을 잃었다.같은 해, Friedrich Loeffler와 Paul Frosch는 유사한 [6]필터를 통해 최초의 동물 바이러스인 아프토바이러스를 통과시켰다.
20세기 초, 영국의 세균학자 프레드릭 트워트는 현재 박테리오파지라고[7] 불리는 박테리아를 감염시키는 바이러스 그룹을 발견했고, 프랑스-캐나다 미생물학자 펠릭스 데렐은 한천 접시에 있는 박테리아에 첨가될 때 죽은 박테리아 영역을 만들 것이라고 바이러스를 설명했습니다.그는 이들 바이러스의 현탁액을 정확히 희석시켜 박테리아를 모두 죽이는 것이 아니라 가장 높은 희석액(바이러스 농도가 낮은 것)이 죽은 유기체의 분리된 영역을 형성한다는 것을 발견했다.이러한 영역을 세고 희석 계수를 곱하면 원래 [8]현탁액에 있는 바이러스 수를 계산할 수 있었다.페이지는 장티푸스나 콜레라와 같은 질병의 잠재적 치료제로 알려졌지만 페니실린의 개발로 그 약속은 잊혀졌다.항생제에 대한 세균 내성의 개발은 박테리오파지의 [9]치료적 사용에 대한 관심을 다시 불러일으켰다.
19세기 말까지 바이러스는 감염성, 필터를 통과할 수 있는 능력, 그리고 살아있는 숙주에 대한 필요성 측면에서 정의되었습니다.바이러스는 식물과 동물에서만 길러졌다.1906년 로스 그랜빌 해리슨은 림프에서 조직을 성장시키는 방법을 발명했고 1913년 E.슈타인하르트, C이스라엘인과 R.A. 램버트는 이 방법을 사용하여 기니피그 각막 조직의 [10]파편에서 백시니아 바이러스를 키웠다.1928년, H. B. Maitland와 M. C. Maitland는 다진 암탉의 신장 부유물에 백시니아 바이러스를 키웠다.폴리오바이러스가 백신 [11]생산을 위해 대규모로 재배된 1950년대까지는 이 방법이 널리 채택되지 않았다.
또 다른 돌파구는 1931년 미국 병리학자인 어니스트 윌리엄 굿파스처와 앨리스 마일스 우드러프가 수정란에서 [12]인플루엔자와 다른 여러 바이러스를 키웠을 때 나왔다.1949년 존 프랭클린 엔더스, 토마스 웰러, 프레데릭 로빈스는 유산된 인간 배아 [13]조직으로부터 배양된 세포에서 폴리오바이러스를 키웠는데, 이것은 단단한 동물 조직이나 난자를 사용하지 않고 자란 최초의 바이러스이다.이 연구로 힐러리 코프로스키와 조나스 소크는 효과적인 소아마비 [14]백신을 만들 수 있었습니다.
바이러스의 첫 이미지는 1931년 독일 엔지니어 에른스트 루스카와 맥스 [15]놀에 의해 전자 현미경의 발명에 의해 획득되었습니다.1935년, 미국의 생화학자이자 바이러스학자인 웬델 메레디스 스탠리는 담배 모자이크 바이러스를 조사했고 그것이 대부분 [16]단백질로 만들어졌다는 것을 발견했다.잠시 후, 이 바이러스는 단백질과 RNA [17]부분으로 분리되었다.담배 모자이크 바이러스는 가장 먼저 결정화되었고 따라서 그 구조가 자세히 설명될 수 있었다.결정화된 바이러스의 첫 X선 회절 사진은 1941년 베르날과 판쿠첸에 의해 입수되었다.로잘린드 프랭클린은 X선 결정학 사진을 [18]바탕으로 1955년 바이러스의 전체 구조를 발견했다.같은 해, 하인즈 프랭켈 콘랏과 로블리 윌리엄스는 정제된 담배 모자이크 바이러스 RNA와 단백질 피막이 기능성 바이러스를 형성하기 위해 스스로 결합할 수 있다는 것을 보여주며, 이 단순한 메커니즘이 아마도 숙주 [19]세포 내에서 바이러스가 생성되는 수단이었을 것이라는 것을 시사했다.
20세기 후반은 바이러스 발견의 황금기였고, 기록된 동물, 식물, 박테리아 바이러스의 대부분은 이 [20]기간 동안 발견되었다.1957년 말기 동맥 바이러스와 소 바이러스 설사(페스티바이러스)의 원인이 발견되었다.1963년 B형 간염 바이러스는 Baruch Blumberg에 [21]의해 발견되었고 1965년 Howard Temin은 최초의 레트로바이러스를 설명했다.레트로바이러스가 RNA의 DNA 복제를 위해 사용하는 효소인 역전사효소는 1970년 테민과 데이비드 볼티모어에 의해 독립적으로 처음 설명되었다.[22]1983년 프랑스 파스퇴르 연구소의 Luc Montagnier의 팀은 현재 [23]HIV라고 불리는 레트로바이러스를 처음 분리했다.1989년 키론사의 마이클 호튼 팀은 [24][25]C형 간염을 발견했다.
바이러스성 질환
바이러스를 연구하게 된 한 가지 주된 동기는 바이러스가 식물과 [26]동물의 많은 전염병을 유발하기 때문이다.바이러스가 질병을 일으키는 방식에 대한 연구는 바이러스 병원성입니다.바이러스가 질병을 일으키는 정도는 그것의 [27]독성이다.이러한 연구 분야는 식물 바이러스학, 동물 바이러스학,[28] 인간 바이러스학 또는 의학 바이러스학이라고 불립니다.
바이러스 검출
바이러스를 검출하는 방법에는 바이러스 입자(바이온)나 그 항원, 핵산 및 감염성 분석의 검출이 포함됩니다.
전자 현미경 검사
바이러스는 전자 현미경이 발명된 1930년대에 처음 발견되었다.이 현미경들은 빛 대신 전자 빔을 사용하는데, 이것은 훨씬 더 짧은 파장을 가지고 있고 빛 현미경으로 볼 수 없는 물체를 탐지할 수 있다.전자현미경을 사용하여 얻을 수 있는 일반적인 배율은 최대 1,000,000배이며[29], 광현미경으로 얻을 수 있는 최대 배율은 약 1,500배입니다.[30]
바이러스학자들은 바이러스의 시각화를 돕기 위해 음성 염색을 사용하는 경우가 많다.이 과정에서 바이러스는 초산우라늄과 같은 금속염 용액에 현탁된다.금속 원자는 전자에 불투명하며 바이러스는 금속 [29]원자의 어두운 배경에서 부유하는 것으로 보인다.이 기술은 1950년대부터 [31]사용되어 왔다.많은 바이러스들이 이 기술을 사용하여 발견되었고 음성 염색 전자 현미경은 바이러스학자의 [32]무기고에서 여전히 귀중한 무기이다.
기존 전자현미경 검사는 바이러스가 전자현미경 내부의 고진공에서 건조해 손상되고 전자빔 자체가 [29]파괴적이라는 단점이 있다.극저온전자현미경법에서는 유리수 환경에 바이러스를 [33]매립함으로써 바이러스의 구조를 보존한다.이를 통해 근원자 [34]분해능으로 생체 분자 구조를 결정할 수 있으며,[35] 바이러스 구조 결정을 위한 X선 결정학 또는 NMR 분광학의 대안으로 접근법에 대한 폭넓은 관심을 끌어왔다.
문화의 성장
바이러스는 필수 세포 내 기생충이며 숙주의 살아있는 세포 안에서만 번식하기 때문에 이러한 세포들은 실험실에서 그것들을 키우기 위해 필요하다.동물을 감염시키는 바이러스(일반적으로 "동물 바이러스"라고 불린다)의 경우 실험실 세포 배양에서 자란 세포가 사용됩니다.과거에는, 가임 닭의 알이 사용되었고 바이러스는 배아를 둘러싼 막에서 자라났다.이 방법은 여전히 일부 백신 제조에 사용되고 있다.세균을 감염시키는 바이러스인 박테리오파지는 시험관에서 자라는 세균을 직접 사용할 수 있다.식물 바이러스의 경우, 천연 숙주 식물을 사용할 수 있으며, 특히 감염이 명백하지 않은 경우에는 감염의 징후를 [36][37]더 명확하게 보이는 소위 지시 식물을 사용할 수 있습니다.
세포 배양에서 성장한 바이러스는 숙주 세포에 미치는 해로운 영향에 의해 간접적으로 검출될 수 있다.이러한 세포병리 효과는 종종 바이러스의 유형에 특징적이다.예를 들어, 단순 헤르페스 바이러스는 전형적으로 인간의 섬유아세포인 세포의 특징적인 "풍선화"를 생성한다.유행성 이하선염 바이러스와 같은 일부 바이러스는 닭의 적혈구가 감염된 세포에 단단히 부착되도록 한다.이것은 "해흡입" 또는 "혈흡입"이라고 불립니다.일부 바이러스는 플라크라고 불리는 세포층에서 국부적인 "결핍증"을 일으키는데, 이것은 정량 측정과 플라크 감소 [38][39]측정을 통한 바이러스 종 식별에 유용하다.
세포 배양에서 자라는 바이러스는 검증된 새로운 [40]항바이러스제에 대한 민감도를 측정하는데 사용된다.
혈청학
바이러스는 항체의 생성을 유도하는 항원으로 이 항체는 실험실에서 바이러스를 연구하는 데 사용될 수 있다.관련된 바이러스들은 종종 서로의 항체와 반응하며, 어떤 바이러스들은 그들이 반응하는 항체에 기초하여 이름이 지어질 수 있다.한 때 동물의 혈청(혈액)에서 독점적으로 추출된 항체를 사용하는 것을 [41]혈청학이라고 한다.일단 검사에서 항체-반응이 발생하면 이를 확인하기 위해 다른 방법이 필요하다.이전 방법에는 보체 고정 테스트,[42] 혈구 응집 억제 및 바이러스 중화 [43]등이 있었다.새로운 방법으로는 효소면역측정(EIA)[44]이 있습니다.
PCR이 발명되기 전 몇 년 동안 면역 형광은 바이러스 감염을 빠르게 확인하기 위해 사용되었다.항체가 사용되기 때문에 바이러스 종에 특화된 감염성 검사입니다.항체에는 발광성 염료가 부착되어 있으며 광원이 변경된 광학 현미경을 사용하면 감염된 세포가 [45]어둠 속에서 빛난다.
중합효소 연쇄반응(PCR) 및 기타 핵산 검출방법
PCR은 동식물을 포함한 모든 종의 바이러스를 검출하는 주요 방법이다.그것은 바이러스 특이 RNA나 DNA의 흔적을 발견함으로써 작동한다.매우 민감하고 특이하지만 오염으로 인해 쉽게 손상될 수 있습니다.수의학 바이러스학 및 의료 바이러스학에서 사용되는 대부분의 테스트는 PCR 또는 전사 매개 증폭과 같은 유사한 방법에 기초합니다.코로나 바이러스와 같은 새로운 바이러스가 출현했을 때, 바이러스 게놈이 배열되고 바이러스 DNA 또는 RNA의 고유한 영역이 [46]확인되는 한, 특정 테스트는 빠르게 고안될 수 있다.미세 유체학 시험에 이러한 시험들 대부분이 국경의 특이성이나 감수성에도 불구하고 automated,[47]도록 허용하는 발명, PCR그건 아마도 최대 21일 동안 잔류 바이러스 핵산은 좀 앉아에서 치우는 것을 허용하기 위하여 지연될 것 가지고 있고 비전 염성 감염 바이러스와"의 시험"차별화하지 않을 경우에 불리한 점이 있다.e는 o감염을 [48]막다.
진단 테스트
실험실에서 바이러스 검출을 위한 진단 테스트의 대부분은 PCR과 같은 핵산 증폭 방법이다.전자현미경 검사와 효소면역측정 등이 있어 바이러스나 그 성분을 검출하는 검사도 있다.소위 "집" 또는 "자체 검사"라고 불리는 장치는 보통 태그가 달린 모노클로널 [49]항체를 사용하여 바이러스를 탐지하는 측면 흐름 검사입니다.이것들은 농업, 식품, 환경 [50]과학에도 사용된다.
정량화 및 바이러스 부하
바이러스(정량화)의 세기는 바이러스학에서 항상 중요한 역할을 해 왔고, 바이러스 [51]부하를 측정하는 인간의 일부 감염을 제어하는 데 있어 핵심이 되었습니다.두 가지 기본적인 방법이 있습니다: 감염성 검사라고 불리는 완전 감염성 바이러스 입자를 세는 방법과 결함이 있는 [29]입자를 포함한 모든 입자를 세는 방법입니다.
감염성 평가
감염성 평가는 알려진 부피의 샘플에서 감염성 바이러스의 양([52]농도)을 측정합니다.숙주 세포의 경우, 박테리아 또는 동물 세포의 식물 또는 배양물이 사용됩니다.생쥐와 같은 실험용 동물들도 특히 수의 바이러스학에서 [53]사용되어 왔다.이러한 평가는 결과가 연속적인 척도로 나타나는 정량적 또는 사건이 발생하거나 발생하지 않는 정량적 측정이다.정량적 분석은 절대값을 제공하고 정량적 분석은 숙주 세포, 식물 또는 동물의 50% 감염을 보장하는 데 필요한 테스트 샘플의 부피와 같은 통계적 확률을 제공한다.이것은 중간 감염 용량 또는 [54]ID라고 불립니다. 감염성 박테리오파지는 배양 접시에 있는 박테리아 "세균"에 씨를 뿌림으로써 셀 수 있습니다.농도가 낮을 때, 바이러스는 셀 수 있는 잔디에 구멍을 형성한다.그리고 나서 바이러스의 수는 플라크 형성 단위로 표현된다.배양할 수 없는 세균에서 번식하는 박테리오파지는 바이러스 부하 측정법을 사용한다.[55]
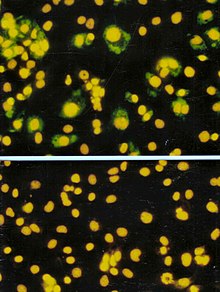
FFA(Focus Forming Assay)는 플라크 분석의 변형이지만 플라크 형성을 검출하기 위해 세포 용해에 의존하는 대신 FFA는 감염된 숙주 세포와 실제 플라크가 형성되기 전에 바이러스 항원에 특정한 형광 라벨이 부착된 항체를 사용하여 면역 항체 기술을 사용한다.FFA는 특히 세포막을 용해시키지 않는 바이러스의 종류를 정량화하는 데 유용합니다. 이러한 바이러스는 플라크 분석에 순응할 수 없기 때문입니다.플라크 분석과 마찬가지로 숙주 세포 단분자는 바이러스 샘플의 다양한 희석물에 감염되어 감염 바이러스의 확산을 제한하는 반고립체 중첩 매체 아래에서 비교적 짧은 잠복 기간(예: 24-72시간) 동안 배양할 수 있으며, 감염된 세포의 국소적 클러스터(foci)를 형성한다.이어서 형광표지 항체로 바이러스 항원에 대해 플레이트를 프로빙하고 형광현미경법을 사용하여 포시 수를 계수 및 정량화한다.FFA 방법은 일반적으로 플라크 또는 50% 조직배양감염선량(TCID50) 분석보다 시간이 짧지만, 필요한 시약과 장비 측면에서 더 비쌀 수 있다.검사 완료 시간도 사용자가 세는 영역의 크기에 따라 달라집니다.면적이 넓을수록 시간이 더 걸리지만 샘플을 더 정확하게 표현할 수 있습니다.FFA 결과는 밀리리터당 포커스 형성 단위(FFU/)[56]로 표현된다.
바이러스 부하 측정
감염성 바이러스 입자의 측정(Plaque assay, Focus assay)을 실시할 때 바이러스 적정량은 감염성 바이러스 입자의 농도를 나타내며, 이는 전체 바이러스 입자와는 다르다.바이러스 부하 측정은 일반적으로 입자의 수보다 존재하는 바이러스 게놈의 수를 세며 [57]PCR과 유사한 방법을 사용합니다.바이러스 부하 검사는 HIV에 [58]의한 감염을 통제하는데 중요하다.이 다용도 방법은 식물 [59][60]바이러스에 사용할 수 있습니다.
분자생물학
분자 바이러스학은 핵산과 단백질 수준의 바이러스를 연구하는 학문이다.분자생물학자들이 발명한 방법들은 모두 바이러스학에서 유용한 것으로 입증되었다.작은 크기와 비교적 단순한 구조는 바이러스를 이러한 기술로 연구하기에 이상적인 후보로 만든다.
바이러스 및 그 컴포넌트 정화
더 많은 연구를 위해, 실험실에서 자란 바이러스는 숙주 세포에서 오염 물질을 제거하기 위해 정화가 필요하다.사용되는 방법은 바이러스를 집중시킬 수 있다는 장점이 있기 때문에 바이러스를 조사하는 것이 쉬워집니다.
원심 분리
원심분리기는 종종 바이러스를 정화하기 위해 사용된다.저속 원심분리기, 즉 최고속도가 분당 10,000회전(rpm)인 원심분리기는 바이러스를 집중시킬 만큼 강력하지 않지만 최고속도가 약 10,000rpm인 초원심분리기는 이 차이를 차등원심분리라고 하는 방법으로 사용한다.이 방법에서는 저속 원심분리에 의해 바이러스 혼합물에서 크고 무거운 오염물질을 제거한다.작고 가벼워 현탁 상태로 방치된 바이러스는 고속 [62]원심분리를 통해 농축된다.
차동 원심분리 후 바이러스 현탁액은 종종 침전계수가 동일하고 절차에 따라 제거되지 않는 파편에 오염된 상태로 남아 있습니다.이러한 경우, 부력 밀도 원심 분리라고 하는 원심 분리의 수정이 사용됩니다.이 방법에서는 미분 원심분리 시 회수된 바이러스를 원심분리 중 튜브 내에서 낮은 것부터 높은 것까지 밀도 구배를 형성하는 설탕 또는 소금의 고밀도 용액에서 몇 시간 동안 매우 빠른 속도로 다시 원심분리한다.어떤 경우에는 밀도가 지속적으로 감소하는 솔루션이 서로 조심스럽게 중첩되는 미리 형성된 구배를 사용한다.사해의 물체처럼, 원심력에도 불구하고, 바이러스 입자는 그들보다 더 밀도가 높은 용액에 가라앉을 수 없고, 그들은 종종 눈에 보이는 농축 바이러스의 분리된 층을 형성합니다.염화세슘은 비교적 불활성이지만 초원심분리기에서 [61]고속으로 원심분리하면 쉽게 구배를 형성하기 때문에 이러한 용액에 자주 사용된다.부력밀도 원심분리법은 핵산이나 [63]단백질과 같은 바이러스의 성분을 정화하는데도 사용될 수 있다.
전기영동
전하를 기반으로 한 분자의 분리는 전기영동이라고 불린다.이 방법으로 바이러스와 그 모든 성분을 분리하여 정제할 수 있다.이것은 보통 아가로스나 폴리아크릴아미드겔과 같은 지지 매체에서 이루어집니다.분리된 분자는 단백질의 경우 쿠마시 블루, 핵산의 경우 브롬화 에티듐과 같은 얼룩을 사용하여 드러난다.어떤 경우에는 바이러스 성분이 [64]전자영동이 오토라디오그래피라고 알려진 프로세스에서 사진필름을 사용하여 밝혀지기 전에 방사성을 띠게 된다.
바이러스 게놈의 배열
대부분의 바이러스는 너무 작아서 빛 현미경으로 볼 수 없기 때문에, 염기서열 분석은 바이러스를 식별하고 연구하기 위한 바이러스학의 주요 도구 중 하나이다.기존의 생어 시퀀싱과 차세대 시퀀싱(NGS)은 새로운 바이러스 감염 진단, 바이러스 병원체의 분자 역학, 약물 내성 테스트뿐만 아니라 기초 및 임상 연구에서 바이러스의 시퀀싱에 사용됩니다.GenBank에는 [65]230만 개 이상의 고유한 바이러스 서열이 있습니다.NGS는 바이러스 [65]게놈을 생성하는 가장 인기 있는 방법으로서 전통적인 생어를 능가했다.바이러스 게놈 배열은 바이러스 역학 및 바이러스 분류의 중심 방법이 되었다.
계통학적 분석
바이러스 게놈 배열의 데이터는 진화적 관계를 결정하기 위해 사용될 수 있으며 이것은 계통학적 [66]분석이라고 불린다.PHYLIP와 같은 소프트웨어는 계통수를 그리는 데 사용됩니다.이 분석은 또한 지역사회에서 바이러스 감염의 확산을 연구하는 [67]데에도 사용된다.
클로닝
진단검사나 백신에 정제된 바이러스나 바이러스 성분이 필요할 경우 바이러스를 [68]키우는 대신 복제를 사용할 수 있다.COVID-19 대유행 시작 시 중증 급성 호흡기 증후군 코로나바이러스 2 RNA 시퀀스의 가용성은 테스트를 신속하게 [69]제조할 수 있게 했다.바이러스 및 그 컴포넌트를 복제하는 검증된 방법이 몇 가지 있습니다.클로닝 벡터라고 불리는 DNA의 작은 조각들이 종종 사용되며 가장 흔한 것은 실험실에서 변형된 플라스미드이다.바이러스 핵산 또는 그 일부가 플라스미드에 삽입되어 박테리아에 의해 여러 번 복제됩니다.이 재조합 DNA는 토종 [70]바이러스의 필요 없이 바이러스 성분을 생산하는 데 사용될 수 있다.
파지 바이러스학
박테리아, 고세균, 곰팡이균에서 번식하는 바이러스는 비공식적으로 "phages"[71]라고 불리며, 박테리아를 감염시키는 바이러스학 및 생물학에서 [72]특히 유용합니다.박테리오파지는 20세기 [73]초에 발견된 첫 번째 바이러스 중 일부였고, 실험실에서 비교적 빨리 자라기 쉽기 때문에, 바이러스에 대한 우리의 이해의 대부분은 그것들을 [73]연구함으로써 비롯되었다.환경에서의 긍정적인 효과로 오랫동안 알려진 박테리오파지는 단백질 DNA 서열을 선별하기 위한 파지 표시 기술에 사용된다.그것들은 분자생물학에서 [74]강력한 도구이다.
유전학
모든 바이러스는 [75]유전학을 사용하여 연구되는 유전자를 가지고 있다.복제, 돌연변이 RNA 소음 생성과 같은 분자 생물학에서 사용되는 모든 기술은 바이러스 [76]유전학에 사용됩니다.
재할 것
재조합은 서로 다른 부모로부터 유전자를 바꾸는 것으로, 인플루엔자 바이러스나 로타바이러스와 같이 분절된 게놈(두 개 이상의 핵산 분자로 조각난)을 가지는 바이러스의 유전학을 연구할 때 특히 유용합니다.혈청형과 같은 특성을 코드하는 유전자는 이런 방식으로 [77]식별될 수 있다.
재결합
종종 재조합과 혼동되는 재조합은 유전자의 혼합이기도 하지만, 메커니즘은 RNA 또는 DNA 복제 주기 동안 완전한 분자가 아닌 DNA 또는 RNA 분자의 연장선이 결합된다는 점에서 다릅니다.재조합은 자연에서 재조합만큼 흔하지는 않지만 바이러스 [78]유전자의 구조와 기능을 연구하기 위한 실험실에서 강력한 도구이다.
역유전학
역유전학은 바이러스학에서 [79]강력한 연구방법이다.이 절차에서는 "감염성 클론"이라고 불리는 바이러스 게놈의 무료 DNA(cDNA) 복사를 사용하여 유전자가 조작된 바이러스를 생산하고, 그 후 독성 또는 전염성의 [80]변화를 테스트할 수 있습니다.
바이러스 분류
바이러스학의 주요 분야는 바이러스 분류이다.진화 계통학이 아니라 바이러스의 [81][82]공유 또는 구별되는 성질을 기반으로 한다는 점에서 인위적이다.유사성에 [83]따라 바이러스의 이름을 붙이고 그룹화하여 바이러스의 다양성을 기술하려고 합니다.1962년 Andre Lwoff, Robert Horne 및 Paul Tournier는 린네의 계층 체계를 [84]기반으로 바이러스 분류 수단을 개발한 최초의 사람이었다.이 체계는 문, 계급, 목, 과, 속, 종을 기준으로 분류한다.바이러스는 숙주의 특성이 아닌 공유 특성 및 [85]게놈을 형성하는 핵산의 유형에 따라 분류되었습니다.1966년, 국제 바이러스 분류 위원회(ICTV)가 결성되었다.Lwoff, Horne, Tournier가 제안한 시스템은 바이러스의 작은 게놈 크기와 높은 돌연변이율로 인해 그들의 조상을 순서를 초월한 것으로 판단하기가 어려웠기 때문에 ICTV에 의해 처음에는 받아들여지지 않았다.이와 같이, 볼티모어 분류 시스템은 보다 전통적인 계층을 [86]보완하기 위해 사용되게 되었다.ICTV는 2018년부터 시간이 지남에 따라 발견된 바이러스 간의 진화적 관계를 인정하기 시작했고 영역과 [87]종에 이르는 15등급 분류 시스템을 채택했습니다.또, 같은 속내의 일부 종들은, 제노그룹으로 [88][89]그룹화된다.
ICTV 분류
ICTV는 현재의 분류 시스템을 개발하고 가족의 일치를 유지하기 위해 특정 바이러스의 특성에 더 큰 비중을 두는 지침을 작성했다.통합 분류 체계(바이러스를 분류하는 보편적인 체계)가 확립되었습니다.바이러스의 총 다양성 중 극히 일부만이 [90]연구되었다.2021년 현재 ICTV는 [91]6개 영역, 10개 왕국, 17개 섬, 2개 섬, 39개 클래스, 65개 아목, 8개 아목, 233개 과, 168개 아과, 2,606개 속, 84개 아속, 10,434종의 바이러스를 정의했다.
분류범위의 일반적인 분류구조와 분류명칭에 사용되는 접미사는 다음과 같다.2021년 현재 서브영역, 서브킹덤 및 서브클래스의 순위는 사용되지 않으며 다른 순위는 [91]모두 사용되고 있습니다.
- 레름(-viria)
볼티모어 분류
노벨상 수상자인 데이비드 볼티모어는 볼티모어 분류 시스템을 [92]고안했다.
볼티모어의 바이러스 분류는 mRNA 생산 메커니즘을 기반으로 합니다.바이러스는 단백질을 생산하고 스스로를 복제하기 위해 게놈에서 mRNA를 생성해야 하지만, 각 바이러스 계열에서 이를 달성하기 위해 다른 메커니즘이 사용된다.바이러스 게놈은 단일가닥(ss) 또는 이중가닥(ds), RNA 또는 DNA일 수 있으며 역전사효소(RT)를 사용할 수도 있고 사용하지 않을 수도 있다.또한 ssRNA 바이러스는 센스(+) 또는 안티센스(-) 중 하나입니다.이 분류에서는 바이러스를 7개의 그룹으로 분류합니다.
- I: dsDNA 바이러스(Adenovirus, Herpesvirus, Poxvirus 등)
- II: ssDNA 바이러스(+ 가닥 또는 "센스") DNA(Parvovirus 등)
- III: dsRNA 바이러스(Reovirus 등)
- IV:(+) ssRNA 바이러스(+ 가닥 또는 감지) RNA(예: Coronavirus, Picornavirus, Togavirus)
- V: (-)ssRNA 바이러스(-가닥 또는 안티센스) RNA(예: Orthomyxovirus, Rhabdovirus)
- VI: 라이프 사이클에서 DNA 중간체를 가진 ssRNA-RT 바이러스(+가닥 또는 감지) RNA(예: 레트로바이러스)
- VII: dsDNA-RT 바이러스(라이프 사이클에서 RNA 중간체)
레퍼런스
- ^ Bordenave G (May 2003). "Louis Pasteur (1822-1895)". Microbes and Infection. 5 (6): 553–60. doi:10.1016/S1286-4579(03)00075-3. PMID 12758285.
- ^ 쇼르 페이지 74, 827
- ^ a b 콜리어 페이지 3
- ^ Dimmock 페이지 4
- ^ Dimmock pp.4–5
- ^ Fenner F (2009). Mahy BW, Van Regenmortal MH (eds.). Desk Encyclopedia of General Virology (1 ed.). Oxford: Academic Press. p. 15. ISBN 978-0-12-375146-1.
- ^ Shors 페이지 827
- ^ D'Herelle F (September 2007). "On an invisible microbe antagonistic toward dysenteric bacilli: brief note by Mr. F. D'Herelle, presented by Mr. Roux. 1917". Research in Microbiology. 158 (7): 553–54. doi:10.1016/j.resmic.2007.07.005. PMID 17855060.
- ^ Domingo-Calap P, Georgel P, Bahram S (March 2016). "Back to the future: bacteriophages as promising therapeutic tools". HLA. 87 (3): 133–40. doi:10.1111/tan.12742. PMID 26891965. S2CID 29223662.
- ^ Steinhardt E, Israeli C, Lambert RA (1913). "Studies on the cultivation of the virus of vaccinia". The Journal of Infectious Diseases. 13 (2): 294–300. doi:10.1093/infdis/13.2.294.
- ^ 콜리어 페이지 4
- ^ Goodpasture EW, Woodruff AM, Buddingh GJ (October 1931). "The cultivation of vaccine and other viruses in the chorioallantoic membrane of chick embryos". Science. 74 (1919): 371–72. Bibcode:1931Sci....74..371G. doi:10.1126/science.74.1919.371. PMID 17810781.
- ^ Thomas Huckle Weller (2004). Growing Pathogens in Tissue Cultures: Fifty Years in Academic Tropical Medicine, Pediatrics, and Virology. Boston Medical Library. p. 57. ISBN 978-0-88135-380-8.
- ^ Rosen FS (October 2004). "Isolation of poliovirus--John Enders and the Nobel Prize". The New England Journal of Medicine. 351 (15): 1481–83. doi:10.1056/NEJMp048202. PMID 15470207.
- ^ Frängsmyr T, Ekspång G, eds. (1993). Nobel Lectures, Physics 1981–1990. Singapore: World Scientific Publishing Co. Bibcode:1993nlp..book.....F.
- 1887년, 뷰스트는 가장 큰 바이러스 중 하나인 바키니아 바이러스를 염색한 후 광학 현미경으로 시각화했다.그 당시에는 백시니아가 바이러스로 알려져 있지 않았다.(Buist J.B.Baccinia와 Variola: 그들의 인생사 연구 Churchill, London)
- ^ Stanley WM, Loring HS (January 1936). "The Isolation of Crystalline Tobacco Mosaic Virus Protein From Diseased Tomato Plants". Science. 83 (2143): 85. Bibcode:1936Sci....83...85S. doi:10.1126/science.83.2143.85. PMID 17756690.
- ^ Stanley WM, Lauffer MA (April 1939). "Disintegration of Tobacco Mosaic Virus in Urea Solutions". Science. 89 (2311): 345–47. Bibcode:1939Sci....89..345S. doi:10.1126/science.89.2311.345. PMID 17788438.
- ^ Creager AN, Morgan GJ (June 2008). "After the double helix: Rosalind Franklin's research on Tobacco mosaic virus". Isis; an International Review Devoted to the History of Science and Its Cultural Influences. 99 (2): 239–72. doi:10.1086/588626. PMID 18702397. S2CID 25741967.
- ^ Dimmock 페이지 12
- ^ Norrby E (2008). "Nobel Prizes and the emerging virus concept". Archives of Virology. 153 (6): 1109–23. doi:10.1007/s00705-008-0088-8. PMID 18446425. S2CID 10595263.
- ^ 콜리에 페이지 745
- ^ Temin HM, Baltimore D (1972). "RNA-directed DNA synthesis and RNA tumor viruses". Advances in Virus Research. 17: 129–86. doi:10.1016/S0065-3527(08)60749-6. ISBN 9780120398171. PMID 4348509.
- ^ Barré-Sinoussi F, Chermann JC, Rey F, Nugeyre MT, Chamaret S, Gruest J, et al. (May 1983). "Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS)". Science. 220 (4599): 868–71. Bibcode:1983Sci...220..868B. doi:10.1126/science.6189183. PMID 6189183.
- ^ Choo QL, Kuo G, Weiner AJ, Overby LR, Bradley DW, Houghton M (April 1989). "Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome". Science. 244 (4902): 359–62. Bibcode:1989Sci...244..359C. CiteSeerX 10.1.1.469.3592. doi:10.1126/science.2523562. PMID 2523562.
- ^ Houghton M (November 2009). "The long and winding road leading to the identification of the hepatitis C virus". Journal of Hepatology. 51 (5): 939–48. doi:10.1016/j.jhep.2009.08.004. PMID 19781804.
- ^ Dolja VV, Koonin EV (November 2011). "Common origins and host-dependent diversity of plant and animal viromes". Current Opinion in Virology. 1 (5): 322–31. doi:10.1016/j.coviro.2011.09.007. PMC 3293486. PMID 22408703.
- ^ Novella IS, Presloid JB, Taylor RT (December 2014). "RNA replication errors and the evolution of virus pathogenicity and virulence". Current Opinion in Virology. 9: 143–7. doi:10.1016/j.coviro.2014.09.017. PMID 25462446.
- ^ BMC Medicine 2020
- ^ a b c d 페인 S.바이러스 연구 방법바이러스.2017;37-52.doi:10.1016/B978-0-12-803109-4.00004-0
- ^ 현미경, 크기, 확대(CCEA) BBC
- ^ Brenner S, Horne RW (July 1959). "A negative staining method for high resolution electron microscopy of viruses". Biochimica et Biophysica Acta. 34: 103–10. doi:10.1016/0006-3002(59)90237-9. PMID 13804200.
- ^ Goldsmith CS, Miller SE (October 2009). "Modern uses of electron microscopy for detection of viruses". Clinical Microbiology Reviews. 22 (4): 552–63. doi:10.1128/CMR.00027-09. PMC 2772359. PMID 19822888.
- ^ Tivol, William F.; Briegel, Ariane; Jensen, Grant J. (October 2008). "An Improved Cryogen for Plunge Freezing". Microscopy and Microanalysis. 14 (5): 375–379. Bibcode:2008MiMic..14..375T. doi:10.1017/S1431927608080781. ISSN 1431-9276. PMC 3058946. PMID 18793481.
- ^ Cheng Y, Grigorieff N, Penczek PA, Walz T (April 2015). "A primer to single-particle cryo-electron microscopy". Cell. 161 (3): 438–449. doi:10.1016/j.cell.2015.03.050. PMC 4409659. PMID 25910204.
- ^ Stoddart, Charlotte (1 March 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. Retrieved 25 March 2022.
- ^ Liu JZ, Richerson K, Nelson RS (August 2009). "Growth conditions for plant virus-host studies". Current Protocols in Microbiology. Chapter 16: Unit16A.1. doi:10.1002/9780471729259.mc16a01s14. PMID 19653216. S2CID 41236532.
- ^ Valmonte-Cortes GR, Lilly ST, Pearson MN, Higgins CM, MacDiarmid RM (January 2022). "The Potential of Molecular Indicators of Plant Virus Infection: Are Plants Able to Tell Us They Are Infected?". Plants (Basel, Switzerland). 11 (2): 188. doi:10.3390/plants11020188. PMC 8777591. PMID 35050076.
- ^ Gauger PC, Vincent AL (2014). "Serum virus neutralization assay for detection and quantitation of serum-neutralizing antibodies to influenza A virus in swine". Animal Influenza Virus. Methods in Molecular Biology (Clifton, N.J.). Vol. 1161. pp. 313–24. doi:10.1007/978-1-4939-0758-8_26. ISBN 978-1-4939-0757-1. PMID 24899440.
- ^ Dimitrova K, Mendoza EJ, Mueller N, Wood H (2020). "A Plaque Reduction Neutralization Test for the Detection of ZIKV-Specific Antibodies". Zika Virus. Methods in Molecular Biology (Clifton, N.J.). Vol. 2142. pp. 59–71. doi:10.1007/978-1-0716-0581-3_5. ISBN 978-1-0716-0580-6. PMID 32367358. S2CID 218504421.
- ^ Lampejo T (July 2020). "Influenza and antiviral resistance: an overview". European Journal of Clinical Microbiology & Infectious Diseases. 39 (7): 1201–1208. doi:10.1007/s10096-020-03840-9. PMC 7223162. PMID 32056049.
- ^ Zainol Rashid Z, Othman SN, Abdul Samat MN, Ali UK, Wong KK (April 2020). "Diagnostic performance of COVID-19 serology assays". The Malaysian Journal of Pathology. 42 (1): 13–21. PMID 32342927.
- ^ Swack NS, Gahan TF, Hausler WJ (August 1992). "The present status of the complement fixation test in viral serodiagnosis". Infectious Agents and Disease. 1 (4): 219–24. PMID 1365549.
- ^ Smith TJ (August 2011). "Structural studies on antibody recognition and neutralization of viruses". Current Opinion in Virology. 1 (2): 150–6. doi:10.1016/j.coviro.2011.05.020. PMC 3163491. PMID 21887208.
- ^ Mahony JB, Petrich A, Smieja M (2011). "Molecular diagnosis of respiratory virus infections". Critical Reviews in Clinical Laboratory Sciences. 48 (5–6): 217–49. doi:10.3109/10408363.2011.640976. PMID 22185616. S2CID 24960083.
- ^ AbuSalah MA, Gan SH, Al-Hatamleh MA, Irekeola AA, Shueb RH, Yean Yean C (March 2020). "Recent Advances in Diagnostic Approaches for Epstein-Barr Virus". Pathogens (Basel, Switzerland). 9 (3): 226. doi:10.3390/pathogens9030226. PMC 7157745. PMID 32197545.
- ^ Zhu H, Zhang H, Xu Y, Laššáková S, Korabečná M, Neužil P (October 2020). "PCR past, present and future". BioTechniques. 69 (4): 317–325. doi:10.2144/btn-2020-0057. PMC 7439763. PMID 32815744.
- ^ Wang X, Hong XZ, Li YW, Li Y, Wang J, Chen P, Liu BF (March 2022). "Microfluidics-based strategies for molecular diagnostics of infectious diseases". Military Medical Research. 9 (1): 11. doi:10.1186/s40779-022-00374-3. PMC 8930194. PMID 35300739.
- ^ Benzigar MR, Bhattacharjee R, Baharfar M, Liu G (April 2021). "Current methods for diagnosis of human coronaviruses: pros and cons". Analytical and Bioanalytical Chemistry. 413 (9): 2311–2330. doi:10.1007/s00216-020-03046-0. PMC 7679240. PMID 33219449.
- ^ 버렐, C.J., 하워드, C.R. & 머피, F.A. (2017).바이러스 질환의 실험실 진단.페너 앤 화이트 의학 바이러스학, 135-154.https://doi.org/10.1016/B978-0-12-375156-0.00010-2
- ^ Koczula KM, Gallotta A (June 2016). "Lateral flow assays". Essays in Biochemistry. 60 (1): 111–20. doi:10.1042/EBC20150012. PMC 4986465. PMID 27365041.
- ^ Lee MJ (October 2021). "Quantifying SARS-CoV-2 viral load: current status and future prospects". Expert Review of Molecular Diagnostics. 21 (10): 1017–1023. doi:10.1080/14737159.2021.1962709. PMC 8425446. PMID 34369836.
- ^ Mistry BA, D'Orsogna MR, Chou T (June 2018). "The Effects of Statistical Multiplicity of Infection on Virus Quantification and Infectivity Assays". Biophysical Journal. 114 (12): 2974–2985. arXiv:1805.02810. Bibcode:2018BpJ...114.2974M. doi:10.1016/j.bpj.2018.05.005. PMC 6026352. PMID 29925033.
- ^ Kashuba C, Hsu C, Krogstad A, Franklin C (January 2005). "Small mammal virology". The Veterinary Clinics of North America. Exotic Animal Practice. 8 (1): 107–22. doi:10.1016/j.cvex.2004.09.004. PMC 7110861. PMID 15585191.
- ^ Cutler TD, Wang C, Hoff SJ, Kittawornrat A, Zimmerman JJ (August 2011). "Median infectious dose (ID50) of porcine reproductive and respiratory syndrome virus isolate MN-184 via aerosol exposure". Veterinary Microbiology. 151 (3–4): 229–37. doi:10.1016/j.vetmic.2011.03.003. PMID 21474258.
- ^ Moon K, Cho JC (March 2021). "Metaviromics coupled with phage-host identification to open the viral "black box"". Journal of Microbiology (Seoul, Korea). 59 (3): 311–323. doi:10.1007/s12275-021-1016-9. PMID 33624268. S2CID 232023531.
- ^ Salgado EN, Upadhyayula S, Harrison SC (September 2017). "Single-Particle Detection of Transcription following Rotavirus Entry". Journal of Virology. 91 (18). doi:10.1128/JVI.00651-17. PMC 5571246. PMID 28701394.
- ^ Yokota I, Hattori T, Shane PY, Konno S, Nagasaka A, Takeyabu K, Fujisawa S, Nishida M, Teshima T (February 2021). "Equivalent SARS-CoV-2 viral loads by PCR between nasopharyngeal swab and saliva in symptomatic patients". Scientific Reports. 11 (1): 4500. Bibcode:2021NatSR..11.4500Y. doi:10.1038/s41598-021-84059-2. PMC 7904914. PMID 33627730.
- ^ Nichols BE, Girdwood SJ, Crompton T, Stewart-Isherwood L, Berrie L, Chimhamhiwa D, Moyo C, Kuehnle J, Stevens W, Rosen S (September 2019). "Monitoring viral load for the last mile: what will it cost?". Journal of the International AIDS Society. 22 (9): e25337. doi:10.1002/jia2.25337. PMC 6742838. PMID 31515967.
- ^ Shirima RR, Maeda DG, Kanju E, Ceasar G, Tibazarwa FI, Legg JP (July 2017). "Absolute quantification of cassava brown streak virus mRNA by real-time qPCR". Journal of Virological Methods. 245: 5–13. doi:10.1016/j.jviromet.2017.03.003. PMC 5429390. PMID 28315718.
- ^ Rubio L, Galipienso L, Ferriol I (2020). "Detection of Plant Viruses and Disease Management: Relevance of Genetic Diversity and Evolution". Frontiers in Plant Science. 11: 1092. doi:10.3389/fpls.2020.01092. PMC 7380168. PMID 32765569.
- ^ a b Beards GM (August 1982). "A method for the purification of rotaviruses and adenoviruses from faeces". Journal of Virological Methods. 4 (6): 343–52. doi:10.1016/0166-0934(82)90059-3. PMID 6290520.
- ^ Zhou Y, McNamara RP, Dittmer DP (August 2020). "Purification Methods and the Presence of RNA in Virus Particles and Extracellular Vesicles". Viruses. 12 (9): 917. doi:10.3390/v12090917. PMC 7552034. PMID 32825599.
- ^ Su Q, Sena-Esteves M, Gao G (May 2019). "Purification of the Recombinant Adenovirus by Cesium Chloride Gradient Centrifugation". Cold Spring Harbor Protocols. 2019 (5): pdb.prot095547. doi:10.1101/pdb.prot095547. PMID 31043560. S2CID 143423942.
- ^ Klepárník K, Boček P (March 2010). "Electrophoresis today and tomorrow: Helping biologists' dreams come true". BioEssays : News and Reviews in Molecular, Cellular and Developmental Biology. 32 (3): 218–226. doi:10.1002/bies.200900152. PMID 20127703. S2CID 41587013.
- ^ a b Castro, Christina; Marine, Rachel; Ramos, Edward; Ng, Terry Fei Fan (22 June 2020). "The effect of variant interference on de novo assembly for viral deep sequencing". BMC Genomics. 21 (1): 421. doi:10.1186/s12864-020-06801-w. PMC 7306937. PMID 32571214.
- ^ Cui J, Li F, Shi ZL (March 2019). "Origin and evolution of pathogenic coronaviruses". Nature Reviews. Microbiology. 17 (3): 181–192. doi:10.1038/s41579-018-0118-9. PMC 7097006. PMID 30531947.
- ^ 고발레냐 AE, 라우버 C바이러스의 계통학바이오메디컬 사이언스 레퍼런스 모듈.2017; B978-0-12-801238-3.95723-4.doi:10.1016/B978-0-12-801238-3.95723-4
- ^ Koch L (July 2020). "A platform for RNA virus cloning". Nature Reviews. Genetics. 21 (7): 388. doi:10.1038/s41576-020-0246-8. PMC 7220607. PMID 32404960.
- ^ Thi Nhu Thao T, Labroussaa F, Ebert N, V'kovski P, Stalder H, Portmann J, Kelly J, Steiner S, Holwerda M, Kratzel A, Gultom M, Schmied K, Laloli L, Hüsser L, Wider M, Pfaender S, Hirt D, Cippà V, Crespo-Pomar S, Schröder S, Muth D, Niemeyer D, Corman VM, Müller MA, Drosten C, Dijkman R, Jores J, Thiel V (June 2020). "Rapid reconstruction of SARS-CoV-2 using a synthetic genomics platform". Nature. 582 (7813): 561–565. Bibcode:2020Natur.582..561T. doi:10.1038/s41586-020-2294-9. PMID 32365353. S2CID 213516085.
- ^ Rosano GL, Morales ES, Ceccarelli EA (August 2019). "New tools for recombinant protein production in Escherichia coli: A 5-year update". Protein Science. 28 (8): 1412–1422. doi:10.1002/pro.3668. PMC 6635841. PMID 31219641.
- ^ Pennazio S (2006). "The origin of phage virology". Rivista di Biologia. 99 (1): 103–29. PMID 16791793.
- ^ Harada LK, Silva EC, Campos WF, Del Fiol FS, Vila M, Dąbrowska K, Krylov VN, Balcão VM (2018). "Biotechnological applications of bacteriophages: State of the art". Microbiological Research. 212–213: 38–58. doi:10.1016/j.micres.2018.04.007. PMID 29853167. S2CID 46921731.
- ^ a b Stone E, Campbell K, Grant I, McAuliffe O (June 2019). "Understanding and Exploiting Phage-Host Interactions". Viruses. 11 (6): 567. doi:10.3390/v11060567. PMC 6630733. PMID 31216787.
- ^ Nagano K, Tsutsumi Y (January 2021). "Phage Display Technology as a Powerful Platform for Antibody Drug Discovery". Viruses. 13 (2): 178. doi:10.3390/v13020178. PMC 7912188. PMID 33504115.
- ^ Ibrahim B, McMahon DP, Hufsky F, Beer M, Deng L, Mercier PL, Palmarini M, Thiel V, Marz M (June 2018). "A new era of virus bioinformatics". Virus Research. 251: 86–90. doi:10.1016/j.virusres.2018.05.009. PMID 29751021.
- ^ Bamford, Dennis; Zuckerman, Mark A. (2021). Encyclopedia of virology. Amsterdam. ISBN 978-0-12-814516-6. OCLC 1240584737.
- ^ McDonald SM, Nelson MI, Turner PE, Patton JT (July 2016). "Reassortment in segmented RNA viruses: mechanisms and outcomes". Nature Reviews. Microbiology. 14 (7): 448–60. doi:10.1038/nrmicro.2016.46. PMC 5119462. PMID 27211789.
- ^ Li J, Arévalo MT, Zeng M (2013). "Engineering influenza viral vectors". Bioengineered. 4 (1): 9–14. doi:10.4161/bioe.21950. PMC 3566024. PMID 22922205.
- ^ Lee CW (2014). "Reverse genetics of influenza virus". Methods in Molecular Biology (Clifton, N.J.). 1161: 37–50. doi:10.1007/978-1-4939-0758-8_4. PMID 24899418.
- ^ Li Z, Zhong L, He J, Huang Y, Zhao Y (April 2021). "Development and application of reverse genetic technology for the influenza virus". Virus Genes. 57 (2): 151–163. doi:10.1007/s11262-020-01822-9. PMC 7851324. PMID 33528730.
- ^ Hull R, Rima B (November 2020). "Virus taxonomy and classification: naming of virus species". Archives of Virology. 165 (11): 2733–2736. doi:10.1007/s00705-020-04748-7. PMID 32740831. S2CID 220907379.
- ^ Pellett PE, Mitra S, Holland TC (2014). "Basics of virology". Handbook of Clinical Neurology. 123: 45–66. doi:10.1016/B978-0-444-53488-0.00002-X. ISBN 9780444534880. PMC 7152233. PMID 25015480.
- ^ Simmonds P, Aiewsakun P (August 2018). "Virus classification - where do you draw the line?". Archives of Virology. 163 (8): 2037–2046. doi:10.1007/s00705-018-3938-z. PMC 6096723. PMID 30039318.
- ^ Lwoff A, Horne RW, Tournier P (June 1962). "[A virus system]". Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences (in French). 254: 4225–27. PMID 14467544.
- ^ Lwoff A, Horne R, Tournier P (1962). "A system of viruses". Cold Spring Harbor Symposia on Quantitative Biology. 27: 51–55. doi:10.1101/sqb.1962.027.001.008. PMID 13931895.
- ^ Fauquet CM, Fargette D (August 2005). "International Committee on Taxonomy of Viruses and the 3,142 unassigned species". Virology Journal. 2: 64. doi:10.1186/1743-422X-2-64. PMC 1208960. PMID 16105179.
- ^ International Committee on Taxonomy of Viruses Executive Committee (May 2020). "The New Scope of Virus Taxonomy: Partitioning the Virosphere Into 15 Hierarchical Ranks". Nat Microbiol. 5 (5): 668–674. doi:10.1038/s41564-020-0709-x. PMC 7186216. PMID 32341570.
- ^ Khan MK, Alam MM (July 2021). "Norovirus Gastroenteritis Outbreaks, Genomic Diversity and Evolution: An Overview". Mymensingh Medical Journal : MMJ. 30 (3): 863–873. PMID 34226482.
- ^ Eberle J, Gürtler L (2012). "HIV types, groups, subtypes and recombinant forms: errors in replication, selection pressure and quasispecies". Intervirology. 55 (2): 79–83. doi:10.1159/000331993. PMID 22286874. S2CID 5642060.
- ^ Delwart EL (2007). "Viral metagenomics". Reviews in Medical Virology. 17 (2): 115–31. doi:10.1002/rmv.532. PMC 7169062. PMID 17295196.
- ^ a b "Virus Taxonomy: 2021 Release". talk.ictvonline.org. International Committee on Taxonomy of Viruses. Retrieved 4 April 2022.
- ^ Koonin EV, Krupovic M, Agol VI (August 2021). "The Baltimore Classification of Viruses 50 Years Later: How Does It Stand in the Light of Virus Evolution?" (PDF). Microbiology and Molecular Biology Reviews. 85 (3): e0005321. doi:10.1128/MMBR.00053-21. PMID 34259570. S2CID 235821748.
참고 문헌
- Collier L, Balows A, Sussman M (1998). Mahy B, Collier LA (eds.). Topley and Wilson's Microbiology and Microbial Infections. Virology. Vol. 1 (Ninth ed.). ISBN 0-340-66316-2.
- Dimmock NJ, Easton AJ, Leppard K (2007). Introduction to Modern Virology (Sixth ed.). Blackwell Publishing. ISBN 978-1-4051-3645-7.
- Shors T (2017). Understanding Viruses. Jones and Bartlett Publishers. ISBN 978-1-284-02592-7.
외부 링크
- ICTV: 국제 바이러스 분류 위원회