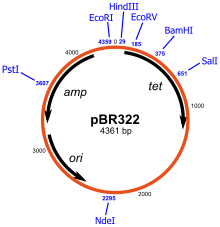
플라스미드
Plasmid플라스미드는 염색체 DNA에서 물리적으로 분리되어 독립적으로 복제할 수 있는 세포 내의 작은 염색체 외 DNA 분자이다.그들은 박테리아에서 작은 원형, 이중 가닥 DNA 분자로 가장 흔하게 발견됩니다; 하지만 플라스미드는 때때로 고세균과 진핵 [1][2]생물에 존재합니다.자연에서 플라스미드는 종종 유기체의 생존에 도움이 되는 유전자를 가지고 있고 항생제 내성과 같은 선택적 이점을 준다.염색체는 크고 정상적인 조건에서 살기 위한 모든 필수 유전 정보를 포함하고 있는 반면, 플라스미드는 보통 매우 작고 특정한 상황이나 조건에서 유용할 수 있는 추가적인 유전자만 포함합니다.인공 플라스미드는 분자 복제에서 벡터로 널리 사용되며 숙주 유기체 내에서 재조합 DNA 서열의 복제를 촉진하는 역할을 한다.실험실에서 플라스미드는 형질전환을 통해 세포에 도입될 수 있다.합성 플라스미드는 인터넷을 [3][4][5]통해 조달할 수 있습니다.
플라스미드는 적절한 숙주 내에서 자율적으로 복제할 수 있는 DNA 단위인 복제자로 간주됩니다.그러나 플라스미드는 바이러스와 마찬가지로 일반적으로 [6]생명체로 분류되지 않는다.플라스미드는 주로 [7]결합을 통해 한 박테리아에서 다른 박테리아로 전염된다.유전자 물질의 숙주 간 이동은 수평 유전자 이동의 메커니즘 중 하나이며 플라스미드는 모빌롬의 일부로 간주됩니다.캡시드라고 불리는 보호 단백질 외피로 유전 물질을 감싸는 바이러스와 달리, 플라스미드는 "나체" DNA이고 새로운 숙주로 유전 물질을 감싸는데 필요한 유전자를 암호화하지 않습니다; 하지만, 어떤 종류의 플라스미드는 그들 자신의 전달에 필요한 결합체 "성" 필러스를 암호화합니다.플라스미드의 크기는 1kbp에서 [8]400kbp 이상으로 다양하며, 단일 세포 내의 동일한 플라스미드의 수는 상황에 따라 1에서 수천까지 다양할 수 있습니다.
역사
플라스미드라는 용어는 1952년 미국의 분자생물학자 조슈아 레더버그에 의해 "염색체 외 유전 결정인자"[9]를 지칭하기 위해 도입되었다.이 용어의 초기 사용에는 복제 주기의 적어도 일부 동안 색소 외적으로 존재하는 박테리아 유전 물질이 포함되었지만, 이 기술이 박테리아 바이러스를 포함하기 때문에 플라스미드의 개념은 시간이 지남에 따라 자율적으로 [10]번식하는 유전 요소를 구성하도록 수정되었습니다.이후 1968년 플라스미드라는 용어를 염색체 외 유전원소를 [11]지칭하는 용어로 채택하기로 결정했고, 바이러스와 구별하기 위해 염색체 밖에 독점적으로 또는 주로 존재하며 [10]자율적으로 복제할 수 있는 유전원소로 정의를 좁혔다.
속성 및 특성
플라스미드가 세포 내에서 독립적으로 복제되기 위해서는, 그들은 복제의 기원으로 작용할 수 있는 DNA의 범위를 가지고 있어야 한다.자기복제 유닛(이 경우 플라스미드)은 레플리콘이라고 불립니다.전형적인 박테리아 리플리콘은 플라스미드 특이 복제 개시 단백질(Rep)의 유전자, 반복 단위인 반복 단위, DnaA 박스 및 인접한 AT 리치 [10]영역과 같은 많은 요소로 구성될 수 있다.더 작은 플라스미드는 그들 자신을 복제하기 위해 숙주 복제 효소를 사용하는 반면, 더 큰 플라스미드는 그러한 플라스미드의 복제에 특정한 유전자를 가지고 있을 수 있습니다.몇몇 종류의 플라스미드가 숙주 염색체에 삽입될 수 있고, 이러한 통합 플라스미드는 원핵생물에서 [12]에피솜으로 언급되기도 한다.
플라스미드는 거의 항상 적어도 하나의 유전자를 가지고 있다.플라스미드에 의해 운반되는 많은 유전자들은 숙주 세포에게 유익하다: 예를 들어 숙주 세포가 그렇지 않으면 치명적이거나 성장에 제약적인 환경에서 생존할 수 있도록 한다.이러한 유전자 중 일부는 항생제 내성 또는 중금속에 대한 내성을 위한 특성을 코드하는 반면, 다른 것들은 박테리아가 숙주를 식민지로 만들고 그것의 방어를 극복하도록 하거나 박테리아가 저항제 또는 t를 분해하는 능력을 포함하여 특정 영양소를 이용할 수 있게 하는 특정한 대사 기능을 가질 수 있습니다.독성 유기 [10]화합물플라스미드는 또한 박테리아에게 질소를 고정시키는 능력을 제공할 수 있다.그러나 일부 플라스미드는 숙주 세포의 표현형에 대해 관찰할 수 없는 영향을 미치거나 숙주 세포에 대한 그 이점을 결정할 수 없으며, 이러한 플라스미드를 [13]암호 플라스미드라고 한다.
자연적으로 발생하는 플라스미드는 물리적 특성이 매우 다양하다.크기는 1킬로베이스 쌍(kbp) 미만의 매우 작은 미니 플라스미드에서 여러 메가베이스 쌍(Mbp)의 매우 큰 메가 플라스미드에 이르기까지 다양합니다.위쪽 끝에는 메가플라스미드와 미니크롬의 차이가 거의 없다.플라스미드는 일반적으로 원형이지만 선형 플라스미드의 예도 알려져 있습니다.이러한 선형 플라스미드는 [10]끝부분을 복제하기 위한 특수 메커니즘을 필요로 합니다.
플라스미드는 1에서 수백까지 다양한 수로 개별 세포에 존재할 수 있습니다.단일 세포에서 발견될 수 있는 플라스미드의 정상적인 복사본 수는 플라스미드 복사본 번호라고 불리며, 복제 개시가 어떻게 조절되고 분자의 크기에 의해 결정됩니다.플라스미드가 클수록 복사 번호가 [12]더 낮은 경향이 있습니다.각 박테리아에 1개 또는 몇 개의 복사본으로만 존재하는 저복사 수 플라스미드는 세포 분열 시 분리 박테리아 중 하나에서 손실될 위험이 있다.이러한 단일 복사 플라스미드는 두 딸 세포에 복사본을 적극적으로 배포하는 시스템을 가지고 있습니다.parABS 시스템 및 parMRC 시스템을 포함하는 이러한 시스템은 종종 플라스미드의 파티션 시스템 또는 파티션 함수라고 불립니다.
분류 및 유형
플라스미드는 여러 가지 방법으로 분류될 수 있다.플라스미드는 크게 켤레플라스미드와 비결합플라스미드로 나눌 수 있다.켤레 플라스미드는 다른 [12]세포들 사이의 성적 켤레를 촉진하는 일련의 전달 유전자를 포함한다.복합 결합 과정에서 플라스미드는 전달 유전자의 일부에 의해 코드된 성필리를 통해 한 박테리아에서 다른 박테리아로 전달될 수 있다(그림 [14]참조).비결합 플라스미드는 결합을 개시할 수 없으므로 결합 플라스미드의 도움에 의해서만 전이될 수 있다.플라스미드의 중간 클래스는 이동 가능하며 전이에 필요한 유전자의 서브셋만을 포함한다.그들은 결합 플라스미드를 기생시킬 수 있고, 오직 존재하는 곳에서만 높은 빈도로 전이될 수 있다.
플라스미드는 또한 비호환성 그룹으로 분류될 수 있다.미생물은 다른 종류의 플라스미드를 숨길 수 있지만, 다른 플라스미드는 호환성이 있을 경우에만 단일 박테리아 세포에 존재할 수 있습니다.만약 두 개의 플라스미드가 양립할 수 없다면, 한쪽은 세포에서 빠르게 손실될 것이다.따라서 서로 공존할 수 있는지 여부에 따라 서로 다른 플라스미드가 서로 다른 비호환성 그룹에 할당될 수 있습니다.호환되지 않는 플라스미드(같은 비호환성 그룹에 속함)는 일반적으로 동일한 복제 또는 파티션 메커니즘을 공유하므로 단일 [15][16]셀에 함께 보관할 수 없습니다.
플라스미드를 분류하는 또 다른 방법은 기능에 의한 것이다.5가지 주요 클래스가 있습니다.
- 트라 유전자를 포함한 다산성 F-플라스미드.그들은 결합이 가능하고 성욕의 발현을 초래한다.
- 항생제 또는 독에 대한 내성을 제공하는 유전자를 포함하는 내성(R) 플라스미드.플라스미드의 성질이 이해되기 전에는 역사적으로 R-요소로 알려져 있었다.
- 콜 플라스미드는 다른 박테리아를 죽일 수 있는 단백질인 박테리오신을 코드하는 유전자를 포함하고 있다.
- 분해 플라스미드: 톨루엔 및 살리실산과 같은 특이한 물질의 소화를 가능하게 합니다.
- 세균을 병원체로 바꾸는 독성 플라스미드.아그로박테륨 투메파시엔스의 Ti 플라스미드
플라스미드는 이러한 기능성 그룹 중 하나 이상에 속할 수 있습니다.
RNA플라스미드
대부분의 플라스미드는 이중사슬 DNA 분자이지만, 일부는 단일사슬 DNA 또는 주로 이중사슬 RNA로 구성됩니다. RNA 플라스미드는 비감염성 외염색체 선형 RNA 복제체로, 곰팡이와 다양한 식물에서 조류에서 육지 식물에 이르기까지 발견되어 왔습니다.그러나 많은 경우 RNA 플라스미드와 RNA 바이러스 및 다른 전염성 [17]RNA를 명확하게 구별하는 것은 어렵거나 불가능할 수 있습니다.
벡터
인공적으로 구축된 플라스미드를 유전공학에서 벡터로서 사용할 수 있다.이 플라스미드는 유전학 및 생명공학 연구실에서 중요한 도구로서 기능하며, 그곳에서 그들은 일반적으로 복제하고 증폭하거나 [18]특정 유전자를 발현하는 데 사용됩니다.다양한 플라스미드가 이러한 용도로 시판되고 있다.복제될 유전자는 일반적으로 플라스미드에 삽입되며, 플라스미드는 일반적으로 그들의 사용을 위한 많은 특징을 가지고 있다.여기에는 특정 항생제에 대한 내성을 부여하는 유전자(암피실린은 박테리아 균주에 가장 자주 사용됨), 박테리아 세포가 플라스미드 DNA를 복제할 수 있도록 하는 복제의 기원, 그리고 복제하기에 적합한 장소(다중 복제 사이트라고 함)가 포함됩니다.
DNA의 구조적 불안정성은 유전 물질의 예기치 않은 재배열, 손실 또는 이득을 초래하는 일련의 자발적 사건으로 정의될 수 있다.이러한 이벤트는 이동 요소의 이동 또는 비표준(비B) 구조와 같은 불안정한 요소의 존재에 의해 자주 발생합니다.세균의 백본과 관련된 부속 영역은 광범위한 구조적 불안정 현상에 관여할 수 있다.유전적 불안정성의 잘 알려진 촉매는 직접, 반전 및 탠덤 반복을 포함하며, 이는 상업적으로 이용 가능한 많은 복제 및 발현 [19]벡터에서 두드러지는 것으로 알려져 있다.삽입 배열은 또한 결실과 [20]재배열, 활성화, 하향 조절 또는 인접 유전자 발현 불활성화를 초래함으로써 플라스미드 기능과 산출량에 심각한 영향을 미칠 수 있다.따라서 외부 비부호화 백본 시퀀스의 감소 또는 완전한 제거는 그러한 사건이 발생하는 경향을 현저하게 감소시키고 결과적으로 플라스미드의 [21][22]전반적인 재조합 잠재성을 감소시킬 것이다.
클로닝
플라스미드는 가장 일반적으로 사용되는 박테리아 복제 [23]벡터이다.이러한 클로닝 벡터는 DNA 조각이 삽입될 수 있는 사이트를 포함합니다. 예를 들어 다중 클로닝 사이트나 DNA 조각이 결합될 수 있는 몇 개의 일반적으로 사용되는 제한 사이트를 가진 폴리링커입니다.관심 있는 유전자를 삽입한 후, 플라스미드는 변형이라고 불리는 과정을 통해 박테리아에 도입된다.이러한 플라스미드는 특정 항생제를 포함한 선택적 성장 배지에서 박테리아에 생존하고 증식할 수 있는 능력을 부여하는 선택 가능한 마커, 보통 항생제 내성 유전자를 포함합니다.형질전환 후의 세포는 선택적 배지에 노출되어 플라스미드를 포함한 세포만이 생존할 수 있다.이와 같이 항생제는 플라스미드 DNA를 포함한 세균만을 선택하는 필터 역할을 한다.이 벡터는 또한 복제된 삽입물로 플라스미드를 쉽게 선택할 수 있도록 다른 마커 유전자 또는 리포터 유전자를 포함할 수 있다.플라스미드를 포함한 세균은 대량으로 배양하여 수확할 수 있으며, 그 후 다양한 플라스미드 제제의 방법을 사용하여 해당 플라스미드를 분리할 수 있다.
플라스미드 클로닝 벡터는 일반적으로 최대 15kbp의 [24]DNA 단편 복제에 사용됩니다.더 긴 길이의 DNA를 복제하기 위해 용원 유전자를 제거한 람다 파지, 우주체, 박테리아 인공 염색체 또는 효모 인공 염색체를 사용한다.
단백질 생산
플라스미드의 또 다른 주요 용도는 많은 양의 단백질을 만드는 것이다.이 경우 연구진은 관심 유전자를 가진 플라스미드를 포함한 박테리아를 배양한다.박테리아가 항생제 내성을 부여하기 위해 단백질을 생성하는 것처럼, 삽입된 유전자로부터 많은 양의 단백질을 생성하도록 유도될 수도 있다.이것은 예를 들어 인슐린과 같이 유전자가 코드하는 단백질을 대량생산하는 싸고 쉬운 방법입니다.
유전자 치료
플라스미드는 또한 세포에 부족한 단백질을 발현시키기 위해 유전자 치료의 잠재적 치료법으로서 유전자 전달에 사용될 수 있다.유전자 치료의 일부 형태는 인간 게놈 내의 미리 선택된 염색체 표적 부위에 치료용 유전자를 삽입해야 한다.플라스미드 벡터는 이러한 목적을 위해 사용될 수 있는 많은 접근법 중 하나입니다.아연 핑거뉴클라아제(ZFNs)는 DNA 게놈에 부위 특이적 이중 가닥을 파괴하고 상동 재조합을 일으키는 방법을 제공한다.ZFN을 코드하는 플라스미드는 세포 손상, 암을 유발하는 돌연변이 또는 면역 반응을 [25]피하기 위해 치료 유전자를 특정 부위에 전달하는 데 도움을 줄 수 있다.
질병 모델
플라스미드는 역사적으로 쥐의 배아줄기세포를 유전적으로 조작하여 쥐의 유전병 모델을 만드는데 사용되었다.플라스미드 기반 기술의 제한적인 효율성으로 인해 보다 정확한 인간 세포 모델을 만들 때 사용이 금지되었다.그러나 아데노 관련 바이러스 재조합 기술과 아연 손가락 핵산 분해효소의 개발로 새로운 세대의 등원성 인간 질병 모델이 탄생했다.
에피솜
에피솜이라는 용어는 1958년 프랑수아 제이콥과 엘리 울만이 자율적으로 복제되거나 [26][27]염색체에 통합될 수 있는 염색체 외 유전 물질을 지칭하기 위해 도입했다.그러나 용어가 도입된 이후 플라스미드가 염색체 외 DNA를 자율적으로 복제하는 데 선호되는 용어가 되면서 용도가 바뀌었다.1968년 런던에서 열린 심포지엄에서 일부 참석자들은 episome이라는 용어를 포기해야 한다고 제안했지만,[28][29] 다른 참석자들은 의미가 바뀌면서 이 용어를 계속 사용했다.
오늘날, 몇몇 작가들은 염색체에 통합될 수 있는 플라스미드를 언급하기 위해 원핵생물 맥락에서 에피솜을 사용한다.통합 플라스미드는 여러 세대를 통해 세포 내에서 복제되고 안정적으로 유지될 수 있지만, 어떤 단계에서는 독립적인 플라스미드 [30]분자로 존재할 것이다.진핵생물의 맥락에서 에피솜이라는 용어는 [31][32]핵에서 복제될 수 있는 비집적 염색체 외 폐쇄 원형 DNA 분자를 의미하기 위해 사용된다.바이러스는 헤르페스바이러스, 아데노바이러스, 폴리오마바이러스와 같이 이것의 가장 흔한 예이지만, 어떤 것들은 플라스미드이다.다른 예로는 인공 유전자 증폭 중 또는 병리학적 과정(예: 암세포 변형)에서 발생할 수 있는 2분 염색체 조각과 같은 비정상적인 염색체 조각이 있다.진핵생물의 에피솜은 DNA가 안정적으로 유지되고 숙주 세포와 복제된다는 점에서 원핵생물의 플라스미드와 유사하게 행동한다.세포질 바이러스 에피솜도 발생할 수 있다.헤르페스 바이러스와 같은 일부 에피솜은 박테리오파지(세균 파지 바이러스)와 유사하게 롤링 서클 메커니즘으로 복제됩니다.그 외는 쌍방향 복제 메커니즘(Theta 타입 플라스미드)을 통해 복제됩니다.두 경우 모두 에피솜은 숙주 세포 염색체로부터 물리적으로 분리된 상태로 남아 있다.Epstein-Barr 바이러스와 Kaposi의 육종 관련 헤르페스 바이러스를 포함한 몇몇 암 바이러스는 암세포의 증식을 촉진하는 종양유전자를 발현하는 암세포에서 잠복하고 염색체적으로 구별되는 에피솜으로 유지된다.암의 경우 세포가 분열할 때 이 에피솜들은 숙주 염색체와 함께 수동적으로 복제된다.이 바이러스 에피솜들이 여러 개의 바이러스 입자를 생성하기 위해 용해 복제를 시작할 때, 그들은 일반적으로 숙주 세포를 죽이는 세포 고유의 면역 방어 메커니즘을 활성화한다.
플라스미드 유지 보수
일부 플라스미드 또는 미생물 숙주는 [33]대장균의 플라스미드 R1의 hok/sok(숙주 살처분/살처분 억제제) 시스템과 같은 중독계 또는 분리 후 살처분계(PSK)를 포함한다.이 변종은 장수독과 단수명 해독제를 모두 생산한다.여러[34] 유형의 플라스미드 중독 시스템(독신/항독소, 대사 기반, ORT 시스템)이 문헌에 기술되어 있으며 생명공학(발효) 또는 생물의학(백신 치료) 응용 분야에 사용되었다.플라스미드의 복사본을 보유한 딸세포는 살아남는 반면 플라스미드를 물려받지 못한 딸세포는 부모세포의 독이 남아 있어 죽거나 성장률이 떨어진다.마지막으로 전체적인 생산성을 향상시킬 수 있습니다.
반면 pUC18, pBR322 및 유도 벡터 등 생명공학에 사용되는 플라스미드는 독소항독소 중독 시스템을 거의 포함하고 있지 않기 때문에 플라스미드 손실을 피하기 위해 항생제 압력을 가할 필요가 있다.
효모플라스미드
효모는 자연적으로 다양한 플라스미드를 함유하고 있다.그 중 주목할 만한 것은 2μm 플라스미드(효모의 유전자 공학에 종종 사용되는 작은 원형 플라스미드)와 킬러 표현형의 [35]원인이 되는 Kluyveromyces 락티스의 선형 pGKL 플라스미드이다.
다른 유형의 플라스미드는 종종 다음과 같은 효모 클로닝 벡터와 관련이 있습니다.
- 효모 통합 플라스미드(YIP)는 생존과 복제를 위해 숙주 염색체로의 통합에 의존하는 효모 벡터이며, 보통 단독 유전자의 기능성을 연구하거나 유전자가 독성이 있을 때 사용된다.또한 피리미딘 뉴클레오티드(T, C)의 생합성과 관련된 효소를 코드하는 유전자 URA3와 연결된다.
- 효모복제플라스미드(YRP)는 복제의 기원을 포함한 염색체 DNA의 염기서열을 운반한다.이 플라스미드는 싹이 트는 동안 없어질 수 있기 때문에 안정성이 떨어진다.
플라스미드 DNA 추출
플라스미드는 게놈의 나머지 부분으로부터 쉽게 정제될 수 있기 때문에 특정 배열을 정화하는 데 종종 사용된다.그들의 벡터로서의 사용과 분자 복제를 위해 플라스미드는 종종 분리될 필요가 있다.
플라스미드 DNA를 박테리아로부터 분리하는 방법은 미니프리프에서 맥시프리프 또는 벌크프리프까지 [18]여러 가지가 있습니다.전자는 몇 가지 박테리아 복제품 중 하나에서 플라스미드가 정확한지 빠르게 알아내기 위해 사용될 수 있다.소량의 불순 플라스미드 DNA는 제한 소화 및 일부 복제 기술에 의해 분석하기에 충분합니다.
후자는 맥시프리프를 실행할 수 있는 훨씬 더 많은 양의 세균 현탁액을 배양한다.본질적으로, 이것은 추가적인 정제 후에 스케일업된 미니프리프입니다.이것은 매우 순수한 플라스미드 DNA의 비교적 많은 양(수백 마이크로그램)을 낳는다.
다양한 스케일, 순도 및 자동화 수준에서 플라스미드 추출을 수행하기 위해 많은 시판 키트가 개발되었습니다.
구성
플라스미드 DNA는 전기영동 중에 겔에서 (특정 크기만큼) 다른 속도로 실행되는 5가지 구성 중 하나로 나타날 수 있습니다.다음으로 전기영동도(특정 인가전압의 속도)를 가장 느린 것부터 가장 빠른 것 순으로 나타냅니다.
- 트인 원형 DNA에 가닥이 하나 잘려 있어요
- 완화된 원형 DNA는 양쪽 가닥을 절단하지 않은 상태에서 완전히 온전하지만 효소적으로 완화된 상태(슈퍼코일 제거)입니다.이것은, 꼬인 연장 코드를 풀어서 느슨하게 한 후, 그 자체에 꽂는 것으로 모델링할 수 있습니다.
- 선형 DNA는 양쪽 가닥이 절단되었거나 DNA가 생체 내에서 선형이었기 때문에 자유단을 가진다.이 모델에는 전기 연장 코드가 꽂혀 있지 않습니다.
- 초코일형(또는 공유 닫힌 원형) DNA는 양쪽 가닥을 절단하지 않고 일체적으로 꼬여 완전히 온전하며, 콤팩트한 형태를 형성한다.연장 코드를 꼬아 본체에 꽂으면 모델을 만들 수 있습니다.
- 초코일 변성 DNA는 초코일 DNA와 비슷하지만, 결합되지 않은 영역이 있어 약간 덜 콤팩트하다. 이것은 플라스미드 준비 중에 과도한 알칼리성으로 인해 발생할 수 있다.
작은 선형 조각의 이동 속도는 낮은 전압에서 인가되는 전압에 정비례합니다.더 높은 전압에서는 더 큰 조각이 지속적으로 증가하지만 다른 속도로 이동합니다.따라서 겔의 분해능은 전압 증가에 따라 감소한다.
소정의 저전압에서 작은 선형 DNA 조각의 이동 속도는 그 길이의 함수이다.큰 선형 fragment(20KB 내외)는 길이에 관계없이 일정한 고정 환율로 이행합니다.이는 분자가 겔 매트릭스를 통해 선단을 따라 '재호흡'하기 때문입니다.제한 다이제스트는 정제 플라스미드를 분석하기 위해 자주 사용된다.이 효소들은 특정 짧은 염기서열에서 DNA를 분해한다.생성된 선형 조각은 겔 전기영동 후에 '밴드'를 형성합니다.겔에서 띠를 잘라내고 겔을 녹여 DNA 조각을 방출함으로써 특정 조각들을 정화할 수 있다.
그것의 엄격한 구조 때문에, 초코일 DNA는 선형 또는 개방된 원형의 DNA보다 겔을 통해 더 빨리 이동한다.
생물정보학 및 설계용 소프트웨어
분자생물학에서의 기법으로 플라스미드를 사용하는 것은 생물정보학 소프트웨어에 의해 지원된다.이 프로그램들은 플라스미드 벡터의 DNA 시퀀스를 기록하고, 제한 효소의 절단 부위를 예측하고, 조작을 계획하는 데 도움을 준다.플라스미드 맵을 처리하는 소프트웨어 패키지의 예로는 ApE, Clone Manager, Gene Construction Kit, Genious, Genome Compiler, LabGenius, Lasergene, MacVector, pDraw32, Serial Cloner, VectorFriends, Vector NTI, WebDSV 등이 있습니다.이 소프트웨어 조각들은 젖은 [36]실험을 하기 전에 실리코에서 전체 실험을 수행하는 데 도움이 됩니다.
플라스미드 컬렉션
수년간 많은 플라스미드가 생성되었으며 연구자들은 비영리 조직 Addgene 및 BCCM/LMBP와 같은 플라스미드 데이터베이스에 플라스미드를 배포했다.이러한 데이터베이스에서는 플라스미드를 찾아 연구를 의뢰할 수 있습니다.연구자들은 또한 종종 특정 플라스미드의 시퀀스를 검색할 수 있는 NCBI 데이터베이스에 플라스미드 시퀀스를 업로드한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Esser K, Kück U, Lang-Hinrichs C, Lemke P, Osiewacz HD, Stahl U, Tudzynski P (1986). Plasmids of Eukaryotes: fundamentals and Applications. Berlin: Springer-Verlag. ISBN 978-3-540-15798-4.
- ^ Wickner RB, Hinnebusch A, Lambowitz AM, Gunsalus IC, Hollaender A, eds. (1987). "Mitochondrial and Chloroplast Plasmids". Extrachromosomal Elements in Lower Eukaryotes. Boston, MA: Springer US. pp. 81–146. ISBN 978-1-4684-5251-8.
- ^ "GenBrick™ Building Blocks for Synthetic Biology".
- ^ "Custom gene synthesis".
- ^ "Invitrogen GeneArt Gene Synthesis".
- ^ Sinkovics J, Horvath J, Horak A (1998). "The origin and evolution of viruses (a review)". Acta Microbiologica et Immunologica Hungarica. 45 (3–4): 349–90. PMID 9873943.
- ^ Smillie C, Garcillán-Barcia MP, Francia MV, Rocha EP, de la Cruz F (September 2010). "Mobility of plasmids". Microbiology and Molecular Biology Reviews. 74 (3): 434–52. doi:10.1128/MMBR.00020-10. PMC 2937521. PMID 20805406.
- ^ Thomas CM, Summers D (2008). "Bacterial Plasmids". Encyclopedia of Life Sciences. doi:10.1002/9780470015902.a0000468.pub2. ISBN 978-0-470-01617-6.
- ^ Lederberg J (October 1952). "Cell genetics and hereditary symbiosis". Physiological Reviews. 32 (4): 403–30. CiteSeerX 10.1.1.458.985. doi:10.1152/physrev.1952.32.4.403. PMID 13003535.
- ^ a b c d e Hayes F (2003). "Chapter 1 – The Function and Organization of Plasmids". In Casali N, Presto A (eds.). E. Coli Plasmid Vectors: Methods and Applications. Methods in Molecular Biology. Vol. 235. Humana Press. pp. 1–5. ISBN 978-1-58829-151-6.
- ^ Falkow S. "Microbial Genomics: Standing on the Shoulders of Giants". Microbiology Society.
- ^ a b c Brown TA (2010). "Chapter 2 – Vectors for Gene Cloning: Plasmids and Bacteriophages". Gene Cloning and DNA Analysis: An Introduction (6th ed.). Wiley-Blackwell. ISBN 978-1405181730.
- ^ Summers DK (1996). "Chapter 1 – The Function and Organization of Plasmids". The Biology of Plasmids (First ed.). Osney, Oxford OX: Wiley-Blackwell. pp. 21–22. ISBN 978-0-632-03436-9.
- ^ Clark DP, Pazdernik NJ (2012). Molecular Biology (2nd ed.). Academic Cell. p. 795. ISBN 978-0123785947.
- ^ Radnedge L, Richards H (January 1999). "Chapter 2: The Development of Plasmid Vectors.". In Smith MC, Sockett RE (eds.). Genetic Methods for Diverse Prokaryotes. Methods in Microbiology. Vol. 29. Academic Press. pp. 51-96 (75-77). ISBN 978-0-12-652340-9.
- ^ "Plasmids 101: Origin of Replication". addgene.org.
- ^ Brown GG, Finnegan PM (January 1989). "RNA plasmids". International Review of Cytology. 117: 1–56. doi:10.1016/s0074-7696(08)61333-9. ISBN 978-0-12-364517-3. PMID 2684889.
- ^ a b Russell DW, Sambrook J (2001). Molecular cloning: a laboratory manual. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory.
- ^ Oliveira PH, Prather KJ, Prazeres DM, Monteiro GA (August 2010). "Analysis of DNA repeats in bacterial plasmids reveals the potential for recurrent instability events". Applied Microbiology and Biotechnology. 87 (6): 2157–67. doi:10.1007/s00253-010-2671-7. PMID 20496146. S2CID 19780633.
- ^ Gonçalves GA, Oliveira PH, Gomes AG, Prather KL, Lewis LA, Prazeres DM, Monteiro GA (August 2014). "Evidence that the insertion events of IS2 transposition are biased towards abrupt compositional shifts in target DNA and modulated by a diverse set of culture parameters" (PDF). Applied Microbiology and Biotechnology. 98 (15): 6609–19. doi:10.1007/s00253-014-5695-6. hdl:1721.1/104375. PMID 24769900. S2CID 9826684.
- ^ Oliveira PH, Mairhofer J (September 2013). "Marker-free plasmids for biotechnological applications - implications and perspectives". Trends in Biotechnology. 31 (9): 539–47. doi:10.1016/j.tibtech.2013.06.001. PMID 23830144.
- ^ Oliveira PH, Prather KJ, Prazeres DM, Monteiro GA (September 2009). "Structural instability of plasmid biopharmaceuticals: challenges and implications". Trends in Biotechnology. 27 (9): 503–11. doi:10.1016/j.tibtech.2009.06.004. PMID 19656584.
- ^ Geoghegan T (2002). "Molecular Applications". In Streips UN, Yasbin RE (eds.). Modern Microbial Genetics (2nd ed.). Wiley-Blackwell. p. 248. ISBN 978-0471386650.
- ^ Preston A (2003). "Chapter 2 – Choosing a Cloning Vector". In Casali N, Preston A (eds.). E. Coli Plasmid Vectors: Methods and Applications. Methods in Molecular Biology. Vol. 235. Humana Press. pp. 19–26. ISBN 978-1-58829-151-6.
- ^ Kandavelou K, Chandrasegaran S (2008). "Plasmids for Gene Therapy". Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- ^ Morange M (December 2009). "What history tells us XIX. The notion of the episome" (PDF). Journal of Biosciences. 34 (6): 845–48. doi:10.1007/s12038-009-0098-z. PMID 20093737. S2CID 11367145.
- ^ Jacob F, Wollman EL (1958), "Les épisomes, elements génétiques ajoutés", Comptes Rendus de l'Académie des Sciences de Paris, 247 (1): 154–56, PMID 13561654
- ^ Hayes W (1969). "What are episomes and plasmids?". In Wolstenholme GE, O'Connor M (eds.). Bacterial Episomes and Plasmids. CIBA Foundation Symposium. pp. 4–8. ISBN 978-0700014057.
- ^ Wolstenholme GE, O'Connor M, eds. (1969). Bacterial Episomes and Plasmids. CIBA Foundation Symposium. pp. 244–45. ISBN 978-0700014057.
- ^ Brown TA (2011). Introduction to Genetics: A Molecular Approach. Garland Science. p. 238. ISBN 978-0815365099.
- ^ Van Craenenbroeck K, Vanhoenacker P, Haegeman G (September 2000). "Episomal vectors for gene expression in mammalian cells". European Journal of Biochemistry. 267 (18): 5665–78. doi:10.1046/j.1432-1327.2000.01645.x. PMID 10971576.
- ^ Colosimo A, Goncz KK, Holmes AR, Kunzelmann K, Novelli G, Malone RW, Bennett MJ, Gruenert DC (August 2000). "Transfer and expression of foreign genes in mammalian cells" (PDF). BioTechniques. 29 (2): 314–18, 320–22, 324 passim. doi:10.2144/00292rv01. PMID 10948433. Archived from the original (PDF) on 24 July 2011.
- ^ Gerdes K, Rasmussen PB, Molin S (May 1986). "Unique type of plasmid maintenance function: postsegregational killing of plasmid-free cells". Proceedings of the National Academy of Sciences of the United States of America. 83 (10): 3116–20. Bibcode:1986PNAS...83.3116G. doi:10.1073/pnas.83.10.3116. PMC 323463. PMID 3517851.
- ^ Kroll J, Klinter S, Schneider C, Voss I, Steinbüchel A (November 2010). "Plasmid addiction systems: perspectives and applications in biotechnology". Microbial Biotechnology. 3 (6): 634–57. doi:10.1111/j.1751-7915.2010.00170.x. PMC 3815339. PMID 21255361.
- ^ Gunge N, Murata K, Sakaguchi K (July 1982). "Transformation of Saccharomyces cerevisiae with linear DNA killer plasmids from Kluyveromyces lactis". Journal of Bacteriology. 151 (1): 462–64. doi:10.1128/JB.151.1.462-464.1982. PMC 220260. PMID 7045080.
- ^ "Vector NTI feedback video". The DNA Lab.
추가 정보
일반 작업
- Klein DW, Prescott LM, Harley J (1999). Microbiology. Boston: WCB/McGraw-Hill.
- Moat AG, Foster JW, Spector MP (2002). Microbial Physiology. Wiley-Liss. ISBN 978-0-471-39483-9.
- Smith CU (2002). "Chapter 5: Manipulating Biomolecules". Elements of Molecular Neurobiology (3rd ed.). Chichester, West Sussex, England: Wiley. pp. 101–11. ISBN 978-0-470-85717-5.
에피솜
- Piechaczek C, Fetzer C, Baiker A, Bode J, Lipps HJ (January 1999). "A vector based on the SV40 origin of replication and chromosomal S/MARs replicates episomally in CHO cells". Nucleic Acids Research. 27 (2): 426–28. doi:10.1093/nar/27.2.426. PMC 148196. PMID 9862961.
- Bode J, Fetzer CP, Nehlsen K, Scinteie M, Hinrichsen BH, Baiker A, et al. (January 2001). "The Hitchhiking principle: Optimizing episomal vectors for the use in gene therapy and biotechnology" (PDF). Gene Therapy and Molecular Biology. 6: 33–46. Archived from the original (PDF) on 30 May 2009.
- Nehlsen K, Broll S, Bode J (2006). "Replicating minicircles: Generation of nonviral episomes for the efficient modification of dividing cells" (PDF). Gene Ther Mol Biol. 10: 233–44. Archived from the original (PDF) on 30 May 2009.
- Ehrhardt A, Haase R, Schepers A, Deutsch MJ, Lipps HJ, Baiker A (June 2008). "Episomal vectors for gene therapy". Current Gene Therapy. 8 (3): 147–61. doi:10.2174/156652308784746440. PMID 18537590. Archived from the original on 26 September 2011.
- Argyros O, Wong SP, Niceta M, Waddington SN, Howe SJ, Coutelle C, Miller AD, Harbottle RP (December 2008). "Persistent episomal transgene expression in liver following delivery of a scaffold/matrix attachment region containing non-viral vector". Gene Therapy. 15 (24): 1593–605. doi:10.1038/gt.2008.113. PMID 18633447.
- Wong SP, Argyros O, Coutelle C, Harbottle RP (August 2009). "Strategies for the episomal modification of cells". Current Opinion in Molecular Therapeutics. 11 (4): 433–41. PMID 19649988. Archived from the original on 17 September 2011.
- Haase R, Argyros O, Wong SP, Harbottle RP, Lipps HJ, Ogris M, Magnusson T, Vizoso Pinto MG, Haas J, Baiker A (March 2010). "pEPito: a significantly improved non-viral episomal expression vector for mammalian cells". BMC Biotechnology. 10: 20. doi:10.1186/1472-6750-10-20. PMC 2847955. PMID 20230618.