구조생물학
Structural biology| 시리즈의 일부 |
| 생화학 |
|---|
 |
구조생물학은 생물학적 고분자의 분자구조와 관련된 분자생물학, 생화학, 그리고 생물물리학의 한 분야이다.기능에 [1]영향을 미칩니다.고분자가 세포의 대부분의 기능을 수행하며, 특정한 3차원 형상에 감겨야만 이러한 기능을 수행할 수 있기 때문에 이 주제는 생물학자들에게 매우 흥미롭다.분자의 "테리어 구조"인 이 구조는 각 분자의 기본 구성, 즉 "1차 구조"에 복잡한 방식으로 의존합니다.
지난 몇 년 동안 매우 정확한 물리적 분자 모델이 생물학적 [2]구조에 대한 실리코 연구를 보완하는 것이 가능해졌다.이러한 모델의 예는 단백질 데이터 뱅크에서 찾을 수 있습니다.
분자 역학 시뮬레이션과 같은 계산 기술은 단백질 구조, 구조 및 기능을 [3]확장하고 연구하기 위해 경험적 구조 결정 전략과 함께 사용될 수 있습니다.

역사
1912년 Max Von Laue는 회절 [4]패턴을 생성하는 결정화된 황산동 X선을 조사했습니다.이러한 실험은 X-레이 결정학의 개발로 이어졌고, 생물학적 [2]구조를 탐구하는 데 있어 X-레이 결정학의 사용으로 이어졌다.1951년, 로잘린드 프랭클린과 모리스 윌킨스는 디옥시리보핵산의 첫 이미지를 포착하기 위해 X선 회절 패턴을 사용했다.프랜시스 크릭과 제임스 왓슨은 1953년 이 기술을 이용해 DNA의 이중 나선 구조를 모형화했고 1962년 [citation needed]윌킨스와 함께 노벨 의학상을 받았다.
1962년 노벨 [5]화학상을 수상한 테오도르 스베드버그는 펩신 결정을 X선 회절에 사용하기 위해 결정화한 최초의 단백질이었다.최초의 3차 단백질 구조인 미오글로빈은 1958년 존 켄드루에 [6]의해 발표되었다.이 기간 동안 단백질 구조의 모델링은 발사 목재 또는 와이어 [7]모델을 사용하여 수행되었습니다.1970년대 [8]후반에 CCP4와 같은 모델링 소프트웨어가 발명되면서, 모델링은 이제 컴퓨터의 도움을 받아 이루어집니다.이 분야의 최근 개발에는 X선 자유 전자 레이저의 발생이 포함되어 이전에 숨겨진 구조를[9] 분석하고 합성 생물학을 [10]지원하는 구조 생물학을 사용할 수 있습니다.
1930년대 후반과 1940년대 초, 이시도르 라비, 펠릭스 블로흐, 에드워드 밀스 퍼셀이 수행한 작업의 조합은 핵자기공명(NMR)의 개발을 이끌었다.현재 고체 NMR은 단백질의 구조와 동적 성질을 결정하기 위해 구조생물학 분야에서 널리 사용되고 있다.[11]1990년 리처드 헨더슨은 극저온 전자 현미경법(cryo-EM)[12]을 사용하여 박테리올호돕신의 3차원 고해상도 이미지를 최초로 제작했습니다.
최근에는 생물학적 구조를 모델링하고 연구하기 위한 계산 방법이 개발되고 있다.예를 들어, 분자역학(MD)은 일반적으로 생물학적 분자의 동적 움직임을 분석하는데 사용된다.1975년 MD를 이용한 생물학적 접힘 과정의 첫 시뮬레이션이 네이처에 [13]발표되었습니다.최근, 단백질 구조 예측은 [14]알파폴드라고 불리는 새로운 기계 학습 방법에 의해 상당히 개선되었다.어떤 사람들은 컴퓨터 접근법이 구조 생물학 [15]연구 분야를 주도하기 시작했다고 주장한다.
기술
생체분자는 너무 작아서 아무리 첨단 광현미경으로도 자세히 볼 수 없다.구조생물학자들이 그들의 구조를 결정하기 위해 사용하는 방법들은 일반적으로 동시에 많은 수의 동일한 분자에 대한 측정을 포함한다.이러한 방법에는 다음이 포함됩니다.
- 질량 분석
- 고분자 결정학
- 중성자 회절
- 단백질 분해
- 단백질핵자기공명분광법(NMR)
- 전자상자기공명(EPR)
- 극저온전자현미경(CryoEM)
- 전자결정학 및 미세결정 전자회절
- 다각도 광산란
- 작은 각도 산란
- 초고속 레이저 분광법
- 2차원 적외선 분광법
- 이중편파간섭법과 원형이색성
대부분의 연구자들은 고분자의 "원래 상태"를 연구하기 위해 그것들을 사용한다.그러나 이러한 방법의 변형은 초기 또는 변성된 분자가 그들의 고유 상태를 가정하거나 재평가하는 것을 보기 위해서도 사용된다.단백질 접힘을 참조하십시오.
구조생물학자들이 구조를 이해하기 위해 취하는 세 번째 접근법은 특정 형태를 발생시키는 다양한 배열들 사이에서 패턴을 찾는 생물정보학이다.연구자들은 종종 소수성 분석에 의해 예측된 막 토폴로지를 바탕으로 통합막 단백질의 구조 측면을 추론할 수 있다.단백질 구조 예측을 참조하십시오.
적용들
구조 생물학자들은 인간 질병의 기초가 되는 분자 성분과 메커니즘을 이해하는 데 중요한 기여를 했다.예를 들어, 저온 전자파(Cryo-EM)와 ssNMR은 알츠하이머병, [16]파킨슨병, 제2형 당뇨병과 관련된 아밀로이드 섬유소의 집적을 연구하기 위해 사용되어 왔다.아밀로이드 단백질 외에도,[17] 과학자들은 알츠하이머 환자의 뇌에서 미래에 더 나은 치료법을 개발하는 데 도움을 줄 수 있는 고해상도 타우 필라멘트의 모델을 만들기 위해 저온 전자파를 사용해 왔다.구조 생물학 도구는 또한 병원체와 숙주 사이의 상호작용을 설명하기 위해 사용될 수 있습니다.예를 들어, 구조 생물학 도구들은 바이러스학자들이 HIV가 인간의 면역 반응을 [18]회피하는 것을 어떻게 허용하는지 이해할 수 있게 해주었다.
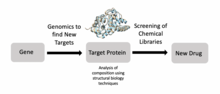
구조생물학 또한 약물 [19]발견의 중요한 구성요소이다.과학자들은 유전체학을 이용하여 대상을 식별하고, 구조생물학을 이용하여 대상을 연구하며, 그러한 목표에 적합한 약을 개발할 수 있다.구체적으로는 리간드-NMR, 질량분석, X선 결정학 등이 약물 발견 과정에서 일반적으로 사용되는 기법이다.예를 들어,[20] 연구자들은 암의 중요한 약물 표적인 Met을 더 잘 이해하기 위해 구조 생물학을 사용해 왔다.HIV [19]표적이 에이즈 환자를 치료하기 위한 유사한 연구가 수행되었습니다.또한 연구자들은 구조 주도의 약물 [19]발견을 이용하여 마이코박테리아 감염을 위한 새로운 항균제를 개발하고 있습니다.
「 」를 참조해 주세요.
- 일차 구조
- 이차 구조
- 3차 구조
- 4차 구조
- 구조 영역
- 구조 모티브
- 단백질 서브유닛
- 분자 모형
- 협동성
- 샤페로닌
- 구조 유전체학
- 입체화학
- 해상도(전자 밀도)
- 단백질 사전 단백질 및 기타 분자의 공동 3D 백과사전.
- 단백질 구조 예측
레퍼런스
- ^ Banaszak LJ (2000). Foundations of Structural Biology. Burlington: Elsevier. ISBN 9780080521848.
- ^ a b Stoddart, Charlotte (1 March 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. Retrieved 25 March 2022.
- ^ Karplus M, McCammon JA (September 2002). "Molecular dynamics simulations of biomolecules". Nature Structural Biology. 9 (9): 646–652. doi:10.1038/nsb0902-646. PMID 12198485. S2CID 590640.
- ^ Curry S (July 2015). "Structural Biology: A Century-long Journey into an Unseen World". Interdisciplinary Science Reviews. 40 (3): 308–328. doi:10.1179/0308018815z.000000000120. PMC 4697198. PMID 26740732.
- ^ Jaskolski M, Dauter Z, Wlodawer A (September 2014). "A brief history of macromolecular crystallography, illustrated by a family tree and its Nobel fruits". The FEBS Journal. 281 (18): 3985–4009. doi:10.1111/febs.12796. PMC 6309182. PMID 24698025.
- ^ Kendrew JC, Bodo G, Dintzis HM, Parrish RG, Wyckoff H, Phillips DC (March 1958). "A three-dimensional model of the myoglobin molecule obtained by x-ray analysis". Nature. 181 (4610): 662–666. Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261. S2CID 4162786.
- ^ Garman EF (March 2014). "Developments in x-ray crystallographic structure determination of biological macromolecules". Science. 343 (6175): 1102–1108. Bibcode:2014Sci...343.1102G. doi:10.1126/science.1247829. PMID 24604194. S2CID 21067016.
- ^ "About CCP4". legacy.ccp4.ac.uk. Retrieved 2021-04-02.
- ^ Waldrop MM (January 2014). "X-ray science: The big guns". Nature. 505 (7485): 604–606. Bibcode:2014Natur.505..604W. doi:10.1038/505604a. PMID 24476872.
- ^ Kiel C, Serrano L (November 2012). "Structural data in synthetic biology approaches for studying general design principles of cellular signaling networks". Structure. 20 (11): 1806–1813. doi:10.1016/j.str.2012.10.002. PMID 23141693.
- ^ Wüthrich K (November 2001). "The way to NMR structures of proteins". Nature Structural Biology. 8 (11): 923–925. doi:10.1038/nsb1101-923. PMID 11685234. S2CID 26153265.
- ^ Henderson R, Baldwin JM, Ceska TA, Zemlin F, Beckmann E, Downing KH (June 1990). "Model for the structure of bacteriorhodopsin based on high-resolution electron cryo-microscopy". Journal of Molecular Biology. 213 (4): 899–929. doi:10.1016/S0022-2836(05)80271-2. PMID 2359127.
- ^ Levitt M, Warshel A (February 1975). "Computer simulation of protein folding". Nature. 253 (5494): 694–698. Bibcode:1975Natur.253..694L. doi:10.1038/253694a0. PMID 1167625. S2CID 4211714.
- ^ Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, et al. (August 2021). "Highly accurate protein structure prediction with AlphaFold". Nature. 596 (7873): 583–589. Bibcode:2021Natur.596..583J. doi:10.1038/s41586-021-03819-2. PMC 8371605. PMID 34265844.
- ^ Nussinov, Ruth; Tsai, Chung-Jung; Shehu, Amarda; Jang, Hyunbum (2019-02-12). "Computational Structural Biology: Successes, Future Directions, and Challenges". Molecules (Basel, Switzerland). 24 (3): E637. doi:10.3390/molecules24030637. ISSN 1420-3049. PMC 6384756. PMID 30759724.
- ^ Iadanza MG, Jackson MP, Hewitt EW, Ranson NA, Radford SE (December 2018). "A new era for understanding amyloid structures and disease". Nature Reviews. Molecular Cell Biology. 19 (12): 755–773. doi:10.1038/s41580-018-0060-8. PMID 30237470. S2CID 52307956.
- ^ Fitzpatrick, Anthony W. P.; Falcon, Benjamin; He, Shaoda; Murzin, Alexey G.; Murshudov, Garib; Garringer, Holly J.; Crowther, R. Anthony; Ghetti, Bernardino; Goedert, Michel; Scheres, Sjors H. W. (2017-07-13). "Cryo-EM structures of tau filaments from Alzheimer's disease". Nature. 547 (7662): 185–190. Bibcode:2017Natur.547..185F. doi:10.1038/nature23002. ISSN 1476-4687. PMC 5552202. PMID 28678775.
- ^ Engelman, Alan; Cherepanov, Peter (2012-03-16). "The structural biology of HIV-1: mechanistic and therapeutic insights". Nature Reviews Microbiology. 10 (4): 279–290. doi:10.1038/nrmicro2747. ISSN 1740-1526.
- ^ a b c Thomas SE, Mendes V, Kim SY, Malhotra S, Ochoa-Montaño B, Blaszczyk M, Blundell TL (August 2017). "Structural Biology and the Design of New Therapeutics: From HIV and Cancer to Mycobacterial Infections: A Paper Dedicated to John Kendrew". Journal of Molecular Biology. John Kendrew’s 100th Anniversary Special Edition. 429 (17): 2677–2693. doi:10.1016/j.jmb.2017.06.014. PMID 28648615.
- ^ Wendt KU, Weiss MS, Cramer P, Heinz DW (February 2008). "Structures and diseases". Nature Structural & Molecular Biology. 15 (2): 117–120. doi:10.1038/nsmb0208-117. PMC 7097323. PMID 18250627.
외부 링크
 Wikimedia Commons 구조생물학 관련 매체
Wikimedia Commons 구조생물학 관련 매체- 네이처: Structure & Molecular Biology 매거진 웹사이트
- 구조 생물학 저널
- 구조생물학 - 생화학, 분자생물학 및 세포생물학 가상도서관
- 유럽의 구조생물학