디코딩 콤플렉스
Decapping complexmRNA decapping complex는 5's cap의 제거를 담당하는 진핵 세포의 단백질 복합체다.[1]디코딩 콤플렉스의 활성효소는 빌로브드 누딕스 계열 효소 Dcp2로, 5'의 캡을 가수분해하여 7m의 GDP와 5'의 단인산 mRNA를 방출한다.[1]이 디코딩된 mRNA는 번역을 위해 억제되며 외부 핵물질에 의해 분해될 것이다.[2]핵심 디코딩 단지는 진핵생물에 보존되어 있다.Dcp2는 디코딩 단백질 1(Dcp1)과 비계 단백질 VCS가 결합한 고농축 진핵생물에서 활성화된다.[3]다른 많은 부속 단백질들과 함께, 분해하는 복잡한 결합체들은 세포질에서 P-beades로 구성된다.
디코딩 콤플렉스의 목적
mRNA는 분해될 필요가 있다. 그렇지 않으면 세포주위를 계속 떠다니며 원하지 않는 단백질을 임의로 생성한다.mRNA 5' 캡은 mRNA가 사용되기 전에 분해되지 않도록 특별히 설계되어 있으므로 mRNA 붕괴 경로가 이를 처리할 수 있도록 제거해야 한다.[4]
디코딩 메커니즘
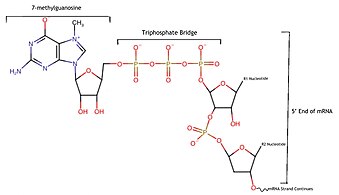
Dcp2는 mRNA를 실제로 분해하는 단백질로, 복합체 내 나머지 단백질은 그 기능을 강화하고 mRNA를 5'캡에 부착하는 화학적 결합을 가수 분해할 수 있게 한다.[5]Dcp2의 누딕스 도메인은 mRNA와 5'의 캡을 함께 고리 모양으로 만드는 3인산 브리지의 결합 중 하나를 가수분해하여 7-메틸과노신 캡이 벗겨지고 세포의 외핵에 의해 mRNA가 분해되도록 열어둔다.[4]
디코딩 콤플렉스의 구조
단세포 유기체와 다세포 유기체 모두 이를 제거하기 위해 mRNA를 분해해야 하지만, 유기체마다 이 과정을 수행하는 단백질이 조금씩 다르다.같은 상태를 유지하는 단백질은 많지만 단세포(이스트)와 다세포(메타조아) 디코딩 콤플렉스의 몇 가지 주요 차이점이 있다.[5]
효모 디코딩 콤플렉스
효모(S. cerebisiae)에서는 Dcp2가 디코딩 활성제 Dcp1, 헬리코아제 Dhh1, 엑소누클리스 Xrn1, 넌센스 매개 붕괴 인자 Upf1, Upf2, Upf3, LSm 콤플렉스, Pat1, 그리고 다양한 단백질로 결합된다.이 단백질들은 모두 P-beades라고 불리는 세포질 구조에 국소화된다.특히 효모에는 P-body 안에 번역인자나 리보솜 단백질이 없다.[6]
메타조안 디코딩 콤플렉스
높은 진핵생물들은 디코딩 콤플렉스의 구성원이 약간 다르다.효소 Dcp2는 여전히 Dcp1과 함께 홀로엔자임(holoenzyme)을 형성하고, Xrn1, Upf1, Upf2, Upf3, LSm 복합체, Dhh1 정형 DDX6 등의 보조 단백질과 상호작용하는 촉매 서브단위다.[5][7][8]식물과 포유류 고유의 단백질로는 베타 프로펠러 단백질 헤들(Hedls)과 Edc3를 해독하는 엔핸서가 있다.[9]연구자들은 이 복합체가 면역복제로 인해 물리적으로 어떻게 연관되는지 알고 있는 반면, 복합체의 각 부분의 구조적 세부사항은 단백질 크리스타일과 연계한 X선 결정학을 이용해 밝혀냈다.이들 단백질은 각각 아래와 같이 디코딩 콤플렉스에 서로 다른 것을 기여한다.
Dcp2
Dcp2는 디코딩 과정의 주요 촉매로서, 그것을 가수 분해하기 위해 5's cap에 맞추어 스스로를 정렬시키기 위해 누딕스 영역이라고 불리는 아미노산의 특정한 패턴에 의존한다.[5]누딕스 영역은 여러 개의 알파 나선 사이에 두 개의 베타 시트를 포장하여 만들어지며, 다양한 길이와 크기가 될 수 있으며, 일반적으로 단백질에 의해 인산염과 나머지 분자 사이의 결합에 물 분자를 삽입하여 인산염을 제거함으로써 탈인산화를 수행하는데 사용된다.[10]Dcp2의 경우 정상 세포 조건에서 음전하를 띠는 글루탐산 사이드 체인을 여러 개 포함하고 있으며, 이를 통해 단백질이 물 분자를 조작하여 mRNA의 5' 끝부분을 7-메틸구아노신 캡으로 연결하는 3-인산 다리를 가수분해 할 수 있다.[5]따라서 누딕스 도메인은 Dcp2가 5'캡을 제거할 수 있게 하는 것으로, 그 결과 GDP 7m, 인산염 2개 그룹이 부착된 7-메틸과노신, mRNA 스트랜드가 생성된다.
누딕스 도메인 이전에 NRD(N-terminal Regulation Domain)는 5'mRNA 캡을 가수 분해하는 데 더 도움이 된다.Nudix 도메인은 Box B라고 불리는 C-단자 영역으로 Dcp2를 RNA에 바인딩하는 것을 돕는다.[5]이 세 가지 주요 모티브로 Dcp2는 5'mRNA 캡을 찾고 단단히 바인딩하며 가수 분해할 수 있다.이것은 Dcp2 결합 및 디코딩 원소라 불리는 캡의 10가지 기본 쌍 내에서 RNA의 헤어핀 루프를 인식하거나, mRNA에서 기본 쌍 패턴을 인식하여 Dcp2-Dcp1 홀로엔자임(holoenzyme)을 직접 모집하는 별도의 단백질에 의해 이루어진다.[8]불행하게도 Dcp2는 느리게 작동하며, 그것과 조정하기 위해 몇 개의 다른 단백질이 필요하다. 그래야 적시에 mRNA를 해독할 수 있다.
Dcp1
Dcp1은 규제 서브 유닛으로 Dcp2와 결합해 mRNA를 제대로 디캡할 수 있는 홀로엔자임(holoenzyme)을 만든다.[11]Dcp1이 없다면 Dcp2가 생체내 어떤 것도 decapp하는 것은 사실상 불가능하며, 그것은 믿을 수 없을 정도로 느린 체외에서만 작용하기 때문에 이 홀로엔자임 형성은 decapping에 필수적인 과정이 된다.[5]
Dcp1의 이차 구조는 7개의 베타 시트와 3개의 알파 헬리크로 구성되어 있다.V자 모양의 3차 구조를 형성하기 위해 모인다.Dcp1의 정의 특징은 EVH1 도메인과 다른 단백질에 대한 proline 리치 시퀀스(PRS)를 인식하는 도메인이다.EVH1 도메인은 앞에서 언급한 Dcp2의 NRD와 직접 상호작용하며, 비록 그렇게 하는 방법은 불분명하지만, 현재는 mRNA의 디코딩을 직접적으로 돕는다고 생각된다.PRS를 인식하는 영역은 대부분 소수성 아미노산으로 이루어져 있으며, Dcp1 구조의 'V'의 구획 내에서 발견된다.Dcp1에 대한 디코딩 콤플렉스의 다른 단백질과 결합하는 데 사용된다.[11]
PNRC2
PNRC2는 Dcp1에 부착되어 Dcp1의 효과를 높여 디코딩을 장려하며, 디코딩 단지에 Upf1을 채용하기도 한다.소수성인 프롤라인 리치 시퀀스를 보유하고 있으며 Dcp1의 균등 소수성 구획에 강하게 달라붙어, Dcp1은 PNRC2의 프롤라인 리치 영역을 묶어 Dcp2의 기능을 더욱 강화한다.현재 연구에서는 PNRC2가 Dcp2와 Dcp1을 함께 연관시켜 Dcp2-Dcp1 홀로엔자임(holenoenzyme)을 더욱 안정화시켜 Dcp2의 효능을 높이는 데 도움이 된다고 제안하고 있으나, 그 방법에 대한 정확한 내용은 모호하다.[12]Upf1의 모집은 해독단지가 난센스 매개 mRNA 붕괴에 참여할 수 있도록 해, PNRC2가 잘못 기록된 mRNA 파괴를 담당하는 규제 경로와 연결되는 길이 된다.[13]
업프1-3
Upf1, Upf2, Upf3는 nonse-mediated mRNA 붕괴의 규제 경로에 관여하는 단백질이지 mRNA의 실제 디코딩은 아니다.Upf1만 디코딩 콤플렉스에 직접 부착하고 Upf2와 Upf3는 mRNA에 부착한 후, Upf1에 부착하여 부정확한 mRNA의 파괴를 용이하게 한다.이들은 잘못 형성된 mRNA에서 콤플렉스를 지시할 수 있지만 실제로 mRNA를 분해하는 데는 도움이 되지 않는다는 점에서 콤플렉스의 활성제다.[8]
DDX6
Dhh1의 직교자인 DDX6는 또한 Dcp2-Dcp1 홀로엔자임(holoenzyme)이 5's cap을 가수 분해하는 동안 효과성을 향상시킨다.[14]헬리코아제인 만큼 dcp2가 5'캡에 더 쉽게 접근할 수 있도록 mRNA의 5' 끝부분을 재구성하는 데 관여하고, 디코딩 단지의 나머지 부분에 부착되었을 때 Dcp2와 더 잘 상호작용하도록 Dcp1을 자극하는 것이 제안된다.[15]
Edc3
Edc3는 Dcp2-Dcp1 홀로엔자임(holoenzyme)을 더욱 활성화시켜 mRNA를 신속하게 해독할 수 있게 한다.N-terminus에 LSm 도메인을 보유하고 있어 Dcp1의 C 종단부에서 발견된 HLM 파편이라는 특정 아미노산 모티브와 상호작용해 Edc3가 결합할 수 있도록 했다.이 단백질의 또 다른 중요한 부분은 FDF 링커로, DDX6와 결합하고 mRNA와 직접 결합하는 것을 막아 대신 해독 콤플렉스의 단백질과 상호작용을 가능하게 하는 길고 구조화되지 않은 아미노산 스트레칭이다.노트의 마지막 영역은 Yjef-N C-terminus 도메인으로, mRNA로 희미해지고 디코딩 단지의 위치 주변에 P-beadies를 만들 수 있도록 돕는다.[5]
P-bodies는 기본적으로 디코딩되거나 억제된 mRNA와 디코딩 콤플렉스 및 nonse-mediated mRNA 붕괴 기계 등의 mRNA 분해계수를 혼합하여 비축되어 있으므로 Dcp2에 의해 변경되는 mRNA의 최종 파괴에 중요하다.[16]Edc3는 디코딩 콤플렉스를 중심으로 P-bape를 생성하기 때문에 dcp2가 mRNA 5' caps를 찾기 쉬워져 콤플렉스 전체의 효과가 높아진다.[17]
팻1
팻1은 디코딩 콤플렉스의 효율을 높이는 또 다른 단백질이다.[17]그것은 세 개의 주요 도메인을 가지고 있다.하나는 mRNA를 해독하는 데 필요하며, Dcp2-Dcp1 홀로엔자임에게 직접 도움을 준다.나머지 두 가지는 단백질이 mRNA를 쉽게 해독할 수 있게 해주지만 인산염 결합의 가수분해에는 직접 관여하지 않는다.[16]pat1은 해독 콤플렉스 내의 다양한 단백질과 많은 상호작용을 하며, 무언가를 해독할 때가 되면 모든 것을 하나로 모아주기 때문에 '삭제 단백질'로 알려져 있다.N-terminus 도메인은 DXX6과 상호 작용하여 Dcp1을 활성화할 수 있도록 가깝게 하고, 다른 부분은 Edc3와 함께 P-beadies를 생성하는데 도움을 주며, C-terminus 도메인은 Dcp1–Dcp2, Lsm1–7 콤플렉스, Xrn1을 단지에 부착한다.[5][18]
Xrn1
Xrn1은 방금 결정된 mRNA를 비하하는 5에서 3의 엑소누클레이저다.그것은 dcp2가 mRNA에 부착하는 3개의 인산염 중 2개와 함께 캡을 수산화하여 7-메틸과노신 캡을 떼어낼 때 남는 mRNA의 5' 단인산염 끝을 대상으로 한다.Xrn1은 Dcp2가 아직 해독하지 않은 어떤 mRNA와 단백질이 상호작용을 하지 못하도록 물리적으로 차단하는 강인한 장애가 있는 구조로 되어 있기 때문에 Xrn1의 구조는 capped mRNA와 상호작용을 허용하지 않는다는 것이 현재의 이론이다.[7]
참조
- ^ a b Mugridge, Jeffrey S; Ziemniak, Marcin; Jemielity, Jacek; Gross, John D (November 2016). "Structural basis of mRNA cap recognition by Dcp1–Dcp2". Nature Structural & Molecular Biology. 23 (11): 987–994. doi:10.1038/nsmb.3301. ISSN 1545-9993. PMC 5113729. PMID 27694842.
- ^ Chantarachot T, Bailey-Serres J (January 2018). "Polysomes, Stress Granules, and Processing Bodies: A Dynamic Triumvirate Controlling Cytoplasmic mRNA Fate and Function". Plant Physiology. 176 (1): 254–269. doi:10.1104/pp.17.01468. PMC 5761823. PMID 29158329.
- ^ Sieburth LE, Vincent JN (2018-12-17). "Beyond transcription factors: roles of mRNA decay in regulating gene expression in plants". F1000Research. 7: 1940. doi:10.12688/f1000research.16203.1. PMC 6305221. PMID 30613385.
- ^ a b Beelman, C. A.; Parker, R. (1995-04-21). "Degradation of mRNA in eukaryotes". Cell. 81 (2): 179–183. doi:10.1016/0092-8674(95)90326-7. ISSN 0092-8674. PMID 7736570. S2CID 9803923.
- ^ a b c d e f g h i Charenton, Clément; Graille, Marc (2018-12-19). "mRNA decapping: finding the right structures". Philosophical Transactions of the Royal Society B: Biological Sciences. 373 (1762): 20180164. doi:10.1098/rstb.2018.0164. PMC 6232594. PMID 30397101.
- ^ Parker R, Sheth U (March 2007). "P bodies and the control of mRNA translation and degradation". Molecular Cell. 25 (5): 635–46. doi:10.1016/j.molcel.2007.02.011. PMID 17349952.
- ^ a b Delorme-Axford, Elizabeth; Abernathy, Emma; Lennemann, Nicholas J.; Bernard, Amélie; Ariosa, Aileen; Coyne, Carolyn B.; Kirkegaard, Karla; Klionsky, Daniel J. (2018-05-04). "The exoribonuclease Xrn1 is a post-transcriptional negative regulator of autophagy". Autophagy. 14 (5): 898–912. doi:10.1080/15548627.2018.1441648. ISSN 1554-8627. PMC 6070002. PMID 29465287.
- ^ a b c Kramer, Susanne; McLennan, Alexander G. (2019). "The complex enzymology of mRNA decapping: Enzymes of four classes cleave pyrophosphate bonds". WIREs RNA. 10 (1): e1511. doi:10.1002/wrna.1511. ISSN 1757-7012. PMID 30345629. S2CID 53044937.
- ^ Fenger-Grøn M, Fillman C, Norrild B, Lykke-Andersen J (December 2005). "Multiple processing body factors and the ARE binding protein TTP activate mRNA decapping" (PDF). Molecular Cell. 20 (6): 905–15. doi:10.1016/j.molcel.2005.10.031. PMID 16364915. Archived from the original (PDF) on 2011-06-06.
- ^ "InterPro". www.ebi.ac.uk. Retrieved 2020-11-14.
- ^ a b She, Meipei; Decker, Carolyn J; Sundramurthy, Kumar; Liu, Yuying; Chen, Nan; Parker, Roy; Song, Haiwei (March 2004). "Crystal structure of Dcp1p and its functional implications in mRNA decapping". Nature Structural & Molecular Biology. 11 (3): 249–256. doi:10.1038/nsmb730. ISSN 1545-9993. PMC 2040073. PMID 14758354.
- ^ Lai, Tingfeng; Cho, Hana; Liu, Zhou; Bowler, Matthew W.; Piao, Shunfu; Parker, Roy; Kim, Yoon Ki; Song, Haiwei (2012-12-05). "Structural Basis of the PNRC2-Mediated Link between mRNA Surveillance and Decapping". Structure. 20 (12): 2025–2037. doi:10.1016/j.str.2012.09.009. ISSN 0969-2126. PMID 23085078.
- ^ Baker, Kristian E; Parker, Roy (2004-06-01). "Nonsense-mediated mRNA decay: terminating erroneous gene expression". Current Opinion in Cell Biology. 16 (3): 293–299. doi:10.1016/j.ceb.2004.03.003. ISSN 0955-0674. PMID 15145354.
- ^ Sharif, Humayun; Ozgur, Sevim; Sharma, Kundan; Basquin, Claire; Urlaub, Henning; Conti, Elena (September 2013). "Structural analysis of the yeast Dhh1–Pat1 complex reveals how Dhh1 engages Pat1, Edc3 and RNA in mutually exclusive interactions". Nucleic Acids Research. 41 (17): 8377–90. doi:10.1093/nar/gkt600. PMC 3783180. PMID 23851565.
- ^ Fischer, Nicole; Weis, Karsten (2002-06-03). "The DEAD box protein Dhh1 stimulates the decapping enzyme Dcp1". The EMBO Journal. 21 (11): 2788–2797. doi:10.1093/emboj/21.11.2788. ISSN 0261-4189. PMC 126031. PMID 12032091.
- ^ a b Pilkington, Guy R.; Parker, Roy (2008-02-15). "Pat1 Contains Distinct Functional Domains That Promote P-Body Assembly and Activation of Decapping". Molecular and Cellular Biology. 28 (4): 1298–1312. doi:10.1128/MCB.00936-07. ISSN 0270-7306. PMC 2258743. PMID 18086885.
- ^ a b Franks, Tobias M.; Lykke-Andersen, Jens (2008-12-05). "The Control of mRNA Decapping and P-Body Formation". Molecular Cell. 32 (5): 605–615. doi:10.1016/j.molcel.2008.11.001. ISSN 1097-2765. PMC 2630519. PMID 19061636.
- ^ Sharif, Humayun; Ozgur, Sevim; Sharma, Kundan; Basquin, Claire; Urlaub, Henning; Conti, Elena (September 2013). "Structural analysis of the yeast Dhh1–Pat1 complex reveals how Dhh1 engages Pat1, Edc3 and RNA in mutually exclusive interactions". Nucleic Acids Research. 41 (17): 8377–8390. doi:10.1093/nar/gkt600. ISSN 0305-1048. PMC 3783180. PMID 23851565.