Cas9
Cas9이 문서에서는 다른 Cas9s, 다른 Class 2 Type II CRISPR 시스템을 포함한 호몰로그에 대한 정보가 누락되어 있습니다.(2021년 9월) |
| CRISPR관련핵산가수분해효소Cas9 | |||||||
|---|---|---|---|---|---|---|---|
 S. pyogenes Cas9의 결정구조와 sgRNA 및 그 표적 DNA가 2.5Ω [1]분해능인 복합체. | |||||||
| 식별자 | |||||||
| 유기체 | |||||||
| 기호. | 케이스9 | ||||||
| Alt. | SpCas9 | ||||||
| 엔트레즈 | 901176 | ||||||
| PDB | 4OO8 | ||||||
| RefSeq(mRNA) | NC_002737.2 | ||||||
| RefSeq(프로트) | NP_269215.1 | ||||||
| 유니프로트 | Q99ZW2 | ||||||
| 기타 데이터 | |||||||
| EC 번호 | 3.1.-.- | ||||||
| 염색체 | 게놈: 0.85~0.86 Mb | ||||||
| |||||||
| Cas9 | |
|---|---|
| 식별자 | |
| 기호. | ? |
| 인터프로 | IPR028629 |
Cas9(CRISPR 관련 단백질 9, Csn1 또는 Csx12)는 DNA 바이러스와 플라스미드에 대한 특정 박테리아의 면역학적 방어에 중요한 역할을 하는 160킬로달톤 단백질로 유전자 공학 응용 분야에서 많이 이용된다.그것의 주된 기능은 DNA를 잘라내서 세포의 게놈을 바꾸는 것이다.CRISPR-Cas9 게놈 편집 기술은 2020년 노벨 화학상을 에마뉴엘 샤펜티에와 제니퍼 [2]도드나에게 수여하는 데 중요한 기여를 했다.
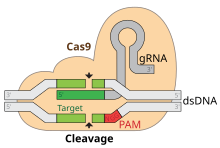
보다 엄밀히 말하면, Cas9은 Streptoccus pyogenes의 [3][4]CRISPR(Clustered Regularly Interspaceed Short Palindromic Repeates) 적응 면역 체계와 관련된 이중 RNA 유도 DNA 핵산 내 효소이다.S. pyogenes는 CRISPR을 사용하여 기억하며 Cas9를 사용하여 박테리오파지 DNA 또는 플라스미드 [4][5][6][7]DNA 침입과 같은 외부 DNA를 추궁하고 절단합니다.Cas9은 외부 DNA를 풀고 가이드 RNA(gRNA)의 20개 뉴클레오티드 스페이서 영역에 상보적인 부위를 체크함으로써 이 질문을 수행한다.DNA 기판이 가이드 RNA에 상보적인 경우 Cas9은 침입한 DNA를 절단한다.이러한 의미에서 CRISPR-Cas9 메커니즘은 진핵생물에서의 RNA 간섭(RNAi) 메커니즘과 많은 유사성을 가진다.
Cas9 단백질은 세균 면역에 대한 본래의 기능 외에도 DNA에서 부위 지향적인 이중 가닥 파괴를 유도하는 게놈 공학적 도구로서 많이 사용되어 왔다.이러한 파괴는 많은 실험실 모델 유기체에서 각각 비호몰로지 말단 결합과 호몰로지 재조합을 통해 유전자 불활성화 또는 이종 유전자의 도입을 초래할 수 있다.아연 핑거뉴클라아제 및 Transcription Activator-like effector nuclease(TALEN) 단백질과 함께 Cas9은 게놈 편집 분야에서 중요한 도구가 되고 있다.
Cas9은 가이드 [4]RNA에 상보적인 거의 모든 배열을 절단할 수 있기 때문에 최근 몇 년 동안 트랙션을 얻고 있습니다. Cas9의 표적 특이성은 가이드 RNA에서 비롯되기 때문입니다.단백질 자체에 대한 수정이 아닌 DNA 상호보완성(TALENs 및 아연 핑거)은 새로운 DNA를 목표로 하는 Cas9을 엔지니어링하는 [8]것은 간단합니다.관련 DNA를 결합하지만 분리하지 않는 Cas9 버전은 전사 활성화 및 [9][10]억제를 제어하기 위해 특정 DNA 배열에 대한 전사 활성화제 또는 억제제를 찾기 위해 사용될 수 있다.Native Cas9은 CRISPR RNA(crRNA)와 트랜스활성화 crRNA(tracrRNA)[3]라는 두 개의 이질적인 RNA로 구성된 가이드 RNA를 필요로 합니다.Cas9 타겟팅은 키메라 단일 가이드 RNA(chiRNA)의 엔지니어링을 통해 단순화되었습니다.과학자들은 Cas9 기반의 유전자 드라이브가 [11]전체 유기체의 게놈을 편집할 수 있을 것이라고 제안했다.2015년,[12] Cas9은 인간 배아의 게놈을 수정하는 데 처음으로 사용되었다.
CRISPR 매개 면역
박테리오파지로 가득 찬 다양한 도전적이고 불우한 서식지에서 살아남기 위해 박테리아와 고세균은 포식성 바이러스를 피하고 물리치는 방법을 발전시켰다.이것은 적응 면역의 CRISPR 시스템을 포함한다.실제로 CRISPR/Cas 시스템은 자체 프로그램 가능한 제한 효소로 작용합니다.CRISPR 궤적은 24~48뉴클레오티드 길이의 CRISPR 반복과 가변 CRISPR 스페이서로 구성된 일정한 간격으로 발생하는 짧은 회문 반복으로 구성됩니다.이러한 CRISPR 흔적은 대개 인접한 CRISPR 관련(cas) 유전자와 함께 나타난다.2005년, 세 개의 별도 그룹에 의해 스페이서 영역이 플라스미드와 바이러스를 포함한 외래 DNA 원소와 상동성이 있다는 것이 발견되었다.이 보고서들은 CRISPR이 면역 체계로서 기능할 수 있다는 최초의 생물학적 증거를 제공했다.
Cas9은 게놈 편집 도구로 자주 사용되어 왔다.Cas9은 바이러스가 숙주의 DNA를 조작하는 것을 막는데 최근 개발에 사용되어 왔다.CRISPR-Cas9은 박테리아 게놈 시스템에서 개발되었기 때문에 바이러스의 유전 물질을 목표로 하는 데 사용될 수 있다.Cas9 효소의 사용은 많은 바이러스 감염의 해결책이 될 수 있다.Cas9은 바이러스 유전 정보의 특정 가닥을 타겟팅함으로써 특정 바이러스를 타겟팅하는 능력을 가지고 있다.보다 구체적으로 Cas9 효소는 바이러스가 정상적인 [13]기능을 수행하는 것을 막는 바이러스 게놈의 특정 부분을 목표로 한다.Cas9은 또한 질병을 일으키는 해로운 DNA와 RNA의 가닥과 돌연변이된 DNA 가닥을 파괴하는 데 사용되어 왔다.Cas9은 이미 HIV-1의 영향을 교란할 가능성을 보여왔다. Cas9은 HIV-1에서 장기간의 말기 반복의 발현을 억제하는 것으로 나타났다.Cas9는 HIV-1 [14][15]게놈에 도입되었을 때 HIV-1의 가닥을 변이시키는 능력을 보여 왔다.Cas9은 B형 간염 바이러스 [16]게놈의 긴 말기 반복의 말단을 목표로 하여 B형 간염 치료에 사용되어 왔다.Cas9은 생쥐의 백내장을 일으키는 돌연변이를 치료하는데 사용되어 왔다.
오래된 정보로 인해 이 문서의 일부(주요 유형 수와 관련된 항목)의 사실 정확도가 저하될 수 있습니다.(2021년 9월) |
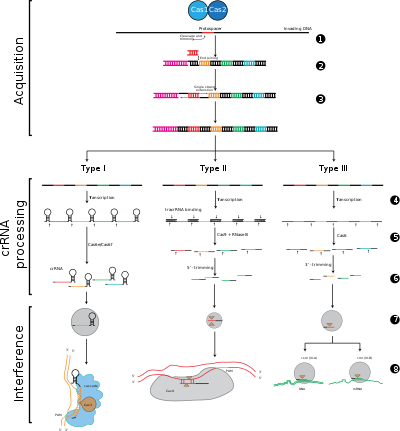
CRISPR-Cas 시스템은 크게 세 가지 유형(타입 I, 타입 II, 타입 III)과 12개의 서브타입으로 구분되며, 이들 서브타입은 유전적 함량과 구조적 차이에 따라 달라집니다.그러나 모든 CRISPR-Cas 시스템의 핵심 정의 특성은 cas 유전자와 그 단백질이다. cas1과 cas2는 유형 및 아형에 걸쳐 보편적이며, cas3, cas9 및 cas10은 각각 유형 I, 유형 II 및 유형 III의 시그니처 유전자이다.
CRISPR-Cas 방어 스테이지
적응.
적응은 CRISPR 궤적에서 인접한 두 반복측정 사이의 스페이서 인식 및 통합을 포함합니다."프로토스피서"는 스페이서에 해당하는 바이러스 게놈의 염기서열을 말한다.보존된 뉴클레오티드의 짧은 스트레치는 프로토스페이서 인접 모티브(PAM)라고 불리는 프로토스페이서 근처에 존재합니다.PAM은 DNA [7]단편 획득에 사용되는 인식 모티브입니다.타입 II에서 Cas9은 기능 스페이서를 [5]획득하기 위해 적응 중에 PAM을 인식합니다.
아라나즈 외 연구진도 스페이서 손실과 여러 그룹의 손실을 관찰했다.2004년 및 Pourcel 등 2007년.이것은 아마도 반복간 물질의 [17]상동 재조합을 통해 발생할 것이다.
CRISPR 처리/바이오제네이션
CRISPR 발현에는 CRISPR RNA(Pre-crRNA)라고 불리는 1차 전사가 포함되며, 이는 RNA 중합효소에 의해 CRISPR 궤적에서 전사된다.그런 다음 특정 엔도리보핵산가수분해효소는 사전 CRNA를 작은 CRISPR RNA(crRNA)[18]로 분해한다.
간섭/면역
간섭은 CASCADE라고 불리는 다단백질 복합체 내의 crRNA를 포함하며, 이것은 상보적인 외래 DNA를 삽입하는 영역을 인식하고 특히 염기쌍을 형성할 수 있다.그런 다음 crRNA-외부 핵산 복합체를 절단하지만 스페이서와 표적 DNA 사이에 불일치가 있거나 PAM에 돌연변이가 있는 경우 분할이 시작되지 않습니다.후자의 시나리오에서는, 외부 DNA는 세포의 공격을 목표로 하지 않기 때문에, 바이러스의 복제가 진행되어 숙주는 바이러스 감염에 면역이 되지 않는다.간섭 단계는 기계적으로나 일시적으로 CRISPR 획득 및 발현과 구별될 수 있지만, 방어 시스템으로서의 완전한 기능을 위해서는 세 단계 모두 [19]기능해야 합니다.
스테이지 1: CRISPR 스페이서 통합.원호스페이서 및 원호스페이서 관련 모티브(빨간색으로 표시)는 숙주 DNA에서 CRISPR 배열의 "리더" 끝에서 획득됩니다.CRISPR 배열은 스페이서 시퀀스(컬러 박스 표시)와 리피트(검은 다이아몬드)로 구성됩니다.이 프로세스에서는 Cas1과 Cas2(및[5] 타입 II의 Cas9)가 필요합니다.이들은 보통 CRISPR 어레이 근처에 있는 cas locus에 부호화되어 있습니다.
스테이지 2: CRISPR 표현.프리 crRNA는 숙주 RNA 중합효소에 의해 리더 영역에서 시작하여 Cas 단백질에 의해 단일 스페이서 및 부분 반복을 포함하는 더 작은 crRNA로 분해된다(색 스페이서를 가진 헤어핀 구조 표시).
스테이지 3: CRISPR 간섭. 유입되는 외래 DNA에 강한 상보성을 가진 스페이서를 가진 crRNA는 Cas 단백질을 필요로 하는 절단 이벤트(가위로 퇴적)를 시작한다.DNA 분열은 바이러스 복제를 방해하고 숙주에 대한 면역성을 제공한다.간섭 단계는 기능적으로나 일시적으로 CRISPR 획득 및 표현(셀을 나누는 흰색 선으로 구분됨)과 구별될 수 있습니다).
dCas9를 사용한 문자 변환 비활성화
또한 엔도핵산가수분해효소결핍 Cas9라고도 불리는 dCas9는 유전자 중 원하는 부분의 전사결합 부위에 적용되었을 때 유전자 발현을 편집하는데 이용될 수 있다.dCas9의 최적 기능은 동작 모드에 기인합니다.뉴클레오티드가 RNA사슬에 더 이상 첨가되지 않아 해당 사슬의 신장이 종료되고 그 결과 전사 과정에 영향을 미칠 때 유전자 발현을 억제한다.이 과정은 dCas9이 대량 생산될 때 일어나 배열 특이 안내 RNA 분자를 통해 주어진 시간에 가장 많은 양의 유전자에 영향을 미칠 수 있다.dCas9은 유전자 발현을 하향 조절하는 것으로 보이기 때문에, 이 작용은 억제성 크로마틴 수식자 [20]도메인과 함께 사용될 때 더욱 증폭된다.dCas9 단백질은 유전자 발현 조절 이외의 다른 기능을 가지고 있다.dCas9 단백질에 프로모터를 첨가하여 DNA 가닥을 따라 서로 다른 배열에서 전사를 시작하거나 정지하는 데 효율적이 되도록 할 수 있다.이 두 단백질은 유전자에 작용하는 부분에서 특이하다.이것은 프로모터와 dCas9가 DNA의 전사 조각을 형성하기 위해 함께 모이는 뉴클레오티드의 폴리머의 신장을 방해하기 위해 함께 정렬할 때 특정 유형의 원핵생물에 널리 퍼져 있습니다.프로모터가 없으면 dCas9 단백질은 그 자체 또는 유전자 [21]본체와 동일한 효과를 발휘하지 못한다.
전사의 억제 효과를 더 조사하면 히스톤의 아미노산 성분인 H3K27은 dCas9와 FOG1이라는 펩타이드의 상호작용을 통해 메틸화된다.본질적으로 이 상호작용은 유전자의 특정 접합부에서 아미노산 복합체의 C+N 말단부에 유전자 억제를 유발하고 그 결과 [22]전사가 종료된다.
dCas9은 또한 질병을 발생시킬 수 있는 특정 단백질을 변화시키는 데 효과적이라는 것을 증명한다.dCas9이 guide-RNA라고 불리는 RNA의 형태에 결합할 때, 그것은 유기체의 게놈에 해로울 수 있는 반복 코돈과 DNA 서열의 증식을 막는다.기본적으로 여러 개의 반복 코돈이 생성되면 반응을 유도하거나 풍부한 dCas9를 모집하여 코돈의 과잉생산을 방지하고 전사의 정지를 초래한다.dCas9은 gRNA와 상승작용을 일으켜 연속 전사에 의해 DNA 중합효소 II에 직접 영향을 준다.
dCas9 단백질이 어떻게 작용하는지에 대한 자세한 설명은 식물에서 유전자 생산을 조절하여 특정 특성을 증가시키거나 감소시키는 식물 게놈의 활용에서 찾을 수 있다.CRISPR-CAS9 시스템은 유전자를 상향 조절하거나 하향 조절하는 기능을 가지고 있다.dCas9 단백질은 CRISPR-CAS9 시스템의 구성 요소이고 이 단백질들은 식물 유전자의 특정 영역을 억제할 수 있다.이는 DCAS9가 억제제 도메인에 결합할 때 발생하며, 식물의 경우 AtCSTF64와 같은 조절 유전자의 비활성화가 발생한다.[23]
박테리아는 dCas9 단백질 사용의 또 다른 초점이다.진핵생물들은 더 큰 DNA 구성과 게놈을 가지고 있기 때문에; 훨씬 더 작은 박테리아는 조작하기 쉽다.그 결과 진핵생물은 dCas9를 사용하여 RNA 중합효소가 유전물질의 [24]전사과정을 지속하는 것을 억제한다.
구조 및 생화학 연구
결정 구조
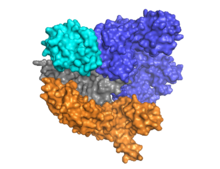
Cas9은 가이드 RNA가 알파 헬리컬 로브(파란색)와 뉴클레아제 로브(cyan, 주황색 및 회색) 사이에 위치한 이중 로브 아키텍처를 특징으로 합니다.이 두 개의 로브는 단일 브리지 나선을 통해 연결됩니다.다중 도메인 핵산가수분해효소 로브에는 비 표적 DNA 가닥을 절단하는 RuvC(회색)와 표적 DNA 가닥을 절단하는 HNH 핵산가수분해효소 도메인(cyan)의 두 가지 핵산가수분해효소 도메인이 있다.RuvC 도메인은 3차 구조에서 상호작용하여 RuvC 분할 도메인을 형성하는 순차적으로 서로 다른 사이트에 의해 인코딩됩니다(오른쪽 그림 참조).
대상 DNA의 특징은 그것은three-nucleotide sequence- NGG로 구성된(PAM) protospacer 인접한 모티프를 포함해야 한다.(PI도메인, 오렌지)Cas9의 C-terminal 끝 근처에 위치한 이 PAM은 PAM-interacting 도메인에 의해 인정 받고 있다.Cas9고 RNA을 RNA반드시 가이드:아포 사이에는 뚜렷한 형태 변화를 겪는다.DNA상태를 묶었다.
Cas9은 stem-loop 구조crRNA-tracrRNA 리보 핵 단백질 복합체의 성숙을 중재하는 CRISPR 활동 중심지에 고유한 인식하고 있다.[26]Cas9 단지에 CRISPR RNA(crRNA)과 trans-activating crRNA과(tracrRNA)더 발전하고 파괴할 대상 dsDNA을 인정하고 있다.[27]그co-crystal 여기 건물에서는 그 crRNA-tracrRNA 단지 자연 RNA단지로 동일한 기능임이 판명된 비현실적인single-guide RNA(sgRNA의 붉은)에 의해 대체된다.[4]그 sgRNA 기본 목표 ssDNA과 쌍을 이루Cas9에 의한 T자형 건축물처럼 정박해 있다.그 DNA-bound Cas9 것 효소의 이 결정 구조가 알파 헬릭스의. 엽의 뉴 클레아 제라에 관한 뚜렷한 형태 변화뿐만 아니라 HNH 도메인의 위치를 드러낸다.단백질은 인식 엽(REC)과 뉴 클레아 제라 엽(NUC)로 구성되어 있다.그 HNH를 제외한 지역 형태 서로와 sgRNA-ssDNA 단지 다른과 긴밀하게 상호 작용, 반면 HNH 도메인이 단백질의 나머지를 연락처를 형성한다.Cas9 단지가 결정 발견된 다른 형태에서, HNH 도메인이 보이지 않는다.이러한 구조체 HNH 도메인의 형태 유연성을 시사하고 있다.
현재까지, 적어도 세개의 수정 구조와 출판된 공부를 해왔다.하나는 apo [25]상태의 Cas9의 Configuration을 나타내고, 다른 하나는 DNA [28][1]결합 상태의 Cas9를 나타냅니다.
sgRNA와의 상호작용
sgRNA-Cas9 복합체에서는 결정구조에 기초하여 REC1, BH 및 PI 도메인은 반복 영역과 스페이서 [1][28]영역 모두에서 백본 또는 베이스와 중요한 접촉을 갖는다.REC1 또는 REC2 도메인 삭제 및 BH의 잔류물 돌연변이를 포함한 여러 Cas9 돌연변이가 테스트되었다.REC1 및 BH 관련 돌연변이는 야생형에 비해 활성이 낮거나 전혀 없으며, 이는 이들 두 도메인이 반복 시퀀스에서의 sgRNA 인식과 전체 복합체의 안정화에 매우 중요하다는 것을 의미한다.스페이서 시퀀스와 Cas9 사이의 상호작용 및 PI 도메인과 반복 영역 간의 상호작용은 추가 연구가 필요하지만, Cas9과 sgRNA 사이의 명확한 인터페이스를 보여줍니다.
DNA분열
이전의 배열 분석 및 생화학 연구는 Cas9가 McrA 유사 HNH 핵산가수분해효소 도메인과 RuvC 유사 핵산가수분해효소 도메인이라는 [29]두 개의 핵산가수분해효소 도메인을 포함하는 것으로 가정했다.이러한 HNH 및 RuvC 유사 핵산가수분해효소 도메인은 각각 [4]상보/대상 DNA 가닥과 비상보/비대상 DNA 가닥의 분열을 담당한다.낮은 배열 유사성에도 불구하고, RNase H와 유사한 배열은 RuvC 폴드(RNase H 패밀리의 1명)를 가지며, HNH 영역은 T4 Endo VII(HNH 엔도핵산가수분해효소 [citation needed]패밀리의 1명)로 접힌다.
야생형 S. pyogenes Cas9은 RNA 매개 DNA 분열을 위해 마그네슘(Mg2+) 보조 인자를 필요로 하지만, Cas9은 다른 2가 금속 [4]이온의 존재 하에서 다양한 수준의 활성을 보이는 것으로 나타났다.예를 들어 망간(Mn2+)이 존재하는 Cas9은 RNA의존성 DNA분열이 [30]가능한 것으로 나타났다.Cas9에 의한 DNA 분열의 역학은 과학계의 큰 관심사였는데, 이 데이터는 반응의 복잡함에 대한 통찰력을 제공하기 때문이다.RNA 결합 Cas9에 의한 DNA의 분할은 비교적 빠른 것으로 나타났지만(k 700 700s−1), 분할 생성물의 방출은 매우 느리며(t1/2 = ln(2)/k 43 43-91 h), 기본적으로 Cas9은 단일 결합 [31]효소로 생성된다.Cas9의 역학에 관한 추가 연구는 Cas9가 [32][33]반응 속도를 변경함으로써 목표 외의 효과를 감소시키는 데 효과적이라는 것을 보여주었다.
Cas9의 분할 효율은 많은 요인에 의해 결정됩니다.주요 요건은 절단 부위 [34]하류의 비표적 스트랜드 3 뉴클레오티드에 유효한 PAM이 존재하는 것이다.S의 표준 PAM 시퀀스. Pyogenes Cas9은 NGG이지만 다른 모티브는 낮은 절단 활성으로 허용된다.와일드 타입 S를 위한 가장 효율적인 대체 PAM 모티브. Pyogenes Cas9은 NAG와 [35][36]NGA입니다.gRNA의 20 뉴클레토드 스페이서 영역을 보완하는 표적 DNA 부위의 배열 조성도 분할 효율에 영향을 미친다.효율성에 영향을 미치는 가장 관련성이 높은 뉴클레오티드 조성 특성은 PAM-근접 [37][38][36]영역에 있는 특성입니다.핵산의 자유 에너지 변화 또한 매우 분열 활동을 정의하는데 관련이 있다.[39]가이드 RNAs DNA에 결합 자유 에너지 변화의 일반적으로 효율적으로 수행하는 극도로 또는 안정적인 약한 바인딩을 배제하는 제한 범위에 속하양면을 형성하는 복사물.[36]안정적인 안내 RNA 접기 입체 배열 또한 분열을 떨어뜨릴 수 있다.[40]
Cas9 편집 시 세균이 일으키는 문제
대부분의 고세균과 박테리아는 Cas9이 그들의 게놈을 편집하는 것을 완강히 거부한다.이것은 그들이 그들에게 영향을 미치지 않는 외래 DNA를 그들의 게놈에 부착할 수 있기 때문이다.이들 셀이 Cas9에 대항하는 또 다른 방법은 RM(Restriction Modification) 시스템 프로세스입니다.박테리오파지가 박테리아나 고세균 세포에 들어가면 RM 시스템에 의해 표적이 됩니다.그리고 나서 RM 시스템은 제한 효소에 의해 박테리오파지 DNA를 분리하여 DNA의 가닥을 더욱 파괴하기 위해 엔도핵산가수분해효소를 사용한다.이는 RM 시스템이 Cas9 [41]프로세스에 의해 추가된 외래 유전자도 대상으로 하기 때문에 Cas9 편집에 문제가 됩니다.
Cas9의 문자 변환 튜닝 적용
dCas9에 의한 전사 간섭
Cas9의 어떤 게놈에서도 본질적으로 어떤 보체 배열에도 결합할 수 있는 독특한 능력 때문에, 연구원들은 다양한 게놈 궤적의 전사를 억제하기 위해 이 효소를 사용하기를 원했다.이를 달성하기 위해 RuvC 및 HNH 도메인의 두 가지 중요한 촉매 잔기는 Cas9의 모든 엔도핵산가수분해효소 활성을 폐지하는 알라닌으로 변이될 수 있다.그 결과 생성된 단백질은 '죽은' Cas9 또는 줄여서 'dCas9'로 만들어졌으며, 여전히 dsDNA와 단단히 결합할 수 있다.이 촉매적으로 비활성화된 Cas9 변종은 Cas9 DNA 질문 결합에 대한 기계적 연구와 일반적인 프로그램 가능한 DNA 결합 RNA-단백질 복합체 모두에 사용되어 왔다.
목표 dsDNA과 dCas9의 상호 작용이 너무 높은 질량 요소 단백질 denaturant 완전히dsDNA 타겟에서 나오는dCas9 RNA-protein 복잡한 분리할 수 없은 꽉 낀다.[42]dCas9는 강화된 단일 가이드 RNAs에 어떤 장소의 dCas9 RNA중합 효소와 프로모터에 transcrip를 막기 위해 경쟁할 수 있개시 사이트 transcription에 표적이 되어 왔다.또한 [43]dCas9는 [43]전사의 신장 단계에서 RNA 중합효소의 억제가 일어나도록 위치 부호화 영역을 목표로 할 수 있다.진핵생물에서는 dCas9를 인핸서 배열로 타겟팅함으로써 유전자 발현 사일런스를 연장할 수 있으며, 여기서 dCas9는 특정 [10]유전자 발현 사일런스로 이어지는 전사인자의 집합을 차단할 수 있다.또한 dCas9에 제공되는 가이드 RNA는 [43]연구자가 관심 유전자에 적용되는 유전자 사일링의 정도를 조정할 수 있도록 하기 위해 dCas9의 상호 작용을 양적으로 약화시키는 상보적 동인지 배열에 특정 불일치를 포함하도록 설계될 수 있다.이 기술은 RNAi와 원칙적으로 유사해 유전자 발현이 RNA 수준에서 조절된다.그러나 dCas9 접근법은 RNAi [44]스크린에 비해 목표 외 효과가 적고 일반적으로 dCas9의 사용을 통해 더 크고 더 재현 가능한 소음 효과가 존재하기 때문에 많은 관심을 끌었다.또한 유전자 사일런싱에 대한 dCas9 접근방식은 정량적으로 제어할 수 있기 때문에 연구자는 관심 있는 유전자가 억제되는 정도를 정밀하게 제어할 수 있게 되어 유전자 조절 및 유전자 화학측정법에 대한 더 많은 의문이 해결된다.
dCas9의 위치 전사상 민감한 위치에 대한 직접 결합을 넘어 dCas9은 다양한 변조 단백질 도메인과 융합하여 무수한 기능을 수행할 수 있다.최근 dCas9은 염색질 리모델링 단백질(HDACs/[43]HATs)에 융합되어 다양한 위치 주변의 염색질 구조를 재구성하고 있다.이는 헤테로크로마틴 구조가 Cas9 결합을 방해하기 때문에 관심 있는 다양한 진핵생물의 유전자를 대상으로 하는 데 중요하다.또한 Cas9은 헤테로크로마틴에 반응할 수 있기 때문에 이 효소는 다양한 [43]자리의 크로마틴 구조를 연구하는 데 더욱 응용할 수 있다는 이론이 있다.또한 dCas9은 유전자 억제의 게놈 와이드 스크린에 사용되어 왔다.수천 개의 유전자를 대상으로 할 수 있는 가이드 RNA의 큰 라이브러리를 사용하여 dCas9를 이용한 게놈 전체 유전자 검사를 실시했습니다.[45]
Cas9로 전사를 소음하는 또 다른 방법은 촉매 활성 Cas9 [46]효소로 mRNA 제품을 직접 분해하는 것이다.이 접근방식은 Cas9 결합을 위한 dsDNA-RNA PAM 사이트를 허용하는 ssRNA에 PAM 보완 시퀀스를 가진 ssDNA를 하이브리드화함으로써 가능합니다.이 기술은 RNA나 RNA 태깅 방법에 대한 화학적 수정을 유도할 필요 없이 세포에서 내인성 RNA 전사를 분리할 수 있는 능력을 가능하게 한다.
dCas9 융합단백질에 의한 전사활성화
사일런트 유전자와 달리 dCas9은 전사활성인자와 [43]융합될 때 유전자를 활성화하는 데 사용될 수도 있다.이러한 인자는 박테리아 RNA 중합효소 II의 서브유닛과 진핵생물에서의 전통적인 전사 인자를 포함한다.최근에는 'CRISPRa'라는 이름의 dCas9 융합을 [45]활성화하는 게놈 전체의 전사 활성화 스크린도 이루어졌다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI, Dohmae N, Ishitani R, Zhang F, Nureki O (February 2014). "Crystal structure of Cas9 in complex with guide RNA and target DNA". Cell. 156 (5): 935–49. doi:10.1016/j.cell.2014.02.001. PMC 4139937. PMID 24529477.
- ^ "The Nobel Prize in Chemistry 2020". NobelPrize.org. Retrieved 2020-10-07.
- ^ a b Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E (March 2011). "CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III". Nature. 471 (7340): 602–607. Bibcode:2011Natur.471..602D. doi:10.1038/nature09886. PMC 3070239. PMID 21455174.
- ^ a b c d e f Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (August 2012). "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity". Science. 337 (6096): 816–21. Bibcode:2012Sci...337..816J. doi:10.1126/science.1225829. PMC 6286148. PMID 22745249.
- ^ a b c Heler R, Samai P, Modell JW, Weiner C, Goldberg GW, Bikard D, Marraffini LA (March 2015). "Cas9 specifies functional viral targets during CRISPR-Cas adaptation". Nature. 519 (7542): 199–202. Bibcode:2015Natur.519..199H. doi:10.1038/nature14245. PMC 4385744. PMID 25707807.
- ^ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (March 2007). "CRISPR provides acquired resistance against viruses in prokaryotes". Science. 315 (5819): 1709–12. Bibcode:2007Sci...315.1709B. doi:10.1126/science.1138140. hdl:20.500.11794/38902. PMID 17379808. S2CID 3888761.
- ^ a b Garneau JE, Dupuis MÈ, Villion M, Romero DA, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán AH, Moineau S (November 2010). "The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA". Nature. 468 (7320): 67–71. Bibcode:2010Natur.468...67G. CiteSeerX 10.1.1.451.9645. doi:10.1038/nature09523. PMID 21048762. S2CID 205222849.
- ^ Mali P, Esvelt KM, Church GM (October 2013). "Cas9 as a versatile tool for engineering biology". Nature Methods. 10 (10): 957–63. doi:10.1038/nmeth.2649. PMC 4051438. PMID 24076990.
- ^ Mali P, Aach J, Stranges PB, Esvelt KM, Moosburner M, Kosuri S, Yang L, Church GM (September 2013). "CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering". Nature Biotechnology. 31 (9): 833–8. doi:10.1038/nbt.2675. PMC 3818127. PMID 23907171.
- ^ a b Gilbert LA, Larson MH, Morsut L, Liu Z, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS (July 2013). "CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes". Cell. 154 (2): 442–51. doi:10.1016/j.cell.2013.06.044. PMC 3770145. PMID 23849981.
- ^ Esvelt KM, Smidler AL, Catteruccia F, Church GM (July 2014). "Concerning RNA-guided gene drives for the alteration of wild populations". eLife. 3. doi:10.7554/eLife.03401. PMC 4117217. PMID 25035423.
- ^ Cyranoski D, Reardon S (22 April 2015). "Chinese scientists genetically modify human embryos". Nature. doi:10.1038/nature.2015.17378. S2CID 87604469.
- ^ Doudna JA, Mali P (2016). CRISPR-Cas: a laboratory manual. Cold Spring Harbor, New York. ISBN 9781621821304. OCLC 922914104.
- ^ Chen W, Page-McCaw PS (March 2019). "CRISPR/Cas9 gene editing". AccessScience. McGraw-Hill Education. doi:10.1036/1097-8542.168060.
- ^ Ebina H, Misawa N, Kanemura Y, Koyanagi Y (2013-08-26). "Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus". Scientific Reports. 3 (1): 2510. Bibcode:2013NatSR...3E2510E. doi:10.1038/srep02510. PMC 3752613. PMID 23974631.
- ^ Li H, Sheng C, Wang S, Yang L, Liang Y, Huang Y, Liu H, Li P, Yang C, Yang X, Jia L, Xie J, Wang L, Hao R, Du X, Xu D, Zhou J, Li M, Sun Y, Tong Y, Li Q, Qiu S, Song H (2017-03-22). "Removal of Integrated Hepatitis B Virus DNA Using CRISPR-Cas9". Frontiers in Cellular and Infection Microbiology. 7: 91. doi:10.3389/fcimb.2017.00091. PMC 5360708. PMID 28382278.
- ^ Deveau H, Garneau JE, Moineau S (2010-10-13). "CRISPR/Cas system and its role in phage-bacteria interactions". Annual Review of Microbiology. Annual Reviews. 64 (1): 475–493. doi:10.1146/annurev.micro.112408.134123. PMID 20528693.
- ^ Horvath P, Barrangou R (January 2010). "CRISPR/Cas, the immune system of bacteria and archaea". Science. 327 (5962): 167–70. Bibcode:2010Sci...327..167H. doi:10.1126/science.1179555. PMID 20056882. S2CID 17960960.
- ^ Karginov FV, Hannon GJ (January 2010). "The CRISPR system: small RNA-guided defense in bacteria and archaea". Molecular Cell. 37 (1): 7–19. doi:10.1016/j.molcel.2009.12.033. PMC 2819186. PMID 20129051.
- ^ Jensen ED, Ferreira R, Jakočiūnas T, Arsovska D, Zhang J, Ding L, et al. (March 2017). "Transcriptional reprogramming in yeast using dCas9 and combinatorial gRNA strategies". Microbial Cell Factories. 16 (1): 46. doi:10.1186/s12934-017-0664-2. PMC 5353793. PMID 28298224.
- ^ Pinto BS, Saxena T, Oliveira R, Méndez-Gómez HR, Cleary JD, Denes LT, McConnell O, Arboleda J, Xia G, Swanson MS, Wang ET (November 2017). "Impeding Transcription of Expanded Microsatellite Repeats by Deactivated Cas9". Molecular Cell. 68 (3): 479–490.e5. doi:10.1016/j.molcel.2017.09.033. PMC 6013302. PMID 29056323.
- ^ O'Geen H, Ren C, Nicolet CM, Perez AA, Halmai J, Le VM, Mackay JP, Farnham PJ, Segal DJ (September 2017). "dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression". Nucleic Acids Research. 45 (17): 9901–9916. doi:10.1093/nar/gkx578. PMC 5622328. PMID 28973434.
- ^ Lowder LG, Paul JW, Qi Y (2017). "Multiplexed Transcriptional Activation or Repression in Plants Using CRISPR-dCas9-Based Systems". Plant Gene Regulatory Networks. Methods in Molecular Biology. Vol. 1629. pp. 167–184. doi:10.1007/978-1-4939-7125-1_12. ISBN 978-1-4939-7124-4. PMID 28623586.
- ^ Barrangou R, Horvath P (June 2017). "A decade of discovery: CRISPR functions and applications". Nature Microbiology. 2 (7): 17092. doi:10.1038/nmicrobiol.2017.92. PMID 28581505. S2CID 19072900.
- ^ a b Jinek M, Jiang F, Taylor DW, Sternberg SH, Kaya E, Ma E, Anders C, Hauer M, Zhou K, Lin S, Kaplan M, Iavarone AT, Charpentier E, Nogales E, Doudna JA (March 2014). "Structures of Cas9 endonucleases reveal RNA-mediated conformational activation". Science. 343 (6176): 1247997. doi:10.1126/science.1247997. PMC 4184034. PMID 24505130.
- ^ Wiedenheft B, Sternberg SH, Doudna JA (February 2012). "RNA-guided genetic silencing systems in bacteria and archaea". Nature. 482 (7385): 331–8. Bibcode:2012Natur.482..331W. doi:10.1038/nature10886. PMID 22337052. S2CID 205227944.
- ^ Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (November 2013). "Genome engineering using the CRISPR-Cas9 system". Nature Protocols. 8 (11): 2281–2308. doi:10.1038/nprot.2013.143. PMC 3969860. PMID 24157548.
- ^ a b Anders C, Niewoehner O, Duerst A, Jinek M (September 2014). "Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease". Nature. 513 (7519): 569–73. Bibcode:2014Natur.513..569A. doi:10.1038/nature13579. PMC 4176945. PMID 25079318.
- ^ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (March 2006). "A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action". Biology Direct. 1 (1): 7. doi:10.1186/1745-6150-1-7. PMC 1462988. PMID 16545108.
- ^ Sundaresan R, Parameshwaran HP, Yogesha SD, Keilbarth MW, Rajan R (December 2017). "RNA-Independent DNA Cleavage Activities of Cas9 and Cas12a". Cell Reports. 21 (13): 3728–3739. doi:10.1016/j.celrep.2017.11.100. PMC 5760271. PMID 29281823.
- ^ Raper AT, Stephenson AA, Suo Z (February 2018). "Functional Insights Revealed by the Kinetic Mechanism of CRISPR/Cas9". Journal of the American Chemical Society. 140 (8): 2971–2984. doi:10.1021/jacs.7b13047. PMID 29442507.
- ^ Lee JK, Jeong E, Lee J, Jung M, Shin E, Kim YH, et al. (August 2018). "Directed evolution of CRISPR-Cas9 to increase its specificity". Nature Communications. 9 (1): 3048. Bibcode:2018NatCo...9.3048L. doi:10.1038/s41467-018-05477-x. PMC 6078992. PMID 30082838.
- ^ Singh D, Wang Y, Mallon J, Yang O, Fei J, Poddar A, et al. (April 2018). "Mechanisms of improved specificity of engineered Cas9s revealed by single-molecule FRET analysis". Nature Structural & Molecular Biology. 25 (4): 347–354. doi:10.1038/s41594-018-0051-7. PMC 6195204. PMID 29622787.
- ^ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (August 2012). "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity". Science. 337 (6096): 816–21. doi:10.1126/science.1225829. PMC 6286148. PMID 22745249.
- ^ Kim N, Kim HK, Lee S, et al. (2020). "Prediction of the sequence-specific cleavage activity of Cas9 variants". Nat Biotechnol. 38: 1328–1336. doi:10.1038/s41587-020-0537-9. PMID 32514125.
- ^ a b c Corsi GI, Qu K, Alkan F, Pan X, Luo Y, Gorodkin J (May 2022). "CRISPR/Cas9 gRNA activity depends on free energy changes and on the target PAM context". Nature Communications. 13 (3006). doi:10.1038/s41467-022-30515-0. PMC 9151727. PMID 35637227.
- ^ Xu H, Xiao T, Chen CH, Li W, Meyer CA, Wu Q, Wu D, Cong L, Zhang F, Liu JS, Brown M, Liu XS (Aug 2015). "Sequence determinants of improved CRISPR sgRNA design". Genome Res. 25 (8). doi:10.1101/gr.191452.115. PMC 4509999. PMID 26063738.
- ^ Xiang X, Corsi, GI, Anthon C, Qu K, Pan X, Liang X, Han P, Dong Z, Liu L, Zhong J, Ma T, Wang J, Zhang X, Jiang H, Xu F, Liu X, Xu X, Wang J, Yang H, Bolund L, Church GM, Lin L, Gorodkin J, Luo Y (May 2021). "Enhancing CRISPR-Cas9 gRNA efficiency prediction by data integration and deep learning". Nature Communications. 12 (1): 3238. doi:10.1038/s41467-021-23576-0. PMC 8163799. PMID 34050182.
- ^ Alkan F, Wenzel A, Anthon C, Havgaard JH, Gorodkin J (October 2018). "CRISPR-Cas9 off-targeting assessment with nucleic acid duplex energy parameters". Genome Biology. 19 (177): 177. doi:10.1186/s13059-018-1534-x. PMC 6203265. PMID 30367669.
- ^ Thyme SB, Akhmetova L, Montague TG, Valen E, Schier AF (Jun 2016). "Internal guide RNA interactions interfere with Cas9-mediated cleavage". Nature Communications. 7 (11750). doi:10.1038/ncomms11750. PMC 4906408. PMID 27282953.
- ^ Kusano K, Naito T, Handa N, Kobayashi I (November 1995). "Restriction-modification systems as genomic parasites in competition for specific sequences". Proceedings of the National Academy of Sciences of the United States of America. 92 (24): 11095–9. Bibcode:1995PNAS...9211095K. doi:10.1073/pnas.92.24.11095. PMC 40578. PMID 7479944.
- ^ Sternberg SH, Redding S, Jinek M, Greene EC, Doudna JA (March 2014). "DNA interrogation by the CRISPR RNA-guided endonuclease Cas9". Nature. 507 (7490): 62–7. Bibcode:2014Natur.507...62S. doi:10.1038/nature13011. PMC 4106473. PMID 24476820.
- ^ a b c d e f Bikard D, Jiang W, Samai P, Hochschild A, Zhang F, Marraffini LA (August 2013). "Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system". Nucleic Acids Research. 41 (15): 7429–37. doi:10.1093/nar/gkt520. PMC 3753641. PMID 23761437.
- ^ Heintze J, Luft C, Ketteler R (2013). "A CRISPR CASe for high-throughput silencing". Frontiers in Genetics. 4: 193. doi:10.3389/fgene.2013.00193. PMC 3791873. PMID 24109485.
- ^ a b Gilbert LA, Horlbeck MA, Adamson B, Villalta JE, Chen Y, Whitehead EH, Guimaraes C, Panning B, Ploegh HL, Bassik MC, Qi LS, Kampmann M, Weissman JS (October 2014). "Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation". Cell. 159 (3): 647–61. doi:10.1016/j.cell.2014.09.029. PMC 4253859. PMID 25307932.
- ^ O'Connell MR, Oakes BL, Sternberg SH, East-Seletsky A, Kaplan M, Doudna JA (December 2014). "Programmable RNA recognition and cleavage by CRISPR/Cas9". Nature. 516 (7530): 263–6. Bibcode:2014Natur.516..263O. doi:10.1038/nature13769. PMC 4268322. PMID 25274302.
추가 정보
- Kennedy EM, Cullen BR (May 2015). "Bacterial CRISPR/Cas DNA endonucleases: A revolutionary technology that could dramatically impact viral research and treatment". Virology. 479–480: 213–20. doi:10.1016/j.virol.2015.02.024. PMC 4424069. PMID 25759096.
- Abbott TR, Dhamdhere G, Liu Y, Lin X, Goudy L, Zeng L, et al. (May 2020). "Development of CRISPR as an Antiviral Strategy to Combat SARS-CoV-2 and Influenza". Cell. 181 (4): 865–876.e12. doi:10.1016/j.cell.2020.04.020. PMC 7189862. PMID 32353252.
- Steven Levy (March 18, 2020). "Could Crispr Be Humanity's Next Virus Killer?". Wired.