UTP-글루코스-1-인산 우리딜릴트랜스퍼라제
UTP—glucose-1-phosphate uridylyltransferase| UTP-글루코스-1-인산 우리딜릴트랜스퍼라제 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 인간 UTP—피몰에서 만들어진 글루코스-1-인산염 유리딜트랜스퍼라제 만화 | |||||||||
| 식별자 | |||||||||
| EC no. | 2.7.7.9 | ||||||||
| CAS no. | 9026-22-6 | ||||||||
| 데이터베이스 | |||||||||
| 인텐즈 | InTEnz 뷰 | ||||||||
| 브렌다 | 브렌다 항목 | ||||||||
| ExPASY | 나이스자임뷰 | ||||||||
| KEGG | KEGG입력 | ||||||||
| 메타사이클 | 대사 경로 | ||||||||
| 프리암 | 옆모습 | ||||||||
| PDB 구조물 | RCSB PDB PDBe PDBsum | ||||||||
| 유전자 온톨로지 | 아미고 / 퀵고 | ||||||||
| |||||||||
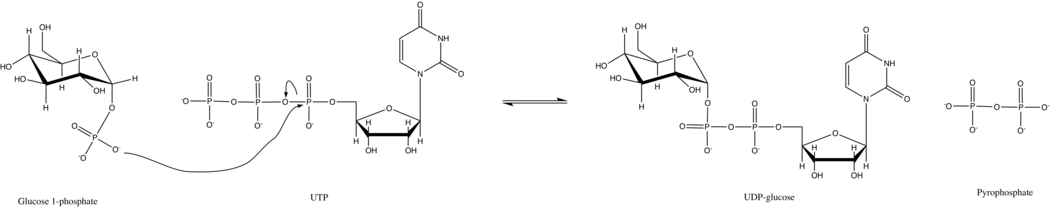
UTP-글루코스-1-인산 유리딜트랜스퍼라제(, UDP-glucose pyrophosphorylase)는 탄수화물 대사에 관여하는 효소입니다. 글루코스-1-인산과 UTP로부터 UDP-글루코스를 합성합니다.
- 글루코스-1-인산염 + ⇌ \harpoons} UDP-글루코스 + 피로인산염
UTP-glucose-1-phosphate uridyltransferase는 당 생성과 세포벽 합성의 핵심 역할을 하기 때문에 세 가지 도메인(세균, 진핵, 고세균)에서 모두 발견되는 효소입니다. 당 대사에서의 당의 역할은 식물의 성장을 이해하고 농업 생산을 증가시키기 위해 식물에서 광범위하게 연구되었습니다. 최근 인간 UTP-glucose-1-phosphate uridyltransferase가 연구되고 결정화되어 이전에 연구된 다른 유기체와 다른 유형의 조절을 밝혀내고 있습니다. 그 중요성은 갈락토스 대사, 글리코겐 합성, 당단백질 합성 및 당지질 합성을 포함한 UDP-포도당의 많은 사용에서 비롯됩니다.[1][2]
구조.
UTP-glucose-1-phosphate uridyltransferase의 구조는 원핵생물과 진핵생물 사이에서 상당히 다르지만 진핵생물 내에서 효소의 1차, 2차, 3차 구조는 상당히 보존되어 있습니다.[3] 많은 종에서 UTP-glucose-1-phosphate uridyltransferase는 대칭적인 4차 구조에서 동일한 소단위로 구성된 단일중합체로 발견됩니다.[4][5] 서브유닛의 수는 종에 따라 다릅니다. 예를 들어, 대장균에서는 효소가 사량체로 발견되는 반면, 부르크홀데리아 제노보란스에서는 효소가 이량체로 발견됩니다.[5][6] 인간과 효모에서 효소는 소단위체 사이의 계면에 소수성 잔기와 함께 서로 적층된 두 개의 사량체로 구성된 팔량체로 활성화됩니다.[7][8] 대조적으로, 식물의 효소는 소단위체 사이의 계면을 형성하는 전하를 띤 잔기를 보존하고 있습니다.
인간의 경우, 각 효소 소단위체는 진핵생물에서 고도로 보존된 여러 잔기(L113, N251, N328)를 포함합니다. Rossman 폴드 모티프는 UTP 뉴클레오티드와 당 결합 도메인(잔기 T286–G293)이 글루코스 고리와 배위하는 결합에 참여합니다.[9] 활성 부위(진핵생물에서 보존되는)를 포함하는 효소 영역의 미스센스 돌연변이(G115D)는 시험관 내에서 효소 활성의 극적인 감소를 유발합니다.[10]
- UTP의 결정구조 - 부르크홀데리아 제노보란스의 글루코스-1-인산 우리딜릴트랜스퍼라제
- 인간 UTP-글루코스-1-인산 우리딜릴트랜스퍼라제는 UDP-글루코스가 결합된 동형 1 서브유닛이다.
예
UTP-glucose-1-phosphate uridyltransferase 활성을 갖는 단백질을 코딩하는 인간 유전자는 각각 56.9 kDa 및 55.7 kDa의 분자량을 갖는 2개의 이소형을 포함합니다.[11]
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||
기능.
UTP-glucose-1-phosphate uridyltransferase는 탄수화물 대사의 중심 화합물인 UDP-glucose 생성에 중요한 역할을 하기 때문에 자연적으로 어디에나 있습니다. 식물 잎에서 UTP-포도당-1-인산 우리딜릴트랜스퍼라제는 UDP-포도당 및 D-과당 6-인산을 수크로스-6-인산으로 전환시키는 수크로스-인산 합성효소에 우리딘 이인산 포도당을 공급하는 수크로스 생합성 경로의 핵심 부분입니다.[12] 또한 UDP-포도당을 사용하는 다른 조직에서 수크로스의 분해에 부분적으로 책임이 있을 수 있습니다.
고등 동물에서 효소는 간과 근육을 포함한 당 생성에 관여하는 조직에서 매우 활성적입니다.[13] 예외적으로 뇌는 글리코겐 수준은 높지만 UTP의 특정 활성은 낮은 글루코스-1-인산 우리딜릴트랜스퍼라제를 가지고 있습니다.[14] 동물 세포에서 UTP-glucose-1-phosphate uridyltransferase는 주로 세포질에서 발견됩니다.
UTP-glucose-1-phosphate uridyltransferase는 동물과 미생물의 갈락토스 대사에도 필요합니다. 갈락토스 대사에서 갈락토스 1-인산 우리딜릴트랜스퍼라제는 UDP-글루코스에서 갈락토스 1-인산으로 인산을 전달하여 UDP-갈락토스를 생성하고, 이를 UDP-글루코스로 전환합니다.[15] UTP-glucose-1-phosphate uridyltransferase가 결함이 있는 박테리아는 갈락토스를 세포벽에 통합할 수 없습니다.[16]
메카니즘
이 효소의 1차 반응에서 글루코스-1-인산 상의 인산기가 UTP 상의 인산기 결합을 대체합니다. 이 반응은 쉽게 가역적이며 깁스 자유 에너지는 0에 가깝습니다. 그러나 일반적인 세포 조건에서 무기 피로포스파타제는 피로인산염 생성물을 빠르게 가수분해하고 르 샤틀리에의 원리에 의해 반응을 진행시킵니다.
UTP-글루코스-1-인산 우리딜릴트랜스퍼라제는 정방향 및 역방향 반응 모두에 대해 순서화된 순차적 BiBi 메커니즘을 사용합니다.[17] 효모에서 효소는 단순한 Michaelis-Menten kinetics를 따르며 팔량체의 소단위체 간의 협력성을 나타내지 않습니다.[8]
다른 당 뉴클레오티딜트랜스퍼라제와 유사하게, UTP-glucose-1-phosphate uridyltransferase 활성은 음전하를 띤 인산기의 결합을 안정화하기 위해 두 개의 2가 양이온이 필요합니다.[18] 마그네슘은 일반적으로 이 역할을 하지만 망간과 같은 다른 이온들이 이 역할을 합니다.II), 코발트()II), 니켈(Nickel)II) 또한 최적의 활성을 ~75% 감소시키는 것으로 대체할 수 있습니다.[19] X선 결정학 실험은 하나의 Mg2+ 이온이 포도당 1-인산의 인산 산소와 UTP의 α-인산 산소에 의해 배위된다는 것을 보여주었습니다.[5] 음전하를 띤 인산염을 안정화시키는 것 외에도 Mg는2+ UTP의 α-인의 친핵성 공격을 위해 포도당 1-인산을 배향하는 것으로 생각됩니다.[20]
규정
UDP-glucose pyrophosphylase는 종 간에 기능적으로 유사하지만 유기체마다 구조와 조절 메커니즘이 다릅니다.
미생물
효모에서 UTP-glucose-1-phosphate uridyltransferase는 PAS kinase에 의한 인산화에 의해 조절됩니다.[21] 이 인산화는 가역적이며 글리코겐과 세포벽 합성을 향한 당 플럭스의 분할을 제어합니다.
식물
UTP-식물에서 글루코스-1-인산 우리딜릴트랜스퍼라제는 올리고머화 및 인산화를 통해 조절됩니다.[22] 보리에서 UDP-glucose pyrophosphylase는 단량체 형태에서만 활성이지만 쉽게 올리고머를 형성하는 것으로 나타났으며, 이는 올리고머화가 효소의 조절 형태일 수 있음을 시사합니다. 쌀에서 저온 스트레스는 효소의 N-글리코실화를 감소시키는데, 이는 추위에 반응하여 효소의 활성을 변화시키는 것으로 생각됩니다.[23]
Arabidopsis에는 UTP의 두 가지 동종효소인 glucose-1-phosphate uridyltransferase가 있습니다: UGP1과 UGP2.[24] 이 두 가지 동종효소는 활성이 거의 같고 32개의 아미노산만 다른데, 모두 활성 부위에서 떨어진 단백질의 바깥쪽 표면에 위치합니다. 이러한 사소한 차이는 동종효소 활성의 차등 알로스테릭 조절을 허용할 수 있습니다. UGP1과 UGP2는 식물의 다른 부분에서 차등적으로 발현됩니다. UGP1 발현은 대부분의 조직에서 광범위하게 발현되는 반면, UGP2는 주로 꽃에서 발현되며, 이는 UGP1이 효소의 주요 형태이고 UGP2가 보조 기능을 한다는 것을 시사합니다. 실제로, UGP2 발현은 인산염 결핍과 같은 스트레스 요인에 대한 반응으로 증가하며, 이는 UGP2가 아마도 식물이 환경 스트레스를 받을 때 UGP1에 대한 백업으로 기능함을 나타냅니다.
동물들
UTP-glucose-1-phosphate uridyltransferase 활성의 조절은 주로 유전적 수단(즉, 전사 및 번역의 조절)에 의해 이루어집니다. 대부분의 효소와 유사하게, UTP-glucose-1-phosphate uridyltransferase는 UDP-glucose의 산물에 의해 억제됩니다. 그러나 이 효소는 다양한 대사 경로에서 UDP-포도당이 널리 사용되는 것을 고려할 때 논리적으로 중요한 알로스테릭 조절을 받지 않습니다.
인간
인간에서 UDP-glucose pyrophosphylase는 옥타머로 활성화되어 있습니다.[7] 효소의 활성 또한 O-글리코실화에 의해 변경됩니다.[25] 다른 포유류 종들과 마찬가지로, 인간에게는 유전자의 대체 접합에 의해 생성되는 두 가지 다른 동형이 있습니다.[3][11][26] 동형은 N-말단에서 단지 11개의 아미노산 차이가 있고 그들의 기능적 활성에 있어서 유의미한 차이는 확인되지 않았습니다.
질병관련성
사람에게서 갈락토오스혈증은 당 갈락토오스를 제대로 대사하지 못해 신생아와 어린이의 발육에 영향을 미치는 질환입니다. UDP-glucose pyrophosphorylase의 과발현은 갈락토스혈증이 있는 사람의 증상을 완화시킬 수 있다고 추측됩니다.[27]
일반적으로 해당과정의 비율이 높고 글리코겐 함량이 감소하는 암세포에서 UTP-glucose-1-phosphate uridyltransferase의 활성은 정상 세포에 비해 최대 50-60%까지 하향 조절되는 경우가 많습니다.[28] UTP-glucose-1-phosphate uridyltransferase의 비정상적으로 낮은 활성은 효소 수준의 감소와 글리코겐 합성효소 및 포스포글루코뮤타제를 포함한 글리코겐 경로의 다른 효소의 하향 조절 때문입니다.
UTP-glucose-1-phosphate uridyltransferase는 박테리아 및 원생동물을 포함한 다양한 병원체에서 중요한 독성 인자로 밝혀졌습니다.[29][30] 예를 들어, 이 효소는 폐렴, 기관지염 및 기타 호흡 문제의 박테리아 원인인 연쇄상구균 폐렴의 중요한 독성 인자인 캡슐 다당류의 생합성에 필요한 것으로 밝혀졌습니다.[31] 그 결과 이 효소는 의약품의 잠재적 표적으로 주목을 받았습니다. 그러나 특이성을 달성하기 위해서는 활성 부위가 종에 걸쳐 고도로 보존되어 있기 때문에 약물이 단백질 표면의 알로스테릭 부위를 특이적으로 표적하도록 설계되어야 합니다.[3]
UDP-glucose pyrophosphylase (UGP2)는 최근 바라캇-페렌탈러 증후군이라고도 알려진 인간의 새로운 신경 발달 장애와 관련이 있는 것으로 밝혀졌습니다.[33] 이 장애는 15개 가족의 22명의 개인에서 처음 기술되었으며, 심각한 간질성 뇌병증, 거의 모든 발달 이정표가 없는 신경 발달 지연, 난치성 발작, 진행성 소두증, 시각 장애 및 유사한 경미한 변형을 나타냈습니다. Barakat와 동료들은 영향을 받은 모든 개체(chr2:64083454A > G)에서 반복적인 동형접합 돌연변이를 확인했으며, 이는 UGP2의 더 짧은 단백질 동형의 번역 시작 부위를 돌연변이시킵니다. 따라서 동형접합 돌연변이를 가진 환자에서는 더 이상 더 짧은 단백질 동형이 생성될 수 없습니다. 같은 그룹의 기능 연구에 따르면 짧은 단백질 동형은 보통 사람의 뇌에서 주로 발현됩니다. 따라서, 반복적인 돌연변이는 뇌에 조직 특이적인 UGP2의 부재를 초래하고, 이는 글리코겐 대사의 변화, 상향 조절된 펼쳐진 단백질 반응 및 조기 신경 분화로 이어집니다. UGP2의 짧고 긴 이소형이 고갈된 인간 배아 줄기세포가 심근세포와 혈액세포에서 분화하지 못하기 때문에 UGP2의 다른 이중 대립 기능 상실 돌연변이는 치명적일 가능성이 높습니다. 따라서, 이 새로운 질병의 확인은 또한 필수 단백질의 조직 관련 이소폼의 발현 손실을 유발하는 이소폼 특이적 시작-손실 돌연변이가 유기체 전체의 단백질 부재가 생명과 양립할 수 없는 경우에도 유전적 질병을 유발할 수 있음을 보여줍니다. 바라캇-페렌탈러 증후군에 대한 치료법은 현재 존재하지 않습니다.
참고문헌
- ^ Sandhoff K, van Echten G, Schröder M, Schnabel D, Suzuki K (August 1992). "Metabolism of glycolipids: the role of glycolipid-binding proteins in the function and pathobiochemistry of lysosomes". Biochemical Society Transactions. 20 (3): 695–9. doi:10.1042/bst0200695. PMID 1426613.
- ^ Alonso MD, Lomako J, Lomako WM, Whelan WJ (September 1995). "A new look at the biogenesis of glycogen". FASEB Journal. 9 (12): 1126–37. doi:10.1096/fasebj.9.12.7672505. PMID 7672505. S2CID 40281321.
- ^ a b c Führing JI, Cramer JT, Schneider J, Baruch P, Gerardy-Schahn R, Fedorov R (April 2015). "A quaternary mechanism enables the complex biological functions of octameric human UDP-glucose pyrophosphorylase, a key enzyme in cell metabolism". Scientific Reports. 5 (1): 9618. Bibcode:2015NatSR...5E9618F. doi:10.1038/srep09618. PMC 5381698. PMID 25860585.
- ^ Kim H, Choi J, Kim T, Lokanath NK, Ha SC, Suh SW, Hwang HY, Kim KK (April 2010). "Structural basis for the reaction mechanism of UDP-glucose pyrophosphorylase". Molecules and Cells. 29 (4): 397–405. doi:10.1007/s10059-010-0047-6. PMID 20238176. S2CID 25022544.
- ^ a b c Thoden JB, Holden HM (July 2007). "Active site geometry of glucose-1-phosphate uridylyltransferase". Protein Science. 16 (7): 1379–88. doi:10.1110/ps.072864707. PMC 2206702. PMID 17567737.
- ^ Disease, Seattle Structural Genomics Center for Infectious (2016). "Crystal structure of UDP-glucose pyrophosporylase / UTP-glucose-1-phosphate uridylyltransferase from Burkholderia xenovorans". Worldwide Protein Data Bank. doi:10.2210/pdb5j49/pdb.
- ^ a b Yu Q, Zheng X (March 2012). "The crystal structure of human UDP-glucose pyrophosphorylase reveals a latch effect that influences enzymatic activity". The Biochemical Journal. 442 (2): 283–91. doi:10.1042/BJ20111598. PMID 22132858.
- ^ a b Roeben A, Plitzko JM, Körner R, Böttcher UM, Siegers K, Hayer-Hartl M, Bracher A (December 2006). "Structural basis for subunit assembly in UDP-glucose pyrophosphorylase from Saccharomyces cerevisiae". Journal of Molecular Biology. 364 (4): 551–60. doi:10.1016/j.jmb.2006.08.079. PMID 17010990.
- ^ Kleczkowski LA, Geisler M, Fitzek E, Wilczynska M (November 2011). "A common structural blueprint for plant UDP-sugar-producing pyrophosphorylases". The Biochemical Journal. 439 (3): 375–9. doi:10.1042/BJ20110730. PMID 21992098.
- ^ Flores-Díaz M, Alape-Girón A, Persson B, Pollesello P, Moos M, von Eichel-Streiber C, Thelestam M, Florin I (September 1997). "Cellular UDP-glucose deficiency caused by a single point mutation in the UDP-glucose pyrophosphorylase gene". The Journal of Biological Chemistry. 272 (38): 23784–91. doi:10.1074/jbc.272.38.23784. PMID 9295324.
- ^ a b "UGP2 - UTP--glucose-1-phosphate uridylyltransferase - Homo sapiens (Human) - UGP2 gene & protein". www.uniprot.org. Retrieved 2017-03-06.
- ^ Mendicino J (December 1960). "Sucrose phosphate synthesis in wheat germ and green leaves". The Journal of Biological Chemistry. 235 (12): 3347–52. doi:10.1016/S0021-9258(18)64469-2. PMID 13769376.
- ^ Turnquist, Richard L.; Hansen, R. Gaurth (1973-01-01). "2 Uridine Diphosphoryl Glucose Pyrophosphorylase". In Boyer, Paul D. (ed.). The Enzymes. Group Transfer Part A: Nucleotidyl Transfer Nucleosidyl Transfer Acyl Transfer Phosphoryl Transfer. Vol. 8. Academic Press. pp. 51–71. doi:10.1016/S1874-6047(08)60062-1. ISBN 9780121227081.
- ^ Villar-Palasi C, Larner J (March 1960). "Insulin-mediated effect on the activity of UDPG-glycogen transglucosylase of muscle". Biochimica et Biophysica Acta. 39: 171–3. doi:10.1016/0006-3002(60)90142-6. PMID 13842294.
- ^ Bosch AM (August 2006). "Classical galactosaemia revisited". Journal of Inherited Metabolic Disease. 29 (4): 516–25. doi:10.1007/s10545-006-0382-0. PMID 16838075. S2CID 16382462.
- ^ Sundararajan TA, Rapin AM, Kalckar HM (December 1962). "Biochemical observations on E. coli mutants defective in uridine diphosphoglucose". Proceedings of the National Academy of Sciences of the United States of America. 48 (12): 2187–93. Bibcode:1962PNAS...48.2187S. doi:10.1073/pnas.48.12.2187. PMC 221142. PMID 13979281.
- ^ Tsuboi KK, Fukunaga K, Petricciani JC (February 1969). "Purification and specific kinetic properties of erythrocyte uridine diphosphate glucose pyrophosphorylase". The Journal of Biological Chemistry. 244 (3): 1008–15. doi:10.1016/S0021-9258(18)91886-7. PMID 5782905.
- ^ Zea CJ, Camci-Unal G, Pohl NL (July 2008). "Thermodynamics of binding of divalent magnesium and manganese to uridine phosphates: implications for diabetes-related hypomagnesaemia and carbohydrate biocatalysis". Chemistry Central Journal. 2 (1): 15. doi:10.1186/1752-153x-2-15. PMC 2490692. PMID 18627619.
- ^ Gustafson GL, Gander JE (March 1972). "Uridine diphosphate glucose pyrophosphorylase from Sorghum vulgare. Purification and kinetic properties". The Journal of Biological Chemistry. 247 (5): 1387–97. doi:10.1016/S0021-9258(19)45571-3. PMID 5012314.
- ^ Sivaraman J, Sauvé V, Matte A, Cygler M (November 2002). "Crystal structure of Escherichia coli glucose-1-phosphate thymidylyltransferase (RffH) complexed with dTTP and Mg2+". The Journal of Biological Chemistry. 277 (46): 44214–9. doi:10.1074/jbc.M206932200. PMID 12171937.
- ^ Rutter J, Probst BL, McKnight SL (October 2002). "Coordinate regulation of sugar flux and translation by PAS kinase". Cell. 111 (1): 17–28. doi:10.1016/s0092-8674(02)00974-1. PMID 12372297. S2CID 6883785.
- ^ Kleczkowski LA, Geisler M, Ciereszko I, Johansson H (March 2004). "UDP-glucose pyrophosphorylase. An old protein with new tricks". Plant Physiology. 134 (3): 912–8. doi:10.1104/pp.103.036053. PMC 523891. PMID 15020755.
- ^ Komatsu S, Yamada E, Furukawa K (January 2009). "Cold stress changes the concanavalin A-positive glycosylation pattern of proteins expressed in the basal parts of rice leaf sheaths". Amino Acids. 36 (1): 115–23. doi:10.1007/s00726-008-0039-4. PMID 18278531. S2CID 1797342.
- ^ Meng M, Geisler M, Johansson H, Harholt J, Scheller HV, Mellerowicz EJ, Kleczkowski LA (May 2009). "UDP-glucose pyrophosphorylase is not rate limiting, but is essential in Arabidopsis". Plant & Cell Physiology. 50 (5): 998–1011. doi:10.1093/pcp/pcp052. PMID 19366709.
- ^ Wells, Lance; Hart, Gerald W. (2003-07-03). "O-GlcNAc turns twenty: functional implications for post-translational modification of nuclear and cytosolic proteins with a sugar". FEBS Letters. 546 (1): 154–158. doi:10.1016/S0014-5793(03)00641-0. ISSN 1873-3468. PMID 12829252. S2CID 24587552.
- ^ Duggleby RG, Chao YC, Huang JG, Peng HL, Chang HY (January 1996). "Sequence differences between human muscle and liver cDNAs for UDPglucose pyrophosphorylase and kinetic properties of the recombinant enzymes expressed in Escherichia coli". European Journal of Biochemistry. 235 (1–2): 173–9. doi:10.1111/j.1432-1033.1996.00173.x. PMID 8631325.
- ^ Lai K, Elsas LJ (May 2000). "Overexpression of human UDP-glucose pyrophosphorylase rescues galactose-1-phosphate uridyltransferase-deficient yeast". Biochemical and Biophysical Research Communications. 271 (2): 392–400. doi:10.1006/bbrc.2000.2629. PMID 10799308.
- ^ Nigam VN, Macdonald HL, Cantero A (February 1962). "Limiting factors for glycogen storage in tumors. I. Limiting enzymes". Cancer Research. 22 (2): 131–8. PMID 14479721.
- ^ Jiang SS, Lin TY, Wang WB, Liu MC, Hsueh PR, Liaw SJ (May 2010). "Characterization of UDP-glucose dehydrogenase and UDP-glucose pyrophosphorylase mutants of Proteus mirabilis: defectiveness in polymyxin B resistance, swarming, and virulence". Antimicrobial Agents and Chemotherapy. 54 (5): 2000–9. doi:10.1128/AAC.01384-09. PMC 2863647. PMID 20160049.
- ^ Klein KA, Fukuto HS, Pelletier M, Romanov G, Grabenstein JP, Palmer LE, Ernst R, Bliska JB (February 2012). "A transposon site hybridization screen identifies galU and wecBC as important for survival of Yersinia pestis in murine macrophages". Journal of Bacteriology. 194 (3): 653–62. doi:10.1128/JB.06237-11. PMC 3264090. PMID 22139502.
- ^ Bonofiglio L, García E, Mollerach M (October 2005). "Biochemical characterization of the pneumococcal glucose 1-phosphate uridylyltransferase (GalU) essential for capsule biosynthesis". Current Microbiology. 51 (4): 217–21. doi:10.1007/s00284-005-4466-0. PMID 16132460. S2CID 13591083.
- ^ Perenthaler E, Nikoncuk A, Yousefi S, Berdowski WM, Alsagob M, Capo I, et al. (March 2020). "Loss of UGP2 in brain leads to a severe epileptic encephalopathy, emphasizing that bi-allelic isoform-specific start-loss mutations of essential genes can cause genetic diseases". Acta Neuropathologica. 139 (3): 415–442. doi:10.1007/s00401-019-02109-6. PMC 7035241. PMID 31820119.
- ^ "#618744: Epileptic Encephalopathy, Early Infantile 83; EIEE83". Online Mendelian Inheritance in Man (OMIM).
외부 링크
- 미국 국립 의학 도서관의 UDP+Glucose+Pyrophosphylase (MeSH)





