코로나바이러스 봉투단백질
Coronavirus envelope protein| 봉투단백질 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 | |||||||||
| 식별자 | |||||||||
| 기호 | CoV_E | ||||||||
| Pfam | PF02723 | ||||||||
| 인터프로 | IPR003873 | ||||||||
| 프로사이트 | PS51926 | ||||||||
| |||||||||
봉투(E) 단백질은 코로나바이러스 바이러스에서 발견된 4대 구조 단백질 중 가장 작고 성질이 덜하다.[2][3][4]110개 아미노산 잔류물보다 긴 일체형 막 단백질로,[2] 코비드-19의 원인제인 SARS-CoV-2에서 E 단백질은 75개 잔류물이다.[5]바이러스 복제에 반드시 필수적인 것은 아니지만, E 단백질의 부재는 비정상적으로 조립된 바이러스 캡시드를 생성하거나 복제를 줄일 수 있다.[2][3]E는 다기능성 단백질로[6] 바이러스 캡시드의 구조적 단백질로서의 역할 외에도 바이러스성 결합에 관여하고, 바이로포린으로서의 기능을 할 가능성이 있는 것으로 생각되며 바이러스성 병원체 발생에 관여한다.[2][5]
구조
E단백질은 짧은 친수성 N단말 영역, 소수성 나선 투과영역, 그리고 다소 친수성 C단말 영역으로 구성되어 있다.SARS-CoV와 SARS-CoV-2에서 C-터미널 영역은 PDZ 도메인 바인딩 모티브(PBM)를 포함하고 있다.[2][5]이 특성은 알파 및 베타 코로나바이러스 그룹에서만 보존되는 것으로 보이며 감마는 보존되지 않는다.[2]베타 및 감마 그룹에서 보존된 프로라인 잔류물이 골기 단백질을 대상으로 하는 데 관여할 가능성이 있는 C-단자 영역에서 발견된다.[2]
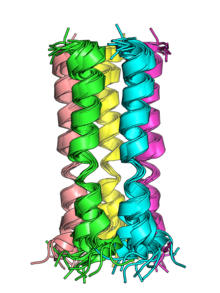
사스-CoV와 사스-CoV-2의 E단백질의 투과성 나선은 과점화될 수 있고 체외에서 양이온 선택적 이온 통로 역할을 하는 중앙 모공을 가진 펜트아메리카 구조를 형성하는 것으로 나타났다.[5]두 바이러스의 E단백질 펜타머는 구조적으로 핵자기공명 분광법에 의해 특징지어졌다.[5][7]
E 단백질의 멤브레인 위상은 일관되지 않은 결과를 가진 다수의 코로나비루스에서 연구되었다. 멤브레인에서 단백질의 방향은 가변적일 수 있다.[3]증거의 균형은 가장 일반적인 방향이 세포질 쪽으로 C-terminus를 향한다는 것을 암시한다.[8]사스-CoV-2 E 단백질에 대한 연구는 이 방향과 일치한다.[5][9]
변환 후 수정
전부는 아니지만 일부 코로나비루스에서는 E단백질이 보존된 시스테인 잔류물에 대한 Palmitoylation에 의해 번역 후 변형된다.[2][8]사스-CoV E 단백질에서는 한 개의 글리코실화 부위가 관찰되어 막 위상에 영향을 줄 수 있으나 E 글리코실화의 기능적 중요성은 불분명하다.[8][2]SARS-CoV E의 유비쿼터스화도 기술되어 있으나, 그 기능적 중요성도 알려져 있지 않다.[2]
표현 및 지역화
 E 유전자의 위치를 나타내는 SARS-CoV-2의 초기 시퀀싱 샘플인 Wuhan-Hu-1의 유전체 조직 | |
| NCBI 게놈 ID | 86693 |
|---|---|
| 게놈 크기 | 29,903 베이스 |
| 준공년도 | 2020 |
| 게놈 브라우저(UCSC) | |
E단백질은 감염된 세포에서 풍부하게 발현된다.그러나 조립된 처녀자리에서는 생성된 총 E단백질 중 소량만 발견된다.[2][4]E단백질은 내소성 망막, 골기 기구, 그리고 코로나바이러스 바이러스 봉투를 발생시키는 세포내 구획인 내소성 망막-망막-골기 중간 구획(ERGIC)에 국부화되어 있다.[2][5]
함수
본질성
다른 코로나비루스의 연구는 E가 바이러스 복제에 필수적인지에 대해 다른 결론에 도달했다.메르스-CoV를 포함한 일부 코로나바이러스에서는 E가 필수적인 것으로 보고되었다.[10]생쥐 코로나바이러스[11], 사스-CoV를 포함한 다른 사례에서 E는 전파 결함을 도입하거나 비정상적인 캡시드 형태학을 유발하여 바이러스 티터를 감소시키지만 필수적인 것은 아니다.[12][2]
바이러스 및 바이러스 어셈블리
E단백질은 조립된 처녀자리에서 발견되는데, 이 처녀자리에서 코로나바이러스막단백질(M)과 단백질-단백질 상호작용을 이루고 있는데, 바이러스 캡시드에 포함된 네 가지 구조 단백질 중 가장 풍부한 단백질이다.[2][4]E와 M의 상호작용은 막의 세포질 쪽에 있는 각각의 C-termini를 통해 일어난다.[2]대부분의 코로나비루스에서 E와 M은 바이러스와 같은 입자를 형성하기에 충분하지만 사스-CoV 역시 N에 의존한다고 보고되었다.[2][4][14]E가 전형적인 구형 코로나바이러스 바이러스 바이러스를 만들기 위해 막 곡률을 유도하는 데 관여하고 있다는 좋은 증거가 있다.[2][15]이 과정에서 E의 역할이 잘 특징지어지지는 않았지만, E가 바이러스 싹트기 또는 디시션에 관여하고 있을 가능성이 높다.[2][4][15]
비로포린
그것의 펜트 아메리카 상태에서, E는 양이온 선택적 이온 채널을 형성하며, 바이로포린으로서 기능할 가능성이 있다.[5]이로 인해 이온 항진증을 교란하고, 막 투과성을 변경하며, 숙주세포의 pH를 변조하여 바이러스 방출을 촉진할 수 있다.[2][4]비로포린으로서의 E 단백질의 역할은 병원체 발생에 관여하는 것으로 보이며, 인플람마솜의 활성화와 관련이 있을 수 있다.[3][16]사스-CoV에서는 E의 이온 채널 기능을 교란시키는 돌연변이는 바이러스성 성장에 거의 영향을 미치지 않는데도 동물 모델에서 병생성이 감쇠되는 결과를 초래한다.[10]
숙주 단백질과의 상호작용
E와 숙주세포의 단백질-단백질 상호작용은 사스-CoV에 가장 잘 설명되어 있으며 C-단자 PDZ 도메인 바인딩 모티브를 통해 발생한다.사스-CoV E 단백질은 5개의 숙주세포 단백질인 Bcl-xL, PALS1, 싱테닌, 나트륨/칼륨(Na+/K+) ATPase α-1 서브유닛, 스토마틴과 상호작용하는 것으로 보고되었다.[2]PALS1과의 상호작용은 엄격한 결합의 결과적 붕괴를 통한 병원생식과 관련될 수 있다.[3][10]이러한 상호작용은 SARS-CoV-2에서도 확인되었다.[17]
진화 및 보존
모든 코로나바이러스군에서 E단백질 순서는 잘 보존되지 않으며 시퀀스 정체성이 30% [12]미만에 이른다.쥐 간염 바이러스에 대한 실험실 실험에서, 서로 다른 그룹의 코로나바이러스의 E 단백질을 대체하는 것은 실행 가능한 바이러스를 생산할 수 있으며, 기능 E 단백질에서 상당한 시퀀스 다양성이 용인될 수 있음을 시사한다.[18]사스-CoV-2 E단백질은 사스-CoV와 매우 유사하며, 3개의 대체제와 1개의 삭제제가 있다.[4]SARS-CoV-2 시퀀스에 대한 연구는 E가 다른 구조 단백질에 비해 상대적으로 천천히 진화하고 있음을 보여준다.[19]
참조
- ^ Solodovnikov, Alexey; Arkhipova, Valeria (2021-07-29). "Достоверно красиво: как мы сделали 3D-модель SARS-CoV-2" [Truly beautiful: how we made the SARS-CoV-2 3D model] (in Russian). N+1. Archived from the original on 2021-07-30. Retrieved 30 July 2021.
- ^ a b c d e f g h i j k l m n o p q r s t Schoeman, Dewald; Fielding, Burtram C. (December 2019). "Coronavirus envelope protein: current knowledge". Virology Journal. 16 (1): 69. doi:10.1186/s12985-019-1182-0. PMC 6537279. PMID 31133031.
- ^ a b c d e Schoeman, Dewald; Fielding, Burtram C. (2020-09-03). "Is There a Link Between the Pathogenic Human Coronavirus Envelope Protein and Immunopathology? A Review of the Literature". Frontiers in Microbiology. 11: 2086. doi:10.3389/fmicb.2020.02086. PMC 7496634. PMID 33013759.
- ^ a b c d e f g h Cao, Yipeng; Yang, Rui; Lee, Imshik; Zhang, Wenwen; Sun, Jiana; Wang, Wei; Meng, Xiangfei (June 2021). "Characterization of the SARS‐CoV ‐2 E Protein: Sequence, Structure, Viroporin, and Inhibitors". Protein Science. 30 (6): 1114–1130. doi:10.1002/pro.4075. ISSN 0961-8368. PMC 8138525. PMID 33813796.
- ^ a b c d e f g h i Mandala, Venkata S.; McKay, Matthew J.; Shcherbakov, Alexander A.; Dregni, Aurelio J.; Kolocouris, Antonios; Hong, Mei (December 2020). "Structure and drug binding of the SARS-CoV-2 envelope protein transmembrane domain in lipid bilayers". Nature Structural & Molecular Biology. 27 (12): 1202–1208. doi:10.1038/s41594-020-00536-8. PMC 7718435. PMID 33177698.
- ^ Liu, D. X.; Yuan, Q.; Liao, Y. (August 2007). "Coronavirus envelope protein: A small membrane protein with multiple functions". Cellular and Molecular Life Sciences. 64 (16): 2043–2048. doi:10.1007/s00018-007-7103-1. PMC 7079843. PMID 17530462.
- ^ Surya, Wahyu; Li, Yan; Torres, Jaume (June 2018). "Structural model of the SARS coronavirus E channel in LMPG micelles". Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6): 1309–1317. doi:10.1016/j.bbamem.2018.02.017. PMC 7094280. PMID 29474890.
- ^ a b c Fung, To Sing; Liu, Ding Xiang (June 2018). "Post-translational modifications of coronavirus proteins: roles and function". Future Virology. 13 (6): 405–430. doi:10.2217/fvl-2018-0008. PMC 7080180. PMID 32201497.
- ^ Duart, Gerard; García-Murria, Mª Jesús; Grau, Brayan; Acosta-Cáceres, José M.; Martínez-Gil, Luis; Mingarro, Ismael (September 2020). "SARS-CoV-2 envelope protein topology in eukaryotic membranes". Open Biology. 10 (9): 200209. doi:10.1098/rsob.200209. PMC 7536074. PMID 32898469.
- ^ a b c DeDiego, Marta L.; Nieto-Torres, Jose L.; Jimenez-Guardeño, Jose M.; Regla-Nava, Jose A.; Castaño-Rodriguez, Carlos; Fernandez-Delgado, Raul; Usera, Fernando; Enjuanes, Luis (December 2014). "Coronavirus virulence genes with main focus on SARS-CoV envelope gene". Virus Research. 194: 124–137. doi:10.1016/j.virusres.2014.07.024. PMC 4261026. PMID 25093995.
- ^ Kuo, Lili; Masters, Paul S. (2003-04-15). "The Small Envelope Protein E Is Not Essential for Murine Coronavirus Replication". Journal of Virology. 77 (8): 4597–4608. doi:10.1128/JVI.77.8.4597-4608.2003. PMC 152126. PMID 12663766.
- ^ a b Ruch, Travis R.; Machamer, Carolyn E. (2012-03-08). "The Coronavirus E Protein: Assembly and Beyond". Viruses. 4 (3): 363–382. doi:10.3390/v4030363. PMC 3347032. PMID 22590676.
- ^ Goodsell, David S.; Voigt, Maria; Zardecki, Christine; Burley, Stephen K. (6 August 2020). "Integrative illustration for coronavirus outreach". PLOS Biology. 18 (8): e3000815. doi:10.1371/journal.pbio.3000815. PMC 7433897. PMID 32760062.
- ^ Siu, Y. L.; Teoh, K. T.; Lo, J.; Chan, C. M.; Kien, F.; Escriou, N.; Tsao, S. W.; Nicholls, J. M.; Altmeyer, R.; Peiris, J. S. M.; Bruzzone, R.; Nal, B. (15 November 2008). "The M, E, and N Structural Proteins of the Severe Acute Respiratory Syndrome Coronavirus Are Required for Efficient Assembly, Trafficking, and Release of Virus-Like Particles". Journal of Virology. 82 (22): 11318–11330. doi:10.1128/JVI.01052-08. PMC 2573274. PMID 18753196.
- ^ a b Alsaadi, Entedar A J; Jones, Ian M (April 2019). "Membrane binding proteins of coronaviruses". Future Virology. 14 (4): 275–286. doi:10.2217/fvl-2018-0144. PMC 7079996. PMID 32201500.
- ^ Nieto-Torres, Jose L.; DeDiego, Marta L.; Verdiá-Báguena, Carmina; Jimenez-Guardeño, Jose M.; Regla-Nava, Jose A.; Fernandez-Delgado, Raul; Castaño-Rodriguez, Carlos; Alcaraz, Antonio; Torres, Jaume; Aguilella, Vicente M.; Enjuanes, Luis (1 May 2014). "Severe Acute Respiratory Syndrome Coronavirus Envelope Protein Ion Channel Activity Promotes Virus Fitness and Pathogenesis". PLOS Pathogens. 10 (5): e1004077. doi:10.1371/journal.ppat.1004077. PMC 4006877. PMID 24788150.
- ^ a b Chai, Jin; Cai, Yuanheng; Pang, Changxu; Wang, Liguo; McSweeney, Sean; Shanklin, John; Liu, Qun (December 2021). "Structural basis for SARS-CoV-2 envelope protein recognition of human cell junction protein PALS1". Nature Communications. 12 (1): 3433. Bibcode:2021NatCo..12.3433C. doi:10.1038/s41467-021-23533-x. PMC 8187709. PMID 34103506.
- ^ Kuo, Lili; Hurst, Kelley R.; Masters, Paul S. (March 2007). "Exceptional Flexibility in the Sequence Requirements for Coronavirus Small Envelope Protein Function". Journal of Virology. 81 (5): 2249–2262. doi:10.1128/JVI.01577-06. PMC 1865940. PMID 17182690.
- ^ Rahman, M. Shaminur; Hoque, M. Nazmul; Islam, M. Rafiul; Islam, Israt; Mishu, Israt Dilruba; Rahaman, Md. Mizanur; Sultana, Munawar; Hossain, M. Anwar (March 2021). "Mutational insights into the envelope protein of SARS-CoV-2". Gene Reports. 22: 100997. doi:10.1016/j.genrep.2020.100997. PMC 7723457. PMID 33319124.