중립 네트워크(진화)
Neutral network (evolution)중립적 네트워크는 기능이나 적합성이 동등한 점 돌연변이에 의해 모두 관련된 유전자의 집합이다.[1] 각 노드는 유전자 서열을 나타내고 각 선은 두 시퀀스를 연결하는 돌연변이를 나타낸다. 중립적 네트워크는 피트니스 환경에서 높고 평평한 고지로 생각할 수 있다. 중립 진화 동안, 유전자는 중립 네트워크를 통해 무작위로 이동할 수 있으며, 강력성과 진화 가능성에 대한 결과를 가져올 수 있는 시퀀스 공간의 영역을 횡단할 수 있다.
유전적 및 분자적 원인
단백질은 돌연변이에 강하기 때문에 중립적 네트워크는 피트니스 환경에 존재한다. 이것은 중립 돌연변이에 의해 연결된 동등한 기능의 유전자의 네트워크를 확장시킨다.[2][3] 단백질은 많은 염기서열이 매우 유사한 구조 접힘으로 접힐 수 있기 때문에 돌연변이에 내성이 있다.[4] 단백질은 컨포머가 펼치거나 잘못 접힌 상태보다 에너지가 낮기 때문에 제한된 자연 순응의 앙상블을 채택한다(접힘의 ΔG.[5][6] 이것은 협력적 상호작용의 분산된 내부 네트워크(수소성, 극성 및 공밸런트)에 의해 달성된다.[7] 단백질 구조 건전성은 단일 돌연변이가 기능을 손상시킬 정도로 충분히 파괴적이기 때문에 발생한다. 부분적으로 접힌 단백질이 결합하여 크고 반복적이며 불용성인 단백질 섬유질과 덩어리를 형성할 수 있기 때문에 단백질도 집적을[8] 피하기 위해 진화해 왔다.[9] 단백질이 구조에서 집적성 베타시트 모티브의 노출을 줄이기 위해 부정적인 디자인 특징을 보인다는 증거가 있다.[10] 또한 대부분의 포인트 돌연변이가 유사한 아미노산(보수적)으로 이어질 수 있도록 유전자 코드 자체가 최적화될 수 있다는 일부 증거가 있다.[11][12] 이러한 요소들은 함께 많은 수의 중립적 돌연변이와 거의 중립적인 돌연변이를 포함하는 돌연변이의 건강 효과의 분포를 만든다.[13]
진화
중립적 네트워크는 동일한 기능을 가진 시퀀스 공간의 시퀀스 중 일부분이며, 따라서 피트니스 풍경에서 넓고 평평한 고원을 형성한다. 따라서 중립 진화는 중성 네트워크를 통해 시퀀스 노드 집합에서 다른 시퀀스 노드 클러스터로 확산되는 모집단으로 시각화할 수 있다. 진화의 대다수가 중립적이라고 생각되기 때문에,[14][15] 유전자 변화의 많은 부분은 확장 중립적 네트워크를 통한 움직임이다.
강건함
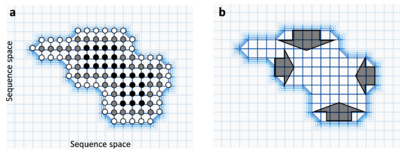
어떤 시퀀스가 더 중립적인 이웃을 가질수록, 돌연변이는 단순히 그것을 동일한 기능적 시퀀스로 중성적으로 변환할 가능성이 더 높기 때문에 그것은 더 강력한 돌연변이가 된다.[1] 실제로 중립적 네트워크 내에서 서로 다른 시퀀스의 중립적 이웃의 수 사이에 큰 차이가 있는 경우, 인구는 이러한 강력한 시퀀스를 향해 진화할 것으로 예측된다. 이것은 때때로 우회중립이라고 불리며, 피트니스 풍경에서 절벽에서 떨어진 인구 이동을 나타낸다.[16]
실리코 모델 외에도, 이러한 과정은 사이토크롬 P450s와[18] B-락타마제의 실험적인 진화에 의해 확인되기 시작하고 있다.[17][19]
진화 가능성
유전적 표류와 선택 사이의 상호작용에 대한 관심은 1930년대 시프트 밸런스 이론이 어떤 상황에서는 유전적 표류가 나중에 적응적 진화를 촉진할 수 있다고 제안했을 때부터 있어 왔다.[20] 이론의 구체적 내용은 대체로 신빙성이 떨어졌지만,[21] 표류가 비록 현재 기능에 중립적이긴 하지만 새로운 기능(진화성)의 선택에 영향을 미칠 수 있는 암호적 변동을 발생시킬 수 있다는 가능성에 주목했다.[22]
정의에 따르면, 중립 네트워크의 모든 유전자는 동등한 기능을 가지고 있지만, 어떤 유전자는 새로운 기능에 대한 적응적 진화의 출발점이 될 수 있는 비규칙적인 활동을 보일 수 있다.[23][24] 시퀀스 공간의 관점에서, 현재의 이론들은 두 가지 다른 활동에 대한 중립적 네트워크가 겹칠 경우, 중성적으로 진화하는 인구가 두 번째 활동에 접근할 수 있는 첫 번째 활동의 중립적 네트워크의 지역으로 확산될 수 있다고 예측한다.[25] 이것은 활동 사이의 거리가 중성적으로 진화하는 인구가 커버할 수 있는 거리보다 작을 때에만 해당될 것이다. 두 네트워크의 상호 접속 정도에 따라 시퀀스 공간에서 비규칙 활동에 대한 암호적 변화가 얼마나 일반적인지를 결정할 것이다.[26]
수학적 프레임워크
중립적인 돌연변이가 아마도 널리 퍼졌을 것이라는 사실은 1965년 프리즈와 요시다에 의해 제안되었다.[27] 이후 기무라 모투는 1968년[28] 킹과 쥬크스가 독자적으로 유사한 이론(1969년)을 제안하면서 중립 진화론을 구체화했다.[29] 기무라는 모집단의 뉴클레오티드 대체율(즉, 게놈 내에서 1개의 염기쌍 대체물이 발생하는 평균 시간)을 계산한 결과 ~ 1.8년이라는 것을 알아냈다. Haldane의 공식에 따르면 이러한 높은 비율은 어떠한 포유류 인구에게도 용인되지 않을 것이다. 따라서 그는 포유류에서 DNA의 중성(또는 거의 중성) 뉴클레오티드 대체 돌연변이가 지배해야 한다고 결론지었다. 그는 그러한 돌연변이가 매년 대략 0-5의 비율로 일어나고 있다고 계산했다.
후년에, RNA를 DNA의 전구 분자로 배치하는 새로운 패러다임이 나타났다. 초기 분자 원리는 크릭에 의해 1968년에 제시되었고,[30] 현재 RNA 세계 가설로 알려져 있다.[31] DNA는 주로 완전한 염기쌍의 이중 나선형으로 발견되는 반면 생물학적 RNA는 단일 좌초되어 복잡한 염기쌍 상호작용을 보이는 경우가 많다. 이것들은 수소 결합을 형성하는 능력이 증가했기 때문인데, 이 사실은 리보스 설탕에 여분의 히드록실 그룹이 존재한다는 데서 기인한다.
1970년대에 스타인과 M. 워터맨은 RNA 2차 구조물의 결합을 위한 기초를 다졌다.[32] Waterman은 RNA 이차 구조와 그 관련 성질에 대한 첫 번째 그래프 이론적 설명을 했고, 이를 사용하여 효율적인 최소 자유 에너지(MFE) 접힘 알고리즘을 만들었다.[33] RNA 이차 구조는 상단 반면에서 비교차 호로 표현되는 그것의 왓슨-크릭 베이스 쌍으로 정점을 표시한 N 위에 다이어그램으로 볼 수 있다. 따라서, 2차 구조는 그것의 암묵적인 베이스 페어링 제약조건과 호환되는 많은 시퀀스를 가진 비계다. 이후 스미스와 워터맨은 로컬 시퀀스 정렬을 수행하는 알고리즘을 개발했다.[34] RNA 2차 구조에 대한 또 다른 예측 알고리즘은 누시노프[35] 누시노프의 알고리즘이 평면 그래프 최적화 문제로서 두 문자 알파벳에 걸친 접기 문제를 기술했는데, 여기서 최대화할 수량은 시퀀스 문자열의 일치 횟수다.
1980년이 다가오는 해, Howell 등은 D 동안 시퀀스의[36] 모든 접힘의 생성 함수를 계산했다. Sankoff(1985)는 유한 염기서열의 정렬, RNA 이차 구조(폴딩)의 예측, 식물유전 트리의 원자서열의 재구성을 위한 알고리즘을 기술했다.[37] 이후 워터맨과 템플(1986)은 일반 RNA 이차 구조를 예측하기 위한 다항 시간 동적 프로그래밍(DP) 알고리즘을 생성하였다.[38] 1990년에 John McCaskill은 RNA 이차 구조의 전체 평형 파티션 함수를 계산하기 위한 다항식 시간 DP 알고리즘을 제시했다.[39]
M. Zuker, Nussinov 외 연구진, [35]Smith 및 Waterman[34], Studnicka 외 연구진 등에 기초한 MFE RNA 2차 구조물의[40] 계산 알고리즘을 구현했다.[41] 나중에 L. 호파커([42]et al., 1994)는 MFE 폴딩과 파티션 함수의 계산을 통합한 소프트웨어 패키지인 The Vienna RNA 패키지와 베이스 페어링 확률을 제시했다.
피터 슈스터와 W. 폰타나(1994)는 구조 지도(genotype-phoptype)에 초점을 시퀀스로 이동시켰다. 그들은 같은 구조를 공유하는 RNA 시퀀스가 시퀀스 공간에 무작위로 분포한다는 계산 증거를 만들기 위해 역 폴딩 알고리즘을 사용했다. 그들은 단지 몇 개의 돌연변이만으로 임의의 순서에서 공통적인 구조물에 도달할 수 있다는 것을 관찰했다. 이 두 가지 사실은 그들이 같은 구조로 접히는 가장 가까운 이웃 돌연변이들의 중립적 네트워크에 의해 시퀀스 공간이 스며들어 있는 것 같다는 결론을 내리게 한다.[43]
1997년에 C. 레이디스 스타들러와 슈스터는 RNA 이차 구조의 중립적 네트워크에 대한 연구와 모델링을 위한 수학적 기초를 마련하였다. 그들은 무작위 그래프 모델을 사용하여 중립 이웃의 일부인 ,에 의해 매개변수화된 구성 공간에서 무작위 하위 그래프를 연결하기 위한 임계값의 존재를 증명했다. 그들은 중립적으로 가장 가까운 이웃의 비율이 임계값인 **을 초과할 경우 네트워크가 연결되고 시퀀스 공간을 초과한다는 것을 보여주었다. 이 임계값 아래로 네트워크는 가장 큰 거대 구성요소와 몇 개의 작은 구성요소로 분할된다. 이 분석의 주요 결과는 슈스터의 형상 공간 추측뿐만 아니라 중립적 네트워크에 대한 밀도와 연결성에 대한 임계값 함수와 관련이 있다.[43][44][45]
참고 항목
참조
- ^ a b van Nimwegen, E; Crutchfield, JP; Huynen, M (Aug 17, 1999). "Neutral evolution of mutational robustness". Proceedings of the National Academy of Sciences of the United States of America. 96 (17): 9716–20. arXiv:adap-org/9903006. Bibcode:1999PNAS...96.9716V. doi:10.1073/pnas.96.17.9716. PMC 22276. PMID 10449760.
- ^ Taverna, DM; Goldstein, RA (Jan 18, 2002). "Why are proteins so robust to site mutations?". Journal of Molecular Biology. 315 (3): 479–84. doi:10.1006/jmbi.2001.5226. PMID 11786027.
- ^ Tokuriki, N; Tawfik, DS (Oct 2009). "Stability effects of mutations and protein evolvability". Current Opinion in Structural Biology. 19 (5): 596–604. doi:10.1016/j.sbi.2009.08.003. PMID 19765975.
- ^ Meyerguz, L; Kleinberg, J; Elber, R (Jul 10, 2007). "The network of sequence flow between protein structures". Proceedings of the National Academy of Sciences of the United States of America. 104 (28): 11627–32. Bibcode:2007PNAS..10411627M. doi:10.1073/pnas.0701393104. PMC 1913895. PMID 17596339.
- ^ Karplus, M (Jun 17, 2011). "Behind the folding funnel diagram". Nature Chemical Biology. 7 (7): 401–4. doi:10.1038/nchembio.565. PMID 21685880.
- ^ Tokuriki, N; Stricher, F; Schymkowitz, J; Serrano, L; Tawfik, DS (Jun 22, 2007). "The stability effects of protein mutations appear to be universally distributed". Journal of Molecular Biology. 369 (5): 1318–32. doi:10.1016/j.jmb.2007.03.069. PMID 17482644.
- ^ Shakhnovich, BE; Deeds, E; Delisi, C; Shakhnovich, E (Mar 2005). "Protein structure and evolutionary history determine sequence space topology". Genome Research. 15 (3): 385–92. arXiv:q-bio/0404040. doi:10.1101/gr.3133605. PMC 551565. PMID 15741509.
- ^ Monsellier, E; Chiti, F (Aug 2007). "Prevention of amyloid-like aggregation as a driving force of protein evolution". EMBO Reports. 8 (8): 737–42. doi:10.1038/sj.embor.7401034. PMC 1978086. PMID 17668004.
- ^ Fink, AL (1998). "Protein aggregation: folding aggregates, inclusion bodies and amyloid". Folding & Design. 3 (1): R9-23. doi:10.1016/s1359-0278(98)00002-9. PMID 9502314.
- ^ Richardson, JS; Richardson, DC (Mar 5, 2002). "Natural beta-sheet proteins use negative design to avoid edge-to-edge aggregation". Proceedings of the National Academy of Sciences of the United States of America. 99 (5): 2754–9. Bibcode:2002PNAS...99.2754R. doi:10.1073/pnas.052706099. PMC 122420. PMID 11880627.
- ^ Müller, MM; Allison, JR; Hongdilokkul, N; Gaillon, L; Kast, P; van Gunsteren, WF; Marlière, P; Hilvert, D (2013). "Directed evolution of a model primordial enzyme provides insights into the development of the genetic code". PLOS Genetics. 9 (1): e1003187. doi:10.1371/journal.pgen.1003187. PMC 3536711. PMID 23300488.
- ^ Firnberg, E; Ostermeier, M (Aug 2013). "The genetic code constrains yet facilitates Darwinian evolution". Nucleic Acids Research. 41 (15): 7420–8. doi:10.1093/nar/gkt536. PMC 3753648. PMID 23754851.
- ^ Hietpas, RT; Jensen, JD; Bolon, DN (May 10, 2011). "Experimental illumination of a fitness landscape". Proceedings of the National Academy of Sciences of the United States of America. 108 (19): 7896–901. Bibcode:2011PNAS..108.7896H. doi:10.1073/pnas.1016024108. PMC 3093508. PMID 21464309.
- ^ 키무라, 모토. (1983). 분자 진화의 중립 이론. 케임브리지
- ^ Kimura, M. (1968). "Evolutionary Rate at the Molecular Level". Nature. 217 (5129): 624–6. Bibcode:1968Natur.217..624K. doi:10.1038/217624a0. PMID 5637732. S2CID 4161261.
- ^ Proulx, SR; Adler, FR (Jul 2010). "The standard of neutrality: still flapping in the breeze?". Journal of Evolutionary Biology. 23 (7): 1339–50. doi:10.1111/j.1420-9101.2010.02006.x. PMID 20492093. S2CID 7774510.
- ^ van Nimwegen E.; Crutchfield J. P.; Huynen M. (1999). "Neutral evolution of mutational robustness". PNAS. 96 (17): 9716–9720. Bibcode:1999PNAS...96.9716V. doi:10.1073/pnas.96.17.9716. PMC 22276. PMID 10449760.
- ^ Bloom, JD; Lu, Z; Chen, D; Raval, A; Venturelli, OS; Arnold, FH (Jul 17, 2007). "Evolution favors protein mutational robustness in sufficiently large populations". BMC Biology. 5: 29. arXiv:0704.1885. doi:10.1186/1741-7007-5-29. PMC 1995189. PMID 17640347.
- ^ Bershtein, Shimon; Goldin, Korina; Tawfik, Dan S. (June 2008). "Intense Neutral Drifts Yield Robust and Evolvable Consensus Proteins". Journal of Molecular Biology. 379 (5): 1029–1044. doi:10.1016/j.jmb.2008.04.024. PMID 18495157.
- ^ Wright, Sewel (1932). "The roles of mutation, inbreeding, crossbreeding and selection in evolution". Proceedings of the Sixth International Congress of Genetics: 356–366.
- ^ Coyne, JA; Barton NH; Turelli M (1997). "Perspective: a critique of Sewall Wright's shifting balance theory of evolution". Evolution. 51 (3): 643–671. doi:10.2307/2411143. JSTOR 2411143. PMID 28568586.
- ^ Davies, E. K. (10 September 1999). "High Frequency of Cryptic Deleterious Mutations in Caenorhabditis elegans". Science. 285 (5434): 1748–1751. doi:10.1126/science.285.5434.1748. PMID 10481013.
- ^ Masel, J (Mar 2006). "Cryptic genetic variation is enriched for potential adaptations". Genetics. 172 (3): 1985–91. doi:10.1534/genetics.105.051649. PMC 1456269. PMID 16387877.
- ^ Hayden, EJ; Ferrada, E; Wagner, A (Jun 2, 2011). "Cryptic genetic variation promotes rapid evolutionary adaptation in an RNA enzyme" (PDF). Nature. 474 (7349): 92–5. doi:10.1038/nature10083. PMID 21637259. S2CID 4390213.
- ^ Bornberg-Bauer, E; Huylmans, AK; Sikosek, T (Jun 2010). "How do new proteins arise?". Current Opinion in Structural Biology. 20 (3): 390–6. doi:10.1016/j.sbi.2010.02.005. PMID 20347587.
- ^ Wagner, Andreas (2011-07-14). The origins of evolutionary innovations : a theory of transformative change in living systems. Oxford [etc.]: Oxford University Press. ISBN 978-0199692590.
- ^ 프리즈, E., 요시다 A. (1965년) 진화에서 돌연변이의 역할. 브라이슨과 H J 보겔에서, 에드. 진화하는 유전자와 단백질, 341-55페이지. 학구적인, 뉴욕.
- ^ Kimura, M (1968). "Evolutionary Rate at the Molecular Level". Nature. 217 (5129): 624–6. Bibcode:1968Natur.217..624K. doi:10.1038/217624a0. PMID 5637732. S2CID 4161261.
- ^ King, JL; Jukes, TH (1969). "Non-Darwinian Evolution". Science. 164 (3881): 788–97. Bibcode:1969Sci...164..788L. doi:10.1126/science.164.3881.788. PMID 5767777.
- ^ Crick, FH (1968). "The origin of the genetic code". Journal of Molecular Biology. 38 (3): 367–79. doi:10.1016/0022-2836(68)90392-6. PMID 4887876.
- ^ Robertson, MP; Joyce, GF (2012). "The origins of the RNA world". Cold Spring Harbor Perspectives in Biology. 4 (5): a003608. doi:10.1101/cshperspect.a003608. PMC 3331698. PMID 20739415.
- ^ Stein, P.R.; Waterman, M.S. (1978). "On some new sequences generalizing the Catalan and Motzkin numbers". Discrete Math. 26 (3): 261–272. doi:10.1016/0012-365x(79)90033-5.
- ^ MS 워터맨 단일 좌초 핵산의 이차 구조 조언. 수학. I (공급), 1:167–212, 1978.
- ^ a b Smith, Temple F.; Waterman, Michael S. (1981). "Identification of common molecular subsequences". Journal of Molecular Biology. 147 (1): 195–197. doi:10.1016/0022-2836(81)90087-5. PMID 7265238.
- ^ a b Nussiniv; et al. (1978). "Algorithms for Loop Matchings". SIAM Journal on Applied Mathematics. 35 (1): 68–82. doi:10.1137/0135006. JSTOR 2101031.
- ^ Howell, J.A.; Smith, T.F.; Waterman, M.S. (1980). "Computation of generating functions for biological molecules". SIAM J. Appl. Math. 39: 119133. doi:10.1137/0139010.
- ^ Sankoff, David (October 1985). "Simultaneous Solution of the RNA Folding, Alignment and Protosequence Problems". SIAM Journal on Applied Mathematics. 45 (5): 810–825. doi:10.1137/0145048.
- ^ Waterman, M.S.; Smith, T.F. (1986). "Rapid dynamic programming algorithms for RNA secondary structure". Adv. Appl. Math. 7 (4): 455–464. doi:10.1016/0196-8858(86)90025-4.
- ^ McCaskill, John (1990). "The Equilibrium Partition Function and Base Pair Binding Probabilities for RNA Secondary Structure". Biopolymers. 29 (6–7): 1105–19. doi:10.1002/bip.360290621. hdl:11858/00-001M-0000-0013-0DE3-9. PMID 1695107. S2CID 12629688.
- ^ Zuker, Michael; Stiegler, Patrick (1981). "Optimal Computer Folding of Large RNA Sequences Using Thermodynamics". Nucleic Acids Research. 9 (1): 133–148. doi:10.1093/nar/9.1.133. PMC 326673. PMID 6163133.
- ^ Studnicka, Gary M.; Rahn, Georgia M.; Cummings, Ian W.; Salser, Winston A. (1978-09-01). "Computer method for predicting the secondary structure of single-stranded RNA". Nucleic Acids Research. 5 (9): 3365–3388. doi:10.1093/nar/5.9.3365. ISSN 0305-1048. PMC 342256. PMID 100768.
- ^ Hofacker, I.L.; Fontana, W.; Stadler, P.F.; et al. (1994). "Fast folding and comparison of RNA secondary structures". Monatsh Chem. 125 (2): 167. doi:10.1007/BF00818163. S2CID 19344304.
- ^ a b Schuster, Peter; Fontana, Walter; Stadler, Peter F.; Hofacker, Ivo L. (1994-03-22). "From Sequences to Shapes and Back: A Case Study in RNA Secondary Structures". Proceedings of the Royal Society of London B: Biological Sciences. 255 (1344): 279–284. Bibcode:1994RSPSB.255..279S. doi:10.1098/rspb.1994.0040. ISSN 0962-8452. PMID 7517565. S2CID 12021473.
- ^ "Neutral networks of RNA Secondary Structures" (PDF).
- ^ Hofacker, Ivo L.; Schuster, Peter; Stadler, Peter F. (1998). "Combinatorics of RNA secondary structures". Discrete Applied Mathematics. 88 (1–3): 207–237. doi:10.1016/s0166-218x(98)00073-0.



