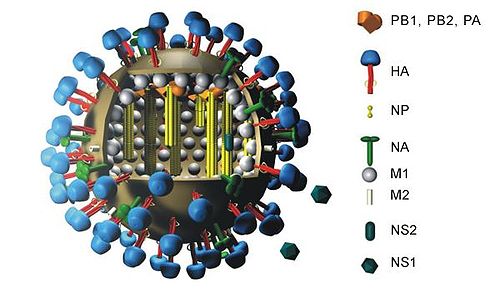
H5N1 유전자 구조
H5N1 genetic structure |
H5N1 유전자 구조는 H5N1 바이러스의 RNA 분자 구조이다.
H5N1은 인플루엔자 A 바이러스의 아형이다.전문가들은 이것이 사람에서 사람으로 쉽게 전염되는 형태로 변이될 수 있다고 믿고 있다.이러한 돌연변이가 발생하면 H5N1 아형으로 남거나 H3N2의 홍콩 독감 변종으로 진화했을 때 H2N2와 같이 아형이 바뀔 수 있다.
H5N1은 항원 표류를 통해 수십 개의 고병원성 변종으로 변이되었지만[1], 현재는 모두 조류 인플루엔자 바이러스 H5N1의 유전자형 Z에 속한다.유전자형 Z는 1996년 중국에서,[2] 1997년 홍콩에서 조류에서 처음 나타난 H5N1의 초기 고병원성 유전자형에서 2002년 재조정을 통해 나타났다."인간의 감염으로 인한 H5N1 바이러스와 2004년과 2005년에 격리된 밀접하게 연관된 조류 바이러스는 단일 유전자형에 속하며 종종 유전자형 [1]Z로 불린다."
이러한 인간 감염은 홍콩의 가금류 개체에서 H5N1 인플루엔자의 유행병과 동시에 발생했다.이 범동물성(특히 넓은 지역에 걸쳐 많은 종의 동물에게 영향을 미치는 질병)의 발생은 그 지역 내의 전체 국내 가금류 개체수가 죽임으로써 중단되었다.H5N1이라는 이름은 바이러스에 존재하는 표면 항원의 하위 유형인 헤마글루티닌 5형과 뉴라미니다아제 1형을 나타냅니다.
H5N1의 유전자형 Z는 현재 H5N1의 지배적인 유전자형이다.유전자형 Z는 동남아시아에 사는 조류들의 풍토병으로 장기적인 대유행의 위협을 나타낸다.
인플루엔자 A 바이러스는 8개의 개별 RNA 분자에 11개의 유전자를 가지고 있다[1].
- PB2(폴리머라아제 기본 2)
- PB1(폴리머라아제 기본 1)
- PB1-F2(PB1 유전자의 5' 말단 부근에 있는 대체 개방 판독 프레임)
- PA(중합효소산)
- HA(혈구응집소)
- NP(핵단백질)
- NA(뉴라미니다아제)
- M1 및 M2(매트릭스)
- NS1(비구조)
- NEP/NS2(vRNP의 핵 수출)
가장 중요한 RNA 분자 중 두 가지는 HA와 PB1이다.HA는 투과성에 특히 중요한 표면 항원을 생성합니다.PB1은 바이러스성 중합효소 분자를 생성하는데, 이 분자는 독성 물질에서 특히 중요하다.
HA RNA 분자는 인플루엔자 바이러스의 표면에서 발견되는 항원성 당단백질이고 감염된 세포에 바이러스를 결합시키는 역할을 하는 헤마글루티닌을 코드하는 HA 유전자를 포함하고 있다.헤마글루티닌은 세포에 바이러스를 부착하는 기능을 하는 독감 바이러스의 표면에 스파이크를 형성한다.이 애착은 헤마글루티닌 단백질에 결합하는 항체에 의해 차단될 수 있는 과정인 독감 바이러스 유전자를 세포로 효율적으로 전달하기 위해 필요하다.
인간 독감 바이러스와 조류 독감 바이러스를 구별하는 한 가지 유전적 요인은 조류 인플루엔자 HA는 알파 2-3 시알산 수용체와 결합하고 인간 인플루엔자 HA는 알파 2-6 시알산 수용체와 결합한다는 것이다.돼지 인플루엔자 바이러스는 두 종류의 시알산 수용체와 결합하는 능력을 가지고 있다.인간은 매우 낮은 밀도로 조류형 수용체를 가지고 있고 닭은 매우 낮은 밀도로 인간형 수용체를 가지고 있다.H5N1에 감염된 사람으로부터 채취된 일부 분리주들은 위치 182, 192, 223, 226 또는 228에서 HA 돌연변이를 갖는 것이 관찰되었으며, 이러한 돌연변이는 앞서 언급한 시알산 조류 및/또는 인간 세포 표면 수용체에 대한 바이러스의 선택적 결합에 영향을 미치는 것으로 나타났다.이것들은 조류독감 바이러스를 독감 대유행 바이러스로 바꿀 수 있는 종류의 돌연변이들이다.
지난 2004년 태국에서 돌았던 조류독감 H5N1 바이러스와 2003년 와이오밍에서 회수된 인간독감 H3N2 바이러스가 실험실에서 교배한 2008년 독성 연구 결과 63개의 바이러스가 생성돼 인간과 조류독감 A형 바이러스의 다양한 조합이 가능했다.5명 중 1명은 저용량으로 생쥐에게 치명적이었다.H5N1과 가장 밀접하게 일치하는 바이러스는 헤마글루티닌(HA), 뉴라미니다아제(NA), PB1 조류독감 바이러스 RNA 분자와 함께 나머지 5개의 RNA 분자(PB2, PA, NP, M, NS)와 인간독감 바이러스의 유전자를 결합시킨 것이다.1957년 대유행과 1968년 대유행의 두 바이러스 모두 조류 독감 바이러스 PB1 유전자를 가지고 있었다.저자들은 조류독감 바이러스 PB1 유전자의 감염이 [3]재촉진을 통해 발생할 수 있는 잠재적인 독감 대유행 바이러스의 중요한 단계가 될 수 있다고 제안합니다."
PB1은 PB1 단백질과 PB1-F2 단백질을 코드합니다.PB1 단백질은 바이러스 중합효소의 중요한 성분이다.PB1-F2 단백질은 PB1 RNA 세그먼트의 대체 개방 판독 프레임에 의해 부호화되어 "아포토시스에 대한 [감작] 세포인 미토콘드리아 투과성 전이 기공 복합체의 2성분인 ANT3 및 VDCA1과 상호 작용한다. [... PB1-F2는 바이러스 병원성에 기여할 가능성이 높고 O의 중요도를 결정하는 데 중요한 역할을 가질 수 있다.유행성 인플루엔자"[4]라고 말했다.이것은 Chen 등에 의해 발견되어 [5]Nature에 보고되었다.1997년 홍콩 H5N1에서 발생한 바이러스를 비교한 결과 PB1-F2 염기서열에서 병원성과 관련된 아미노산 변화(N66S)가 1개 검출됐다.1918년 대유행 A/Brevig Mission/18 [6]바이러스의 PB1-F2 단백질에서도 동일한 아미노산 변화(N66S)가 발견됐다.
용어.
오르토믹소바이러스과에는 7개의 속(속)이 있다.
"RNA 바이러스"는 "음성 ssRNA 바이러스"를 포함하며, 핵단백질(NP 및 M) 항원의 변이에 따라 분류되는 5개의 속을 포함하는 "Orthomyxoviridae" 패밀리를 포함합니다.이들 중 하나는 "인플루엔자 바이러스"라고 불리는 단일 종으로 구성된 "인플루엔자 바이러스 A"속이다. 그 하위 유형 중 하나는 H5N1이다.
H5N1(다른 조류독감 바이러스와 마찬가지로)에는 "고병원성"(HP)과 "저병원성"(LP)이라고 불리는 변종이 있습니다.HPAI를 일으키는 조류 인플루엔자 바이러스는 매우 치명적이며 감염된 양떼의 사망률은 종종 100%에 육박한다.LPAI 바이러스는 일반적으로 독성이 낮지만, 이러한 바이러스는 HPAI 바이러스의 시조로 작용할 수 있습니다.아시아 지역 국내 조류 폐사의 원인이 되는 H5N1의 현재 변종은 HPAI 변종이다. 세계 다른 곳에서 발생하는 H5N1의 다른 변종은 덜 치명적이기 때문에 LPAI 변종으로 분류된다.현재까지 확인된 모든 HPAI 균주는 H5 및 H7 서브타입과 관련되어 있습니다.그 구별은 인간이 아닌 가금류의 병원성에 관한 것이다.보통 고병원성 조류 바이러스는 인간이나 독새에게 고병원성이 아니다.이 H5N1 변종은 많은 종들에게 치명적이라는 점에서 이례적이다.
"인플루엔자"와 "A"는 둘 다 명사 "바이러스"의 형용사로 사용될 수 있고, "인플루엔자 A 바이러스"라는 명사를 대문자화하면 고유명사 인플루엔자 A 바이러스가 되는데, 인플루엔자 A 바이러스는 명사 구가 또한 그 종의 이름을 지칭한다.
맥락
- 바이러스
바이러스는 생물학적 유기체의 세포를 감염시키는 미생물 기생충의 한 종류이다.
- 오르토믹소바이러스과
Orthomyxoviridae는 척추동물을 감염시키는 RNA 바이러스 과이다.인플루엔자를 일으키는 바이러스도 포함되어 있습니다.이 계열의 바이러스는 7~8개의 선형 음성 감지 단일 가닥 RNA 세그먼트를 포함합니다.
- 인플루엔자바이러스
"인플루엔자 바이러스"는 독감을 일으키는 오르토믹소바이러스과의 하위 집합이다.이 분류학적 범주는 계통유전학에 기초하지 않는다.
- 인플루엔자A바이러스
인플루엔자 A 바이러스는 8개의 개별 RNA 분자에 10개의 유전자를 가지고 있으며, 위의 이유로 PB2, PA, HA, NP, NA, M, NS HA, NA, NA, M으로 명명된다.(최근에 발견된 11번째 유전자인 PB1-F2는 때때로 단백질을 생성하지만 일부 인플루엔자 바이러스 분리에는 없다.)[5]인플루엔자 게놈의 이러한 분할은 동시에 [1]두 개의 다른 인플루엔자 바이러스에 감염된 숙주의 세그먼트 재보정에 의해 유전자 재조합을 촉진합니다.인플루엔자 A 바이러스는 Orthomyxoviridae의 인플루엔자 바이러스 A 속 중 유일한 종이며 음성의 단일 가닥, 분할된 RNA 바이러스입니다.
"인플루엔자 바이러스 RNA 중합효소는 바이러스 핵단백질 NP와 함께 바이러스 mRNA 합성과 [7]복제에 필요한 최소한의 보체를 형성하는 세 가지 바이러스 단백질 PB1, PB2, PA로 구성된 다기능 복합체입니다."
표면 부호화 유전자 세그먼트
- 유전자 세그먼트(RNA 분자): (HA, NA)
- HA는 인플루엔자 바이러스의 표면에서 발견되는 항원성 당단백질인 헤마글루티닌의 코드이며, 바이러스를 감염된 세포에 결합시키는 역할을 한다.헤마글루티닌은 세포에 바이러스를 부착하는 기능을 하는 독감 바이러스의 표면에 스파이크를 형성한다.이 애착은 헤마글루티닌 단백질에 결합하는 항체에 의해 차단될 수 있는 과정인 독감 바이러스 유전자를 세포로 효율적으로 전달하기 위해 필요하다.인간 독감 바이러스와 조류 독감 바이러스를 구별하는 한 가지 유전적 요인은 "조류 인플루엔자 HA는 알파 2-3 시알산 수용체를 결합하고 인간 인플루엔자 HA는 알파 2-6 시알산 수용체를 결합한다"는 것이다.돼지 인플루엔자 바이러스는 두 종류의 시알산 [8]수용체와 결합하는 능력을 가지고 있습니다."2006년 터키에서 발견된 돌연변이는 헤모글루티닌 수용체 단백질의 위치 223에 있는 아미노산 샘플의 치환을 포함한다.이 단백질은 독감 바이러스가 숙주의 세포 표면에 있는 수용체에 결합하도록 한다.이 돌연변이는 2003년 홍콩의 한 아버지와 아들에게서, 지난해 베트남에서 한 건의 치명적인 사례에서 두 번 관찰된 적이 있다.그것은 인간 수용체에 결합하는 바이러스의 능력을 증가시키고, 가금류 수용체에 대한 친화력을 감소시켜, 이 돌연변이를 가진 변종이 인간에게 [according to whom?]더 잘 적응하도록 만듭니다."위치 153에 있는 동일한 샘플의 또 다른 돌연변이는 아직 알려지지 않은 [9]효과를 가진다.HA 수용체 결합 포켓의 위치 226과 228에 있는 아미노산 잔류물은 세포 표면 수용체에 대한 결합 친화력을 결정하고 조류(시알산 -2,3-NeuAcGal) 또는 인간(시알산 -2,6-NeuAcGal) 세포 표면 수용체에 대한 바이러스의 선택적 결합에 영향을 미치는 것으로 보인다.인간 A/HK/212/03 및 A/HK/213/03 분리주체는 조류 수용체 결합과 관련된 시그니처를 유지하지만, 수용체 결합 포켓 내에는 밀접하게 관련된 A/Gs/HK/739.2/02(Z+형)[10]에도 존재하지 않는 독특한 아미노산 치환(Ser227Ile)을 가지고 있다.최근의 연구는 인간이 매우 낮은 밀도로 조류형 수용체를 가지고 있고 닭은 매우 낮은 [11]밀도로 인간형 수용체를 가지고 있다는 것을 보여준다.연구진은 "182와 192로 확인된 유전자 내 두 곳의 돌연변이가 바이러스가 조류와 인간의 [12][13]수용체 모두에 결합할 수 있게 한다는 것을 발견했다."H5N1 인플루엔자 바이러스(American Association for Hemagglutin)의 구조와 수용체 특이성(Centre for Disease Control and Prevention, 2006) 및 H5N1 인플루엔자 바이러스(American Association)의 Hemaglutinin 구조 및 수용체 특이성 참조에임스 스티븐스, 올라 블릭스트, 테런스 M텀페이, 제프리 K토번버거, 제임스 C 폴슨, 이안 에이Wilson)을 참조하십시오.
- NA는 인플루엔자 바이러스의 표면에서 발견되는 항원성 당단백질 효소인 뉴라미니다아제 코드입니다.그것은 감염된 세포에서 자손 바이러스를 방출하는 것을 돕는다.독감 치료제인 타미플루와 레렌자는 일부 변종의 뉴라미니다아제를 억제함으로써 작용한다.그들은 N2와 N9에 기반하여 개발되었습니다. "N1 단백질 형태에서, 150-루프라고 불리는 작은 부분이 반전되어, N2와 N9 단백질에는 존재하지 않는 중공의 주머니를 만듭니다. [...] 연구자들이 기존 약물이 N1 단백질과 어떻게 상호작용하는지를 보았을 때, 그들은 뉴라미니드 억제제의 존재에서 발견하였습니다.op은 N2 [14]및 N9 단백질의 것과 유사한 구조를 변경했습니다."
내부 부호화 유전자 세그먼트
- 유전자 세그먼트(RNA 분자): (M, NP, NS, PA, PB1, PB2)
유전자 세그먼트를 코드하는 매트릭스
- 두 개의 표면 단백질(헤마글루티닌 및 뉴라미니다아제)과 함께 바이러스의 캡시드(보호막)를 구성하는 매트릭스 단백질(M1 및 M2)의 M코드.동일한 RNA 세그먼트의 다른 판독 프레임을 사용하여 인코딩합니다.
유전자 세그먼트를 코드하는 핵단백질.
- NP는 핵단백질을 코드한다.
- NS: NS는 두 개의 비구조 단백질(NS1 및 NS2 - 이전에는 NEP라고 불림)을 코드합니다.인플루엔자 바이러스의 병원성은 H5N1/97 바이러스의 비구조적(NS) 유전자와 관련이 있었다.[18]
- NS1: 비구조적: 핵. 세포 RNA 수송, 스플라이싱, 번역에 미치는 영향.항인터페론 [19]단백질"동남아시아의 가금류와 물새에 유통되는 고병원성 조류 H5N1 바이러스의 NS1은 인간 대식세포에서 이러한 바이러스에 의해 유발되는 향상된 염증성 사이토카인 반응(특히 TNFa)의 원인이 될 수 있다."[4]H5N1 NS1은 위치 92에서 단일 아미노산 변화를 특징으로 한다.연구진은 글루탐산에서 아스파라긴산으로 아미노산을 변화시킴으로써 H5N1 NS1의 효과를 없앨 수 있었다.NS1 유전자의 단일 아미노산 변화는 H5N1 인플루엔자 [20]바이러스의 병원성을 크게 증가시켰다.
- NEP: "핵 수출 단백질(NEP, NS2 단백질)은 vRNP의 수출을 중개합니다."[21]
유전자 세그먼트를 코드하는 중합효소
- 바이러스 중합효소의 중요한 성분인 PA 단백질의 PA 코드.
- PB1은 PB1 단백질과 PB1-F2 단백질을 코드합니다.
- PB1 단백질은 바이러스 중합효소의 중요한 성분이다.
- PB1-F2 단백질은 PB1 RNA 세그먼트의 대체 개방 판독 프레임에 의해 부호화되어 "아포토시스에 대한 [감작] 세포인 미토콘드리아 투과성 전이 기공 복합체의 2성분인 ANT3 및 VDCA1과 상호작용한다. [...PB1-F2는 바이러스 병원성에 기여할 가능성이 높고 결정의 중요도를 가질 수 있다.대유행 [4]인플루엔자"이것은 Chen 등에 의해 발견되어 [5]Nature에 보고되었다.1997년 홍콩 H5N1에서 발생한 바이러스를 비교한 결과 PB1-F2 염기서열에서 병원성과 관련된 아미노산 변화(N66S)가 1개 검출됐다.1918년 대유행 A/Brevig Mission/18 [6]바이러스의 PB1-F2 단백질에서도 동일한 아미노산 변화(N66S)가 발견됐다.
- PB2는 바이러스 중합효소의 중요한 성분인 PB2 단백질을 코드한다.2005년 현재, 베트남에서 격리된 H5N1 인간 바이러스 중 75%가 PB2 단백질의 잔류물 627에 리신으로 구성된 돌연변이를 가지고 있으며, 이는 높은 수준의 [22]독성을 유발하는 것으로 여겨진다.H5N1까지 알려진 모든 조류 인플루엔자 바이러스는 위치 627에 글루를 가지고 있었고, 인간 인플루엔자 바이러스는 모두 리신을 가지고 있었다.2007년 현재, "EMA[EMA=유럽, 중동, 아프리카] 계층에서 3개 이상의 기질이 출현한 것은 조류 인플루엔자(H5N1)가 인간 대유행 변종으로 진화할 수 있는 여러 새로운 기회를 나타낸다.동남아시아에서 유통되는 균주와 달리, EMA 바이러스는 PB2 627K 돌연변이를 가진 전구체로부터 유래한다.이들 바이러스는 포유류에서 복제 특성이 강화될 것으로 예상되며, 실제로 EMA의 확산은 터키, 이집트, 이라크, 지부티의 인간과 독일, 오스트리아, 이라크의 고양이를 포함한 포유동물에서 빠르게 나타나고 있다.불행히도 EMA형 바이러스는 아시아에서만 볼 수 있는 변종만큼 치명적인 것으로 보입니다.2006년 중반까지 아시아 이외의 34명의 감염자 중 15명이 [23]사망했습니다.PB2–627의 Lys는 조류 H5N1 바이러스에 [24]포유류의 상·하부 호흡기에서 효율적인 성장의 이점을 부여하는 것으로 여겨진다.
돌연변이
인플루엔자 바이러스는 RNA 바이러스의 특징인 돌연변이율이 상대적으로 높다.인플루엔자 게놈의 분할은 동시에 두 개의 다른 인플루엔자 바이러스에 감염된 숙주의 세그먼트 재보정에 의해 유전자 재조합을 촉진한다.H5N1 바이러스는 돼지, 조류, 인간과 같은 숙주 유기체를 공동 감염시키는 다른 변종과 유전자를 재조합할 수 있고 인간 사이에서 쉽게 전달될 수 있는 형태로 변이시킬 수 있다.이것은 대유행을 일으킬 수 있는 많은 방법 중 하나이다.
다양한 인플루엔자 균주가 종 선택성을 보이는 능력은 주로 헤마글루티닌 유전자의 변화 때문이다.단일 아미노산 치환을 일으키는 헤마글루티닌 유전자의 유전자 돌연변이는 바이러스 헤마글루티닌 단백질이 숙주 세포의 표면에 있는 수용체에 결합하는 능력을 크게 변화시킬 수 있다.조류 H5N1 바이러스의 이러한 돌연변이는 바이러스 변종을 인간 세포를 감염시키는데 비효율적인 것에서 인간 인플루엔자 바이러스의 종류만큼 [25]인간 감염을 유발하는 데 효율적인 것으로 바꿀 수 있다.이것은 하나의 아미노산 치환으로 대유행을 일으킬 수 있다는 것을 의미하지는 않지만, 하나의 아미노산 치환으로 인해 사람에게 병원성이 없는 조류 독감 바이러스가 사람에게 병원성이 될 수 있다는 것을 의미한다.
H3N2(스위인플루엔자)는 중국 돼지들의 풍토병으로 베트남 돼지에서도 검출돼 신종 변종 출현에 대한 우려가 커지고 있다.2006년 1월 H3N2는 항바이러스제인 아만타딘과 리만타딘에 내성이 있다.H5N1과 H3N2가 재조합을 통해 유전자를 교환할 가능성은 주요 관심사다.H5N1의 재배치가 발생하면 H2N2가 H3N2의 홍콩 독감 변종으로 진화했을 때와 마찬가지로 H5N1 아형으로 남아 있거나 아형으로 전환될 수 있습니다.
H2N2와 H3N2 대유행 변종 모두 조류 인플루엔자 바이러스 RNA 세그먼트를 포함하고 있었다.1957년(H2N2)과 1968년(H3N2)의 대유행 인플루엔자 바이러스는 인간과 조류 바이러스의 재분배를 통해 분명히 발생했지만 1918년 스페인 독감을 일으킨 인플루엔자 바이러스는 전적으로 조류에서 [26]유래한 것으로 보인다.
2004년 7월, 중국 하얼빈에 있는 하얼빈 수의학 연구소의 H. 덩과 로버트 G 교수가 이끌었다. 세인트 웹스터입니다 테네시주 멤피스의 주드아동연구병원은 1999년과 2002년 사이 쥐가 중국에서 오리에게서 얻은 확인된 H5N1 변종 21개에 피폭된 실험 결과를 보고했다.그들은 "점진적으로 증가하는 병원성의 분명한 시간적 패턴"[27]을 발견했다.박사님이 보고한 결과2005년 7월 웹스터는 쥐의 병원성과 오리에 의한 바이러스 유출이 더 길어지는 것을 밝혀냈다.
아시아 계통의 HPAI A(H5N1)는 2개의 항원군으로 나뉜다."1번 구역에는 베트남, 태국, 캄보디아에서 사람과 새가 격리되고 라오스, 말레이시아에서 새가 격리됩니다.Clade 2 바이러스는 중국, 인도네시아, 일본, 한국의 조류 분리주에서 처음 발견되어 서쪽으로 중동, 유럽, 아프리카로 확산되었다.WHO에 따르면, Clade 2 바이러스는 2005년 말과 2006년 사이에 발생한 인간 H5N1 감염의 주된 원인이었다.유전자 분석 결과, 6개의 하위 분류군 2가 확인되었으며, 그 중 3개는 뚜렷한 지리적 분포를 보이며 인간 감염과 관련이 있다.지도
EMA 서브레이드에 초점을 맞춘 2007년 연구는 EMA 돌연변이를 더욱 명확히 했다."여기서 보고된 36개의 새로운 분리주들은 최근의 조류 인플루엔자(H5N1) 분리주로부터 이용 가능한 전체 유전자 배열 데이터의 양을 크게 늘렸습니다.프로젝트 전 GenBank는 2004~2006년 동안 유럽의 다른 완전한 게놈을 5개만 포함하고 중동이나 북아프리카의 게놈 전체를 포함하고 있지 않았습니다.우리의 분석 결과 몇 가지 새로운 결과가 나왔다.첫째, 모든 유럽, 중동 및 아프리카 표본은 1997년 홍콩 종족과 공통의 조상을 공유하는 다른 동시대 아시아 계통과는 다른 계통으로 분류된다.8개의 세그먼트 각각에 구축되어 있는 계통수는 그림 1과 같이 3개의 계통의 일관된 그림을 보여준다.두 개의 분지에는 베트남산 분지가 독점적으로 포함되어 있습니다.이 중 작은 분지에는 5개의 분리가 있으며 V1이라는 라벨을 붙입니다.큰 분지에는 9개의 분지가 있습니다.나머지 22개의 분리막은 유럽, 중동 및 아프리카 샘플로 구성된 EMA라는 라벨이 붙은 세 번째 명확하게 구별되는 분류군에 속합니다.다른 7개의 세그먼트의 트리는 동일한 토폴로지를 표시하며, 클래스 V1, V2 및 EMA는 각각의 케이스에서 명확하게 구분됩니다.사용 가능한 모든 완전 인플루엔자(H5N1) 게놈과 589개의 HA 염기서열을 분석한 결과 EMA군이 중화인민공화국, 인도네시아, 동남아시아에서 [23]유통되는 주요 군락과는 구별되는 것으로 나타났습니다."
HA 유전자를 기반으로 한 1,342개의 H5N1 바이러스의 유전자 트리에 대해서는 https://web.archive.org/web/20090709040039/http://who.int/csr/disease/avian_influenza/H5CompleteTree.pdf를 참조하십시오.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c 세계 보건 기구 글로벌 인플루엔자 프로그램 감시 네트워크(2005년)."H5N1바이러스의 아시아에서 진화하는".신흥 전염병. 11(10):1515–1526. doi:10.3201/eid1110.050644.PMC3366754.PMID 16318689.이 기사의 그림 1은 바이러스의 다양한 소이에서 아시아 H5N1바이러스 혈구 응집소 바이러스 유전자의 유전적 유대 관계의diagramatic 표현을 준다.
- ^ WHO (October 28, 2005). "H5N1 avian influenza: timeline" (PDF). Archived from the original (PDF) on July 27, 2011.
- ^ 캐나다 언론 기사 연구에 따르면 조류 독감과 인간 독감 바이러스의 잡종이 잘 들어맞는 것으로 나타났습니다.2008년 6월 1일 Wayback Machine에서 발행되었습니다.2008년 6월 5일 아카이브,
- ^ a b c García-Sastre A (January 2006). "Antiviral response in pandemic influenza viruses". Emerging Infect. Dis. 12 (1): 44–7. doi:10.3201/eid1201.051186. PMC 3291409. PMID 16494716.
- ^ a b c Weisan Chen; Paul A. Calvo; et al. (2001). "A novel influenza A virus mitochondrial protein that induces cell death". Nature Medicine. 7 (12): 1306–12. doi:10.1038/nm1201-1306. PMID 11726970. S2CID 723233.
- ^ a b Scientist Live Archived 2007-10-20 at the Wayback Machine article A Single Mutation in the PB1-F2 of H5N1 (HK/97) and 1918 Influenza A Viruses Contributes to Increased Virulence published October 18, 2007
- ^ M. T. Michael Lee, Konrad Bishop, Liz Medcalf, Debra Elton, Paul Digard, and Laurence Tiley (January 15, 2002). "Definition of the minimal viral components required for the initiation of unprimed RNA synthesis by influenza virus RNA polymerase". Nucleic Acids Research. 30 (2): 429–438. doi:10.1093/nar/30.2.429. PMC 99831. PMID 11788704.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Alex Greninger (July 16, 2004). "The Definition and Measurement of Dangerous Research" (PDF). CISSM Working Paper. Archived from the original (PDF) on November 8, 2006. Retrieved 2006-12-09.
- ^ Butler D (January 2006). "Alarms ring over bird flu mutations". Nature. 439 (7074): 248–9. Bibcode:2006Natur.439..248B. doi:10.1038/439248a. PMID 16421529.
- ^ H5N1 인플루엔자: 프로테아 대유행 위협
- ^ 인플루엔자 리포트 2006 온라인 북 페이지 51
- ^ CIDRAP 기사 H5N1의 인체 감염을 돕는 돌연변이 2종 발견 2006년 11월 21일 발행
- ^ 블룸버그 뉴스 기사 "두 개의 조류 독감 유전자 돌연변이가 더 빠른 인간 확산으로 이어질 수 있다" 2006년 11월 15일 발행
- ^ Scidev.net 뉴스 기사 조류독감 단백질의 '독감'은 2006년 8월 16일 발행된 더 나은 약물에 영감을 줄 수 있다.
- ^ 그 위협 전염병이 독감 발생의:.우리는 준비가 됐나요?페이지 118
- ^ 의학 미생물학에 독감 바이러스 복제, 4판 사무엘 바론에 의해 편집. 1996년장 58이다.아이 에스비엔 0-9631172-1-1.
- ^ Guan Y, Poon LL, Cheung CY, et al. (May 2004). "H5N1 influenza: a protean pandemic threat". Proc. Natl. Acad. Sci. U.S.A. 101 (21): 8156–61. Bibcode:2004PNAS..101.8156G. doi:10.1073/pnas.0402443101. PMC 419573. PMID 15148370.
- ^ Lee CW, Suarez DL, Tumpey TM, et al. (March 2005). "Characterization of highly pathogenic H5N1 avian influenza A viruses isolated from South Korea". J. Virol. 79 (6): 3692–702. doi:10.1128/JVI.79.6.3692-3702.2005. PMC 1075707. PMID 15731263.
또한, 전염병이 인플루엔자그 승객을 머신 센터 전염병 질병 연구 및, 정책 학술 의료 센터--미네소타 대학에서 2010-01-06 Archived. - ^ NS1 Inhibition의 NS1 단백질-강화된virulence/viral 발병에 바이러스 숙주 세포형 인터페론 방어 시스템 Pathobiologics 국제 무장을 해제할 수 있도록 함으로써 설명한다.
- ^ "The Definition and Measurement of Dangerous Research by Alex Greninger" (PDF). Archived from the original (PDF) on 2006-03-06. Retrieved 2006-04-10.
- ^ Paragas J, Talon J, O'Neill RE, Anderson DK, García-Sastre A, Palese P (August 2001). "Influenza B and C virus NEP (NS2) proteins possess nuclear export activities". J. Virol. 75 (16): 7375–83. doi:10.1128/JVI.75.16.7375-7383.2001. PMC 114972. PMID 11462009.
- ^ 그 위협 전염병이 독감 발생의:.우리는 준비가 됐나요?페이지 126
- ^ a b Salzberg SL, Kingsford C, Cattoli G, et al. (May 2007). "Genome analysis linking recent European and African influenza (H5N1) viruses". Emerging Infect. Dis. 13 (5): 713–8. doi:10.3201/eid1305.070013. PMC 2432181. PMID 17553249.
- ^ 2007년 10월 18일 발행된 Wayback Machine의 Scientist Live Archived 2007-10-18 기사 H5N1 인플루엔자 A 바이러스 증가
- ^ Gambaryan A, Tuzikov A, Pazynina G, Bovin N, Balish A, Klimov A (2006). "Fatal Evolution of the receptor binding phenotype of influenza A (H5) viruses". Virology. 344 (2): 432–438. doi:10.1016/j.virol.2005.08.035. PMID 16226289.
- ^ Harder, T. C.; Werner, O. (2006). "Avian Influenza". In Kamps, B. S.; Hoffman, C.; Preiser, W. (eds.). Influenza Report 2006. Paris, France: Flying Publisher. ISBN 3-924774-51-X. Retrieved 2006-04-18.
이 전자책은 지속적으로 개정되고 있으며 조류 인플루엔자에 대한 훌륭한 안내서이다. - ^ Chen H, Deng G, Li Z, et al. (July 2004). "The evolution of H5N1 influenza viruses in ducks in southern China". Proc. Natl. Acad. Sci. U.S.A. 101 (28): 10452–7. Bibcode:2004PNAS..10110452C. doi:10.1073/pnas.0403212101. PMC 478602. PMID 15235128.
- ^ Webster RG, Govorkova EA (November 2006). "H5N1 Influenza — Continuing Evolution and Spread". N. Engl. J. Med. 355 (21): 2174–7. doi:10.1056/NEJMp068205. PMID 17124014.
- ^ "WHO changes H5N1 strains for pandemic vaccines, raising concern over virus evolution". CIDRAP. August 18, 2006.
- ^ "Antigenic and genetic characteristics of H5N1 viruses and candidate H5N1 vaccine viruses developed for potential use as pre-pandemic vaccines" (PDF). WHO. August 18, 2006. Archived from the original (PDF) on August 24, 2006.
추가 정보
- 인플루엔자 보고서 2006 온라인 책.
- Ghedin E, Sengamalay NA, Shumway M, Zaborsky JFeldblyum T, Subbu V, Spiro는 DJ(2005년 11월)."인간의 독감의 대규모 염기 서열 결정 법 바이러스 유전체 진화의 동적 성격이 드러나".자연.437(7062):1162–1166. Bibcode:2005Natur.437.1162G. doi:10.1038/nature04239.PMID 16208317. 무엇이 신종 플루 게놈 시퀀싱 프로젝트에서 발견됐다의 개요를 제공한다.
- 요약 및 전문 링크 및 설명 이 조류 인플루엔자 출판물의 참고 목록은 USGS 국립 야생동물 보건 센터와 야생동물 질병 정보 노드의 협력을 통해 작성되었습니다.
- H5N1에 대한 연구 출판물 검색: Entez PubMed
- Beigel JH, Farrar J, Han AM, et al.; Writing Committee of the World Health Organization (WHO) Consultation on Human Influenza A/H5. (September 2005). "Avian influenza A (H5N1) infection in humans". N. Engl. J. Med. 353 (13): 1374–85. doi:10.1056/NEJMra052211. hdl:10722/45195. PMID 16192482.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - H5N1의 진화적 "Tree of Life" :
- 인플루엔자 바이러스 헤마글루티닌 유전자 세그먼트의 계통수입니다.아미노산은 인플루엔자 바이러스 헤마글루티닌 단백질 세그먼트 HA1의 세 계통(새, 돼지, 사람)에서 변화한다.
- 2002년 Z 유전자형을 만든 1999년부터 2004년까지 H5N1의 재배치에 의한 진화를 나타내는 나무입니다.
- 2002년 이후 항원 표류에 의한 진화를 나타내는 나무는 조류독감 바이러스 H5N1의 고병원성 유전자형 수십 종을 만들어 냈으며, 그 중 일부는 포유류에 점점 적응하고 있다.
- WHO(PDF)에는 H5N1에 대한 최신 진화적 "Tree of Life" 기사가 수록되어 있습니다.H5N1 바이러스와 후보 H5N1 백신 바이러스의 항원성 및 유전적 특성은 2006년 8월 18일 발행되었습니다.
- Hiromoto Y, Yamazaki Y, Fukushima T, et al. (May 2000). "Evolutionary characterization of the six internal genes of H5N1 human influenza A virus". J. Gen. Virol. 81 (Pt 5): 1293–303. doi:10.1099/0022-1317-81-5-1293. PMID 10769072.
- 게놈 데이터베이스 페이지는 인플루엔자 A 바이러스(A/Goose/Guangdong/1/96(H5N1) 게놈의 전체 시퀀스로 연결됩니다.