뉴클레오이드
Nucleoid핵상핵은 원핵세포 내의 불규칙한 형태의 영역으로 유전물질의 [1][2][3]전부 또는 대부분을 포함하고 있다.원핵생물의 염색체는 원형이고, 그 길이는 세포 치수에 비해 매우 크기 때문에 맞추기 위해 압축되어야 한다.진핵세포의 핵과는 대조적으로 핵막으로 둘러싸여 있지 않다.대신, 핵이드는 염색체 구조 단백질과 RNA 분자와 DNA 슈퍼코일의 도움으로 응축과 기능적 배열에 의해 형성된다.게놈의 길이는 매우 다양하며(일반적으로 적어도 몇 백만 개의 염기쌍), 세포는 게놈의 여러 복사본을 포함할 수 있습니다.
박테리아 핵이체로 알려진 고해상도 구조는 아직 없지만, 모델 유기체로 대장균의 주요 특징이 연구되었다.대장균에서 염색체 DNA는 평균적으로 음의 슈퍼코일화되어 다른 물리적 영역에 국한되어 서로 확산되는 경우가 거의 없는 플렉토네믹 루프로 접혀 있다.이러한 루프는 매크로 도메인이라고 불리는 메가베이스 크기의 영역으로 공간적으로 구성되어 있으며, 이 영역 내에서 DNA 사이트는 자주 상호 작용하지만 상호 작용은 드물다.응축되고 공간적으로 조직된 DNA는 방사상으로 세포에 갇힌 나선 타원체를 형성합니다.누조이드 내 DNA의 3D 구조는 조건에 따라 달라 보이며 유전자 발현과 연계되어 있어 핵체 구조와 유전자 전사가 밀접하게 상호의존하며 상호 작용한다.

배경
많은 박테리아에서, 염색체는 유전 정보를 반수체 형태로 암호화하는 단일 공유 폐쇄(원형) 이중 가닥 DNA 분자입니다.DNA의 크기는 [2]유기체에 따라 500에서 수천 개의 유전자를 코드하는 50만에서 수백만 개의 염기쌍(bp)으로 다양하다.염색체 DNA는 진핵세포처럼 [6]핵막으로 둘러싸여 있지 않은 핵소체라고 불리는 매우 작고 조직화된 형태로 세포에 존재한다.분리된 핵소체는 DNA 80%, 단백질 10%, RNA 10%를 [7][8]중량으로 함유하고 있다.
그램 음성 박테리아 대장균은 염색체 DNA가 핵이 되는 방법, 관련된 요소, 구조에 대해 알려진 것, 그리고 DNA 구조적 측면의 일부가 유전자 [2][3]발현에 어떻게 영향을 미치는지에 대한 핵상 연구의 모델 시스템이다.
핵상 형성에는 큰 DNA가 작은 세포 공간으로 응축되는 것과 3차원 형태의 DNA의 기능적 구성이라는 두 가지 필수적인 측면이 있다.대장균의 반수체 원형 염색체는 약 4.6 x6 10 bp로 구성되어 있습니다.만약 DNA가 B 형태로 완화된다면, 그것의 둘레는 약 1.5 밀리미터(0.332 nm x 4.6 x 106)가 될 것이다.그러나 대장균 염색체 DNA와 같은 큰 DNA 분자는 [5]현탁액에서 직선 강체 분자로 남아 있지 않다.브라운 운동은 DNA에 곡률과 굴곡을 일으킬 것이다.브라운 운동으로 인한 굴곡에 저항하여 이중 나선형 DNA가 직선을 유지하는 최대 길이는 ~50 nm 또는 150 bp로, 지속 길이라고 불립니다.따라서, 순수한 DNA는 추가적인 요인 없이 실질적으로 응축된다; 열 평형에서, 그것은 무작위 코일 [4][5]형태를 취한다.대장균 염색체 DNA의 무작위 코일은 회전 반지름(Rg = (θN a)/θ6)에서 계산한 약 523 µm의3 부피(4/3 µr3)를 차지하며, 여기서 a는 쿤 길이(2x 지속성 길이)이고, N은 DNA([5]DNA의 총 길이)로 나눈 쿤 길이 세그먼트 수이다.DNA는 이미 무작위 코일 형태로 응축되어 있지만, 여전히 1미크론 미만의 핵이드의 부피를 가정할 수는 없다.따라서, DNA의 고유한 특성은 충분하지 않다. 추가 인자는 DNA를 103(임의 코일의 부피를 핵체 부피로 나눈 값) 정도로 더 응축하도록 도와야 한다.핵양체 형성의 두 번째 필수적인 측면은 DNA의 기능적 배열이다.염색체 DNA는 응축되어 있을 뿐만 아니라 복제, 재조합, 분리, [9][10][11]전사와 같은 DNA 거래 과정과 양립할 수 있는 방식으로 기능적으로 구성되어 있다.1971년에 [7]시작된 거의 50년간의 연구는 핵이드의 최종 형태가 DNA의 계층적 구조에서 나온다는 것을 보여주었다.가장 작은 규모(1kb 이하)에서 핵소체 관련 DNA 구조단백질은 DNA를 굽힘, 루프, 브리징 또는 랩함으로써 DNA를 응축하고 조직한다.더 큰 규모(10kb 이상)에서 DNA는 초코일에 의해 유도되는 DNA의 편조 형태인 플렉토네믹 루프를 형성한다.메가베이스 규모에서 플렉토네믹 루프는 공간적으로 조직된 6개의 도메인(매크로 도메인)으로 결합되며, 다른 매크로 도메인 [12]간보다 동일한 매크로 도메인 내 DNA 사이트 간의 물리적 상호작용에 의해 정의된다.매크로 도메인 내부 및 사이에 형성된 장기 및 단거리 DNA-DNA 연결은 응축과 기능 구성에 기여한다.마지막으로 핵이드는 세로축에 [13][14][15]고응축 DNA 영역이 있는 나선 타원체이다.
응축 및 구성
뉴클레오이드관련단백질(NAPs)
진핵생물에서, 게놈 DNA는 [16][17][18]뉴클레오솜이라고 불리는 DNA-단백질 입자의 반복 배열의 형태로 응축됩니다.
뉴클레오솜은 히스톤 단백질의 팔중합체 주위를 감싸고 있는 약 146bp의 DNA로 구성됩니다.박테리아는 히스톤을 가지고 있지 않지만 넓은 의미에서 히스톤과 기능적으로 유사한 핵결합단백질(NAPs)로 언급되는 DNA결합단백질군을 가지고 있다.NAP는 매우 풍부하고 핵이드의 [19]단백질 성분의 상당한 비율을 구성한다.
NAP의 특징 중 하나는 특정(배열 특이적 또는 구조 특이적) 및 비배열 특이적 방식으로 DNA를 결합하는 능력이다.결과적으로, NAP는 이중 [20]기능 단백질이다.NAPs의 특이적 결합은 대부분 유전자 특이적 전사, DNA 복제, 재조합, 그리고 [9][10][11]복구와 관련이 있다.그 풍부함의 절정기에, 많은 NAP의 분자 수는 [20]게놈의 특정 결합 부위의 수보다 몇 배나 더 많다.따라서 NAP는 대부분 비배열 특이 모드에서 염색체 DNA에 결합하며 염색체 압축에 중요한 것은 이 모드이다.NAP의 이른바 비시퀀스 고유 바인딩이 완전히 랜덤하지는 않을 수 있습니다.배열 의존성 DNA 배치 또는 다른 NAP에 [18]의해 생성된 DNA 배치로 인해 낮은 배열 특이성 및 구조적 특이성이 있을 수 있습니다.
NAP가 DNA를 생체 내에서 응축하는 방법의 분자 메커니즘은 잘 이해되지 않지만, 광범위한 체외 연구에 따르면 NAP는 다음 메커니즘을 통해 염색체 압축에 참여하는 것으로 보인다. NAP는 DNA의 굴곡을 유도하고 안정화하며, 따라서 지속성 [20]길이를 줄임으로써 DNA 응축에 도움을 준다.NAP는 염색체의 가까운 DNA 세그먼트 또는 먼 DNA 세그먼트 간에 발생할 수 있는 브리징, 랩핑 및 번들로 DNA를 응축합니다.NAP가 염색체 압축에 참여하는 또 다른 메커니즘은 DNA에서 음의 슈퍼코일을 억제함으로써 [20]염색체의 위상 구성에 기여하는 것이다.
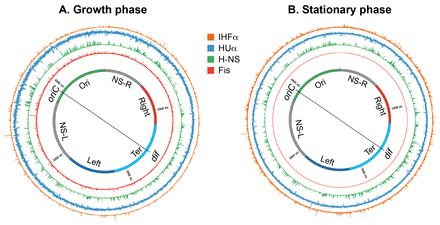
대장균에서 [20]확인된 NAP는 최소 12개이며, 그 중 가장 광범위하게 연구된 것은 HU, IHF, H-NS 및 Fis이다.이들의 풍부함, DNA 결합 특성 및 DNA 응축 및 구성에 대한 영향은 아래 [20]표에 요약되어 있습니다.
| 단백질 | 분자량(kDa) | 네이티브 기능 유닛 | 성장1 국면의 풍부함 | 정지상의1 풍부함 |
|---|---|---|---|---|
| HUα 및 HUβ | ~ 9 | 호모이합체와 헤테로 이합체 | 55,000 (23) | 30,000 (12.5) |
| IHFα 및 IHFβ | ~ 11 | 헤테로다이머 | 12,000 (5) | 55,000 (23) |
| H-NS | ~ 15 | 호모디머 | 20,000 (8) | 15,000 (6) |
| 피스 | ~ 11 | 호모디머 | 60,000 (25) | 검출할 수 없다 |
| DPS | ~ 19 | 도데카메루 | 6,000 (0.4) | 180,000 (12.5) |
1 풍부성(분자/세포) 데이터는 [21]다음과 같다.괄호 안의 숫자는 (원어민 기능 단위 수/아보가드로 수) x (1/셀 부피(리터 단위) x3 10) 공식을 사용하여 계산된 마이크로몰 농도입니다.리터 단위의 세포 부피−15(2 x 10)는 대장균 세포의 부피를 2μm로3 [21]가정하여 측정되었다.
| 단백질 | 바인딩 모티브 | 특이적 DNA결합친화성1 | 랜덤 DNA결합친화성1 |
|---|---|---|---|
| HU | DNA의[22][23] 구부러짐과 꼬임으로 정의된 구조적 모티브 | 7.5 x 10−9[24] | 4.0 x 10−7[24] |
| IHF | 와트[25] | 1.5 x 10−9[26] | 1.7 x 10−6[26] |
| H-NS | TCGATAATT[27] | 10-15 x 10−9[28] | 6 x 10−8[28] |
| 피스 | 동작하지[29] 않다 | 0.2~1.0 x 10−9[29][30] | 8.0 x 10−6[30] 이상 |
| DPS | ND | ND | 1.65 x 10−7[31] |
| MatP | GTGACRNYGTCAC[32] | 8.0 x 10−9 | ND |
| 묵베프 | ND | ND | ND |
1 결합 친화력은 몰 단위(M)의 평형 해리 상수(Kd)를 말한다. ND = 결정되지 않음
HU
대장균주 U93(HU)의 히스톤 유사 단백질은 박테리아에서 [33][34]진화적으로 보존된 단백질이다.HU는 69% 아미노산 [35]정체성을 공유하는 두 개의 서브유닛 HUα와 HUβ의 호모 및 헤테로디머로 대장균에 존재한다.히스톤 유사 단백질로 언급되지만 진핵생물에서 HU의 가까운 기능적 친척은 [36][37]히스톤이 아닌 고이동성 그룹(HMG) 단백질이다.HU는 비배열 특이 DNA 결합 단백질이다.그것은 어떤 선형 DNA에도 낮은 선호도로 결합한다.단, 이것은 구조적으로 [38][39][40][41][42][24]왜곡된 DNA에 고친화도로 우선적으로 결합한다. 왜곡된 DNA 기질의 예로는 십자형 DNA, 불룩한 DNA, 칼집, 틈새, 포크 등의 단가닥 절단을 포함한 dsDNA 등이 있다.또한 HU는 단백질 매개 DNA [43]루프를 특이적으로 결합 및 안정화한다.구조특이적 DNA결합모드에서 HU는 [22][44][23]변형에 의해 생성된 굴곡 또는 꼬임에 의해 정의된 공통의 구조적 모티브를 인식함과 동시에 인산염 [45]골격을 고정함으로써 선형 DNA에 결합한다.부위 특이적 재조합, DNA 복구, DNA 복제 개시, 유전자 [9][10][11]조절 등 HU의 특수 기능에 고선호도 구조 특이적 결합이 필요하지만, 저선호도 일반 결합은 DNA [45]응축에 관여하는 것으로 보인다.염색질-면역 침강과 DNA 염기서열결정(ChIP-Seq)에서 HU는 특정 결합 [46]이벤트를 나타내지 않는다.대신, 그것은 게놈 전체에 걸쳐 대부분 약한 비배열 특이 결합을 반영하는 균일한 결합을 나타내며,[46] 따라서 생체 내 고친화성 결합을 마스킹한다.
HU가 결핍된 균주에서, 핵이드는 DNA [47]압축에서 HU의 역할과 일관되게 "축합"된다.다음의 시험관내 연구는 HU가 어떻게 생체 내에서 DNA를 응축하고 구성할 수 있는지에 대한 가능한 메커니즘을 제시한다.HU는 휘어진 DNA에 안정적으로 결합할 뿐만 아니라 100nM 이하의 농도에서도 선형 DNA에서도 유연한 휘어짐을 유도한다.대조적으로, HU는 더 높은 생리적 관련 [45][9][10][11][47][48]농도에서 DNA에 반대되는 구조적 효과를 보여준다.굽힘이 아닌 DNA의 협착을 일으키는 단단한 핵단백질 필라멘트를 형성합니다.필라멘트는 비배열 특이적 DNA [45]결합에 의해 유발되는 HU-HU 멀티머라이제이션으로 인해 측면 및 내측 모두에서 확장 가능한 DNA 네트워크(DNA 번칭)를 더욱 형성할 수 있습니다.
세포 내에서 HU의 이러한 행동들은 어떻게 관련이 있는가?필라멘트의 형성에는 DNA에 HU의 고밀도 결합이 필요하며, 9-20 bp DNA당 1 HU 이합체입니다.그러나 세포당 30,000 HU 이합체 추정량(4600,000 bp / 30,000)[21]에 근거하여 염색체 DNA의 최대 150 bp마다 HU 이합체가 하나만 있다.이는 유연한 벤딩이 생체 내에서 발생할 가능성이 더 높다는 것을 나타냅니다.유연한 굽힘은 DNA 결합 [48][49]단백질에 의한 단일 DNA 분자의 축합을 연구할 수 있는 자기 핀셋 실험에서 보여지듯이 DNA의 지속 길이 감소로 인해 축합을 야기할 것이다.그러나, 협력성 때문에, 염색체의 일부 영역에서 단단한 필라멘트와 네트워크가 형성될 수 있다.필라멘트 형성만으로는 [48]응축이 유도되지 않지만, DNA 네트워킹 또는 번칭은 멀리 있거나 가까운 염색체 세그먼트를 하나로 [45]묶어 응축에 크게 기여할 수 있다.
IHF
Integration Host Factor(IHF)는[51] 구조적으로는 HU와 거의 동일하지만 많은 면에서 HU와 다르게 동작합니다.배열에 관계없이 구조 모티브에 우선적으로 결합하는 HU와 달리 IHF는 배열의존성 DNA 구조와 변형성을 통해 특이성이 발생하더라도 특정 DNA 배열에 우선적으로 결합한다.동일 부위에서 IHF의 특이적 결합은 DNA를 160도 [51]이상 급격히 구부린다.대장균 [46]게놈에서 동종 배열 모티브의 발생은 약 3000이다.성장 단계에서 IHF의 추정 풍부도는 세포당 약 6000 이합체입니다.하나의 IHF 이합체가 단일 모티브에 결합하고 기하급수적인 성장 단계에서 핵이 하나 이상의 게놈 당량을 포함한다고 가정하면, 대부분의 IHF 분자는 게놈의 특정 부위를 차지하고 급격한 [46]굴곡을 유도함으로써 DNA를 응축시킬 수 있을 것이다.
특정 DNA 배열에 대한 우선 결합 외에 IHF는 또한 HU와 유사한 친화력을 가진 비배열 특이적 방식으로 DNA에 결합한다. IHF의 비특이적 결합의 역할은 정상상 및 추가 I에서 5배 증가하기 때문에 정상상에서는 중요한 것으로 보인다.HF 이합체는 염색체 DNA에 [21][52][53]비특이적으로 결합할 가능성이 있다.HU와 달리 IHF는 고농도에서는 두꺼운 강성 필라멘트를 형성하지 않습니다.그 대신 비특이적 결합은 특정 부위보다 휘어지는 정도가 훨씬 작고 [54]저농도의 선형 DNA에서 HU에 의해 유도되는 유연한 휘어지는 것과 유사하지만 DNA 휘어지는 것을 유도한다.체외에서 IHF의 비특이적 결합에 의해 유도되는 굽힘은 DNA 축합을 일으킬 수 있으며 염화칼륨과 [54]염화마그네슘의 농도에 따라 고차 핵단백질 복합체의 형성을 촉진한다.생체 내 IHF에 의한 고차 DNA 구성은 아직 [54]불분명하다.
H-NS
히스톤 유사 또는 열안정 핵구축단백질(H-NS)[55][56][57][58]과 다른 NAP와의 구별 가능한 특징은 비교적 낮은 농도(1 x 10−5 M 미만)에서 호모디머 형태에서 보다 높은 [59][60]레벨의 올리고머 상태로 전환하는 능력이다.올리고머화 특성 때문에, H-NS는 고친화성 부위가 핵 형성 [61][62][28]중심 역할을 하는 핵 생성 반응에서 AT가 풍부한 DNA를 따라 횡방향으로 확산된다.DNA에서 H-NS의 확산은 반응에서 마그네슘 농도에 따라 두 가지 상반된 결과를 초래한다.낮은 마그네슘 농도(< 2 mM)에서는 H-NS가 단단한 핵단백질 필라멘트를 형성하고, 높은 마그네슘 농도(> 5 mM)[63][64][65][66][67]에서는 분자간 및 분자내 브릿지를 형성한다.단단한 필라멘트의 형성은 응축 없이 DNA를 곧게 펴는 결과를 낳는 반면, 브리징은 상당한 DNA [66]접힘을 일으킨다.ChIP-Seq 분석에 의한 게놈 내 H-NS 결합 분석은 생체 내 DNA에서 H-NS의 확산에 대한 간접 증거를 제공했다.H-NS는 [50]게놈의 458개 영역에 선택적으로 결합한다.H-NS는 DNA[61][68] 배열에서 반복되는 A-트랙에 의해 형성된 곡면 DNA를 선호하는 것으로 입증되었지만, 선택적 결합의 기초는 AT가 풍부한 [27]영역에서 발견되는 보존된 배열 모티프의 존재이다.보다 중요한 것은 협력 단백질-단백질 상호작용을 다시 강화할 수 있는 H-NS 결합 영역 내에서 배열 모티프가 자주 발생하고 결합 영역의 길이가 비정상적으로 긴 것은 단백질의 확산과 일치한다.필라멘트 형성 또는 DNA 브리징이 생체 내에서 유행하는지 여부는 세포 [66][69]내 마그네슘의 생리적인 농도에 따라 달라집니다.마그네슘 농도가 균일하게 낮으면(< 5 mM), H-NS는 [66]생체 내에서 단단한 핵단백질 필라멘트를 형성한다.또는 세포 내에 마그네슘의 불균일한 분포가 있는 경우, DNA 브리징과 경화를 촉진할 수 있지만 핵이체의 [66]다른 영역이다.
또한 H-NS는 수평으로 전달되는 유전자의 전사를 우선적으로 억제하는 글로벌 유전자 소음기로 가장 잘 알려져 있으며 유전자 소음화를 이끄는 [70][71]강직 필라멘트이다.종합하면, 경직 필라멘트의 형성은 유전자 침묵을 유도하지만 DNA 축합을 유도하지 않는 생체 내 H-NS-DNA 상호작용의 가장 가능성이 높은 결과인 것으로 보인다.일관되게 H-NS가 없다고 해서 핵체 [72]부피가 변화하지는 않는다.그러나 대장균은 일부 환경조건에서 마그네슘 농도가 높을 수 있다.이러한 조건에서 H-NS는 필라멘트 유도 형태에서 DNA 응축 및 [66]구성에 기여하는 브리지 유도 형태로 전환할 수 있습니다.
피스
반전 자극 인자(Fis)는 15-bp 대칭 [29][30][73]모티브를 포함하는 특정 DNA 배열에 결합하는 배열 특이적 DNA 결합 단백질이다.IHF처럼, Fis는 같은 부위에서 DNA 굽힘을 유도합니다.DNA를 구부리는 능력은 Fis 호모디머의 구조에서 명백하다.Fis 호모디머는 각 단량체로부터 1개씩 2개의 헬릭스-턴-헬릭스(HTH) 모티브를 가진다.HTH 모티브는 일반적으로 DNA 주요 홈을 인식합니다.그러나 Fis homodimer에서 두 HTH 모티브의 DNA 인식 나선 사이의 거리는 25Ω으로 표준 B-DNA의 피치보다 약 8Ω 짧으며,[74][75] 이는 단백질이 안정적으로 결합하기 위해 DNA를 구부리거나 비틀어야 함을 나타낸다.일관되게 Fis-DNA 복합체의 결정 구조는 인식 나선 사이의 거리가 변하지 [30]않는 반면, DNA는 60~75도 범위에서 곡선을 그린다.대장균 게놈에는 1464개의 Fis 결합 영역이 분포되어 있으며, 계산상 확인된 결합 모티브는 알려진 15-bp [50][76]모티브와 일치한다.그러한 부위에서 Fis의 특이적 결합은 DNA의 굴곡을 유도하고, 따라서 DNA의 지속 길이를 줄임으로써 DNA 응축에 기여할 것이다.또한 많은 Fis 결합 부위는 안정적인 RNA 촉진제, 예를 들어 rRNA 오퍼론 rrnB의 P1 프로모터와 같이 동시에 발생한다.탠덤 부위에서 Fis에 의한 일관성 있는 굽힘은 DNA [77]응축에 더 기여할 수 있는 DNA 마이크로 루프를 만들 가능성이 있다.
Fis는 관련 부위에 대한 고친화도 특이적 결합 외에 임의의 DNA 배열에 결합할 수 있다.비특이적 DNA 결합은 Fis가 성장 단계에서 HU만큼 풍부하기 때문에 중요하다.따라서 대부분의 Fis 분자는 비배열 특이적인 방식으로 DNA와 결합할 것으로 예상된다.자기 핀셋 실험은 Fis의 비특이 결합이 DNA 응축과 [78][79]구성에 기여할 수 있다는 것을 보여준다.Fis는 1mM 미만에서는 단일 DNA 분자의 경미한 축합을 유발하지만, 1mM 이상에서는 평균 크기가 800bp 이하인 DNA 루프의 형성을 통해 상당한 접힘을 유도한다.자기 핀셋 실험의 루프는 염기서열 비의존적 결합에 의해 달성된 고밀도 DNA-단백질 복합체의 형성이 필요하기 때문에 동일 부위의 일관된 DNA 굽힘에 의해 생성된 마이크로 루프와는 구별된다.이러한 루프의 발생은 생체 내에서 입증되어야 하지만, Fis의 고밀도 결합은 특정 결합과 비특이 결합의 일치된 작용을 통해 생체 내에서 발생할 수 있다.특정 부위의 인텐덤 발생은 H-NS와 유사한 핵생성 반응을 일으킬 수 있으며, 비특이적 결합은 국부적인 고밀도 Fis 어레이의 형성을 초래할 수 있다.이들 지역 간의 브리징에 의해 대규모 DNA [79]루프가 발생할 수 있습니다.Fis는 성장 단계에만 존재하며 정지 단계에 [80][81]있지 않다.따라서 Fis에 의한 염색체 응축에서의 역할은 성장하는 [81]세포에 한정되어야 한다.
뉴클레오이드 관련 RNA(naRNA)
격리된 핵체에 대한 RNase A 처리의 영향을 검사하는 초기 연구는 RNA가 응축된 상태에서 [82]핵체의 안정화에 참여했음을 보여주었다.또한 RNase A에 의한 처리는 DNA 섬유를 얇은 섬유로 교란시켰으며, 이는 "기질 용융법"[83]을 이용한 핵소체의 원자력 현미경 검사에서 관찰되었다.이러한 연구 결과는 핵구조에 RNA가 관여하는 것을 보여주었지만,[47] RNA 분자의 정체는 최근까지 알려지지 않았다.HU에 대한 대부분의 연구는 HU의 DNA [83]결합에 초점을 맞췄다.그러나 HU는 선형 dsDNA와 [84]유사한 낮은 친화력으로 dsRNA 및 RNA-DNA 하이브리드에도 결합한다.또한 HU는 2차 구조를 포함한 RNA와 해당 RNA에 니크 또는 오버행이 [84][85]포함된 RNA-DNA 하이브리드에 우선적으로 결합한다.이러한 RNA 기질에 대한 HU의 결합 친화력은 왜곡된 DNA에 결합하는 친화력과 유사하다.HU-bound RNA의 면역 침강 정제된 손상되지 않은 nucleoids HU.[47]들은non-coding RNAs 몇몇과 상호 작용하는nucleoid-associated은 RNA분자 식별되 전사와 마이크로 어레이(RIP-Chip)연구뿐만 아니라 RNA분석 되돌리며 이러한 RNAnaRNA4(nucleoid-associated RNA4)에 이름을 붙였다 짝을 진 r을 에 박혀 있epeTitive extergenic palindrome(REP325).REP325가 결여된 변종에서는 핵이드는 [47]HU가 결여된 변종이기 때문에 축합된다.naRNA4는 [86]HU가 존재하는 상태에서 DNA 세그먼트를 연결함으로써 DNA 축합에 참여할 가능성이 높다.최근 연구는 NARNA4가 DNA-DNA 연결을 확립하는 방법에 대한 분자 메커니즘에 대한 통찰력을 제공한다.RNA는 십자형 구조를 포함하는 DNA의 영역을 목표로 하며 DNA-DNA [87]연결을 확립하는 데 중요한 RNA-DNA 복합체를 형성합니다.놀랍게도 HU는 복합체 형성에 도움을 주지만 최종 복합체에는 존재하지 않아 촉매(보호자)로서의 잠재적 역할을 나타낸다.RNA-DNA 복합체의 형성은 광범위한 왓슨/크릭 염기쌍을 포함하지 않지만 RNA-DNA 하이브리드에서 RNA를 분해하고 복합체가 RNA-DNA 하이브리드에 [47][83][84]특이적인 항체에 결합하는 RNase H에 민감하기 때문에 RNA-DNA 복합체의 특성은 여전히 곤혹스럽다.
슈퍼코일
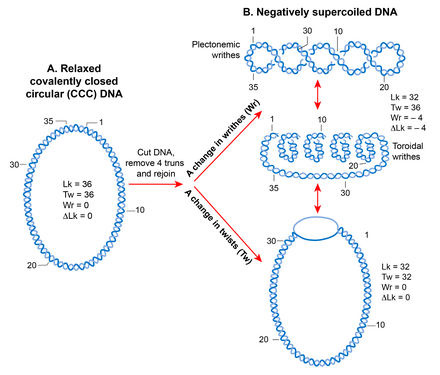
나선형 구조 때문에 이중가닥 DNA 분자는 자유단의 [88]회전을 제거하는 공유 폐쇄 원형으로 위상적으로 구속된다.위상적으로 제약된 DNA에서 두 가닥이 서로 교차하는 횟수를 연결수(Lk)라고 하는데, 이는 원형 [89]분자의 나선 회전수 또는 비틀림 횟수에 해당합니다.위상 DNA의 Lk는 DNA 분자가 어떻게 변형되든, 어느 가닥도 [90][91]끊어지지 않는 한 변하지 않습니다.
완화된 형태의 DNA의 Lk는 Lk로0 정의된다.임의의 DNA에 대해 Lk는0 DNA의 길이(bp 단위)를 나선 회전당 bp 수로 나누어 계산할 수 있다.이는 완화 B형 DNA의 경우 10.4bp와 같습니다.Lk로부터의0 편차는 DNA의 슈퍼코일을 일으킨다.링크 수가 감소하면(Lk < Lk0), 링크 수가 증가하면(Lk> Lk0) 양의 슈퍼 [92][90]코일이 생성됩니다.
슈퍼코일 상태(Lk가 Lk와0 동일하지 않은 경우)는 DNA 구조의 전이를 가져오며, 이는 트위스트 수 변화(음성 <10.4 bp/턴, 회전당 양 > 10.4 bp) 및/또는 슈퍼코일이라고 불리는 글씨 형성의 변화로 나타날 수 있다.따라서, Lk는 두 기하학적 매개변수, 트위스트와 라이트(writte)의 부호 의존 합계로 수학적으로 정의된다.DNA 분자의 크기와 무관한 슈퍼코일의 정량적 측정은 δ = δLk/[91]Lk인0 슈퍼코일 밀도(supercoiling density)이다.
글씨는 플렉토네임과 솔레노이드 또는 트로이드의 두 가지 구조를 채택할 수 있습니다.나선축의 상호 감김으로부터 플렉토네믹 구조가 발생한다.트로이덜 슈퍼코일은 DNA가 전화선처럼 [90]축 주위에 여러 개의 나선형을 형성하고 서로 교차하지 않을 때 발생합니다.플렉토넴 형태의 글씨는 각각 양수 또는 음수 초코일 DNA의 오른손잡이와 왼손잡이이다.트로이덜 슈퍼코일의 손길은 플렉토넴의 손길과 반대입니다.플렉토넴과 트로이덜 슈퍼코일은 모두 자유형태이거나 단백질에 의해 결합형태로 억제될 수 있다.생물학에서 결합된 트로이덜 초코일의 가장 좋은 예는 DNA가 히스톤을 [17]감싸는 진핵 핵소체이다.
대장균의 플렉톤혈증 슈퍼코일
대부분의 박테리아에서 DNA는 초코일 형태로 존재한다.대장균 염색체의 원형 성질은 위상적으로 제약된 분자로 추정 평균 초코일 밀도(θ)가 -0.[93]05인 음의 초코일 분자가 대부분이다.진핵생물 염색질에서 DNA는 주로 뉴클레오솜 형성을 통해 히스톤에 의해 억제되고 정의되는 트로이덜 형태로 발견된다.이와는 대조적으로, 대장균의 핵에서 염색체 DNA의 약 절반은 유리 플렉톤혈증 [94][95][96]슈퍼코일의 형태로 구성되어 있다.나머지 DNA는 NAP와 같은 단백질과의 상호작용에 의해 트로이덜 형태를 포함하지만 이에 국한되지 않는 총체적 형태 또는 대체 형태로 억제된다.따라서 플렉톤혈증 슈퍼코일은 응축과 구성을 담당하는 대장균 게놈의 효과적인 슈퍼코일을 나타냅니다.플렉톤혈증과 트로이덜 초코일은 모두 DNA 응축에 도움이 됩니다.주목할 점은 플렉톤혈구조의 분기로 인해 트로이덜 구조보다 DNA 응축이 적다는 것이다.동일한 초코일 밀도를 가진 동일한 크기의 DNA 분자는 플렉톤혈증 형태보다 트로이덜 형태로 더 콤팩트하다.슈퍼코일은 DNA를 응축하는 것 외에도 DNA 형성을 돕는다.그것은 [97]담금질 확률을 줄임으로써 DNA의 분리를 촉진한다.슈퍼 코일링은 또한 DNA의 두 먼 지점을 근접하게 함으로써 DNA의 [91]다른 부분들 사이의 잠재적인 기능적 상호작용을 촉진하는 것을 돕는다.
대장균의 슈퍼코일 발생원
(i) 토포이소머라아제 활성, (ii) 전사 작용, (ii) NAP의 [95]세 가지 인자가 대장균에서 염색체 DNA 슈퍼코일을 생성하고 유지하는 데 기여한다.
토포이소머라아제
토포이소머라아제(topoisomerase)는 DNA 대사 [98]효소의 특정 범주로, DNA 가닥을 끊고 다시 결합함으로써 슈퍼코일을 생성하거나 제거합니다.대장균은 4가지 토포이소머라아제를 가지고 있다.DNA 자이라아제는 ATP가 있을 때 음의 슈퍼코일을 도입하고 ATP가 [99]없을 때 양의 슈퍼코일을 제거한다.모든 형태의 생명체에서, DNA 자이라제는 음의 슈퍼코일을 만들 수 있는 유일한 토포이소머라아제이고 박테리아 게놈이 자유 음의 슈퍼코일을 가지고 있는 독특한 능력 때문이다; DNA 자이라아제는 모든 박테리아에서 발견되지만 고등 진핵생물에서는 발견되지 않는다.반면 Topo I은 음의 초코일을 [100][101]가진 DNA를 완화함으로써 DNA 자이라제에 반대한다.DNA 자이라아제와 Topo I의 상반된 활성 사이의 균형이 [100][102]대장균의 평균 음성 슈퍼헬리시티의 정상적 수준을 유지하는 데 책임이 있다는 것을 암시하는 유전적 증거가 있다.두 효소 모두 대장균의 생존에 필수적이다.Topo I를 코드하는 유전자인 topA의 늘주는 DNA 자이라제를 [100][102]코드하는 유전자 내에 억제체 돌연변이가 존재하기 때문에만 존속한다.이러한 돌연변이는 자이라아제 활성을 감소시켜 Topo I의 부재로 인한 과도한 음의 슈퍼코일이 DNA 자이라아제의 음의 슈퍼코일 활성을 감소시킴으로써 보상된다는 것을 암시한다.Topo III는 대장균에서 분리할 수 있으며 [103]대장균에서 슈퍼코일링에 어떠한 역할도 하는 것으로 알려져 있지 않다.Topo IV의 주요 기능은 자매 염색체를 분해하는 것이다.그러나 Topo [104][105]I과 함께 부극성 슈퍼코일을 완화함으로써 부극성 슈퍼코일의 정상 상태 수준에도 기여하는 것으로 나타났다.
| 토포이소머라아제 | 유형 | 기능. | 단가닥 또는 이중가닥 절단 |
|---|---|---|---|
| 토포이소머라아제 I | IA | (-) 슈퍼 코일 제거 | SS |
| 토포이소머라아제II | IA | (-) 슈퍼 코일 제거 | SS |
| 토포이소머라아제 IV | IIA | (-) 슈퍼 코일 제거 | DS |
| DNA자이라아제 | IIA | (-) 슈퍼코일 작성 및 (+) 슈퍼코일 삭제 | DS |
문자 변환
Liu와 Wang에 의해 제안된 쌍둥이 슈퍼코일 도메인 모델은 전사 중 DNA 이중나선의 풀림이 [106]에서와 같이 DNA의 슈퍼코일을 유도한다고 주장했다.그들의 모델에 따르면, RNA 중합효소(RNAP)가 DNA를 따라 미끄러지는 것은 DNA가 나선 축에서 회전하도록 강요한다.위상 제약으로 인해 DNA의 자유 회전에 장애가 발생하여 RNAP 앞의 DNA가 과도하게 비틀리고(양적으로 슈퍼코일), RNAP 뒤의 DNA가 과소 비틀림(음성 슈퍼코일)될 수 있다.RNAP은 선형 [107]DNA 템플릿에서도 슈퍼코일을 발생시키는 충분한 토크를 발생시키기 때문에 위상 제약이 필요하지 않은 것으로 밝혀졌다.DNA가 이미 음의 슈퍼코일을 감싼 경우, 이 작용은 RNAP보다 먼저 양의 슈퍼코일을 축적하기 전에 기존의 음의 슈퍼코일을 완화시키고 더 많은 음의 슈퍼코일을 RNAP 뒤에 도입합니다.원칙적으로 DNA 자이라아제와 Topo I은 과잉 양성과 음성의 슈퍼코일을 각각 제거해야 하지만, RNAP 신장률이 두 효소의 회전을 초과하면 전사가 [107]슈퍼코일의 정상 수준에 기여한다.
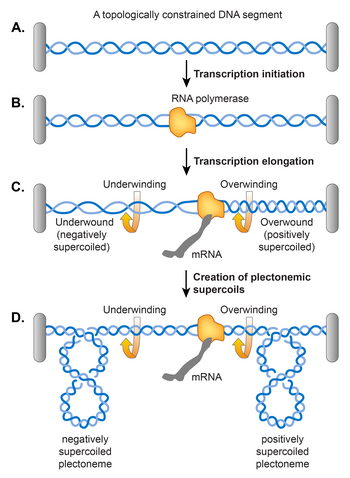
NAP에 의한 슈퍼 코일 제어
진핵생물 크로마틴에서, 뉴클레오솜은 히스톤에 대한 DNA의 긴밀한 결합을 통해 거의 모든 음성 슈퍼코일을 억제하기 때문에 DNA는 자유 슈퍼코일 형태로 거의 존재하지 않는다.마찬가지로 대장균에서도 NAP에 의해 형성된 핵단백질 복합체는 핵이드의 [93][96]초코일 밀도의 절반을 억제한다.다시 말해, NAP가 핵단백질 복합체에서 분리되면, DNA는 자유 플렉톤혈증 형태를 취할 것이다.HU, Fis 및 H-NS의 DNA 결합은 완화되었지만 위상적으로 제약된 [108][109][110][111][112]DNA에서 음성 슈퍼코일을 억제하는 것으로 실험적으로 나타났다.그들은 DNA의 나선 피치를 바꾸거나 DNA를 구부리고 감싸는 것에 의해 트로이덜의 비틀림을 발생시킴으로써 그렇게 할 수 있다.또는 NAP는 십자형 구조 및 분기된 플렉토넴과 같은 언더와이드 DNA의 다른 형태에 우선적으로 결합하고 안정화시킬 수 있다.Fis는 교차 영역과의 결합을 통해 분기된 플렉토넴을 구성하고 HU는 십자형 [112]구조에 우선적으로 결합하는 것으로 보고되었다.
NAP는 또한 DNA 슈퍼코일을 간접적으로 조절합니다.Fis는 DNA 자이라제를 [113]코드하는 유전자의 전사를 억제함으로써 슈퍼코일을 변조할 수 있다.HU가 DNA 자이라제를 자극하고 Topo [114][115]I의 활성을 감소시킴으로써 초코일 수준을 조절한다는 것을 암시하는 유전적 증거가 있다.유전자 연구를 뒷받침하기 위해 HU는 DNA 자이라아제 촉매 [116]체외 DNA 분해를 자극하는 것으로 나타났다.HU가 자이라스와 토포I의 활동을 어떻게 조절하는지는 기계적으로 불분명하다.HU는 DNA 자이라아제 및 Topo I과 물리적으로 상호작용할 수 있으며, DNA 굽힘과 같은 HU의 DNA 구성 활동은 각각 [114][116]DNA 자이라아제와 Topo I의 작용을 촉진하거나 억제할 수 있다.
플렉톤혈증 슈퍼코일은 여러 토폴로지 도메인으로 구성됩니다.
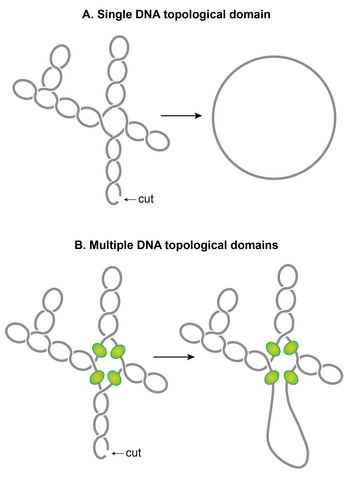
핵이체의 두드러진 특징 중 하나는 플렉톤혈증 슈퍼코일이 여러 위상 [117]도메인으로 구성된다는 것이다.즉, 한 도메인에서 한 컷만 잘라도 해당 도메인이 완화되고 다른 도메인은 완화되지 않습니다.위상 도메인은 초코일 확산 장벽에 의해 형성된다.서로 다른 방법을 사용하는 독립적인 연구에서는 위상 도메인의 크기가 10에서 400kb까지 [95][117][118]다양하다고 보고하였다.이러한 연구에서 일반적으로 관찰되는 장벽의 무작위 배치는 [117]도메인 크기의 광범위한 가변성을 설명하는 것으로 보인다.
도메인 장벽의 동일성은 아직 확립되지 않았지만, 장벽 형성에 관여하는 가능한 메커니즘은 다음과 같다. (i) 슈퍼코일을 억제하는 능력을 가진 단백질이 위상적으로 분리된 DNA 루프 또는 도메인을 형성하는 염색체 상의 두 개의 다른 부위에 동시에 결합할 때 도메인 장벽이 형성될 수 있다.초코일 DNA에서 단백질 매개 루프가 위상 [119][120]도메인을 형성할 수 있다는 것이 실험적으로 증명되었다.H-NS 및 Fis와 같은 NAP는 DNA 루프 능력 및 결합 부위의 분포에 기초해 잠재적 후보이다. (ii) 세균 간 산란 모자이크 요소(BIME)도 도메인 장벽의 잠재적 후보로 나타난다.BIME은 보통 유전자 사이에서 발견되는 회문 반복 배열이다.BIME은 대장균 [121]염색체에 삽입된 합성 설계 위상 카세트에서 슈퍼코일의 확산을 방해하는 것으로 나타났다.게놈 전체에 600개의 BIME이 분포되어 있으며, 염색체를 600개의 위상 [122]도메인으로 나눌 수 있다.(iii) 장벽은 DNA와 막 양쪽에 결합하는 단백질 또는 초기 전사와 막 앵커링 단백질의 번역을 통해 세포막에 DNA가 부착되는 경우에도 발생할 수 있다. (iv) 전사 활성은 초코일 확산 장벽을 생성할 수 있다.활성 전사 RNAP은 플렉톤혈증 슈퍼코일의 방산을 차단하여 초코일 확산 [123][124][125]장벽을 형성하는 것으로 나타났다.
성장상 의존성 핵소체 역학
뉴클레오이드는 정지상 세포에서 재구성되어 뉴클레오이드 구조가 세포의 생리적 상태에 의해 결정되는 매우 역동적이라는 것을 암시합니다.핵소체의 고해상도 접촉 맵을 비교한 결과,[126] 성장기에 비해 Ter 매크로 도메인의 장거리 접촉이 정상 단계에서 증가하였다.또한 정상기 CID 경계는 성장기 CID 경계와 달랐다.마지막으로, 핵상 형태학은 장기간에 걸친 정지상 [127]동안 대규모 변환을 거친다; 핵상 형태들은 [128]질서정연한 트로이덜 구조를 나타낸다.
핵상 구조의 성장 단계 특이적 변화는 핵상 관련 DNA 구조 단백질(NAPs 및 Muk subunits), 초코일링 및 전사 활성 수준의 변화에 의해 야기될 수 있다.NAP와 Muk 서브유닛의 풍부함은 세균의 성장주기에 따라 변화한다.Fis 및 또 다른 NAP인 기아 유발 DNA 결합 단백질 Dps는 각각 성장기와 정상기에 거의 독점적으로 존재한다.피스 레벨은 지수 단계로 진입하면 상승하고 셀이 지수 단계에 있는 동안 급격히 감소하여 정지 [129]단계에서 검출할 수 없는 수준에 도달한다.FIS 레벨은 감소하기 시작하는 반면, DPS 레벨은 상승하기 시작하고 정지 [21]단계에서 최대치에 도달한다.장기 정지상에서 관측된 핵상 구조의 극적인 전이는 주로 Dps에 기인한다.그것은 [128]기아가 일어나는 동안 존재하는 DNA 손상 물질로부터 핵을 보호하는 역할을 하는 DNA/결정 조립체를 형성합니다.
HU, IHF 및 H-NS는 성장기와 정상기 [21]모두에 존재합니다.그러나 이들의 풍부성은 크게 변화하여 HU와 Fis가 성장 단계에서 가장 풍부한 NAP인 반면 IHF와 Dps는 정상 [21]단계에서 가장 풍부한 NAP가 된다.HUα는 초기 지수 단계에서 지배적인 형태인 반면, 이단성 형태는 소량의 호모디머와 [130]함께 정지 단계에서 지배적이다.두 형태가 DNA를 다르게 구성하고 응축하는 것처럼 보이기 때문에 이러한 변화는 핵구조와 관련된 기능적 결과를 가지고 있다; 호모이머와 헤테로이머는 모두 필라멘트를 형성하지만, 오직 호모이머만이 [45]DNA 네트워크를 형성하기 위해 여러 개의 DNA 세그먼트를 함께 가져올 수 있다.MukB의 복사 수는 정지 상태에서 [131][132]2배로 증가합니다.MukB 분자의 증가는 DNA 루프 압출 인자로서 MukBEF 복합체의 처리능력에 영향을 미칠 수 있으며 결과적으로 더 많은 루프가 [131][132]발생할 수 있습니다.
슈퍼코일은 DNA 구조 단백질과 조합된 방식으로 작용하여 핵을 재구성할 수 있다.전체 슈퍼코일 수준은 정지 단계에서 감소하며 슈퍼코일 수준은 지역 [133]수준에서 다른 패턴을 보입니다.초코일의 변화는 핵이드의 위상 구조를 바꿀 수 있다.또한 전사활성이 높은 염색체 영역은 CID 경계를 형성하기 때문에 서로 다른 성장단계에서의 전사활성의 변화는 CID 경계 형성을 변화시켜 핵이드의 공간구조를 변화시킬 수 있다.정상 단계에서 관찰된 CID 경계 변화는 성장 [126]단계에 비해 정상 단계에서 다른 유전자 세트의 높은 발현에 기인할 수 있다.
핵상 구조 및 유전자 발현
NAP 및 유전자 발현
대장균의 염색체 구조와 유전자 발현이 상호 영향을 미치는 것으로 보인다.한편, CID 경계와 높은 전사 활성의 상관관계는 염색체 조직이 전사에 의해 움직이는 것을 나타낸다.한편, 모든 스케일에서 핵상 내 DNA의 3D 구조는 유전자 발현과 관련될 수 있다.첫째, 대장균에서 핵이드의 3D 구조를 재구성하면 세포 전사 [134]패턴을 동적으로 변조할 수 있는 것으로 나타났다.HUa의 돌연변이는 염색체 DNA의 양성 슈퍼헬리시티 증가에 의해 핵이드를 매우 많이 응축시켰다.그 결과, 많은 유전자가 억제되었고, 많은 정지 상태의 유전자가 발현되었다.또한 단백질 매개 국소 구조 변화가 유전자 전사를 변화시키는 많은 특정 사례가 있다.예를 들어 H-NS에 의한 경질핵단백질 필라멘트의 형성은 프로모터에 대한 RNAP 접근을 차단하여 유전자 [135]전사를 방지한다.유전자 사일런싱을 통해 H-NS는 수평으로 전달되는 유전자의 [50][27]전사를 우선적으로 억제하는 글로벌 레프레서 역할을 한다.다른 예에서 갈오페론에서의 HU의 특이적 결합은 유도제가 [136]없는 경우에도 갈오페론을 억제하는 DNA 루프의 형성을 촉진한다.안정적인 RNA 촉진제에서 Fis에 의해 DNA의 일관성 있는 굽힘에 의해 생성된 위상적으로 구별되는 DNA 마이크로루프는 [77]전사를 활성화한다.IHF에 의한 DNA 굽힘은 대장균 [137][138]내 ilvGMEDA 오퍼론의 2개의 탠덤 프로모터로부터의 전사를 차별적으로 제어한다.NAP에 의한 특정한 위상 변화는 유전자 전사를 조절할 뿐만 아니라 DNA 복제 시작, 재조합, [9][10][11]전이와 같은 다른 과정에도 관여한다.특정 유전자 조절과는 대조적으로, 고차 염색체 구조와 그 역학이 분자 수준에서 전 세계적으로 유전자 발현에 어떤 영향을 미치는지 [139]밝혀내야 한다.
DNA 슈퍼코일링 및 유전자 발현
DNA 슈퍼코일과 유전자 [139]전사 사이에는 쌍방향 상호연결이 존재한다.프로모터 영역의 네거티브 슈퍼코일은 프로모터 용융을 촉진하고 단백질 조절기의 DNA 결합 친화성을 증가시킴으로써 전사를 촉진할 수 있다.전사의 확률적 폭발은 고도로 발현된 유전자의 일반적인 특성으로 보이며, DNA 템플릿의 초코일 수준은 전사 [140]폭발의 원인이 된다.쌍둥이 슈퍼코일 도메인 모델에 따르면 유전자의 전사는 슈퍼코일 릴레이를 통해 근처의 다른 유전자의 전사에 영향을 줄 수 있다.그러한 예 중 하나는 leu-500 [139]프로모터의 활성화이다.슈퍼코일은 유전자 고유의 변화뿐만 아니라 유전자 발현에 있어서도 큰 변화를 일으킨다.핵양체의 위상학적 구성은 다른 위상 영역에서 초코일에 민감한 유전자의 독립적인 발현을 가능하게 할 수 있다.제한되지 않은 슈퍼코일링의 게놈 스케일 맵은 게놈 영역이 다른 정상 상태의 슈퍼코일링 밀도를 가지고 있다는 것을 보여주며, 슈퍼코일링의 수준은 개별 [133]토폴로지 영역에 따라 다르다는 것을 보여준다.그 결과 슈퍼코일의 변화는 각 [133]도메인의 슈퍼코일의 수준에 따라 도메인 특이적 유전자 발현을 초래할 수 있다.
유전자 발현에 대한 슈퍼코일의 영향은 슈퍼코일에 직간접적으로 영향을 미치는 NAP에 의해 매개될 수 있다.유전자 발현에 대한 HU의 영향은 슈퍼코일의 변화와 아마도 고차 DNA 구성을 포함하는 것으로 보인다.DNA 자이라아제 결합과 HU의 부재로 인한 유전자의 상향조절 사이의 양의 상관관계는 슈퍼코일의 변화가 미분발현에 책임이 있음을 시사한다.HU는 또한 전사에 의해 [141]유도되는 슈퍼코일을 억제함으로써 전사 단위를 절연함으로써 유전자 발현에 대한 위치적 영향을 미치는 것으로 밝혀졌다.HUa의 점 돌연변이는 대장균의 유전자 발현 프로파일을 극적으로 변화시켜 대장균의 형태, 생리학, 신진대사를 변화시켰다.그 결과, 돌연변이 변종은 포유동물 세포에 [134][142]더 침습적이었다.이러한 극적인 효과는 핵소체 압축과 양성 슈퍼코일 [45][143]증가를 수반했다.돌연변이 단백질은 옥타머였고 야생형 이합체와는 대조적이었다.야생형 [143]HU와 달리 양성 슈퍼코일을 억제하면서 오른손으로 DNA를 감싸고 있습니다.이러한 연구는 HU의 아미노산 치환이 핵상 구조에 극적인 영향을 미칠 수 있으며, 이는 결국 상당한 표현형 [143]변화를 가져온다는 것을 보여준다.
MukB와 HU가 장거리 DNA 상호작용에서 중요한 주체로 부상했기 때문에, 이 두 단백질 각각이 전세계 유전자 [144]발현에 미치는 영향을 비교할 가치가 있을 것이다.HU는 초코일 밀도를 조절해 유전자 발현을 조절하는 것으로 보이지만 정확한 분자 메커니즘은 알려지지 않았으며 MukB가 유전자 발현에 미치는 영향은 아직 [144][145]분석되지 않았다.
공간 구성
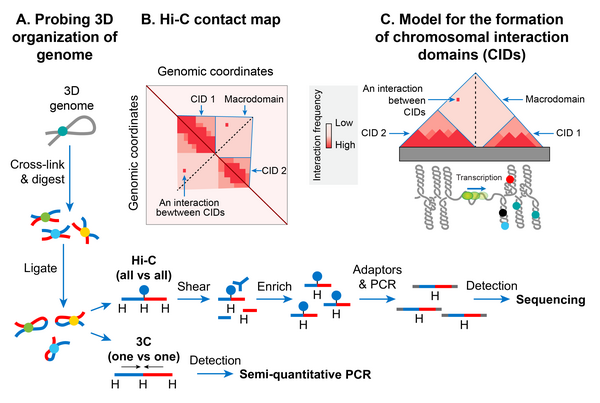
염색체 상호작용 도메인
최근 몇 년 동안, 염색체 배치 포착이라고 불리는 분자 방법의 등장은 박테리아와 진핵 [146]생물 모두에서 염색체의 고해상도 공간 구성을 연구할 수 있게 해 주었다.3C 및 딥 시퀀싱(Hi-C)[147]과 결합된 버전은 3D 공간의 두 게놈 위치 사이의 물리적 근접성을 결정합니다.대장균 염색체를 포함한 세균 염색체의 고해상도 접촉 지도는 세균 염색체가 염색체 상호작용 도메인(CIDs)[126][148][149]이라고 불리는 많은 자기 상호작용 영역으로 분할된다는 것을 밝혀냈다.CID는 많은 진핵생물 [150]염색체에서 관찰되는 위상적으로 연관된 도메인(TAD)과 동등하며, CID의 형성은 게놈 구성의 일반적인 현상임을 시사한다.CID 또는 TAD는 2가지 특성으로 정의됩니다.첫째, CID의 게놈 영역은 해당 CID의 외부 또는 인접 CID의 게놈 영역보다 물리적으로 더 자주 상호 작용한다.둘째, 인접한 2개의 [126]CID의 게놈 영역 간의 물리적 상호작용을 방지하는 CID 간의 경계 존재.
대장균 염색체는 성장 단계에서 31개의 CID로 구성되었다.CID의 사이즈는 40 ~300KB입니다플렉톤혈증 DNA 루프를 위상 도메인으로 분리하는 역할을 하는 초코일 확산 장벽은 대장균과 다른 많은 박테리아에서 CID 경계로 기능하는 것으로 보인다.즉, 초코일 확산 장벽의 존재에 의해 CID의 형성이 정의된다.대장균, Caulobacter crescentus 및 Bacillus subtilis의 염색체 Hi-C 프로브의 소견은 NAP의 DNA 조직 활동과 함께 유전체 위치 간의 물리적 상호작용을 촉진하기 때문에 CID가 형성되는 모델에 수렴되며, CID 경계는 이러한 유전체 위치(FRP)를 방지하는 것으로 구성된다.상호 작용.RNAP를 적극적으로 전사함으로써 DNA를 헬리컬하게 풀면 플렉톤혈증 슈퍼코일이 억제되기 때문에 전사활성이 높아 PFR이 생성된다.그 결과 슈퍼코일 방산도 차단되어 슈퍼코일 확산 장벽이 형성된다.이 모델에 대한 간접적인 증거는 대장균 염색체를 포함한 세균 염색체의 CID가 그 경계에서 고도로 전사된 유전자를 나타내며, CID 경계 [126][148]형성에 있어 전사의 역할을 나타내는 관찰로부터 나온 것이다.보다 직접적인 증거는 경계가 존재하지 않는 위치에 고도로 전사된 유전자를 배치하는 것이 C. 초승달 [148]염색체에 새로운 CID 경계를 만들었다는 발견으로부터 나왔다.그러나 모든 CID 경계가 대장균 염색체의 고도로 전사된 유전자와 상관관계가 있는 것은 아니며, 이는 다른 알려지지 않은 요인들도 CID 경계의 형성 및 초코일 확산 [148]장벽의 원인이라는 것을 시사한다.
매크로 도메인
위상 도메인 또는 CID로 구성된 플렉토네믹 DNA 루프는 매크로 도메인(MD)이라고 불리는 공간적으로 구별되는 큰 도메인을 형성하기 위해 더욱 결합하는 것으로 보입니다.대장균에서 MD는 처음에 형광 인 시트 하이브리제이션([151][152]FISH) 연구에서 함께 국소화된(공동 국재화된) DNA 마커를 가진 게놈의 큰 세그먼트로 확인되었다.ORIC(염색체 복제의 기원) 궤적을 덮는 큰 게놈 영역(~1-Mb)이 함께 국재화되어 Ori 매크로 도메인으로 불렸다.마찬가지로, 복제 말단 영역(ter)을 덮는 큰 게놈 영역(~1-Mb)이 공동 국재화되어 Ter 매크로 도메인으로 불렸다.MD는 염색체의 다양한 먼 위치에 삽입된 람다 사이트 쌍이 서로 얼마나 자주 재결합했는지를 바탕으로 나중에 확인되었다.이 재조합 기반 방법에서 MD는 DNA 부위가 주로 서로 재결합할 수 있는 큰 게놈 영역으로 정의되지만 MD 이외의 부위와는 재결합할 수 없습니다.재조합 기반 방법은 FISH 연구에서 확인된 Ori MD와 Ter MD를 확인하고 두 [12][153]개의 MD를 추가로 식별했습니다.
2개의 추가 MD는 Ter 옆에 있는 ~1 Mb의 추가 영역에 의해 형성되어 좌우로 불리고 있습니다.이들 4개의 MD(Ori, Ter, Left, Right)는 오리강에 인접한 두 개의 게놈 영역을 제외한 대부분의 게놈을 구성했다.이들 두 영역(NS-L 및 NS-R)은 MD에 비해 DNA 부위가 양쪽 MD에 위치한 DNA 부위와 재결합하기 때문에 MD에 비해 유연하고 비구조적이었다.예를 들어, ORIC에 가장 가까운 유전체 영역은 DNA 배열에 관계없이 항상 NS로 동작하고 멀리 떨어진 영역은 항상 MD로 동작하기 때문에 ORIC의 유전자 위치가 MD의 [154]형성을 지시하는 것으로 보인다.
Hi-C 기법은 매크로 [126]도메인의 형태로 CID의 계층적 공간 구성을 추가로 확인했다.즉, 매크로 도메인의 CID는 인접 매크로 도메인의 CID 또는 매크로 도메인 외부의 게놈 위치보다 물리적으로 더 자주 상호 작용했다.Hi-C 데이터는 대장균 염색체가 두 개의 뚜렷한 도메인으로 분할되고 있음을 보여주었다.ter를 둘러싼 영역은 이전에 확인된 Ter MD와 겹치는 절연 도메인을 형성했습니다. 이 도메인에서 DNA-DNA 접촉은 최대 280kb 범위에서만 발생했습니다.염색체의 나머지 부분은 단일 도메인을 형성했고, 게놈 궤적은 >280-kb의 [126]범위에서 접촉했다.이 도메인의 대부분의 접점은 최대 500kb까지로 제한되었지만, 게놈 궤적이 더 먼 거리(최대 1Mb)에서 접점을 형성한 두 개의 느슨한 영역이 있었다.이러한 느슨한 영역은 이전에 식별된 유연하고 덜 구조화된 영역(NS)에 해당합니다.Ter와 Hi-C 방법으로 식별된 느슨한 두 영역을 포함하는 절연 영역의 경계는 전체 염색체를 재조합 기반 분석에 [126]의해 정의된 네 개의 MD와 두 개의 NS 영역에 해당하는 여섯 개의 영역으로 분할했다.
매크로 도메인 형성을 촉진하는 단백질
MatP
매크로 도메인 형성에 관여하는 단백질의 탐색은 매크로 도메인 Ter 단백질(MatP)의 식별으로 이어졌다.MatP는 macrodomain ter sequence(matS)[32]라고 불리는 13-bp 모티브를 인식함으로써 Ter MD에 거의 독점적으로 결합합니다.Ter 도메인에는 23개의 matS 사이트가 있으며, 평균적으로 35KB마다 1개의 사이트가 있습니다.Ter 도메인에서 MatP 결합의 추가 증거는 MatP의 형광 이미징에서 나온다.Ter 도메인 DNA [32]마커와 함께 국소화된 이산 MatP foci가 관찰되었다.Ter MD에서의 ChIP-Seq 신호의 강한 농도는 [32]또한 이 도메인에 대한 MatP의 우선 결합을 뒷받침한다.
MatP는 Ter 도메인에서 DNA를 응축하는데, MatP가 부족하면 Ter 도메인에서 100kb 떨어져 있는 두 개의 형광 DNA 마커 사이의 거리가 증가하기 때문이다.또한 MatP는 염색체의 [126]나머지 부분으로부터 Ter 도메인을 절연하는 데 중요한 역할을 한다.Ter 도메인 내에서 DNA-DNA 접촉을 촉진하지만 Ter 도메인의 DNA 위치와 측면 영역의 DNA 위치 사이의 접촉을 방지한다.MatP는 DNA를 어떻게 응축하고 DNA-DNA 접촉을 촉진합니까?실험 결과가 상반되고 있다.MatP는 체외에서 두 개의 MatS 부위 사이에 DNA 루프를 형성할 수 있으며, 그 DNA 루핑 활동은 MatP 4중합에 의존한다.사분자화는 [156]DNA에 결합된 두 개의 MatP 분자 사이의 코일 코일 상호작용을 통해 일어난다.시험관내 결과에 기초한 한 가지 분명한 모델은 MatP가 matS 부위의 브리징을 통해 생체 내 DNA-DNA 접촉을 촉진한다는 것이다.그러나 Hi-C 스터디에서는 MatP가 원격 사이트를 연결했지만 특별히 MatS 사이트를 연결하지는 않았습니다.또, 4량체를 형성할 수 없었던 MatP 돌연변이는 야생형처럼 행동했다.이러한 결과는 Ter 조직의 MatS 브리징 모델에 반하는 것이므로 MatP 액션의 메커니즘은 이해하기 어렵습니다.하나의 가능성은 MatP가 프라이머리 matS 결합 사이트에서 인근 DNA 세그먼트로 확산되어 4중합에 [156]의존하지 않는 메커니즘을 통해 먼 사이트를 브리지하는 것입니다.
묵베프
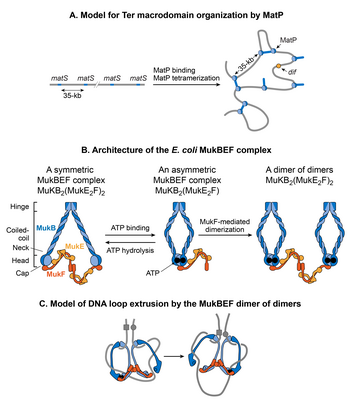
MukB는 [145]진핵생물에서 고차 염색체 구성에 참여하는 염색체 단백질의 구조 유지라고 불리는 ATPase 계열에 속합니다.두 개의 MukB 모노머는 연속적인 역평행 코일-코일 상호작용을 통해 결합되어 100-nm 길이의 강체 로드를 형성합니다.로드 중앙에는 유연한 [162][163]힌지 영역이 있습니다.힌지 영역의 유연성으로 인해 MukB는 SMC 계열의 특징적인 V자 형태를 채택하고 있습니다.MukB와 관련된 비 SMC 서브유닛은 MukE와 MukF입니다.연결은 V 형성을 닫고 큰 고리 모양의 구조를 생성합니다.MukE와 MukF는 대장균의 [164]같은 오퍼론으로 MukB와 함께 부호화됩니다.두 서브유닛 중 하나를 삭제하면 MukBEF 복합체가 생체 [160]내 기능 단위임을 시사하는 동일한 표현형이 된다.복합체의 DNA 결합 활성은 MukB 서브유닛에 있는 반면, MukE와 MukF는 MukB [164]활성을 조절한다.
MukBEF 콤플렉스는 Topo IV와 함께 새로 복제된 [165][166][167][168][155]oriC의 분리 및 위치 변경에 필요합니다.MukBEF의 역할은 DNA 복제 중에 제한되지 않습니다.그것은 비복제 [131]세포에서도 DNA를 구성하고 응축시킨다.최근 MukB 결손 대장균 균주의 고해상도 염색체 배치 지도는 MukB가 Ter [126]도메인을 제외한 전체 염색체에서 DNA-DNA 상호작용 형성에 관여한다는 것을 보여준다.MukB는 어떻게 Ter 도메인에서 동작하지 못하게 되어 있습니까?MatP는 물리적으로 MukB와 대화하기 때문에 MukB가 Ter 도메인에 [155]현지화되지 않습니다.이것은 Ter 도메인에서 MatP와 MukB의 DNA 결합에서 명백하다.MatP의 DNA 결합은 Ter 도메인에서 농축되는 반면, MukB의 DNA 결합은 게놈의 나머지 부분에 비해 감소한다.또한 MatP가 이미 결핍된 균주에서는 MukB가 결핍되면 Ter [126]도메인을 포함한 염색체 전체의 DNA 접촉이 감소한다.이 결과는 MatP가 Ter [126]도메인에서 MukB를 대체한다는 견해와 일치합니다.
MukBEF 복합체는 대장균 염색체를 구성하는 기능을 어떻게 합니까?현재 시각에 따르면 SMC 복합체는 DNA [169]루프를 밀어내 염색체를 형성한다.SMC 복합체는 DNA를 따라 이동해 시스 매너(같은 DNA 분자)에서 루프를 밀어냅니다. 여기서 루프의 크기는 복합체의 처리 능력에 따라 달라집니다.서로 다른 유기체의 SMC 복합체는 루프 [169]압출 메커니즘이 다르다.대장균에서 MukBEF의 단일 분자 형광 현미경 연구는 생체 내 최소 기능 단위가 이합체임을 [160]시사한다.이 단위는 MukF 매개 이합체를 통해 두 개의 ATP 결합 MukBEF 복합체가 결합함으로써 형성된다.MukBEF는 세포의 장축에 평행하게 긴 1-3개의 클러스터로 세포에 국재한다.각 클러스터에는 평균 8~10개의 다이머가 포함되어 있습니다.현재 모델에 따르면 MukBEF는 "암벽 등반"[160][170] 방식으로 DNA 루프를 밀어낸다.이합체의 이합체는 염색체에서 분리되지 않고 DNA의 한 부분을 방출하고 새로운 DNA 부분을 포착한다.DNA 루프 외에 음의 슈퍼코일과 인비보 MukBEF 사이의 연결은 MukB 서브유닛이 음의 슈퍼코일을 체외에서 구속하는 능력과 함께 MukBEF가 슈퍼코일을 [171][172][173]생성함으로써 DNA를 구성하는 것을 시사한다.
NAP 및 naRNA의 역할
NAP는 DNA를 더 작은 규모(~1-kb)로 구부리고, 브리징하고, 루프함으로써 염색체 압축에 기여하는 것 외에, 장랭 DNA-DNA 접촉을 촉진함으로써 DNA 응축과 조직화에 참여한다.두 NAP, Fis와 HU는 [126]염색체 전체에서 발생하는 장거리 DNA-DNA 접촉을 촉진하는 주요 주체로 떠올랐다.더 작은 규모(~1-kb)로 잘 알려진 Fis와 HU의 DNA 구성 활동이 어떻게 장거리 DNA-DNA 상호작용을 형성하는지 연구해야 한다.그럼에도 불구하고, 일부 HU 매개 DNA 상호작용은 naRNA4의 [86]존재를 필요로 한다. naRNA4는 또한 장거리 DNA 접촉에 참여한다.HU는 RNA가 DNA 접촉 형성에 다른 NAP와 함께 참여한다는 것을 암시하는 것이 아니라 일부 접촉의 촉매 역할을 한다.HU는 또한 장거리 DNA-DNA 상호작용을 촉진하기 위해 MukB와 함께 작용하는 것으로 보인다.이러한 견해는 HU 또는 MukB의 부재로 동일한 DNA-DNA 접촉이 감소했다는 관찰에 기초하고 있다.MukB와 HU가 DNA-DNA 상호작용을 촉진하는데 잠재적으로 어떻게 함께 작용하는지 불분명하다.두 단백질이 물리적으로 상호작용하는 것이 가능하다.또는 MukBEF가 큰 DNA 루프를 압출하는 동안 HU는 이러한 [169][48]루프를 응축하고 정리합니다.
대장균의 기능성 유전자는 유전적 거리상 멀리 떨어져 있지만 염색체 내 3차원 공간에 물리적으로 함께 있다는 보고가 있다.기능 관련 유전자의 공간적 근접성은 생물학적 기능을 더욱 세분화하고 효율적으로 만들 뿐만 아니라 핵이드의 접힘과 공간적 구성에 기여할 것이다.특정 DNA 위치를 검출하기 위해 형광 마커를 사용한 최근의 연구는 유전적으로 서로 분리된 7개의 rRNA 오퍼론 사이의 쌍방향 물리적 거리를 조사했다.그것은 rrnC를 제외한 모든 운영자들이 물리적으로 [174][175]근접해 있었다고 보고했다.놀랍게도, 3C-seq 연구는 rrn 오퍼론의 물리적 군집을 나타내지 않았으며, 이는 형광 기반 [126]연구의 결과와 배치된다.따라서 이러한 모순된 관찰을 해결하기 위해서는 추가적인 조사가 필요하다.또 다른 예에서 GalR은 염색체에 [176]산재한 GalR 결합 부위의 상호작용 네트워크를 형성한다.GalR은 당 D-갈락토스의 [177]수송 및 대사를 위한 효소를 코드하는 유전자로 구성된 갈락토오스 레귤레이터의 전사 조절제이다.GalR은 셀 내[176] 1~2개의 Foci에만 존재하며 대규모 구조물로 [178]자가 조립할 수 있습니다.따라서 DNA 결합 GalR은 장거리 상호작용을 [176][178]형성하기 위해 다중화하는 것으로 보인다.
글로벌 형태 및 구조
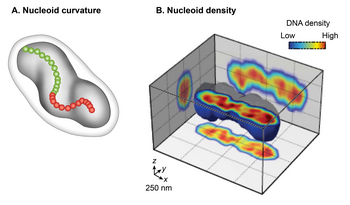
화학적으로 고정된 대장균 세포의 기존 투과전자현미경법(TEM)은 이 핵소체를 불규칙한 형태의 소기관으로 묘사했다.그러나 살아 있는 핵소체를 3D로 광시야 형광 촬영한 결과 이산 타원체 형태가 [3][14][15]밝혀졌다.세포의 위상 대비 영상과 핵양체의 형광 영상의 중첩은 세포 주변까지 핵양체의 전체 길이를 따라 반경 차원에서만 근접한 병렬을 보였다.이 소견은 핵이 [13]방사상으로 구속되어 있음을 나타낸다.장축에 수직으로 교차한 후 3D 형광 영상을 자세히 검사한 결과, 곡률 및 세로 고밀도 영역의 두 가지 전역적 특징이 추가로 발견되었습니다.각 단면의 강도 중심을 연결하여 핵의 중심선의 키랄리티를 검사한 결과, 전체 핵의 형태가 [15]곡선으로 되어 있었다.단면의 형광 강도 분포는 중심핵의 곡선형 고밀도 영역 또는 다발과 [13][14]주변부의 저밀도 영역으로 구성된 밀도 하위구조를 나타냈다.반지름 구속의 한 가지 의미는 그것이 핵양체의 곡선 형태를 결정한다는 것이다.한 모델에 따르면 핵이드는 구부릴 수 있는 길이(지속성 길이)[13]보다 반경이 작은 원통형 대장균 세포에 갇히기 때문에 강제로 구부러진다.이 모델은 세포벽 제거 또는 세포벽 합성의 저해가 세포 반지름을 증가시키고 나선 반지름의 증가 및 [13]핵이드의 나선 피치의 감소를 수반하는 관찰에 의해 뒷받침되었다.
핵양막결합
DNA-막 접합에 의한 팽창력은 핵이드의 최적의 응축 수준을 유지하기 위해 응축력과 반대로 기능하는 것으로 보인다.세포 분절과 전자 현미경 연구는 DNA-막 연결의 [179][180]가능성을 처음으로 나타냈다.현재 DNA-막 연결의 몇 가지 알려진 예가 있습니다.전이(transertion)는 일시적인 DNA-막 [181]접점을 형성하는 초기 막 단백질의 동시 전사, 번역, 삽입 메커니즘이다.두 개의 막 단백질 LacY와 TetA의 전이는 염색체 자리의 [182]막 쪽으로의 위치 변경을 일으키는 것으로 입증되었다.핵막 결합의 또 다른 메커니즘은 막에 고정된 전사 조절제와 염색체 내 표적 부위 사이의 직접 접촉을 통해 이루어진다.대장균의 전사조절제와 같은 예로는 CadC가 있다.CadC는 증배성 감각 도메인과 세포질 DNA 결합 도메인을 포함한다.CadC의 표적 유전자의 전사를 활성화하는 CadC의 DNA 결합 활성을 자극하는 주증압성 감각 영역에 의한 산성 환경의 감지.[183]막앵커 부착 전사조절기에 의해 조절되는 유전자의 막 국재성은 아직 입증되지 않았다.그럼에도 불구하고, 이러한 조절자에 의한 염색체 내 표적 유전자의 활성화는 동적 접촉이지만 핵-막 접촉의 결과를 가져올 것으로 예상된다.이들 예 외에 염색체는 DNA 결합 단백질(예를 들어 SlmA, MatP)과 디비좀 사이의 [184][185]단백질-단백질 상호작용을 통해 세포막에 특이적으로 고정된다.유전자를 코드하는 막-단백질이 게놈 전체에 분포하기 때문에 전이를 통한 동적 DNA-막 접촉이 핵상 팽창력으로 작용할 수 있다.이 팽창력은 최적의 응축 수준을 유지하기 위해 응축력과 반대로 기능한다.대장균 세포가 클로람페니콜에 노출되었을 때 고도로 응축된 핵소체가 형성되어 번역이 차단되며,[186][187] 이는 투석을 통해 형성된 일시적인 DNA-막 접촉의 팽창력을 뒷받침한다.클로람페니콜 처리 후 과도하게 응축된 핵소체의 둥근 모양은 또한 핵소체의 [187]타원체 형태를 정의하는 데 있어 투과 매개 DNA-막 접촉에 대한 역할을 시사한다.
시각화
핵소체는 전자 현미경으로 매우 높은 배율로 선명하게 시각화할 수 있으며, 외관은 다를 수 있지만 세포질에 [188]대해 명확하게 볼 수 있다.때때로 DNA로 생각되는 가닥도 보인다.특이적으로 DNA를 염색하는 Feulgen 염색으로 염색함으로써 광현미경으로도 [189]핵소체를 볼 수 있다.DNA 인터컬레이션 염색체 DAPI와 브롬화 에티듐은 핵체의 형광 현미경 검사에 널리 사용된다.그것은 불규칙한 모양을 하고 있고 원핵 [13][14]세포에서 발견된다.
DNA 손상 및 복구
DNA 손상 조건에 노출된 후 세균 및 고세균의 핵상 구조 변화가 관찰된다.아틸리스균과 대장균균의 핵소체는 모두 자외선 조사 [190][191]후 현저하게 작아진다.대장균에서 콤팩트 구조를 형성하려면 특정 RecA-DNA [192]상호작용을 통한 RecA 활성화가 필요하다.RecA 단백질은 DNA 손상의 상동 재조합 수복에 중요한 역할을 한다.
위의 B. subtilis 및 대장균과 유사하게, 손상 DNA가 핵의 압축과 재조직화를 야기한다는 것을 강조하기 위해 고세균 Haloferax 화산의 노출은 핵의 [193]재조합을 야기한다.압축은 DNA의 이중 가닥 파괴의 상동 재조합 수복의 초기 단계를 촉매하는 Mre11-Rad50 단백질 복합체에 의존합니다.핵소체 압축은 DNA가 목표물의 위치를 찾기 위해 단백질을 복구하는 것을 돕고 상동 [193]재조합 동안 온전한 DNA 서열을 찾는 것을 용이하게 함으로써 세포 회복을 가속화하는 DNA 손상 반응의 일부라고 제안되었다.
「 」를 참조해 주세요.
레퍼런스
![]() 이 기사는 CC BY 4.0 면허(2019)에 따라 다음 출처에서 수정되었다(검토자 보고서). Subhash Verma; Zhong Qian; Sankar L Adhya (December 2019). "Architecture of the Escherichia coli nucleoid". PLOS Genetics. 15 (12): e1008456. doi:10.1371/JOURNAL.PGEN.1008456. ISSN 1553-7390. PMC 6907758. PMID 31830036. Wikidata Q84825966.
이 기사는 CC BY 4.0 면허(2019)에 따라 다음 출처에서 수정되었다(검토자 보고서). Subhash Verma; Zhong Qian; Sankar L Adhya (December 2019). "Architecture of the Escherichia coli nucleoid". PLOS Genetics. 15 (12): e1008456. doi:10.1371/JOURNAL.PGEN.1008456. ISSN 1553-7390. PMC 6907758. PMID 31830036. Wikidata Q84825966.
- ^ Thanbichler M, Wang SC, Shapiro L (October 2005). "The bacterial nucleoid: a highly organized and dynamic structure". Journal of Cellular Biochemistry. 96 (3): 506–21. doi:10.1002/jcb.20519. PMID 15988757.
- ^ a b c Dame RT, Tark-Dame M (June 2016). "Bacterial chromatin: converging views at different scales". Current Opinion in Cell Biology. 40: 60–65. doi:10.1016/j.ceb.2016.02.015. PMID 26942688.
- ^ a b c Kleckner N, Fisher JK, Stouf M, White MA, Bates D, Witz G (December 2014). "The bacterial nucleoid: nature, dynamics and sister segregation". Current Opinion in Microbiology. 22: 127–37. doi:10.1016/j.mib.2014.10.001. PMC 4359759. PMID 25460806.
- ^ a b Bloomfield VA (1997). "DNA condensation by multivalent cations". Biopolymers. 44 (3): 269–82. doi:10.1002/(SICI)1097-0282(1997)44:3<269::AID-BIP6>3.0.CO;2-T. PMID 9591479.
- ^ a b c d Trun NJ, Marko JF (1998). "Architecture of a bacterial chromosome" (PDF). American Society of Microbiology News. 64 (5): 276–283.
- ^ Surovtsev, Ivan V.; Jacobs-Wagner, Christine (March 2018). "Subcellular Organization: A Critical Feature of Bacterial Cell Replication". Cell. 172 (6): 1271–1293. doi:10.1016/j.cell.2018.01.014. PMC 5870143. PMID 29522747.
- ^ a b Stonington OG, Pettijohn DE (January 1971). "The folded genome of Escherichia coli isolated in a protein-DNA-RNA complex". Proceedings of the National Academy of Sciences of the United States of America. 68 (1): 6–9. Bibcode:1971PNAS...68....6S. doi:10.1073/pnas.68.1.6. PMC 391088. PMID 4924971.
- ^ Worcel A, Burgi E (November 1972). "On the structure of the folded chromosome of Escherichia coli". Journal of Molecular Biology. 71 (2): 127–47. doi:10.1016/0022-2836(72)90342-7. PMID 4564477.
- ^ a b c d e Kano Y, Goshima N, Wada M, Imamoto F (1989). "Participation of hup gene product in replicative transposition of Mu phage in Escherichia coli". Gene. 76 (2): 353–8. doi:10.1016/0378-1119(89)90175-3. PMID 2666261.
- ^ a b c d e Ogura T, Niki H, Kano Y, Imamoto F, Hiraga S (January 1990). "Maintenance of plasmids in HU and IHF mutants of Escherichia coli". Molecular & General Genetics. 220 (2): 197–203. doi:10.1007/bf00260482. PMID 2183003. S2CID 10701528.
- ^ a b c d e Hwang DS, Kornberg A (November 1992). "Opening of the replication origin of Escherichia coli by DnaA protein with protein HU or IHF". The Journal of Biological Chemistry. 267 (32): 23083–6. doi:10.1016/S0021-9258(18)50059-4. PMID 1429655.
- ^ a b Valens M, Penaud S, Rossignol M, Cornet F, Boccard F (October 2004). "Macrodomain organization of the Escherichia coli chromosome". The EMBO Journal. 23 (21): 4330–41. doi:10.1038/sj.emboj.7600434. PMC 524398. PMID 15470498.
- ^ a b c d e f g Fisher JK, Bourniquel A, Witz G, Weiner B, Prentiss M, Kleckner N (May 2013). "Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells". Cell. 153 (4): 882–95. doi:10.1016/j.cell.2013.04.006. PMC 3670778. PMID 23623305.
- ^ a b c d e Le Gall A, Cattoni DI, Guilhas B, Mathieu-Demazière C, Oudjedi L, Fiche JB, et al. (July 2016). "Bacterial partition complexes segregate within the volume of the nucleoid". Nature Communications. 7: 12107. Bibcode:2016NatCo...712107L. doi:10.1038/ncomms12107. PMC 4935973. PMID 27377966.
- ^ a b c Hadizadeh Yazdi N, Guet CC, Johnson RC, Marko JF (December 2012). "Variation of the folding and dynamics of the Escherichia coli chromosome with growth conditions". Molecular Microbiology. 86 (6): 1318–33. doi:10.1111/mmi.12071. PMC 3524407. PMID 23078205.
- ^ Olins AL, Olins DE (January 1974). "Spheroid chromatin units (v bodies)". Science. 183 (4122): 330–2. Bibcode:1974Sci...183..330O. doi:10.1126/science.183.4122.330. PMID 4128918. S2CID 83480762.
- ^ a b Luger K, Mäder AW, Richmond RK, Sargent DF, Richmond TJ (September 1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature. 389 (6648): 251–60. Bibcode:1997Natur.389..251L. doi:10.1038/38444. PMID 9305837. S2CID 4328827.
- ^ a b Khorasanizadeh S (January 2004). "The nucleosome: from genomic organization to genomic regulation". Cell. 116 (2): 259–72. doi:10.1016/s0092-8674(04)00044-3. PMID 14744436. S2CID 15504162.
- ^ Talukder A, Ishihama A (September 2015). "Growth phase dependent changes in the structure and protein composition of nucleoid in Escherichia coli". Science China Life Sciences. 58 (9): 902–11. doi:10.1007/s11427-015-4898-0. PMID 26208826.
- ^ a b c d e f Azam TA, Ishihama A (November 1999). "Twelve species of the nucleoid-associated protein from Escherichia coli. Sequence recognition specificity and DNA binding affinity". The Journal of Biological Chemistry. 274 (46): 33105–13. doi:10.1074/jbc.274.46.33105. PMID 10551881. S2CID 9807664.
- ^ a b c d e f g Ali Azam T, Iwata A, Nishimura A, Ueda S, Ishihama A (October 1999). "Growth phase-dependent variation in protein composition of the Escherichia coli nucleoid". Journal of Bacteriology. 181 (20): 6361–70. doi:10.1128/JB.181.20.6361-6370.1999. PMC 103771. PMID 10515926.
- ^ a b Swinger KK, Lemberg KM, Zhang Y, Rice PA (July 2003). "Flexible DNA bending in HU-DNA cocrystal structures". The EMBO Journal. 22 (14): 3749–60. doi:10.1093/emboj/cdg351. PMC 165621. PMID 12853489.
- ^ a b Guo F, Adhya S (March 2007). "Spiral structure of Escherichia coli HUalphabeta provides foundation for DNA supercoiling". Proceedings of the National Academy of Sciences of the United States of America. 104 (11): 4309–14. Bibcode:2007PNAS..104.4309G. doi:10.1073/pnas.0611686104. PMC 1838598. PMID 17360520.
- ^ a b c Pinson V, Takahashi M, Rouviere-Yaniv J (April 1999). "Differential binding of the Escherichia coli HU, homodimeric forms and heterodimeric form to linear, gapped and cruciform DNA". Journal of Molecular Biology. 287 (3): 485–97. doi:10.1006/jmbi.1999.2631. PMID 10092454.
- ^ Craig NL, Nash HA (December 1984). "E. coli integration host factor binds to specific sites in DNA". Cell. 39 (3 Pt 2): 707–16. doi:10.1016/0092-8674(84)90478-1. PMID 6096022. S2CID 26758055.
- ^ a b Ou HD, Phan S, Deerinck TJ, Thor A, Ellisman MH, O'Shea CC (July 2017). "ChromEMT: Visualizing 3D chromatin structure and compaction in interphase and mitotic cells". Science. 357 (6349): eaag0025. doi:10.1126/science.aag0025. PMC 5646685. PMID 28751582.
- ^ a b c Lang B, Blot N, Bouffartigues E, Buckle M, Geertz M, Gualerzi CO, et al. (September 2007). "High-affinity DNA binding sites for H-NS provide a molecular basis for selective silencing within proteobacterial genomes". Nucleic Acids Research. 35 (18): 6330–7. doi:10.1093/nar/gkm712. PMC 2094087. PMID 17881364.
- ^ a b c Gulvady R, Gao Y, Kenney LJ, Yan J (November 2018). "A single molecule analysis of H-NS uncouples DNA binding affinity from DNA specificity". Nucleic Acids Research. 46 (19): 10216–10224. doi:10.1093/nar/gky826. PMC 6212787. PMID 30239908.
- ^ a b c Shao Y, Feldman-Cohen LS, Osuna R (February 2008). "Functional characterization of the Escherichia coli Fis-DNA binding sequence". Journal of Molecular Biology. 376 (3): 771–85. doi:10.1016/j.jmb.2007.11.101. PMC 2292415. PMID 18178221.
- ^ a b c d Stella S, Cascio D, Johnson RC (April 2010). "The shape of the DNA minor groove directs binding by the DNA-bending protein Fis". Genes & Development. 24 (8): 814–26. doi:10.1101/gad.1900610. PMC 2854395. PMID 20395367.
- ^ Narayan K, Subramaniam S (November 2015). "Focused ion beams in biology". Nature Methods. 12 (11): 1021–31. doi:10.1038/nmeth.3623. PMC 6993138. PMID 26513553.
- ^ a b c d Mercier R, Petit MA, Schbath S, Robin S, El Karoui M, Boccard F, Espéli O (October 2008). "The MatP/matS site-specific system organizes the terminus region of the E. coli chromosome into a macrodomain" (PDF). Cell. 135 (3): 475–85. doi:10.1016/j.cell.2008.08.031. PMID 18984159. S2CID 3582710.
- ^ Rouvière-Yaniv J, Gros F (September 1975). "Characterization of a novel, low-molecular-weight DNA-binding protein from Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 72 (9): 3428–32. Bibcode:1975PNAS...72.3428R. doi:10.1073/pnas.72.9.3428. PMC 433007. PMID 1103148.
- ^ Suryanarayana T, Subramanian AR (September 1978). "Specific association of two homologous DNA-binding proteins to the native 30-S ribosomal subunits of Escherichia coli". Biochimica et Biophysica Acta (BBA) - Nucleic Acids and Protein Synthesis. 520 (2): 342–57. doi:10.1016/0005-2787(78)90232-0. PMID 213117.
- ^ Mende L, Timm B, Subramanian R (December 1978). "Primary structures of two homologous ribosome-associated DNA-binding proteins of Escherichia coli". FEBS Letters. 96 (2): 395–8. doi:10.1016/0014-5793(78)80446-3. PMID 215461. S2CID 39245157.
- ^ Megraw TL, Chae CB (June 1993). "Functional complementarity between the HMG1-like yeast mitochondrial histone HM and the bacterial histone-like protein HU". The Journal of Biological Chemistry. 268 (17): 12758–63. doi:10.1016/S0021-9258(18)31453-4. PMID 8509411.
- ^ Paull TT, Johnson RC (April 1995). "DNA looping by Saccharomyces cerevisiae high mobility group proteins NHP6A/B. Consequences for nucleoprotein complex assembly and chromatin condensation". The Journal of Biological Chemistry. 270 (15): 8744–54. doi:10.1074/jbc.270.15.8744. PMID 7721780.
- ^ Kamashev D, Rouviere-Yaniv J (December 2000). "The histone-like protein HU binds specifically to DNA recombination and repair intermediates". The EMBO Journal. 19 (23): 6527–35. doi:10.1093/emboj/19.23.6527. PMC 305869. PMID 11101525.
- ^ Shindo H, Furubayashi A, Shimizu M, Miyake M, Imamoto F (April 1992). "Preferential binding of E.coli histone-like protein HU alpha to negatively supercoiled DNA". Nucleic Acids Research. 20 (7): 1553–8. doi:10.1093/nar/20.7.1553. PMC 312237. PMID 1579448.
- ^ Pontiggia A, Negri A, Beltrame M, Bianchi ME (February 1993). "Protein HU binds specifically to kinked DNA". Molecular Microbiology. 7 (3): 343–50. doi:10.1111/j.1365-2958.1993.tb01126.x. PMID 8459763. S2CID 39362449.
- ^ Bonnefoy E, Takahashi M, Yaniv JR (September 1994). "DNA-binding parameters of the HU protein of Escherichia coli to cruciform DNA". Journal of Molecular Biology. 242 (2): 116–29. doi:10.1006/jmbi.1994.1563. PMID 8089835.
- ^ Castaing B, Zelwer C, Laval J, Boiteux S (April 1995). "HU protein of Escherichia coli binds specifically to DNA that contains single-strand breaks or gaps". The Journal of Biological Chemistry. 270 (17): 10291–6. doi:10.1074/jbc.270.17.10291. PMID 7730334.
- ^ Lyubchenko YL, Shlyakhtenko LS, Aki T, Adhya S (February 1997). "Atomic force microscopic demonstration of DNA looping by GalR and HU". Nucleic Acids Research. 25 (4): 873–6. doi:10.1093/nar/25.4.873. PMC 146491. PMID 9016640.
- ^ Swinger KK, Rice PA (January 2007). "Structure-based analysis of HU-DNA binding". Journal of Molecular Biology. 365 (4): 1005–16. doi:10.1016/j.jmb.2006.10.024. PMC 1945228. PMID 17097674.
- ^ a b c d e f g Hammel M, Amlanjyoti D, Reyes FE, Chen JH, Parpana R, Tang HY, et al. (July 2016). "HU multimerization shift controls nucleoid compaction". Science Advances. 2 (7): e1600650. Bibcode:2016SciA....2E0650H. doi:10.1126/sciadv.1600650. PMC 4966879. PMID 27482541.
- ^ a b c d e Prieto AI, Kahramanoglou C, Ali RM, Fraser GM, Seshasayee AS, Luscombe NM (April 2012). "Genomic analysis of DNA binding and gene regulation by homologous nucleoid-associated proteins IHF and HU in Escherichia coli K12". Nucleic Acids Research. 40 (8): 3524–37. doi:10.1093/nar/gkr1236. PMC 3333857. PMID 22180530.
- ^ a b c d e f Macvanin M, Edgar R, Cui F, Trostel A, Zhurkin V, Adhya S (November 2012). "Noncoding RNAs binding to the nucleoid protein HU in Escherichia coli". Journal of Bacteriology. 194 (22): 6046–55. doi:10.1128/JB.00961-12. PMC 3486375. PMID 22942248.
- ^ a b c d e van Noort J, Verbrugge S, Goosen N, Dekker C, Dame RT (May 2004). "Dual architectural roles of HU: formation of flexible hinges and rigid filaments". Proceedings of the National Academy of Sciences of the United States of America. 101 (18): 6969–74. Bibcode:2004PNAS..101.6969V. doi:10.1073/pnas.0308230101. PMC 406450. PMID 15118104.
- ^ Sarkar R, Rybenkov VV (2016-12-06). "A Guide to Magnetic Tweezers and Their Applications". Frontiers in Physics. 4: 48. Bibcode:2016FrP.....4...48S. doi:10.3389/fphy.2016.00048. S2CID 44183628.
- ^ a b c d Kahramanoglou C, Seshasayee AS, Prieto AI, Ibberson D, Schmidt S, Zimmermann J, et al. (March 2011). "Direct and indirect effects of H-NS and Fis on global gene expression control in Escherichia coli". Nucleic Acids Research. 39 (6): 2073–91. doi:10.1093/nar/gkq934. PMC 3064808. PMID 21097887.
- ^ a b Rice PA, Yang S, Mizuuchi K, Nash HA (December 1996). "Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn". Cell. 87 (7): 1295–306. doi:10.1016/s0092-8674(00)81824-3. PMID 8980235. S2CID 9291279.
- ^ Murtin C, Engelhorn M, Geiselmann J, Boccard F (December 1998). "A quantitative UV laser footprinting analysis of the interaction of IHF with specific binding sites: re-evaluation of the effective concentration of IHF in the cell". Journal of Molecular Biology. 284 (4): 949–61. doi:10.1006/jmbi.1998.2256. PMID 9837718.
- ^ Ditto MD, Roberts D, Weisberg RA (June 1994). "Growth phase variation of integration host factor level in Escherichia coli". Journal of Bacteriology. 176 (12): 3738–48. doi:10.1128/jb.176.12.3738-3748.1994. PMC 205563. PMID 8206852.
- ^ a b c Lin J, Chen H, Dröge P, Yan J (2012). "Physical organization of DNA by multiple non-specific DNA-binding modes of integration host factor (IHF)". PLOS ONE. 7 (11): e49885. Bibcode:2012PLoSO...749885L. doi:10.1371/journal.pone.0049885. PMC 3498176. PMID 23166787.
- ^ Jacquet M, Cukier-Kahn R, Pla J, Gros F (December 1971). "A thermostable protein factor acting on in vitro DNA transcription". Biochemical and Biophysical Research Communications. 45 (6): 1597–607. doi:10.1016/0006-291x(71)90204-x. PMID 4942735.
- ^ Cukier-Kahn R, Jacquet M, Gros F (December 1972). "Two heat-resistant, low molecular weight proteins from Escherichia coli that stimulate DNA-directed RNA synthesis". Proceedings of the National Academy of Sciences of the United States of America. 69 (12): 3643–7. Bibcode:1972PNAS...69.3643C. doi:10.1073/pnas.69.12.3643. PMC 389839. PMID 4566454.
- ^ Spassky A, Buc HC (November 1977). "Physico-chemical properties of a DNA binding protein: Escherichia coli factor H1". European Journal of Biochemistry. 81 (1): 79–90. doi:10.1111/j.1432-1033.1977.tb11929.x. PMID 338303.
- ^ Varshavsky AJ, Nedospasov SA, Bakayev VV, Bakayeva TG, Georgiev GP (August 1977). "Histone-like proteins in the purified Escherichia coli deoxyribonucleoprotein". Nucleic Acids Research. 4 (8): 2725–45. doi:10.1093/nar/4.8.2725. PMC 342604. PMID 333393.
- ^ Falconi M, Gualtieri MT, La Teana A, Losso MA, Pon CL (May 1988). "Proteins from the prokaryotic nucleoid: primary and quaternary structure of the 15-kD Escherichia coli DNA binding protein H-NS". Molecular Microbiology. 2 (3): 323–9. doi:10.1111/j.1365-2958.1988.tb00035.x. PMID 3135462. S2CID 36215353.
- ^ Ueguchi C, Suzuki T, Yoshida T, Tanaka K, Mizuno T (October 1996). "Systematic mutational analysis revealing the functional domain organization of Escherichia coli nucleoid protein H-NS". Journal of Molecular Biology. 263 (2): 149–62. doi:10.1006/jmbi.1996.0566. PMID 8913298.
- ^ a b Rimsky S, Zuber F, Buckle M, Buc H (December 2001). "A molecular mechanism for the repression of transcription by the H-NS protein". Molecular Microbiology. 42 (5): 1311–23. doi:10.1046/j.1365-2958.2001.02706.x. PMID 11886561. S2CID 32623528.
- ^ Bouffartigues E, Buckle M, Badaut C, Travers A, Rimsky S (May 2007). "H-NS cooperative binding to high-affinity sites in a regulatory element results in transcriptional silencing". Nature Structural & Molecular Biology. 14 (5): 441–8. doi:10.1038/nsmb1233. PMID 17435766. S2CID 43768346.
- ^ Dame RT, Wyman C, Goosen N (September 2000). "H-NS mediated compaction of DNA visualised by atomic force microscopy". Nucleic Acids Research. 28 (18): 3504–10. doi:10.1093/nar/28.18.3504. PMC 110753. PMID 10982869.
- ^ Amit R, Oppenheim AB, Stavans J (April 2003). "Increased bending rigidity of single DNA molecules by H-NS, a temperature and osmolarity sensor". Biophysical Journal. 84 (4): 2467–73. Bibcode:2003BpJ....84.2467A. doi:10.1016/S0006-3495(03)75051-6. PMC 1302812. PMID 12668454.
- ^ Dame RT, Noom MC, Wuite GJ (November 2006). "Bacterial chromatin organization by H-NS protein unravelled using dual DNA manipulation". Nature. 444 (7117): 387–90. Bibcode:2006Natur.444..387D. doi:10.1038/nature05283. PMID 17108966. S2CID 4314858.
- ^ a b c d e f Liu Y, Chen H, Kenney LJ, Yan J (February 2010). "A divalent switch drives H-NS/DNA-binding conformations between stiffening and bridging modes". Genes & Development. 24 (4): 339–44. doi:10.1101/gad.1883510. PMC 2816733. PMID 20159954.
- ^ van der Valk RA, Vreede J, Qin L, Moolenaar GF, Hofmann A, Goosen N, Dame RT (September 2017). "Mechanism of environmentally driven conformational changes that modulate H-NS DNA-bridging activity". eLife. 6. doi:10.7554/eLife.27369. PMC 5647153. PMID 28949292.
- ^ Yamada H, Muramatsu S, Mizuno T (September 1990). "An Escherichia coli protein that preferentially binds to sharply curved DNA". Journal of Biochemistry. 108 (3): 420–5. doi:10.1093/oxfordjournals.jbchem.a123216. PMID 2126011.
- ^ Martin-Orozco N, Touret N, Zaharik ML, Park E, Kopelman R, Miller S, et al. (January 2006). "Visualization of vacuolar acidification-induced transcription of genes of pathogens inside macrophages". Molecular Biology of the Cell. 17 (1): 498–510. doi:10.1091/mbc.e04-12-1096. PMC 1345685. PMID 16251362.
- ^ Winardhi RS, Yan J, Kenney LJ (October 2015). "H-NS Regulates Gene Expression and Compacts the Nucleoid: Insights from Single-Molecule Experiments". Biophysical Journal. 109 (7): 1321–9. Bibcode:2015BpJ...109.1321W. doi:10.1016/j.bpj.2015.08.016. PMC 4601063. PMID 26445432.
- ^ Walthers D, Li Y, Liu Y, Anand G, Yan J, Kenney LJ (January 2011). "Salmonella enterica response regulator SsrB relieves H-NS silencing by displacing H-NS bound in polymerization mode and directly activates transcription". The Journal of Biological Chemistry. 286 (3): 1895–902. doi:10.1074/jbc.M110.164962. PMC 3023485. PMID 21059643.
- ^ Gao Y, Foo YH, Winardhi RS, Tang Q, Yan J, Kenney LJ (November 2017). "Charged residues in the H-NS linker drive DNA binding and gene silencing in single cells". Proceedings of the National Academy of Sciences of the United States of America. 114 (47): 12560–12565. doi:10.1073/pnas.1716721114. PMC 5703333. PMID 29109287.
- ^ Hancock SP, Stella S, Cascio D, Johnson RC (2016). "DNA Sequence Determinants Controlling Affinity, Stability and Shape of DNA Complexes Bound by the Nucleoid Protein Fis". PLOS ONE. 11 (3): e0150189. Bibcode:2016PLoSO..1150189H. doi:10.1371/journal.pone.0150189. PMC 4784862. PMID 26959646.
- ^ Kostrewa D, Granzin J, Koch C, Choe HW, Raghunathan S, Wolf W, et al. (January 1991). "Three-dimensional structure of the E. coli DNA-binding protein FIS". Nature. 349 (6305): 178–80. Bibcode:1991Natur.349..178K. doi:10.1038/349178a0. PMID 1986310. S2CID 4318862.
- ^ Kostrewa D, Granzin J, Stock D, Choe HW, Labahn J, Saenger W (July 1992). "Crystal structure of the factor for inversion stimulation FIS at 2.0 A resolution". Journal of Molecular Biology. 226 (1): 209–26. doi:10.1016/0022-2836(92)90134-6. PMID 1619650.
- ^ Cho BK, Knight EM, Barrett CL, Palsson BØ (June 2008). "Genome-wide analysis of Fis binding in Escherichia coli indicates a causative role for A-/AT-tracts". Genome Research. 18 (6): 900–10. doi:10.1101/gr.070276.107. PMC 2413157. PMID 18340041.
- ^ a b Travers A, Muskhelishvili G (June 1998). "DNA microloops and microdomains: a general mechanism for transcription activation by torsional transmission". Journal of Molecular Biology. 279 (5): 1027–43. doi:10.1006/jmbi.1998.1834. PMID 9642081.
- ^ Skoko D, Yan J, Johnson RC, Marko JF (November 2005). "Low-force DNA condensation and discontinuous high-force decondensation reveal a loop-stabilizing function of the protein Fis". Physical Review Letters. 95 (20): 208101. Bibcode:2005PhRvL..95t8101S. doi:10.1103/PhysRevLett.95.208101. PMID 16384101. S2CID 37405026.
- ^ a b Skoko D, Yoo D, Bai H, Schnurr B, Yan J, McLeod SM, et al. (December 2006). "Mechanism of chromosome compaction and looping by the Escherichia coli nucleoid protein Fis". Journal of Molecular Biology. 364 (4): 777–98. doi:10.1016/j.jmb.2006.09.043. PMC 1988847. PMID 17045294.
- ^ Johnson RC, Simon MI (July 1985). "Hin-mediated site-specific recombination requires two 26 bp recombination sites and a 60 bp recombinational enhancer". Cell. 41 (3): 781–91. doi:10.1016/s0092-8674(85)80059-3. PMID 2988787. S2CID 34572809.
- ^ a b Kahmann R, Rudt F, Koch C, Mertens G (July 1985). "G inversion in bacteriophage Mu DNA is stimulated by a site within the invertase gene and a host factor". Cell. 41 (3): 771–80. doi:10.1016/S0092-8674(85)80058-1. PMID 3159478.
- ^ Pettijohn DE, Hecht R (1974). "RNA molecules bound to the folded bacterial genome stabilize DNA folds and segregate domains of supercoiling". Cold Spring Harbor Symposia on Quantitative Biology. 38: 31–41. doi:10.1101/sqb.1974.038.01.006. PMID 4598638.
- ^ a b c Ohniwa RL, Morikawa K, Takeshita SL, Kim J, Ohta T, Wada C, Takeyasu K (October 2007). "Transcription-coupled nucleoid architecture in bacteria". Genes to Cells. 12 (10): 1141–52. doi:10.1111/j.1365-2443.2007.01125.x. PMID 17903174.
- ^ a b c Balandina A, Kamashev D, Rouviere-Yaniv J (August 2002). "The bacterial histone-like protein HU specifically recognizes similar structures in all nucleic acids. DNA, RNA, and their hybrids". The Journal of Biological Chemistry. 277 (31): 27622–8. doi:10.1074/jbc.M201978200. PMID 12006568.
- ^ Balandina A, Claret L, Hengge-Aronis R, Rouviere-Yaniv J (February 2001). "The Escherichia coli histone-like protein HU regulates rpoS translation". Molecular Microbiology. 39 (4): 1069–79. doi:10.1046/j.1365-2958.2001.02305.x. PMID 11251825.
- ^ a b Qian Z, Macvanin M, Dimitriadis EK, He X, Zhurkin V, Adhya S (August 2015). "A New Noncoding RNA Arranges Bacterial Chromosome Organization". mBio. 6 (4): e00998-15. doi:10.1128/mBio.00998-15. PMC 4550694. PMID 26307168.
- ^ Qian Z, Zhurkin VB, Adhya S (November 2017). "Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 114 (46): 12225–12230. doi:10.1073/pnas.1711285114. PMC 5699063. PMID 29087325.
- ^ Bauer WR, Crick FH, White JH (July 1980). "Supercoiled DNA". Scientific American. 243 (1): 100–13. PMID 6256851.
- ^ "Linking coefficient". www.encyclopediaofmath.org. Encyclopedia of Mathematics. Retrieved 2020-02-22.
- ^ a b c Bates AD, Maxwell A (2005). DNA topology (2nd ed.). Oxford: Oxford University Press. ISBN 978-0-19-856709-7. OCLC 56793994.
- ^ a b c Vologodskii, Aleksander Vadimovich. (1992). Topology and physics of circular DNA. CRC Press. ISBN 0-8493-4228-7. OCLC 635611152.
- ^ Sinden RR (2006). DNA structure and function. Elsevier. ISBN 978-0-12-645750-6. OCLC 698461259.
- ^ a b Sinden RR, Carlson JO, Pettijohn DE (October 1980). "Torsional tension in the DNA double helix measured with trimethylpsoralen in living E. coli cells: analogous measurements in insect and human cells". Cell. 21 (3): 773–83. doi:10.1016/0092-8674(80)90440-7. PMID 6254668. S2CID 2503376.
- ^ Griffith JD (February 1976). "Visualization of prokaryotic DNA in a regularly condensed chromatin-like fiber". Proceedings of the National Academy of Sciences of the United States of America. 73 (2): 563–7. Bibcode:1976PNAS...73..563G. doi:10.1073/pnas.73.2.563. PMC 335950. PMID 1108025.
- ^ a b c Postow L, Hardy CD, Arsuaga J, Cozzarelli NR (July 2004). "Topological domain structure of the Escherichia coli chromosome". Genes & Development. 18 (14): 1766–79. doi:10.1101/gad.1207504. PMC 478196. PMID 15256503.
- ^ a b Bliska JB, Cozzarelli NR (March 1987). "Use of site-specific recombination as a probe of DNA structure and metabolism in vivo". Journal of Molecular Biology. 194 (2): 205–18. doi:10.1016/0022-2836(87)90369-x. PMID 3039150.
- ^ Holmes VF, Cozzarelli NR (February 2000). "Closing the ring: links between SMC proteins and chromosome partitioning, condensation, and supercoiling". Proceedings of the National Academy of Sciences of the United States of America. 97 (4): 1322–4. Bibcode:2000PNAS...97.1322H. doi:10.1073/pnas.040576797. PMC 34294. PMID 10677457.
- ^ Champoux JJ (2001). "DNA topoisomerases: structure, function, and mechanism". Annual Review of Biochemistry. 70: 369–413. doi:10.1146/annurev.biochem.70.1.369. PMID 11395412. S2CID 18144189.
- ^ Gellert M, Mizuuchi K, O'Dea MH, Nash HA (November 1976). "DNA gyrase: an enzyme that introduces superhelical turns into DNA". Proceedings of the National Academy of Sciences of the United States of America. 73 (11): 3872–6. Bibcode:1976PNAS...73.3872G. doi:10.1073/pnas.73.11.3872. PMC 431247. PMID 186775.
- ^ a b c Depew RE, Liu LF, Wang JC (January 1978). "Interaction between DNA and Escherichia coli protein omega. Formation of a complex between single-stranded DNA and omega protein". The Journal of Biological Chemistry. 253 (2): 511–8. doi:10.1016/S0021-9258(17)38239-X. PMID 338610.
- ^ Kirkegaard K, Wang JC (October 1978). "Escherichia coli DNA topoisomerase I catalyzed linking of single-stranded rings of complementary base sequences". Nucleic Acids Research. 5 (10): 3811–20. doi:10.1093/nar/5.10.3811. PMC 342711. PMID 214763.
- ^ a b Raji A, Zabel DJ, Laufer CS, Depew RE (June 1985). "Genetic analysis of mutations that compensate for loss of Escherichia coli DNA topoisomerase I". Journal of Bacteriology. 162 (3): 1173–9. doi:10.1128/JB.162.3.1173-1179.1985. PMC 215900. PMID 2987184.
- ^ Dean F, Krasnow MA, Otter R, Matzuk MM, Spengler SJ, Cozzarelli NR (1983). "Escherichia coli type-1 topoisomerases: identification, mechanism, and role in recombination". Cold Spring Harbor Symposia on Quantitative Biology. 47 (2): 769–77. doi:10.1101/sqb.1983.047.01.088. PMID 6305585.
- ^ Zechiedrich EL, Khodursky AB, Bachellier S, Schneider R, Chen D, Lilley DM, Cozzarelli NR (March 2000). "Roles of topoisomerases in maintaining steady-state DNA supercoiling in Escherichia coli". The Journal of Biological Chemistry. 275 (11): 8103–13. doi:10.1074/jbc.275.11.8103. PMID 10713132.
- ^ Kato J, Nishimura Y, Imamura R, Niki H, Hiraga S, Suzuki H (October 1990). "New topoisomerase essential for chromosome segregation in E. coli". Cell. 63 (2): 393–404. doi:10.1016/0092-8674(90)90172-b. PMID 2170028. S2CID 42122316.
- ^ Liu LF, Wang JC (October 1987). "Supercoiling of the DNA template during transcription". Proceedings of the National Academy of Sciences of the United States of America. 84 (20): 7024–7. Bibcode:1987PNAS...84.7024L. doi:10.1073/pnas.84.20.7024. PMC 299221. PMID 2823250.
- ^ a b Kouzine F, Liu J, Sanford S, Chung HJ, Levens D (November 2004). "The dynamic response of upstream DNA to transcription-generated torsional stress". Nature Structural & Molecular Biology. 11 (11): 1092–100. doi:10.1038/nsmb848. PMID 15502847. S2CID 28956216.
- ^ Rouvière-Yaniv J, Yaniv M, Germond JE (June 1979). "E. coli DNA binding protein HU forms nucleosomelike structure with circular double-stranded DNA". Cell. 17 (2): 265–74. doi:10.1016/0092-8674(79)90152-1. PMID 222478. S2CID 28092421.
- ^ Broyles SS, Pettijohn DE (January 1986). "Interaction of the Escherichia coli HU protein with DNA. Evidence for formation of nucleosome-like structures with altered DNA helical pitch". Journal of Molecular Biology. 187 (1): 47–60. doi:10.1016/0022-2836(86)90405-5. PMID 3514923.
- ^ Kundukad B, Cong P, van der Maarel JR, Doyle PS (September 2013). "Time-dependent bending rigidity and helical twist of DNA by rearrangement of bound HU protein". Nucleic Acids Research. 41 (17): 8280–8. doi:10.1093/nar/gkt593. PMC 3783175. PMID 23828037.
- ^ Tupper AE, Owen-Hughes TA, Ussery DW, Santos DS, Ferguson DJ, Sidebotham JM, et al. (January 1994). "The chromatin-associated protein H-NS alters DNA topology in vitro". The EMBO Journal. 13 (1): 258–68. doi:10.1002/j.1460-2075.1994.tb06256.x. PMC 394800. PMID 8306968.
- ^ a b Schneider R, Lurz R, Lüder G, Tolksdorf C, Travers A, Muskhelishvili G (December 2001). "An architectural role of the Escherichia coli chromatin protein FIS in organising DNA". Nucleic Acids Research. 29 (24): 5107–14. doi:10.1093/nar/29.24.5107. PMC 97572. PMID 11812843.
- ^ Schneider R, Travers A, Kutateladze T, Muskhelishvili G (December 1999). "A DNA architectural protein couples cellular physiology and DNA topology in Escherichia coli". Molecular Microbiology. 34 (5): 953–64. doi:10.1046/j.1365-2958.1999.01656.x. PMID 10594821.
- ^ a b Bensaid A, Almeida A, Drlica K, Rouviere-Yaniv J (February 1996). "Cross-talk between topoisomerase I and HU in Escherichia coli". Journal of Molecular Biology. 256 (2): 292–300. doi:10.1006/jmbi.1996.0086. PMID 8594197.
- ^ Malik M, Bensaid A, Rouviere-Yaniv J, Drlica K (February 1996). "Histone-like protein HU and bacterial DNA topology: suppression of an HU deficiency by gyrase mutations". Journal of Molecular Biology. 256 (1): 66–76. doi:10.1006/jmbi.1996.0068. PMID 8609614.
- ^ a b Marians KJ (July 1987). "DNA gyrase-catalyzed decatenation of multiply linked DNA dimers". The Journal of Biological Chemistry. 262 (21): 10362–8. doi:10.1016/S0021-9258(18)61121-4. PMID 3038875.
- ^ a b c Sinden RR, Pettijohn DE (January 1981). "Chromosomes in living Escherichia coli cells are segregated into domains of supercoiling". Proceedings of the National Academy of Sciences of the United States of America. 78 (1): 224–8. Bibcode:1981PNAS...78..224S. doi:10.1073/pnas.78.1.224. PMC 319024. PMID 6165987.
- ^ Higgins NP, Yang X, Fu Q, Roth JR (May 1996). "Surveying a supercoil domain by using the gamma delta resolution system in Salmonella typhimurium". Journal of Bacteriology. 178 (10): 2825–35. doi:10.1128/jb.178.10.2825-2835.1996. PMC 178017. PMID 8631670.
- ^ Yan Y, Ding Y, Leng F, Dunlap D, Finzi L (May 2018). "Protein-mediated loops in supercoiled DNA create large topological domains". Nucleic Acids Research. 46 (9): 4417–4424. doi:10.1093/nar/gky153. PMC 5961096. PMID 29538766.
- ^ Leng F, Chen B, Dunlap DD (December 2011). "Dividing a supercoiled DNA molecule into two independent topological domains". Proceedings of the National Academy of Sciences of the United States of America. 108 (50): 19973–8. Bibcode:2011PNAS..10819973L. doi:10.1073/pnas.1109854108. PMC 3250177. PMID 22123985.
- ^ Moulin L, Rahmouni AR, Boccard F (January 2005). "Topological insulators inhibit diffusion of transcription-induced positive supercoils in the chromosome of Escherichia coli". Molecular Microbiology. 55 (2): 601–10. doi:10.1111/j.1365-2958.2004.04411.x. PMID 15659173.
- ^ Dimri GP, Rudd KE, Morgan MK, Bayat H, Ames GF (July 1992). "Physical mapping of repetitive extragenic palindromic sequences in Escherichia coli and phylogenetic distribution among Escherichia coli strains and other enteric bacteria". Journal of Bacteriology. 174 (14): 4583–93. doi:10.1128/jb.174.14.4583-4593.1992. PMC 206253. PMID 1624447.
- ^ Deng S, Stein RA, Higgins NP (March 2004). "Transcription-induced barriers to supercoil diffusion in the Salmonella typhimurium chromosome". Proceedings of the National Academy of Sciences of the United States of America. 101 (10): 3398–403. Bibcode:2004PNAS..101.3398D. doi:10.1073/pnas.0307550101. PMC 373473. PMID 14993611.
- ^ Deng S, Stein RA, Higgins NP (September 2005). "Organization of supercoil domains and their reorganization by transcription". Molecular Microbiology. 57 (6): 1511–21. doi:10.1111/j.1365-2958.2005.04796.x. PMC 1382059. PMID 16135220.
- ^ Booker BM, Deng S, Higgins NP (December 2010). "DNA topology of highly transcribed operons in Salmonella enterica serovar Typhimurium". Molecular Microbiology. 78 (6): 1348–64. doi:10.1111/j.1365-2958.2010.07394.x. PMID 21143310.
- ^ a b c d e f g h i j k l m n Lioy VS, Cournac A, Marbouty M, Duigou S, Mozziconacci J, Espéli O, et al. (February 2018). "Multiscale Structuring of the E. coli Chromosome by Nucleoid-Associated and Condensin Proteins". Cell. 172 (4): 771–783.e18. doi:10.1016/j.cell.2017.12.027. PMID 29358050.
- ^ Almirón M, Link AJ, Furlong D, Kolter R (December 1992). "A novel DNA-binding protein with regulatory and protective roles in starved Escherichia coli". Genes & Development. 6 (12B): 2646–54. doi:10.1101/gad.6.12b.2646. PMID 1340475. S2CID 10308477.
- ^ a b Frenkiel-Krispin D, Ben-Avraham I, Englander J, Shimoni E, Wolf SG, Minsky A (January 2004). "Nucleoid restructuring in stationary-state bacteria". Molecular Microbiology. 51 (2): 395–405. doi:10.1046/j.1365-2958.2003.03855.x. PMID 14756781.
- ^ Ball CA, Osuna R, Ferguson KC, Johnson RC (December 1992). "Dramatic changes in Fis levels upon nutrient upshift in Escherichia coli". Journal of Bacteriology. 174 (24): 8043–56. doi:10.1128/jb.174.24.8043-8056.1992. PMC 207543. PMID 1459953.
- ^ Claret L, Rouviere-Yaniv J (October 1997). "Variation in HU composition during growth of Escherichia coli: the heterodimer is required for long term survival". Journal of Molecular Biology. 273 (1): 93–104. doi:10.1006/jmbi.1997.1310. PMID 9367749.
- ^ a b c Badrinarayanan A, Lesterlin C, Reyes-Lamothe R, Sherratt D (September 2012). "The Escherichia coli SMC complex, MukBEF, shapes nucleoid organization independently of DNA replication". Journal of Bacteriology. 194 (17): 4669–76. doi:10.1128/JB.00957-12. PMC 3415497. PMID 22753058.
- ^ a b c Petrushenko ZM, Lai CH, Rybenkov VV (November 2006). "Antagonistic interactions of kleisins and DNA with bacterial Condensin MukB". The Journal of Biological Chemistry. 281 (45): 34208–17. doi:10.1074/jbc.M606723200. PMC 1634889. PMID 16982609.
- ^ a b c Lal A, Dhar A, Trostel A, Kouzine F, Seshasayee AS, Adhya S (March 2016). "Genome scale patterns of supercoiling in a bacterial chromosome". Nature Communications. 7: 11055. Bibcode:2016NatCo...711055L. doi:10.1038/ncomms11055. PMC 4820846. PMID 27025941.
- ^ a b Kar S, Edgar R, Adhya S (November 2005). "Nucleoid remodeling by an altered HU protein: reorganization of the transcription program". Proceedings of the National Academy of Sciences of the United States of America. 102 (45): 16397–402. Bibcode:2005PNAS..10216397K. doi:10.1073/pnas.0508032102. PMC 1283455. PMID 16258062.
- ^ Lim CJ, Lee SY, Kenney LJ, Yan J (2012). "Nucleoprotein filament formation is the structural basis for bacterial protein H-NS gene silencing". Scientific Reports. 2: 509. Bibcode:2012NatSR...2E.509L. doi:10.1038/srep00509. PMC 3396134. PMID 22798986.
- ^ Aki T, Choy HE, Adhya S (February 1996). "Histone-like protein HU as a specific transcriptional regulator: co-factor role in repression of gal transcription by GAL repressor". Genes to Cells. 1 (2): 179–88. doi:10.1046/j.1365-2443.1996.d01-236.x. PMID 9140062.
- ^ Pagel JM, Winkelman JW, Adams CW, Hatfield GW (April 1992). "DNA topology-mediated regulation of transcription initiation from the tandem promoters of the ilvGMEDA operon of Escherichia coli". Journal of Molecular Biology. 224 (4): 919–35. doi:10.1016/0022-2836(92)90460-2. PMID 1569580.
- ^ Parekh BS, Hatfield GW (February 1996). "Transcriptional activation by protein-induced DNA bending: evidence for a DNA structural transmission model". Proceedings of the National Academy of Sciences of the United States of America. 93 (3): 1173–7. Bibcode:1996PNAS...93.1173P. doi:10.1073/pnas.93.3.1173. PMC 40051. PMID 8577735.
- ^ a b c Dorman CJ, Dorman MJ (September 2016). "DNA supercoiling is a fundamental regulatory principle in the control of bacterial gene expression". Biophysical Reviews. 8 (3): 209–220. doi:10.1007/s12551-016-0205-y. PMC 5425793. PMID 28510224.
- ^ Chong S, Chen C, Ge H, Xie XS (July 2014). "Mechanism of transcriptional bursting in bacteria". Cell. 158 (2): 314–326. doi:10.1016/j.cell.2014.05.038. PMC 4105854. PMID 25036631.
- ^ Berger M, Gerganova V, Berger P, Rapiteanu R, Lisicovas V, Dobrindt U (August 2016). "Genes on a Wire: The Nucleoid-Associated Protein HU Insulates Transcription Units in Escherichia coli". Scientific Reports. 6: 31512. Bibcode:2016NatSR...631512B. doi:10.1038/srep31512. PMC 4992867. PMID 27545593.
- ^ Koli P, Sudan S, Fitzgerald D, Adhya S, Kar S (2011). "Conversion of commensal Escherichia coli K-12 to an invasive form via expression of a mutant histone-like protein". mBio. 2 (5). doi:10.1128/mBio.00182-11. PMC 3172693. PMID 21896677.
- ^ a b c Kar S, Choi EJ, Guo F, Dimitriadis EK, Kotova SL, Adhya S (December 2006). "Right-handed DNA supercoiling by an octameric form of histone-like protein HU: modulation of cellular transcription". The Journal of Biological Chemistry. 281 (52): 40144–53. doi:10.1074/jbc.M605576200. PMID 17062578.
- ^ a b Kar S, Choi EJ, Guo F, Dimitriadis EK, Kotova SL, Adhya S (December 2006). "Right-handed DNA supercoiling by an octameric form of histone-like protein HU: modulation of cellular transcription". The Journal of Biological Chemistry. 281 (52): 40144–53. doi:10.1074/jbc.M605576200. PMID 17062578.
- ^ a b Uhlmann F (July 2016). "SMC complexes: from DNA to chromosomes". Nature Reviews Molecular Cell Biology. 17 (7): 399–412. doi:10.1038/nrm.2016.30. PMID 27075410. S2CID 20398243.
- ^ Dekker J, Rippe K, Dekker M, Kleckner N (February 2002). "Capturing chromosome conformation". Science. 295 (5558): 1306–11. Bibcode:2002Sci...295.1306D. doi:10.1126/science.1067799. PMID 11847345. S2CID 3561891.
- ^ Lieberman-Aiden E, van Berkum NL, Williams L, Imakaev M, Ragoczy T, Telling A, et al. (October 2009). "Comprehensive mapping of long-range interactions reveals folding principles of the human genome". Science. 326 (5950): 289–93. Bibcode:2009Sci...326..289L. doi:10.1126/science.1181369. PMC 2858594. PMID 19815776.
- ^ a b c d Le TB, Imakaev MV, Mirny LA, Laub MT (November 2013). "High-resolution mapping of the spatial organization of a bacterial chromosome". Science. 342 (6159): 731–4. Bibcode:2013Sci...342..731L. doi:10.1126/science.1242059. PMC 3927313. PMID 24158908.
- ^ Wang X, Le TB, Lajoie BR, Dekker J, Laub MT, Rudner DZ (August 2015). "Condensin promotes the juxtaposition of DNA flanking its loading site in Bacillus subtilis". Genes & Development. 29 (15): 1661–75. doi:10.1101/gad.265876.115. PMC 4536313. PMID 26253537.
- ^ Dekker J, Heard E (October 2015). "Structural and functional diversity of Topologically Associating Domains". FEBS Letters. 589 (20 Pt A): 2877–84. doi:10.1016/j.febslet.2015.08.044. PMC 4598308. PMID 26348399.
- ^ Niki H, Hiraga S (April 1998). "Polar localization of the replication origin and terminus in Escherichia coli nucleoids during chromosome partitioning". Genes & Development. 12 (7): 1036–45. doi:10.1101/gad.12.7.1036. PMC 316681. PMID 9531540.
- ^ Niki H, Yamaichi Y, Hiraga S (January 2000). "Dynamic organization of chromosomal DNA in Escherichia coli". Genes & Development. 14 (2): 212–23. doi:10.1101/gad.14.2.212. PMC 316355. PMID 10652275.
- ^ Boccard F, Esnault E, Valens M (July 2005). "Spatial arrangement and macrodomain organization of bacterial chromosomes". Molecular Microbiology. 57 (1): 9–16. doi:10.1111/j.1365-2958.2005.04651.x. PMID 15948945.
- ^ Duigou S, Boccard F (May 2017). "Long range chromosome organization in Escherichia coli: The position of the replication origin defines the non-structured regions and the Right and Left macrodomains". PLOS Genetics. 13 (5): e1006758. doi:10.1371/journal.pgen.1006758. PMC 5441646. PMID 28486476.
- ^ a b c Nolivos S, Upton AL, Badrinarayanan A, Müller J, Zawadzka K, Wiktor J, et al. (January 2016). "MatP regulates the coordinated action of topoisomerase IV and MukBEF in chromosome segregation". Nature Communications. 7: 10466. Bibcode:2016NatCo...710466N. doi:10.1038/ncomms10466. PMC 4738335. PMID 26818444.
- ^ a b Dupaigne P, Tonthat NK, Espéli O, Whitfill T, Boccard F, Schumacher MA (November 2012). "Molecular basis for a protein-mediated DNA-bridging mechanism that functions in condensation of the E. coli chromosome". Molecular Cell. 48 (4): 560–71. doi:10.1016/j.molcel.2012.09.009. PMC 7505563. PMID 23084832.
- ^ Wang S, Cosstick R, Gardner JF, Gumport RI (October 1995). "The specific binding of Escherichia coli integration host factor involves both major and minor grooves of DNA". Biochemistry. 34 (40): 13082–90. doi:10.1021/bi00040a020. PMID 7548068.
- ^ Karas VO, Westerlaken I, Meyer AS (October 2015). "The DNA-Binding Protein from Starved Cells (Dps) Utilizes Dual Functions To Defend Cells against Multiple Stresses". Journal of Bacteriology. 197 (19): 3206–15. doi:10.1128/JB.00475-15. PMC 4560292. PMID 26216848.
- ^ a b Woo JS, Lim JH, Shin HC, Suh MK, Ku B, Lee KH, et al. (January 2009). "Structural studies of a bacterial condensin complex reveal ATP-dependent disruption of intersubunit interactions" (PDF). Cell. 136 (1): 85–96. doi:10.1016/j.cell.2008.10.050. PMID 19135891. S2CID 4608756.
- ^ a b c d e Badrinarayanan A, Reyes-Lamothe R, Uphoff S, Leake MC, Sherratt DJ (October 2012). "In vivo architecture and action of bacterial structural maintenance of chromosome proteins". Science. 338 (6106): 528–31. Bibcode:2012Sci...338..528B. doi:10.1126/science.1227126. PMC 3807729. PMID 23112333.
- ^ Kumar R, Grosbart M, Nurse P, Bahng S, Wyman CL, Marians KJ (October 2017). "The bacterial condensin MukB compacts DNA by sequestering supercoils and stabilizing topologically isolated loops". The Journal of Biological Chemistry. 292 (41): 16904–16920. doi:10.1074/jbc.M117.803312. PMC 5641887. PMID 28842486.
- ^ Niki H, Imamura R, Kitaoka M, Yamanaka K, Ogura T, Hiraga S (December 1992). "E.coli MukB protein involved in chromosome partition forms a homodimer with a rod-and-hinge structure having DNA binding and ATP/GTP binding activities". The EMBO Journal. 11 (13): 5101–9. doi:10.1002/j.1460-2075.1992.tb05617.x. PMC 556988. PMID 1464330.
- ^ Melby TE, Ciampaglio CN, Briscoe G, Erickson HP (September 1998). "The symmetrical structure of structural maintenance of chromosomes (SMC) and MukB proteins: long, antiparallel coiled coils, folded at a flexible hinge". The Journal of Cell Biology. 142 (6): 1595–604. doi:10.1083/jcb.142.6.1595. PMC 2141774. PMID 9744887.
- ^ a b Yamazoe M, Onogi T, Sunako Y, Niki H, Yamanaka K, Ichimura T, Hiraga S (November 1999). "Complex formation of MukB, MukE and MukF proteins involved in chromosome partitioning in Escherichia coli". The EMBO Journal. 18 (21): 5873–84. doi:10.1093/emboj/18.21.5873. PMC 1171653. PMID 10545099.
- ^ Li Y, Stewart NK, Berger AJ, Vos S, Schoeffler AJ, Berger JM, et al. (November 2010). "Escherichia coli condensin MukB stimulates topoisomerase IV activity by a direct physical interaction". Proceedings of the National Academy of Sciences of the United States of America. 107 (44): 18832–7. doi:10.1073/pnas.1008678107. PMC 2973889. PMID 20921377.
- ^ Vos SM, Stewart NK, Oakley MG, Berger JM (November 2013). "Structural basis for the MukB-topoisomerase IV interaction and its functional implications in vivo". The EMBO Journal. 32 (22): 2950–62. doi:10.1038/emboj.2013.218. PMC 3832749. PMID 24097060.
- ^ Nicolas E, Upton AL, Uphoff S, Henry O, Badrinarayanan A, Sherratt D (February 2014). "The SMC complex MukBEF recruits topoisomerase IV to the origin of replication region in live Escherichia coli". mBio. 5 (1): e01001-13. doi:10.1128/mBio.01001-13. PMC 3950513. PMID 24520061.
- ^ Zawadzki P, Stracy M, Ginda K, Zawadzka K, Lesterlin C, Kapanidis AN, Sherratt DJ (December 2015). "The Localization and Action of Topoisomerase IV in Escherichia coli Chromosome Segregation Is Coordinated by the SMC Complex, MukBEF". Cell Reports. 13 (11): 2587–2596. doi:10.1016/j.celrep.2015.11.034. PMC 5061553. PMID 26686641.
- ^ a b c Baxter J, Oliver AW, Schalbetter SA (January 2019). "Are SMC Complexes Loop Extruding Factors? Linking Theory With Fact". BioEssays. 41 (1): e1800182. doi:10.1002/bies.201800182. PMID 30506702.
- ^ Zawadzka K, Zawadzki P, Baker R, Rajasekar KV, Wagner F, Sherratt DJ, Arciszewska LK (January 2018). "MukB ATPases are regulated independently by the N- and C-terminal domains of MukF kleisin". eLife. 7. doi:10.7554/eLife.31522. PMC 5812716. PMID 29323635.
- ^ Sawitzke JA, Austin S (February 2000). "Suppression of chromosome segregation defects of Escherichia coli muk mutants by mutations in topoisomerase I". Proceedings of the National Academy of Sciences of the United States of America. 97 (4): 1671–6. Bibcode:2000PNAS...97.1671S. doi:10.1073/pnas.030528397. PMC 26494. PMID 10660686.
- ^ Adachi S, Hiraga S (July 2003). "Mutants suppressing novobiocin hypersensitivity of a mukB null mutation". Journal of Bacteriology. 185 (13): 3690–5. doi:10.1128/jb.185.13.3690-3695.2003. PMC 161581. PMID 12813060.
- ^ Petrushenko ZM, Lai CH, Rai R, Rybenkov VV (February 2006). "DNA reshaping by MukB. Right-handed knotting, left-handed supercoiling". The Journal of Biological Chemistry. 281 (8): 4606–15. doi:10.1074/jbc.M504754200. PMC 1633270. PMID 16368697.
- ^ Gaal T, Bratton BP, Sanchez-Vazquez P, Sliwicki A, Sliwicki K, Vegel A, et al. (October 2016). "Colocalization of distant chromosomal loci in space in E. coli: a bacterial nucleolus". Genes & Development. 30 (20): 2272–2285. doi:10.1101/gad.290312.116. PMC 5110994. PMID 27898392.
- ^ Jin DJ, Cabrera JE (November 2006). "Coupling the distribution of RNA polymerase to global gene regulation and the dynamic structure of the bacterial nucleoid in Escherichia coli". Journal of Structural Biology. 156 (2): 284–91. doi:10.1016/j.jsb.2006.07.005. PMID 16934488.
- ^ a b c Qian Z, Dimitriadis EK, Edgar R, Eswaramoorthy P, Adhya S (July 2012). "Galactose repressor mediated intersegmental chromosomal connections in Escherichia coli". Proceedings of the National Academy of Sciences of the United States of America. 109 (28): 11336–41. Bibcode:2012PNAS..10911336Q. doi:10.1073/pnas.1208595109. PMC 3396475. PMID 22733746.
- ^ Weickert MJ, Adhya S (October 1993). "The galactose regulon of Escherichia coli". Molecular Microbiology. 10 (2): 245–51. doi:10.1111/j.1365-2958.1993.tb01950.x. PMID 7934815. S2CID 6872903.
- ^ a b Agerschou ED, Christiansen G, Schafer NP, Madsen DJ, Brodersen DE, Semsey S, Otzen DE (June 2016). "The transcriptional regulator GalR self-assembles to form highly regular tubular structures". Scientific Reports. 6: 27672. Bibcode:2016NatSR...627672A. doi:10.1038/srep27672. PMC 4899725. PMID 27279285.
- ^ Kavenoff R, Ryder OA (March 1976). "Electron microscopy of membrane-associated folded chromosomes of Escherichia coli". Chromosoma. 55 (1): 13–25. doi:10.1007/bf00288323. PMID 767075. S2CID 31879250.
- ^ Worcel A, Burgi E (January 1974). "Properties of a membrane-attached form of the folded chromosome of Escherichia coli". Journal of Molecular Biology. 82 (1): 91–105. doi:10.1016/0022-2836(74)90576-2. PMID 4594427.
- ^ Roggiani M, Goulian M (2015). "Chromosome-Membrane Interactions in Bacteria". Annual Review of Genetics. 49: 115–29. doi:10.1146/annurev-genet-112414-054958. PMID 26436460.
- ^ Libby EA, Roggiani M, Goulian M (May 2012). "Membrane protein expression triggers chromosomal locus repositioning in bacteria". Proceedings of the National Academy of Sciences of the United States of America. 109 (19): 7445–50. Bibcode:2012PNAS..109.7445L. doi:10.1073/pnas.1109479109. PMC 3358875. PMID 22529375.
- ^ Brameyer S, Rösch TC, El Andari J, Hoyer E, Schwarz J, Graumann PL, Jung K (2019). "DNA-binding directs the localization of a membrane-integrated receptor of the ToxR family". Communications Biology. 2: 4. doi:10.1038/s42003-018-0248-7. PMC 6320335. PMID 30740540.
- ^ Espéli O, Borne R, Dupaigne P, Thiel A, Gigant E, Mercier R, Boccard F (May 2012). "A MatP-divisome interaction coordinates chromosome segregation with cell division in E. coli". The EMBO Journal. 31 (14): 3198–211. doi:10.1038/emboj.2012.128. PMC 3400007. PMID 22580828.
- ^ Bernhardt TG, de Boer PA (May 2005). "SlmA, a nucleoid-associated, FtsZ binding protein required for blocking septal ring assembly over Chromosomes in E. coli". Molecular Cell. 18 (5): 555–64. doi:10.1016/j.molcel.2005.04.012. PMC 4428309. PMID 15916962.
- ^ Woldringh CL, Jensen PR, Westerhoff HV (September 1995). "Structure and partitioning of bacterial DNA: determined by a balance of compaction and expansion forces?" (PDF). FEMS Microbiology Letters. 131 (3): 235–42. doi:10.1111/j.1574-6968.1995.tb07782.x. PMID 7557335.
- ^ a b Van Helvoort JM, Huls PG, Vischer NO, Woldringh CL (May 1998). "Fused nucleoids resegregate faster than cell elongation in Escherichia coli pbpB(Ts) filaments after release from chloramphenicol inhibition". Microbiology. 144 (5): 1309–17. doi:10.1099/00221287-144-5-1309. PMID 9611806.
- ^ Eltsov, Mikhail; Zuber, Benoît (2006). "Transmission electron microscopy of the bacterial nucleoid". Journal of Structural Biology. 156 (2): 246–254. doi:10.1016/j.jsb.2006.07.007. ISSN 1047-8477. PMID 16978880.
- ^ Robinow, C; Kellenberger, E (1994). "The bacterial nucleoid revisited". Microbiological Reviews. 58 (2): 211–232. doi:10.1128/mmbr.58.2.211-232.1994. ISSN 0146-0749. PMC 372962. PMID 7521510.
- ^ Smith BT, Grossman AD, Walker GC (January 2002). "Localization of UvrA and effect of DNA damage on the chromosome of Bacillus subtilis". Journal of Bacteriology. 184 (2): 488–93. doi:10.1128/jb.184.2.488-493.2002. PMC 139587. PMID 11751826.
- ^ Odsbu I, Skarstad K (October 2009). "A reduction in ribonucleotide reductase activity slows down the chromosome replication fork but does not change its localization". PLOS ONE. 4 (10): e7617. Bibcode:2009PLoSO...4.7617O. doi:10.1371/journal.pone.0007617. PMC 2773459. PMID 19898675.
- ^ Levin-Zaidman S, Frenkiel-Krispin D, Shimoni E, Sabanay I, Wolf SG, Minsky A (June 2000). "Ordered intracellular RecA-DNA assemblies: a potential site of in vivo RecA-mediated activities". Proceedings of the National Academy of Sciences of the United States of America. 97 (12): 6791–6. Bibcode:2000PNAS...97.6791L. doi:10.1073/pnas.090532397. PMC 18741. PMID 10829063.
- ^ a b Delmas S, Duggin IG, Allers T (January 2013). "DNA damage induces nucleoid compaction via the Mre11-Rad50 complex in the archaeon Haloferax volcanii". Molecular Microbiology. 87 (1): 168–79. doi:10.1111/mmi.12091. PMC 3565448. PMID 23145964.