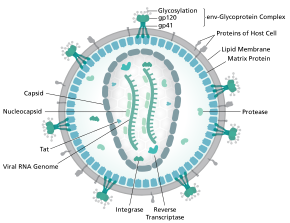
HIV의 구조와 게놈
Structure and genome of HIV인간 면역 결핍 바이러스의 게놈과 [1][2]단백질은 1983년 바이러스가 발견된 이후 광범위한 연구 대상이 되어 왔다."원인 물질을 찾는 과정에서, 이 바이러스는 그 당시 인간의 면역 체계에 영향을 미쳐 특정 백혈병을 일으키는 것으로 알려진 인간 T세포 백혈병 바이러스(HTLV)의 한 형태라고 처음에는 믿었습니다.그러나 파리 파스퇴르 연구소의 연구진은 에이즈 환자들에게서 이전에 알려지지 않은 유전적으로 구별된 레트로바이러스를 분리했다.이 레트로바이러스는 나중에 [3]HIV로 명명되었다." 각각의 바이러스 외피와 캡시드를 둘러싼 관련 매트릭스로 구성되어 있다. 각각의 바이러스 외피에는 외가닥 RNA 게놈의 두 복사본과 여러 효소가 들어있다.이 바이러스의 발견은 에이즈 관련 질병의 [4][5]첫 번째 주요 사례가 보고된 지 2년 후에 이루어졌다.
구조.
감염 바이러스로부터 추출된 HIV-1 게놈의 완전한 배열이 단일 핵 [6]분해능으로 해결되었다.HIV 게놈은 소수의 바이러스 단백질을 암호화하여 HIV 단백질과 HIV와 숙주 단백질 간의 협력 관계를 항상 확립하고 숙주 세포를 침범하여 내부 기구를 [7]탈취합니다.HIV는 다른 레트로바이러스와 구조가 다르다.HIV바이리온은 지름이 100nm까지이다.그것의 가장 안쪽에 있는 부분은 (긍정적인) ssRNA 게놈의 두 개의 복사본, 역전사효소, 인테그라아제, 단백질 분해효소, 그리고 몇몇 작은 단백질,[8] 그리고 주요 핵심 단백질을 포함하는 원뿔 모양의 핵으로 구성되어 있습니다.인간면역결핍바이러스(HIV)의 게놈은 HIV [7]라이프사이클에서 필수적인 역할을 하는 8가지 바이러스 단백질을 암호화한다.
HIV-1은 렌즈 바이러스의 [9][10]전형적인 바이러스 단백질 p24로 구성된 원추형 캡시드로 둘러싸인 비공유적으로 연결된, 결합되지 않은, 양성 감각의 단일 가닥 RNA의 두 복사본으로 구성되어 있습니다.RNA 가닥의 두 복사본은 바이러스 복제의 역전사 동안 발생하는 HIV-1 재조합에 기여하는 데 필수적이다.바이러스리온 내에 두 개의 단일 가닥 RNA 복사본이 포함되지만, 단 하나의 DNA 프로바이러스의 생산은 의사 [11]증배라고 불립니다.RNA 성분은 9749 뉴클레오티드의[12][13] 길이로 5' 캡(Gppp), 3' 폴리(A) 꼬리 및 많은 ORF([14]Open Reading Frame)를 가지고 있습니다.바이러스 구조 단백질은 긴 ORF에 의해 부호화되는 반면, 더 작은 ORF는 바이러스 수명 주기의 조절제: 부착, 막 융합, 복제 및 [14]조립을 부호화합니다.
단일 가닥 RNA는 p7 핵캡시드 단백질, 후기 조립 단백질 p6 및 역전사효소 및 인테그레이스 등 바이러스 발생에 필수적인 효소와 밀접하게 결합되어 있다.리신 tRNA는 마그네슘 의존성 역전사효소의 [9]프라이머이다.뉴클레오캡시드는 게놈 RNA(헥사머당 1개 분자)와 결합하고 뉴클레오카아제(nuclearase)에 의한 소화로부터 RNA를 보호합니다.또, Vif, Vpr, Nef, 바이러스 [citation needed]프로테아제도 포함되어 있다.비리온의 외피는 바이러스 p17 단백질로 구성된 매트릭스에 의해 지지되는 숙주 세포 기원의 플라즈마 막에 의해 형성되어 비리온 입자의 무결성을 보장합니다.비리온 표면에는, gp120, gp41의 헤테로디머에 의해서 형성되는 트리머인 HIV의 외피당단백질(Env)의 수가 한정되어 있다.Env는 1차 숙주 수용체인 CD4와 그 공동 수용체(주로 CCR5 또는 CXCR4)에 결합하는 역할을 하며, 표적 [15]세포로 바이러스 진입을 이끈다.
바이러스 표면의 유일한 단백질로서, 외피당단백질(gp120 및 gp41)은 HIV 백신 [16]노력의 주요 목표물이다.삼량체 외피 스파이크의 질량의 절반 이상이 N-연결 글리칸이다.글리칸은 항체에 의한 중성화로부터 기초 바이러스 단백질을 보호하므로 밀도가 높다.이것은 알려진 가장 조밀한 글리코실화 분자 중 하나이며, 그 밀도가 충분히 높아서 소포체 [17][18]및 골지 기구의 생물 형성 중에 글리칸의 정상적인 성숙 과정을 막을 수 있다.따라서 글리칸의 대부분은 보통 분비되거나 세포 표면인 인간 [19]당단백질에는 존재하지 않는 미성숙한 '고만노스' 글리칸으로 정체된다.비정상적인 처리와 고밀도는 지금까지 식별된 거의 모든(수개월에서 수년 동안 감염된 환자의 하위 집합으로부터) 광범위하게 중화되는 항체가 이러한 외피 [20]글리칸에 결합하거나 이에 대처하기 위해 조정된다는 것을 의미한다.
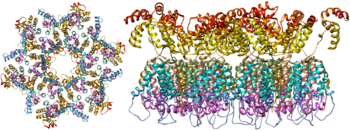
바이러스 스파이크의 분자 구조는 이제 X선 결정학과[21] 극저온 [22]전자 현미경으로 결정되었다.구조생물학의 이러한 발전은 gp41에서 [23]돌연변이를 일으키는 서브유닛 간 이황화 결합과 이소류신의 도입으로 바이러스 스파이크의 안정적인 재조합 형태가 개발되었기 때문에 가능했다.이른바 SOSIP 트리머는 토종 바이러스 스파이크의 항원 특성을 재현할 뿐만 아니라 토종 [24]바이러스와 같은 정도의 미성숙한 글리칸을 나타낸다.재조합 삼량체 바이러스 스파이크는 표적 에피토프에 [25]대한 면역반응을 억제하는 역할을 하는 재조합 단량체 gp120보다 덜 중화 에피토프를 나타내기 때문에 유망한 백신 후보이다.
게놈 구성
HIV는 모든 레트로바이러스에서 발견되는 구조 단백질을 코드하는 몇 가지 주요 유전자뿐만 아니라 [26]HIV에 고유한 몇 가지 비구조적 "액세서리" 유전자도 가지고 있습니다.HIV 게놈은 15개의 바이러스 [27]단백질을 코드하는 9개의 유전자를 포함하고 있다.이것들은 가리온 내부를 위한 단백질을 생성하는 고단백질, 그룹 특이 항원, 바이러스 효소(Pol, 중합효소) 또는 비리온 환경의 당단백질로 합성됩니다.[28]이것들 외에도, HIV는 특정한 조절 및 보조 기능을 가진 단백질을 암호화합니다.[28]HIV-1에는, 다음의 2개의 중요한 규제 요소가 있습니다.특정 조직의 [28]복제에 필수적이지 않은 Nef, Vpr, Vif 및 Vpu와 같은 몇 가지 중요한 보조 단백질과 Tat 및 Rev.개그 유전자는 바이러스의 기본적인 물리적 인프라를 제공하고, 폴은 레트로바이러스가 번식하는 기본 메커니즘을 제공하는 반면, 다른 유전자는 HIV가 숙주 세포에 들어가 번식을 강화하도록 돕는다.돌연변이에 의해 변화될 수 있지만, tev를 제외한 모든 유전자는 HIV의 알려진 모든 변종에 존재합니다.[citation needed] HIV의 유전적 변이를 참조하십시오.
HIV는 10kb 미만의 [29]게놈에서 9개의 다른 유전자 제품을 얻기 위해 정교한 차등 RNA 스플라이싱 시스템을 사용합니다.HIV에는 개그와 폴 전구체를 코드하는 9.2kb의 무절제 게놈 트랜스크립트, env, Vif, Vpr 및 Vpu의 경우 단일 스플라이스 4.5kb 인코딩, Tat, Rev 및 Nef의 [29]경우 다중 스플라이스 2kb mRNA 인코딩이 있습니다.
| 학급 | 유전자명 | 주요 단백질 제품 | 단백질 가공품 |
|---|---|---|---|
| 바이러스 구조단백질 | 재갈을 물리다 | 개그 폴리단백질 | MA, CA, SP1, NC, SP2, P6 |
| 폴 | 폴 폴리단백질 | RT, RNAase H, IN, PR | |
| 부러워하다 | gp160 | gp120, gp41 | |
| 필수 규제 요소 | 두드리다 | 타트 | |
| 리비전 | 리비전 | ||
| 보조조절단백질 | 네프 | 네프 | |
| vpr | Vpr | ||
| 비디오 | 비프 | ||
| VPU | Vpu |
바이러스 구조단백질
- MA(매트릭스 단백질, p17), CA(캡시드 단백질, p24), SP1(스페이서 펩타이드 1, p2), NC(뉴클레오캡시드 단백질, p7), SP2(스페이서 펩타이드 2, p1 및 P6 [30]단백질로 성숙하는 동안 바이러스 단백질 효소에 의해 처리되는 전구체 개그 폴리단백질 코드.
- 바이러스 효소 역전사효소(RT)와 RNAase H, 인테그레이스(IN) 및 HIV 단백질분해효소(PR)[28]에 대한 폴 코드.HIV단백질가수분해효소는 전구체 Gag 폴리단백질을 분해하여 구조단백질을 생성하고, RT는 RNA 템플릿에서 DNA를 전사하고, IN은 이중가닥 바이러스 DNA를 숙주 [26]게놈에 통합하기 위해 필요하다.
- env ("gp160"에 대한 코드)는 숙주 세포의 소포체 내에서 숙주 단백질 분해효소인 furin에 의해 분해된다.번역 후 처리는 림프구에 존재하는 CD4 수용체에 부착하는 표면 당단백질 gp120 또는 SU와 바이러스가 표적 [26][30]세포에 부착 및 융합할 수 있도록 바이러스 엔벨로프에 내장되는 gp41 또는 TM을 생성한다.
필수 규제 요소
- tat(HIV 트랜스 활성화제)는 바이러스 게놈 RNA의 역전사를 조절하고 바이러스 mRNA의 효율적인 합성을 보장하며 감염된 [28]세포에서 바이러스 이온의 방출을 조절하는 데 중요한 역할을 한다.Tat은 72-아미노산 1-exon Tat과 86-101-아미노산 2-exon Tat으로 발현되며 HIV 감염 초기에 중요한 역할을 한다.Tat(14~15kDa)는 트랜스활성화반응요소([9][28]TAR)를 형성하는 5' LTR 영역 근처에서 팽창된 게놈 RNA 스템루프 2차 구조에 결합한다.
- rev(바이리온 단백질 발현 억제):Rev 단백질은 아르기닌이 풍부한 RNA 결합 모티브를 통해 바이러스 게놈에 결합하며, NLS([28]핵 국재 신호)로서도 작용하며, 바이러스 복제 중에 Rev를 세포질에서 핵으로 운반하는 데 필요합니다.Rev는 HIV Rev 응답 요소(RRE)[9][28]로 알려진 Tat 및 Rev의 인트론 분리 코딩 exon에 위치한 mRNA 환경의 복잡한 스템 루프 구조를 인식합니다.Rev는 주요 바이러스 단백질의 합성에 중요하기 때문에 바이러스 [citation needed]복제에 필수적이다.
보조조절단백질
- vpr (렌티바이러스 단백질 R): Vpr은 바이러스 관련 핵세포질 차단 조절 [28]단백질이다.바이러스의 복제, 구체적으로는 통합 전 복합체의 핵 수입에 중요한 역할을 하는 것으로 생각된다.Vpr은 또한 숙주 세포가 G2 단계에서 세포 주기를 정지시키는 것으로 보인다.이 체포는 바이러스 [9]DNA의 통합을 가능하게 할 수 있는 숙주 DNA 복구 기계를 작동시킨다.HIV-2와 SIV는 Vpr과 [28]연계하여 기능하는 Vpx라고 불리는 추가적인 Vpr 관련 단백질을 암호화한다.
- vif - Vif는 고도로 보존된 23kDa 인단백질로서 세포 [9]유형에 따라 HIV-1 바이러스온의 감염성에 중요하다.HIV-1은 Vif가 림프구, 대식세포 및 특정 인간 세포주에서 감염성 바이러스를 합성해야 하는 것으로 밝혀졌다.특히 Hela [28]셀이나 COS 셀에서 동일한 프로세스에 Vif를 필요로 하지 않는 것으로 보입니다.
- nef-Nef, 음인자는 N 말단 미리스토일화막 관련 인산단백질이다.바이러스 복제 주기 동안 여러 기능에 관여합니다.그것은 세포자멸에 중요한 역할을 하고 바이러스 감염성을 [28]증가시킨다고 믿어진다.
- vpu(바이러스 단백질 U): Vpu는 HIV-1에 고유합니다.그것은 많은 생물학적 기능을 가진 I급 올리고머 일체막 인단백질이다.Vpu는 유비퀴틴 프로테아솜 경로를 포함하는 CD4 분해와 감염된 [9][28]세포에서 성공적으로 바이러스 방출에 관여한다.
- tev: 이 유전자는 소수의 HIV-1 분리주에만 존재합니다.이것은 tat, env, rev 유전자의 일부와 tat의 특성 중 일부를 가진 단백질의 코드들의 융합이지만,[31] rev의 특성은 거의 또는 전혀 없다.
RNA 이차 구조
| HIV pol-1 스템루프 | |
|---|---|
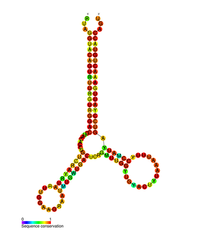 HIV pol-1 스템 루프의 예측된 2차 구조 | |
| 식별자 | |
| 기호. | 폴 |
| Rfam | RF01418 |
| 기타 데이터 | |
| RNA형 | 시스레그 |
| PDB 구조 | PDBe |
몇 가지 보존된 2차 구조 요소는 HIV RNA 게놈 내에서 확인되었다.5'UTR 구조는 작은 [10]링커에 의해 연결된 일련의 스템 루프 구조로 구성됩니다.이들 스템루프(5'~3')는 유전자 [10][32][33]내 게놈의 5' 말단 내에 위치한 트랜스활성화영역(TAR) 요소, 5' 폴리아데닐화 신호[poly(A)], PBS, DIS, 주요 SD 및 γ 헤어핀 구조 및 HIV Rev 응답 요소(RRE)를 포함한다.확인된 또 다른 RNA 구조는 바이러스 [34][35]포장에 관여하는 것으로 생각되는 개그 스템 루프 3(GSL3)이다.RNA 2차 구조는 식별된 모든 요소가 기능을 [citation needed]할당받은 것은 아니지만 HIV 단백질 분해효소 및 역전사효소의 기능을 변경함으로써 HIV 라이프 사이클에 영향을 미치는 것으로 제안되었다.
SHAPE 분석에 의해 결정되는 RNA 2차 구조는 3개의 줄기 루프를 포함하고 있으며 HIV 단백질 분해효소 유전자와 역전사효소 유전자 사이에 위치한다.이 시스 조절 RNA는 HIV 패밀리를 통해 보존되는 것으로 나타났으며 [36]바이러스 라이프 사이클에 영향을 미치는 것으로 생각됩니다.
V3 루프
세 번째 가변 루프 또는 V3 루프는 인간 면역 결핍 바이러스의 일부 또는 영역입니다.바이러스 외피당단백질 gp120의 V3 루프는 HIV의 [37]변종에 따라 CCR5 세포나 CXCR4 세포와 같은 표적 인간 면역 세포상의 사이토카인 수용체에 결합함으로써 인간 면역 세포를 감염시킵니다.엔벨로프 당단백(Env) gp 120/41은 HIV-1 세포 진입에 필수적입니다.Env는 HIV-1 감염자를 치료하는 의약품의 분자 표적이자 에이즈 백신을 개발하기 위한 면역원 공급원이다.그러나 기능성 Env 트리머의 구조는 여전히 [38]명확하지 않습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Barré-Sinoussi F, Chermann JC, Rey F, Nugeyre MT, Chamaret S, Gruest J, Dauguet C, Axler-Blin C, Vézinet-Brun F, Rouzioux C, Rozenbaum W, Montagnier L (May 1983). "Isolation of a T-lymphotropic retrovirus from a patient at risk for acquired immune deficiency syndrome (AIDS)". Science. 220 (4599): 868–71. Bibcode:1983Sci...220..868B. doi:10.1126/science.6189183. PMID 6189183. S2CID 390173.
- ^ Gallo RC, Sarin PS, Gelmann EP, Robert-Guroff M, Richardson E, Kalyanaraman VS, Mann D, Sidhu GD, Stahl RE, Zolla-Pazner S, Leibowitch J, Popovic M (May 1983). "Isolation of human T-cell leukemia virus in acquired immune deficiency syndrome (AIDS)". Science. 220 (4599): 865–7. Bibcode:1983Sci...220..865G. doi:10.1126/science.6601823. PMID 6601823.
- ^ Churi C, Ross MW (2015). "Hiv/Aids". In Whelehan P, Bolin A (eds.). The international encyclopedia of human sexuality. Wiley. ISBN 9781405190060. OCLC 949701914.
- ^ Centers for Disease Control (June 1981). "Pneumocystis pneumonia--Los Angeles". MMWR. Morbidity and Mortality Weekly Report. 30 (21): 250–2. PMID 6265753.
- ^ Centers for Disease Control (CDC) (July 1981). "Kaposi's sarcoma and Pneumocystis pneumonia among homosexual men--New York City and California" (PDF). MMWR. Morbidity and Mortality Weekly Report. 30 (25): 305–8. PMID 6789108. Archived from the original on October 22, 2012. Retrieved September 15, 2017.
{{cite journal}}: CS1 유지보수: 부적합한 URL(링크) - ^ Watts JM, Dang KK, Gorelick RJ, Leonard CW, Bess JW, Swanstrom R, Burch CL, Weeks KM (August 2009). "Architecture and secondary structure of an entire HIV-1 RNA genome". Nature. 460 (7256): 711–6. Bibcode:2009Natur.460..711W. doi:10.1038/nature08237. PMC 2724670. PMID 19661910.
- ^ a b Li G, De Clercq E (September 2016). "HIV Genome-Wide Protein Associations: a Review of 30 Years of Research". Microbiology and Molecular Biology Reviews. 80 (3): 679–731. doi:10.1128/MMBR.00065-15. PMC 4981665. PMID 27357278.
- ^ Singleton P, Sainsbury D, eds. (2006). "Hiv". Dictionary of microbiology & molecular biology (3rd ed.). Hoboken, NJ: Wiley. ISBN 9780470035450. OCLC 71223221.
- ^ a b c d e f g Montagnier L (1999). "Human Immunodeficiency Viruses (Retroviridae)". Encyclopedia of Virology (2nd ed.). pp. 763–774.
- ^ a b c Lu K, Heng X, Summers MF (July 2011). "Structural determinants and mechanism of HIV-1 genome packaging". Journal of Molecular Biology. 410 (4): 609–33. doi:10.1016/j.jmb.2011.04.029. PMC 3139105. PMID 21762803.
- ^ Hwang CK, Svarovskaia ES, Pathak VK (October 2001). "Dynamic copy choice: steady state between murine leukemia virus polymerase and polymerase-dependent RNase H activity determines frequency of in vivo template switching". Proceedings of the National Academy of Sciences of the United States of America. 98 (21): 12209–14. Bibcode:2001PNAS...9812209H. doi:10.1073/pnas.221289898. PMC 59793. PMID 11593039.
- ^ Wain-Hobson S, Sonigo P, Danos O, Cole S, Alizon M (January 1985). "Nucleotide sequence of the AIDS virus, LAV". Cell. 40 (1): 9–17. doi:10.1016/0092-8674(85)90303-4. PMID 2981635. S2CID 33055050.
- ^ Ratner L, Haseltine W, Patarca R, Livak KJ, Starcich B, Josephs SF, Doran ER, Rafalski JA, Whitehorn EA, Baumeister K (1985). "Complete nucleotide sequence of the AIDS virus, HTLV-III". Nature. 313 (6000): 277–84. Bibcode:1985Natur.313..277R. doi:10.1038/313277a0. PMID 2578615. S2CID 4316242.
- ^ a b Castelli JC, Levy A (2002). "HIV (Human Immunodeficiency Virus)". Encyclopedia of Cancer. Vol. 2 (2nd ed.). pp. 407–415.
- ^ Checkly MA, Freed EO (22 July 2011). "HIV-1 envelope glycoprotein biosynthesis, trafficking, and incorporation". Journal of Molecular Biology. 410 (4): 582–608. doi:10.1016/j.jmb.2011.04.042. PMC 3139147. PMID 21762802.
- ^ National Institute of Health (June 17, 1998). "Crystal structure of key HIV protein reveals new prevention, treatment targets" (Press release). Archived from the original on February 19, 2006. Retrieved September 14, 2006.
- ^ Behrens AJ, Vasiljevic S, Pritchard LK, Harvey DJ, Andev RS, Krumm SA, Struwe WB, Cupo A, Kumar A, Zitzmann N, Seabright GE, Kramer HB, Spencer DI, Royle L, Lee JH, Klasse PJ, Burton DR, Wilson IA, Ward AB, Sanders RW, Moore JP, Doores KJ, Crispin M (March 2016). "Composition and Antigenic Effects of Individual Glycan Sites of a Trimeric HIV-1 Envelope Glycoprotein". Cell Reports. 14 (11): 2695–706. doi:10.1016/j.celrep.2016.02.058. PMC 4805854. PMID 26972002.
- ^ Pritchard LK, Spencer DI, Royle L, Bonomelli C, Seabright GE, Behrens AJ, Kulp DW, Menis S, Krumm SA, Dunlop DC, Crispin DJ, Bowden TA, Scanlan CN, Ward AB, Schief WR, Doores KJ, Crispin M (June 2015). "Glycan clustering stabilizes the mannose patch of HIV-1 and preserves vulnerability to broadly neutralizing antibodies". Nature Communications. 6: 7479. Bibcode:2015NatCo...6.7479P. doi:10.1038/ncomms8479. PMC 4500839. PMID 26105115.
- ^ Pritchard LK, Harvey DJ, Bonomelli C, Crispin M, Doores KJ (September 2015). "Cell- and Protein-Directed Glycosylation of Native Cleaved HIV-1 Envelope". Journal of Virology. 89 (17): 8932–44. doi:10.1128/JVI.01190-15. PMC 4524065. PMID 26085151.
- ^ Crispin M, Doores KJ (April 2015). "Targeting host-derived glycans on enveloped viruses for antibody-based vaccine design". Current Opinion in Virology. Viral pathogenesis • Preventive and therapeutic vaccines. 11: 63–9. doi:10.1016/j.coviro.2015.02.002. PMC 4827424. PMID 25747313.
- ^ Julien JP, Cupo A, Sok D, Stanfield RL, Lyumkis D, Deller MC, Klasse PJ, Burton DR, Sanders RW, Moore JP, Ward AB, Wilson IA (December 2013). "Crystal structure of a soluble cleaved HIV-1 envelope trimer". Science. 342 (6165): 1477–83. Bibcode:2013Sci...342.1477J. doi:10.1126/science.1245625. PMC 3886632. PMID 24179159.
- ^ Lyumkis D, Julien JP, de Val N, Cupo A, Potter CS, Klasse PJ, Burton DR, Sanders RW, Moore JP, Carragher B, Wilson IA, Ward AB (December 2013). "Cryo-EM structure of a fully glycosylated soluble cleaved HIV-1 envelope trimer". Science. 342 (6165): 1484–90. Bibcode:2013Sci...342.1484L. doi:10.1126/science.1245627. PMC 3954647. PMID 24179160.
- ^ Sanders RW, Derking R, Cupo A, Julien JP, Yasmeen A, de Val N, Kim HJ, Blattner C, de la Peña AT, Korzun J, Golabek M, de Los Reyes K, Ketas TJ, van Gils MJ, King CR, Wilson IA, Ward AB, Klasse PJ, Moore JP (September 2013). "A next-generation cleaved, soluble HIV-1 Env trimer, BG505 SOSIP.664 gp140, expresses multiple epitopes for broadly neutralizing but not non-neutralizing antibodies". PLOS Pathogens. 9 (9): e1003618. doi:10.1371/journal.ppat.1003618. PMC 3777863. PMID 24068931.
- ^ Pritchard LK, Vasiljevic S, Ozorowski G, Seabright GE, Cupo A, Ringe R, Kim HJ, Sanders RW, Doores KJ, Burton DR, Wilson IA, Ward AB, Moore JP, Crispin M (June 2015). "Structural Constraints Determine the Glycosylation of HIV-1 Envelope Trimers". Cell Reports. 11 (10): 1604–13. doi:10.1016/j.celrep.2015.05.017. PMC 4555872. PMID 26051934.
- ^ de Taeye SW, Ozorowski G, Torrents de la Peña A, Guttman M, Julien JP, van den Kerkhof TL, Burger JA, Pritchard LK, Pugach P, Yasmeen A, Crampton J, Hu J, Bontjer I, Torres JL, Arendt H, DeStefano J, Koff WC, Schuitemaker H, Eggink D, Berkhout B, Dean H, LaBranche C, Crotty S, Crispin M, Montefiori DC, Klasse PJ, Lee KK, Moore JP, Wilson IA, Ward AB, Sanders RW (December 2015). "Immunogenicity of Stabilized HIV-1 Envelope Trimers with Reduced Exposure of Non-neutralizing Epitopes". Cell. 163 (7): 1702–15. doi:10.1016/j.cell.2015.11.056. PMC 4732737. PMID 26687358.
- ^ a b c Mushahwar IK (2007). "Human Immunodeficiency Viruses: Molecular Virology, pathogenesis, diagnosis and treatment". Perspectives in Medical Virology. 13: 75–87. doi:10.1016/S0168-7069(06)13005-0. ISBN 9780444520739.
- ^ Li G, Piampongsant S, Faria NR, Voet A, Pineda-Peña AC, Khouri R, Lemey P, Vandamme AM, Theys K (February 2015). "An integrated map of HIV genome-wide variation from a population perspective". Retrovirology. 12 (1): 18. doi:10.1186/s12977-015-0148-6. PMC 4358901. PMID 25808207.
- ^ a b c d e f g h i j k l m Votteler J, Schubert U (2008). "Human Immunodeficiency Viruses: Molecular Biology". Encyclopedia of Virology (3rd ed.). pp. 517–525.
- ^ a b Feinberg Mark B, Greene Warner C (1992). "Molecular Insights into human immunodeficiency virus type1 pathogenesis". Current Opinion in Immunology. 4 (4): 466–474. doi:10.1016/s0952-7915(06)80041-5. PMID 1356348.
- ^ a b King Steven R (1994). "HIV: Virology and Mechanisms of disease". Annals of Emergency Medicine. 24 (3): 443–449. doi:10.1016/s0196-0644(94)70181-4. PMID 7915889.
- ^ Benko DM, Schwartz S, Pavlakis GN, Felber BK (June 1990). "A novel human immunodeficiency virus type 1 protein, tev, shares sequences with tat, env, and rev proteins". Journal of Virology. 64 (6): 2505–18. doi:10.1128/JVI.64.6.2505-2518.1990. PMC 249426. PMID 2186172.
- ^ Berkhout B (January 1992). "Structural features in TAR RNA of human and simian immunodeficiency viruses: a phylogenetic analysis". Nucleic Acids Research. 20 (1): 27–31. doi:10.1093/nar/20.1.27. PMC 310321. PMID 1738599.
- ^ Paillart JC, Skripkin E, Ehresmann B, Ehresmann C, Marquet R (February 2002). "In vitro evidence for a long range pseudoknot in the 5'-untranslated and matrix coding regions of HIV-1 genomic RNA". The Journal of Biological Chemistry. 277 (8): 5995–6004. doi:10.1074/jbc.M108972200. PMID 11744696.
- ^ Damgaard CK, Andersen ES, Knudsen B, Gorodkin J, Kjems J (February 2004). "RNA interactions in the 5' region of the HIV-1 genome". Journal of Molecular Biology. 336 (2): 369–79. doi:10.1016/j.jmb.2003.12.010. PMID 14757051.
- ^ Rong L, Russell RS, Hu J, Laughrea M, Wainberg MA, Liang C (September 2003). "Deletion of stem-loop 3 is compensated by second-site mutations within the Gag protein of human immunodeficiency virus type 1". Virology. 314 (1): 221–8. doi:10.1016/S0042-6822(03)00405-7. PMID 14517075.
- ^ Wang Q, Barr I, Guo F, Lee C (December 2008). "Evidence of a novel RNA secondary structure in the coding region of HIV-1 pol gene". RNA. 14 (12): 2478–88. doi:10.1261/rna.1252608. PMC 2590956. PMID 18974280.
- ^ "The interactions of the gp120 V3 loop of different HIV-1 strains with the potent anti-HIV human monoclonal antibody 447-52D". Weizmann Institute of Science: Department of Structural Biology. Archived from the original on 2007-07-18. Retrieved 2017-04-18.
- ^ Takeda S, Takizawa M, Miyauchi K, Urano E, Fujino M, Murakami T, Murakami T, Komano J (June 2016). "Conformational properties of the third variable loop of HIV-1AD8 envelope glycoprotein in the liganded conditions". Biochemical and Biophysical Research Communications. 475 (1): 113–8. doi:10.1016/j.bbrc.2016.05.051. PMID 27178216.
외부 링크
- HIV pol-1 스템루프의 Rfam 엔트리
- HIV1 바이러스 전체의 3D 모델
- Liu J, Wright ER, Winkler H (2010). "3D Visualization of HIV Virions by Cryoelectron Tomography". Cryo-EM, Part C: Analyses, Interpretation, and Case studies. Methods in Enzymology. Vol. 483. pp. 267–90. doi:10.1016/S0076-6879(10)83014-9. ISBN 9780123849939. PMC 3056484. PMID 20888479.