핵산 구조 결정
Nucleic acid structure determinationRNA와 DNA와 같은 핵산의 구조를 결정하는 실험적인 접근법은 크게 생물물리학적 방법과 생화학적 방법으로 분류할 수 있다. 생물물리학적 방법은 X선 결정학, NMR, 극저온 전자파 등 구조 결정에 분자의 기본적인 물리적 특성을 이용한다. 생화학적 방법은 특정 시약과 조건을 사용하여 핵산의 화학적 특성을 이용하여 핵산의 구조를 검증한다.[1] 이러한 방법은 특정 시약으로 화학적 탐색을 하거나 고유 화학 또는 아날로그 화학에 의존할 수 있다. 다른 실험 접근방식은 고유한 장점을 가지고 있으며 다른 실험 목적에 적합하다.
생물물리학적 방법
엑스선 결정학
X선 결정학은 DNA도 RNA도 쉽게 결정체를 형성하지 않기 때문에 핵산에만 흔히 있는 것이 아니다. 이는 핵산 구조에서 내인성 장애와 역동성이 더 크고 음전하(디옥시)인산염 등뼈가 서로 근접하게 밀어내기 때문이다. 그러므로 결정화된 핵산은 관심 있는 단백질과 복합되어 구조 질서를 제공하고 음전하를 중화시키는 경향이 있다.[citation needed]
핵자기공명분광기(NMR)
핵산 NMR은 DNA나 RNA와 같은 핵산 분자의 구조와 역학에 대한 정보를 얻기 위해 NMR 분광법을 사용하는 것이다. 2003년 현재 알려진 RNA 구조물의 거의 절반이 NMR 분광법에 의해 결정되었다.[2]
핵산 NMR은 단백질 NMR과 유사한 기술을 사용하지만 몇 가지 차이가 있다. 핵산은 수소 원자의 비율이 적은데, 수소 원자는 NMR에서 주로 관찰되는 원자로 핵산 이중 나선은 경직되고 대략 선형적이기 때문에 스스로 접히지 않아 '장거리' 상관관계를 부여한다.[3] NMR의 종류 보통 핵산과 사를 감지할 수 있는 있경수소 또는 양성자 NMR,13C NMR,15N NMR, 31P NMR. Two-dimensional NMR방법 거의 항상, 상관 분광학(COSY)와 전체의 일관성 전달 분광 법(TOCSY)through-bond 원자 연결 장치를 감지하기 위해 사용됩니다, 그리고 핵오버하우저 효과 분광 법(NOESY) 해uplin우주에서 서로 가까운 핵 사이의 [4]gs
주로 NOESY 교차 피크와 연결 상수인 스펙트럼에서 추출한 매개변수를 사용하여 글리코시드 결합 각도, 이음각(Karplus 방정식 사용) 및 슈가 퍼커 일치와 같은 국소 구조 특성을 결정할 수 있다. 대규모 구조물의 경우 이중나선(double helix)을 통과함에 따라 오류가 더해지고 단백질과 달리 이중나선(double helix)은 컴팩트한 내부를 가지지 못하며 스스로 접히지 않기 때문에 이러한 국소적 매개변수를 다른 구조적 가정이나 모델로 보완해야 한다. NMR은 또한 구부러진 헬리컬, 비 왓슨-크릭 베이스파이어링, 동축 적층 등과 같은 비표준 기하학적 구조를 조사하는 데도 유용하다. 줄기-루프, 가성비 등 복잡한 순응을 채택하는 경향이 있는 천연 RNA 올리고뉴클레오티드의 구조를 조사하는데 특히 유용했다. NMR은 또한 다른 분자의 결합에 따라 어떤 공진이 이동되는지를 확인함으로써 단백질이나 약물과 같은 다른 분자에 대한 핵산 분자의 결합을 조사하는 데 유용하다.[4]
극저온 전자 현미경(cryo-EM)
극저온 전자현미경(cryo-EM)은 전자빔을 이용해 수용액에 극저온적으로 보존된 샘플을 영상화하는 기법이다. 액체 샘플은 작은 금속 격자에 피펫팅되어 액체 질소 욕조에 의해 극도로 차갑게 유지되는 액체 에탄/프로판 용액에 담근다. 이 동결 과정에서 표본의 물 분자는 얼음에서 발견되는 것처럼 육각형 격자를 형성할 충분한 시간이 없기 때문에 표본이 유리처럼 물처럼 보존되어(혹은 유리화 얼음이라고도 함) 전자빔을 사용하여 이러한 표본을 이미지화하기 쉽다. X선 결정술에 비해 극저온 전자파(Cryo-EM)의 장점은 샘플이 수성 용액 상태로 보존되고 샘플의 결정체를 형성하여 동요하지 않는다는 것이다. 한 가지 단점은 75킬로달톤보다 작은 핵산이나 단백질 구조를 해결하기 어렵다는 것인데, 부분적으로 이 유리화된 수용액에서 입자를 찾는데 충분한 대비를 갖기 어렵기 때문이다. 또 다른 단점은 샘플에 대한 원자 수준의 구조 정보를 얻기 위해서는 많은 이미지(흔히 전자 마이크로그래프라고 함)를 촬영해야 하고, 단일 입자 재구성이라고 하는 과정에서 그러한 이미지들에 대한 평균을 내야 한다는 것이다. 이것은 계산적으로 집약적인 과정이다.
Cryo-EM은 더 새롭고 덜 동요하는 전송 전자 현미경이다. 표본이 표면에 건조되지 않기 때문에 덜 동요하며, 이 건조 과정은 음극-스테인 TEM에서 종종 수행되며, 크라이오-EM은 중금속 염류(예: 천왕성 아세테이트 또는 포쇼퉁스트산)와 같은 조영제를 필요로 하지 않기 때문에 생체 분자의 구조에도 영향을 미칠 수 있다. 전송전자현미경은 샘플이 전자빔과 상호작용을 하고 전자빔과 상호작용을 하지 않는 샘플의 일부만이 전자 검출 시스템으로 '전송'할 수 있다는 사실을 기법으로 활용한다. 일반적으로 TEM은 1960년대 이후 핵산 구조를 결정하는데 유용한 기술이었다.[5][6] 이중 가닥 DNA(dsDNA) 구조는 전통적으로 구조로 간주되지 않을 수 있지만, 일반적으로 단일 가닥 영역과 이중 가닥 영역의 교차 세그먼트가 존재한다는 점에서, 실제로 dsDNA는 단순히 DNA의 열변동 및 대체 구조로 인해 길이의 모든 위치에서 완벽하게 정렬된 이중 나선 구조는 아니다.오므는 G-플렉스처럼 보인다. 핵산의 크라이오EM은 바이러스 내 리보솜,[7] 바이러스 RNA,[8] 단일 가닥 RNA 구조물에 대해 실시되어 왔다.[9][10] 이러한 연구들은 뉴클레오바제 수준(2-3 앵스트롬)에서 3차 구조 모티브(나노미터보다 큼)까지 다양한 분해능으로 구조적 특성을 해결했다.
화학 프로빙
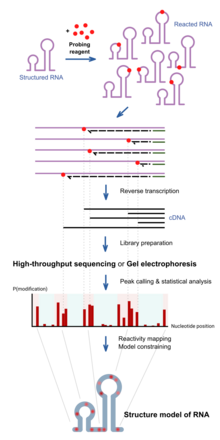
RNA 화학적 프로빙은 RNA와 반응하는 화학물질을 사용한다. 중요한 것은, 그들의 반응성은 국소 RNA 구조(예: 베이스 페어링 또는 접근성)에 따라 달라진다. 따라서 반응성의 차이는 순서에 따른 구조의 발자국으로 작용할 수 있다. 다른 시약들은 RNA 구조의 다른 위치에서 반응하며 반응성의 스펙트럼이 다르다.[1] 최근의 발전은 많은 RNA의 구조와 세포 환경에서 RNA 분자의 직접 측정(세포 내 프로빙)[11]을 동시에 연구할 수 있게 한다.[12]
구조화된 RNA는 주어진 배양 시간 동안 먼저 프로빙 시약과 반응한다. 이 시약들은 반응 부위에서 RNA에 공밸런트 유도체를 형성할 것이다. 역분해효소를 이용해 RNA를 역번사하면 효소가 유도체에 의해 차단되기 때문에 생성된 DNA가 반응 위치에서 잘린다. 따라서 다양한 잘린 길이의 DNA 분자의 집합은 RNA를 따라 구조 프로파일을 반영하는 모든 기본 위치에서의 반응 빈도를 알려준다. 이것은 전통적으로 젤 위에서 DNA를 실행함으로써 분석되며, 밴드의 강도는 각 위치에서 잘린 부분을 관찰하는 빈도를 알려준다. 최근의 접근법은 처리량과 민감도가 더 높은 동일한 목적을 달성하기 위해 높은 처리량 시퀀싱을 사용한다.
반응도 프로파일은 특정 가설의 특정 위치에서 구조물의 정도를 연구하기 위해 사용하거나, 완전한 실험적으로 지원되는 구조 모델을 만들기 위해 연산 알고리즘과 함께 사용할 수 있다.[13]
사용된 화학 시약에 따라 수산화기와 같은 일부 시약은 RNA 분자를 대신 분해할 수 있다. 잘린 DNA의 결과는 같다. DMS와 같은 일부 시약들은 역성격화효소를 차단하지 않고 대신 DNA 사본의 현장에서 실수를 유발하기도 한다. 이들은 고투과 시퀀싱 방법을 사용할 때 검출될 수 있으며, 돌연변이 프로파일링(MaP)으로 프로빙의 결과 개선을 위해 사용되기도 한다.[14][15]
RNA 상의 위치는 국소 구조뿐만 아니라 그 위치 위의 결합 단백질로도 시약으로부터 보호될 수 있다. 이것은 단백질 결합을 분석하기 위해 화학적 탐침을 사용하는 몇몇 작업을 이끌어냈다.[16]
히드록실 래디컬 프로빙
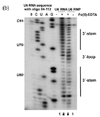
히드록실산소는 용액이 짧기 때문에 실험 시 생성해야 한다. 이것은 HO22, 아스코르브산, Fe(II)-EDTA 복합체를 사용하여 수행할 수 있다. 이 시약들은 펜턴 화학으로 수산화기를 생성하는 시스템을 형성한다. 히드록실산기는 핵산 분자와 반응할 수 있다.[17] 히드록실산소는 리보스/데옥시리보스 링을 공격하고 이로 인해 당인산 등뼈가 부러진다. 결합 단백질이나 RNA 3차 구조로부터 보호되고 있는 부위는 낮은 비율로 히드록실 라디칼에 의해 분해될 것이다.[17] 따라서 이러한 위치는 젤에 밴드가 없거나 시퀀싱을 통한 낮은 신호로 나타날 수 있다.[17][18]
DMS
DMS로 알려진 디메틸황산염은 2차 구조를 결정하기 위해 핵산을 수정하는 데 사용할 수 있는 화학물질이다. DMS와의 반응은 메틸화합물이라고 알려진 현장에서 메틸화합물을 첨가한다. 특히 DMS 메틸레이트는 아데닌(A)의 N1과 시토신(C)의 N3으로 베이스페어링 시 천연 수소결합 현장에 모두 위치한다.[19] 따라서 수정은 나선형 끝에서 쌍을 이룬 단일 가닥의 A와 C 뉴클레오바제에서만 발생할 수 있으며, GU 흔들림 쌍 또는 옆에 있는 베이스 쌍에서만 발생할 수 있으며, 후자는 베이스 쌍이 가끔 열릴 수 있는 위치다. 더욱이 수정된 사이트는 기저부위를 손상시킬 수 없기 때문에, 수정 사이트는 RT-PCR에 의해 검출될 수 있다. 여기서 역대본효소가 메틸화 베이스에서 떨어져 다른 잘린 cDNA를 생산한다. 잘린 cDNA는 젤 전기영양 또는 고투과 시퀀싱을 통해 식별할 수 있다.
잘라내기 기반 방법을 개선하여, 시퀀싱(DMS-MaPseq)을 사용한 DMS 돌연변이 프로파일링은 단일 RNA 분자에서 여러 DMS 수정을 감지할 수 있어 판독당 더 많은 정보를 얻을 수 있다(150nt, 0에서 1개의 절단 부위가 아니라 일반적으로 2~3개의 돌연변이 부위가 읽히는 경우) 저분자의 구조를 결정할 수 있다.RNA를 춤추고, 대체 2차 구조로 RNA의 하위 집단을 식별한다.[20] DMS-MaPseq는 DMS에 의해 메틸화 된 베이스 메틸화물과 마주쳤을 때 cDNA에서 돌연변이(잘림 대신)를 생성하는 온도조절 그룹 II intron 역분해효소(TGIRT)를 사용하지만, 그렇지 않으면 높은 충실도로 트랜시브를 역전시킨다. 결과 cDNA의 염기서열을 분석하면 역전사 중에 어떤 염기서열이 변이되었는지 알 수 있다. 이러한 염기서열은 원래 RNA에서 염기서열화되었을 수 없다.
DMS 수정은 예를 들어 DNA-단백질 상호작용에 사용될 수 있다.[21]
모양
프라이머 확장에 의해 분석된 선택적 2′-하이드록실 아틸레이션(SHAPE)은 구조적으로 유연한 영역에서 RNA의 백본을 우선적으로 수정하는 시약을 활용한다.
N-메틸리사토 무수화물(NMIA) 및 1-메틸-7-니트로이사토 무수화물(1M7)과 같은 시약은 2'히드록실 그룹과 반응하여 RNA 백본의 2'히드록실 위에 유도체를 형성한다. 다른 RNA 프로빙 기법에 사용되는 화학물질에 비해 이들 시약은 대체로 염기성 정체성에 치우치지 않고 순응성 역학에는 매우 민감하게 남아 있다는 장점이 있다. (대개 염기쌍화에 의해) 구속되는 뉴클레오티드는 손상되지 않은 뉴클레오티드에 비해 유도형성이 적다. 역분해효소를 가진 보완 DNA 프라이머를 확장하고 수정되지 않은 제어장치의 파편과 비교함으로써 주어진 RNA에서 각 뉴클레오티드에 대해 유도 형성을 정량화한다.[23] 그러므로 SHAPE는 개별 뉴클레오티드 수준에서 RNA 구조에 대해 보고한다. 이 데이터는 매우 정확한 2차 구조 모델을 생성하기 위해 입력으로 사용될 수 있다.[24] SHAPE는 전체 HIV-1 게놈을 포함한 다양한 RNA 구조를 분석하기 위해 사용되어 왔다.[25] 가장 좋은 방법은 화학적 검사 시약과 실험 데이터를 조합하여 사용하는 것이다.[26] SAFE-Seq SAFE는 RNA-Seq와 결합된 바코드 기반 멀티플렉싱에 의해 확장되며 고투과 방식으로 수행될 수 있다.[27]
카르보디미데스

또한 카르보디미드 모이티(carbodiimide moiety)는 노화되지 않은 N에 의한 핵포착 공격 시 요라실인 노출된 뉴클레오베이스와 더 작은 범위에서 구아닌에서 공밸런트 유도체를 형성할 수 있다. 이들은 주로 요람의 N3과 구아닌의 N1에 반응하여 염기서 수소 결합을 담당하는 두 부위를 수정한다.[19]
1-사이클로헥실-(2-모르폴리노에틸)카르보디미드 메토-p-톨루엔 술폰산염(CMCT 또는 CMC)[29][30]은 RNA 구조 탐사에 가장 많이 사용되는 카르보디미드다. DMS와 유사하게, 역전사 후 젤 전기영동 또는 고투과 시퀀싱으로 검출할 수 있다. G와 U에 반응하기 때문에 A와 C에 정보를 제공하는 DMS 프로빙 실험의 데이터를 보완하는 데 사용할 수 있다.[31]
1-ethyl-3-(3-dimethylaminopropyl)카르보디미드는 EDC라고도 하며 CMC와 유사한 반응성을 보이는 수용성 카르보디미드로서 RNA 구조의 화학적 프로빙에도 사용된다. EDC는 세포에 침투할 수 있으며, 따라서 원시 환경에서 RNA의 직접 세포 내 프로빙에 사용된다.[32][28]
케톡살, 글리옥살 및 파생상품


일부 1,2-디카르보닐 화합물은 N1과 N2에서 단일 가닥 구아닌(G)과 반응할 수 있으며, 왓슨-크릭 면에 5-엠베드 링 인덕트를 형성하고 있다.
1,1-Dihydroxy-3-ethoxy-2-butanone, also known as kethoxal, has a structure related to 1,2-dicarbonyls, and was the first in this category used extensively for the chemical probing of RNA. Kethoxal causes the modification of guanine, specifically altering the N1 and the exocyclic amino group (N2) simultaneously by covalent interaction.[35]
모두 키 1,2-디카르보닐 모이티를 운반하는 글리옥살, 메틸글리옥살, 페닐글리옥살은 모두 케톡살과 유사한 자유 구아닌과 반응하며, 구조화된 RNA에서 손상된 구아닌 염기를 탐사하는 데 사용할 수 있다. 화학적 특성 때문에, 이 시약들은 세포에 쉽게 침투할 수 있고, 따라서 그들의 고유 세포 환경에서 RNA를 분석하는 데 사용될 수 있다.[34]
레이저 또는 NAZ 프로빙
RNA(Light-Activated Structural Examination of RNA) 프로빙은 UV광선을 활용하여 니코티노일 아지드(NAz)를 활성화하여 물속에서 고반응 니트로늄 양이온을 생성하는데, 이 니트로늄은 무장애 프리델-크래프트 반응을 통해 C-8 위치에서 RNA의 용매 접근성 구아노신과 반응한다. 레이저 프로빙은 용제에 접근할 수 있는 한 단일 가닥 잔류물과 이중 가닥 잔류물을 모두 대상으로 한다. 히드록실 래디컬 프로빙은 체내 RNA의 용매 접근성을 측정하기 위해 싱크로트론 방사선이 필요하기 때문에 많은 연구소의 경우 세포 내 발자국 RNA에 히드록실 래디컬 프로빙을 적용하기 어렵다. 이와는 대조적으로 레이저 프로빙은 휴대용 UV 램프(20W)를 흥분용으로 활용하며, RNA 용매 접근성을 연구하기 위해 체내 연구에 레이저 프로빙을 적용하는 것이 훨씬 쉽다. 이 화학적 프로빙 방법은 경량 조절이 가능하며, 세포 내 RNA 결합 단백질의 발자국이 드러난 뉴클레오바제의 용제 접근성을 프로브한다.[36]
인라인 프로빙

인라인 프로빙은 RNA 구조를 수정하기 위해 어떤 종류의 화학 물질이나 시약으로도 처리하지 않는다. 이러한 유형의 프로빙 측정은 RNA의 구조 의존적인 분할을 사용한다. 단일 좌초 부위는 더 유연하고 불안정하며 시간이 지남에 따라 성능이 저하된다.[38] 인라인 프로빙 프로세스는 리간드 결합에 의한 구조물의 변화를 결정하는 데 종종 사용된다. 리간드를 결합하면 여러 가지 갈라진 무늬가 생길 수 있다. 인라인 프로빙의 과정은 장기간에 걸쳐 구조 또는 기능 RNA의 배양과 관련이 있다. 이 기간은 며칠이 될 수 있지만 각 실험마다 다르다. 인큐베이팅된 제품들은 겔로 실행되어 밴드를 시각화한다. 이 실험은 흔히 리간드가 없는 상태에서 1) 리간드로, 2) 두 가지 다른 조건을 사용하여 행해진다.[37] 갈라짐은 밴드 길이가 짧아지고 베이스가 손상되지 않은 영역을 나타낸다. 베이스가 손상된 부위는 자발적인 갈라짐에 덜 민감하기 때문이다.[38] 인라인 프로빙은 리간드 결합에 대응하여 RNA의 구조적 변화를 결정하는 데 사용할 수 있는 기능 검사다. 그것은 리간드에 반응하는 RNA 영역의 유연성과 결합의 변화를 직접적으로 보여줄 수 있을 뿐만 아니라, 그 반응을 아날로그 리간드와 비교할 수도 있다. 이 검사는 특히 리보스위치를 검사할 때 동적 연구에 일반적으로 사용된다.[38]
뉴클레오티드 아날로그 간섭 매핑(NAIM)
뉴클레오티드 아날로그 간섭 매핑(NAIM)은 뉴클레오티드와 어떤 면에서는 비슷하지만 기능이 부족한 분자인 뉴클레오티드 아날로그를 사용하여 RNA 분자의 각 위치에서 기능 그룹의 중요성을 결정하는 과정이다.[39][40] NAIM의 과정은 단일 뉴클레오티드 아날로그를 고유한 부지에 삽입하는 것이다. 이것은 T7 RNA 중합효소를 사용하여 짧은 RNA를 변환한 다음, 특정 위치에서 아날로그가 포함된 짧은 올리고뉴클레오티드를 합성한 다음, 리가제를 사용하여 DNA 템플릿에 함께 묶는 방법으로 할 수 있다.[39] 뉴클레오티드 아날로그는 인광로티오테이트로 태그되고, RNA 모집단의 활성 멤버는 비활성 멤버와 구별되며, 비활성 멤버는 인광로티오테이트 태그를 제거하며, 아날로그 사이트는 젤 전기영양과 자동 방사선 촬영으로 식별된다.[39] 이것은 요오드에 의한 인광로티오테이트의 갈라짐으로 인해 뉴클레오티드 아날로그 삽입 부위에서 분해되는 RNA가 발생하기 때문에 기능적으로 중요한 뉴클레오티드를 나타낸다. experiment[40]단지 통합 결과를 언제 어디서나 겔, 기능 RNAs는 아날로그 그 위치에 통합된 밴드를 선물 가지고 있을 것입니다에 나왔으나, 달리기가 아날로그 결과 중요성의 위치를 가리킨다라고 지시했다 겔에 이러한 불완전한 RNA분자함으로써 관심의 nucleotide 염기 서열 결정 법에 대한 확인될 수 있다. 에서 비기능성, 기능 RNA 분자가 겔 위에서 실행될 때, 겔의 그 위치에 해당하는 대역은 없을 것이다.[41] 이 프로세스는 아날로그가 단일 뉴클레오티드에 의해 다른 현장 고유 위치에 배치되는 전체 영역을 평가하는데 사용될 수 있으며, 이후 기능 RNA가 분리되어 젤 위에서 실행될 때 밴드가 생성되는 모든 영역은 비필수 뉴클레오티드를 나타내지만, 기능 RNA에 밴드가 없는 영역은 삽입하는 것을 나타낸다. 그 위치에 있는 뉴클레오티드 아날로그는 RNA 분자를 비기능적으로[39] 만들었다.
참조
- ^ a b Weeks, Kevin (2010). "Advances in RNA structure analysis by chemical probing". Current Opinion in Structural Biology. 20 (3): 295–304. doi:10.1016/j.sbi.2010.04.001. PMC 2916962. PMID 20447823.
- ^ Fürtig B, Richter C, Wöhnert J, Schwalbe H (October 2003). "NMR spectroscopy of RNA". ChemBioChem. 4 (10): 936–62. doi:10.1002/cbic.200300700. PMID 14523911. S2CID 33523981.
- ^ Addess, Kenneth J.; Feigon, Juli (1996). "Introduction to 1H NMR Spectroscopy of DNA". In Hecht, Sidney M. (ed.). Bioorganic Chemistry: Nucleic Acids. New York: Oxford University Press. ISBN 0-19-508467-5.
- ^ a b Wemmer, David (2000). "Chapter 5: Structure and Dynamics by NMR". In Bloomfield, Victor A.; Crothers, Donald M.; Tinoco, Ignacio (eds.). Nucleic acids: Structures, Properties, and Functions. Sausalito, California: University Science Books. ISBN 0-935702-49-0.
- ^ [고마토스 PJ, 스토에케니우스 W. 전자현미경 연구 RNAVIOVIRS RNA. 미국 국립과학원 절차. 1964년 12월 52:1449-1455. DOI: 10.1073/pnas.52.6.1449]
- ^ [ 마이클 맥주와 리처드 조벨(1961) "전자 얼룩 II: 얼룩진 DNA 분자의 가시성에 관한 전자 현미경 연구" J. 몰. 비올. 제3권, 제6권, 1961년 12월, 페이지 717–726, IN3–IN5"]
- ^ Frank, J; Penczek, P; Grassucci, R; Srivastava, S (Nov 1991). "Three-dimensional reconstruction of the 70S Escherichia coli ribosome in ice: the distribution of ribosomal RNA". The Journal of Cell Biology. 115 (3): 597–605. doi:10.1083/jcb.115.3.597. PMC 2289182. PMID 1918155.
- ^ Gopal, A; Zhou, ZH; Knobler, CM; Gelbart, WM (2012). "Visualizing large RNA molecules in solution". RNA. 18 (2): 284–299. doi:10.1261/rna.027557.111. PMC 3264915. PMID 22190747.
- ^ 코닝, R, 고메즈-블랑코, J, 아코프자나, 아이 외 비대칭 크라이오-EM 재구성 페이징 MS2는 게놈 구조를 현장에서 드러낸다. 냇 코뮌 7, 12524 (2016) https://doi.org/10.1038/ncomms12524
- ^ Dai, X.; Li, Z.; Lai, M.; et al. (2017). "In situ structures of the genome and genome-delivery apparatus in a single-stranded RNA virus". Nature. 541 (7635): 112–116. Bibcode:2017Natur.541..112D. doi:10.1038/nature20589. PMC 5701785. PMID 27992877.
- ^ Kwok, Chun Kit; Tang, Yin; Assmann, Sarah; Bevilacqua, Philip (April 2015). "The RNA structurome: transcriptome-wide structure probing with next-generation sequencing". Trends in Biochemical Sciences. 40 (4): 221–232. doi:10.1016/j.tibs.2015.02.005. PMID 25797096.
- ^ Kubota, M; Tran, C; Spitale, R (2015). "Progress and challenges for chemical probing of RNA structure inside living cells". Nature Chemical Biology. 11 (12): 933–941. doi:10.1038/nchembio.1958. PMC 5068366. PMID 26575240.
- ^ Mathews, DH; Disney, MD; Childs, JL; Schroeder, SJ; Zuker, M; Turner DH (2004). "Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure". Proceedings of the National Academy of Sciences. 101 (19): 7287–7292. Bibcode:2004PNAS..101.7287M. doi:10.1073/pnas.0401799101. PMC 409911. PMID 15123812.
- ^ Siegfried, N; Busan, S; Weeks, K (2014). "RNA motif discovery by SHAPE and mutational profiling (SHAPE-MaP)". Nature Methods. 11 (9): 959–965. doi:10.1038/nmeth.3029. PMC 4259394. PMID 25028896.
- ^ Sexton, A; Wang, P; Rutenberg-Schoenberg, M; Simon, M (2017). "Interpreting Reverse Transcriptase Termination and Mutation Events for Greater Insight into the Chemical Probing of RNA". Biochemistry. 56 (35): 4713–3721. doi:10.1021/acs.biochem.7b00323. PMC 5648349. PMID 28820243.
- ^ Smola, M; Calabrese, J; Weeks, K (2015). "Detection of RNA–Protein Interactions in Living Cells with SHAPE". Biochemistry. 54 (46): 6867–6875. doi:10.1021/acs.biochem.5b00977. PMC 4900165. PMID 26544910.
- ^ a b c d Karaduman R, Fabrizio P, Hartmuth K, Urlaub H, Luhrmann R (2006). "RNA structure and RNA-protein interactions in purified yeast U6 snRNPs". J. Mol. Biol. 356 (5): 1248–1262. doi:10.1016/j.jmb.2005.12.013. hdl:11858/00-001M-0000-0012-E5F8-4. PMID 16410014.
- ^ Tullius, T. D.; Dombroski, B. A. (1986). "Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein". Proceedings of the National Academy of Sciences. 83 (15): 5469–5473. Bibcode:1986PNAS...83.5469T. doi:10.1073/pnas.83.15.5469. PMC 386308. PMID 3090544.
- ^ a b Tijerina P, Mohr S, Russell R (2007). "DMS footprinting of structured RNAs and RNA-protein complexes". Nat Protoc. 2 (10): 2608–23. doi:10.1038/nprot.2007.380. PMC 2701642. PMID 17948004.
- ^ Zubradt, Meghan; Gupta, Paromita; Persad, Sitara; Lambowitz, Alan; Weissman, Jonathan; Rouskin, Silvi (2017). "DMS-MaPseq for genome-wide or targeted RNA structure probing in vivo". Nature Methods. 14 (1): 75–82. doi:10.1038/nmeth.4057. PMC 5508988. PMID 27819661.
- ^ Albert S. Baldwin Jr.; Marjorie Oettinger & Kevin Struhl (1996). "Unit 12.3: Methylation and Uracil Interference Assays for Analysis of Protein-DNA Interactions". Current Protocols in Molecular Biology. Vol. Chapter 12. Wiley. pp. Unit 12.3. doi:10.1002/0471142727.mb1203s36. PMID 18265086. S2CID 45581978.
- ^ Mortimer SA, Weeks KM (2007). "A Fast-Acting Reagent for Accurate Analysis of RNA Secondary and Tertiary Structure by SHAPE Chemistry". J Am Chem Soc. 129 (14): 4144–45. doi:10.1021/ja0704028. PMID 17367143.
- ^ Merino EJ, Wilkinson KA, Coughlan JL, Weeks KM (2005). "RNA structure analysis at single nucleotide resolution by selective 2′-hydroxyl acylation and primer extension (SHAPE)". J Am Chem Soc. 127 (12): 4223–31. doi:10.1021/ja043822v. PMID 15783204.
- ^ Deigan KE, Li TW, Mathews DH, Weeks KM (2009). "Accurate SHAPE-directed RNA structure determination". Proc Natl Acad Sci USA. 106 (1): 97–102. Bibcode:2009PNAS..106...97D. doi:10.1073/pnas.0806929106. PMC 2629221. PMID 19109441.
- ^ Watts JM, Dang KK, Gorelick RJ, Leonard CW, Bess JW Jr, Swanstrom R, Burch CL, Weeks KM (2009). "Architecture and secondary structure of an entire HIV-1 RNA genome". Nature. 460 (7256): 711–6. Bibcode:2009Natur.460..711W. doi:10.1038/nature08237. PMC 2724670. PMID 19661910.
- ^ Wipapat Kladwang; Christopher C. VanLang; Pablo Cordero; Rhiju Das (7 Sep 2011). "Understanding the errors of SHAPE-directed RNA structure modeling". Biochemistry. 50 (37): 8049–8056. arXiv:1103.5458. Bibcode:2011arXiv1103.5458K. doi:10.1021/bi200524n. PMC 3172344. PMID 21842868.
- ^ Lucks JB, Mortimer SA, Trapnell C, Luo S, Aviran S, Schroth GP, Pachter L, Doudna JA, Arkin AP (2011). "Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq)". Proc Natl Acad Sci USA. 108 (27): 11063–8. Bibcode:2011PNAS..10811063L. doi:10.1073/pnas.1106501108. PMC 3131332. PMID 21642531.
- ^ a b Wang, PY; Sexton, AN; Culligan, WJ; Simon, MD (2019). "Carbodiimide reagents for the chemical probing of RNA structure in cells". RNA. 25 (1): 135–146. doi:10.1261/rna.067561.118. PMC 6298570. PMID 30389828.
- ^ Fritz JJ, Lewin A, Hauswirth W, Agarwal A, Grant M, Shaw L (2002). "Development of hammerhead ribozymes to modulate endogenous gene expression for functional studies". Methods. 28 (2): 276–285. doi:10.1016/S1046-2023(02)00233-5. PMID 12413427.
- ^ Metz, D; Brown, G (1969). "Investigation of nucleic acid secondary structure by means of chemical modification with a carbodiimide reagent. II. Reaction between N-cyclohexyl-N'-β-(4-methylmorpholinium) ethylcarbodiimide and transfer ribonucleic acid". Biochemistry. 8 (6): 2329–2342. doi:10.1021/bi00834a013. PMID 4895018.
- ^ Incarnato, D; Neri, F; Anselmi, F; Oliviero, S (2014). "Genome-wide profiling of mouse RNA secondary structures reveals key features of the mammalian transcriptome". Genome Biology. 15 (491): 491. doi:10.1186/s13059-014-0491-2. PMC 4220049. PMID 25323333.
- ^ Mitchell, D; Renda, A; Douds, C; Babitzke, P; Assmann, S; Bevilacqua, P (2019). "In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC)". RNA. 25 (1): 147–157. doi:10.1261/rna.067868.118. PMC 6298566. PMID 30341176.
- ^ Noller HF, Chaires JB (1972). "Functional modification of 16S ribosomal RNA by kethoxal". Proc. Natl. Acad. Sci. USA. 69 (11): 3115–3118. Bibcode:1972PNAS...69.3115N. doi:10.1073/pnas.69.11.3115. PMC 389716. PMID 4564202.
- ^ a b Mitchell, D; Ritchey, L; Park, H; Babitzke, P; Assmann, S; Bevilacqua, P (2018). "Glyoxals as in vivo RNA structural probes of guanine base-pairing". RNA. 24 (1): 114–124. doi:10.1261/rna.064014.117. PMC 5733565. PMID 29030489.
- ^ Litt, M; Hancock, V (1967). "Kethoxal—a potentially useful reagent for the determination of nucleotide sequences in single-stranded regions of transfer ribonucleic acid". Biochemistry. 6 (6): 1848–1854. doi:10.1021/bi00858a036. PMID 6035923.
- ^ Feng C, Chan D, Joseph J, Muuronen M, Coldren WH, Dai N, Correa Jr IR, Furche F, Hadad CM, Spitale RC (2018). "Light-activated chemical probing of nucleobase solvent accessibility inside cells". Nat Chem Biol. 14 (3): 276–283. doi:10.1038/nchembio.2548. PMC 6203945. PMID 29334380.
- ^ a b Muhlbacher J, Lafontaine DA (2007). "Ligand recognition determinants of guanine riboswitches". Nucleic Acids Research. 35 (16): 5568–5580. doi:10.1093/nar/gkm572. PMC 2018637. PMID 17704135.
- ^ a b c Regulski, E; Breaker, R (2008). Wilusz, J (ed.). In-Line Probing Analysis of Riboswitches. Post-Transcriptional Gene Regulation. Methods in Molecular Biology. Vol. 419. Totowa, NJ: Humana Press. pp. 53–67. doi:10.1007/978-1-59745-033-1_4. ISBN 978-1-58829-783-9. PMID 18369975.
- ^ a b c d Ryder SP, Strobel SA (1999). "Nucleotide Analog Interference Mapping". Methods. 18 (1): 38–50. doi:10.1006/meth.1999.0755. PMID 10208815.
- ^ a b Waldsich C (2008). "Dissecting RNA folding by nucleotide analog interference mapping (NAIM)". Nature Protocols. 3 (5): 811–823. doi:10.1038/nprot.2008.45. PMC 2873565. PMID 18451789.
- ^ Strobel SA, Shetty K (1997). "Defining the chemical groups essential for Tetrahymena group I intron function by nucleotide analog interference mapping". Proc. Natl. Acad. Sci. USA. 94 (7): 2903–2908. Bibcode:1997PNAS...94.2903S. doi:10.1073/pnas.94.7.2903. PMC 20295. PMID 9096319.