캡시드
Capsid

캡시드는 바이러스의 단백질 껍질로 유전 물질을 감싸고 있다.그것은 프로토머라고 불리는 단백질로 만들어진 몇 개의 올리고머(반복하는) 구조 서브 유닛으로 구성됩니다.개별 단백질에 대응하거나 대응하지 않을 수 있는 관측 가능한 3차원 형태학적 소단위들을 캡소미어라고 한다.캡시드를 구성하는 단백질은 캡시드 단백질 또는 바이러스 피복 단백질이라고 불립니다.캡시드와 내부 게놈은 뉴클레오캡시드라고 불린다.
캡시드는 그 구조에 따라 크게 분류된다.대부분의 바이러스는 나선형 또는 20면체[2][3] 구조의 캡시드를 가지고 있다.박테리오파지와 같은 일부 바이러스는 탄성과 [4]정전기의 제약으로 인해 더 복잡한 구조를 발달시켰다.20개의 정삼각형 면으로 이루어진 이십면체 모양은 구에 가깝고, 나선 모양은 스프링 모양을 닮아 원통형 공간을 차지하지만 원통형 자체는 [5]아니다.캡시드 표면은 하나 이상의 단백질로 구성될 수 있다.예를 들어, 구제역 바이러스 캡시드는 VP1-3이라는 [6]이름의 세 개의 단백질로 구성된 얼굴을 가지고 있다.
어떤 바이러스들은 포락되어 있는데, 이것은 캡시드가 바이러스 포락선으로 알려진 지질막으로 덮여 있다는 것을 의미한다.외피는 바이러스의 숙주의 세포 내막에서 캡시드에 의해 획득됩니다; 예로는 내부 핵막, 골지막,[7] 그리고 세포의 외막을 포함합니다.
일단 바이러스가 세포를 감염시키고 자기 복제를 시작하면, 새로운 캡시드 서브유닛은 세포의 단백질 생합성 메커니즘을 사용하여 합성된다.나선형 캡시드를 가진 바이러스, 특히 RNA 게놈을 가진 바이러스를 포함한 일부 바이러스에서 캡시드 단백질은 그들의 게놈과 함께 결합됩니다.다른 바이러스, 특히 이중가닥 DNA 게놈을 가진 더 복잡한 바이러스에서 캡시드 단백질은 하나의 정점에 특화된 간문 구조를 포함하는 빈 전구체 프로캡시드로 모인다.이 포털을 통해 바이러스 DNA가 캡시드로 [8]이동한다.
주요 캡시드 단백질(MCP) 아키텍처의 구조 분석을 사용하여 바이러스를 계통별로 분류했습니다.예를 들어 박테리오파지 PRD1, 조류바이러스 Paramecium bursaria Chlorella virus-1(PBCV-1), 미미바이러스 및 포유동물 아데노바이러스는 같은 [9][10][11][12]계통에 배치되고 꼬리 이중사슬 DNA 박테리오파지(Caudovirales) 및 헤르페스바이러스는 제2계통에 속한다.
특정 모양
이십면체

이십면체 구조는 바이러스들 사이에서 매우 흔하다.20면체는 12개의 5중 꼭지점으로 구분된 20개의 삼각형 면으로 구성되며 60개의 비대칭 단위로 구성됩니다.따라서, 20면체 바이러스는 60N개의 단백질 서브유닛으로 구성되어 있다.Donald Caspar와 Aaron Klug가 [13]제안한 "준등가 원리"를 사용하여 정십면체 캡시드의 캡소미어의 수와 배열을 분류할 수 있다.골드버그 다면체와 마찬가지로, 정십면체 구조는 오각형과 육각형으로 구성되어 있다고 볼 수 있다.구조물은 h1 { h \ 1} k 0 2개의 정수 h 및 k 0으로 색인화할 수 있습니다.구조물은 펜타머 가장자리에서h 스텝을 밟아 시계 반대 방향으로 60도 회전시킨 후 다음 펜타머에 도달하기 위해k 스텝을 밟는 것으로 생각할 수 있습니다.capsid의 삼각형 번호T는 다음과 같이 정의됩니다.
이 방법에서 정십면체 캡시드는 12개의 펜타머와 10([14][15]T - 1)개의 헥사머를 포함한다.T 번호는 캡시드의 [16]크기와 복잡성을 나타냅니다.h, k 및 T의 많은 값에 대한 기하학적 예는 측지학 다면체 목록과 골드버그 다면체에서 찾을 수 있습니다.
이 규칙에는 다음과 같은 많은 예외가 있습니다.예를 들어 폴리오마바이러스와 파필로마바이러스는 준T=7 격자상의 6가 위치에 6가 위치하는 대신 펜타머를 가진다.레오바이러스, 로타바이러스 및 박테리오파지 δ6를 포함한 이중결합 RNA 바이러스 계통의 구성원은 비대칭 단위로 T=2캡시드에 해당하는 120개의 캡시드 단백질 복사본으로 구성된 캡시드를 가지고 있다.마찬가지로, 많은 작은 바이러스들은 T = 3 (또는 P = 3) Capsid를 가지고 있으며, 이것은 T = 3 격자에 따라 구성되지만, 세 개의 준등가적인 위치를 차지하고 있는 별개의 폴리펩타이드를 가지고 있다.
T-number는 다양한 방법으로 표현될 수 있다. 예를 들어 T = 1은 이십면체 또는 이십면체의 종류에 따라 T = 3은 잘린 12면체, 이십면체 또는 잘린 이십면체, 삼면체, 삼면체로 표현될 수 있다.또는 5면체 [18][clarification needed]12면체입니다.
프롤레이트
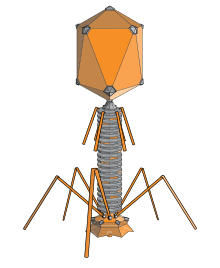
길쭉한 20면체는 박테리오파지의 머리부분에서 흔히 볼 수 있는 형태이다.이러한 구조는 양 끝에 캡이 있는 실린더로 구성됩니다.실린더는 10개의 가늘고 긴 삼각형 면으로 구성되어 있습니다.임의의 양의 [19]정수일 수 있는 Q 숫자(또는mid T)는 실린더의 10개의 삼각형을 구성하는 비대칭 하위 단위로 구성된 삼각형의 수를 지정합니다.대문자 수는 T([20]또는end T) 번호로 분류됩니다.
대장균은 프롤레이트 머리 구조를 가진 박테리오파지 T4의 숙주이다.박테리오파지 부호화 gp31 단백질은 대장균 샤페론 단백질 GroES와 기능적으로 상동성이 있으며 [21]감염 중 박테리오파지 T4 비리온의 집합에서 이를 대체할 수 있는 것으로 보인다.gp31은 GroES와 마찬가지로 박테리오파지 T4 주요 캡시드 단백질 gp23의 [21]생체 내 접힘 및 조립에 절대적으로 필요한 GroEL 샤페로닌과 안정된 복합체를 형성한다.
헬리컬
많은 막대 모양과 필라멘트 모양의 식물 바이러스들은 나선형 [22]대칭을 가진 캡시드를 가지고 있다.나선 구조는 n배축 [23]대칭에 의해 관련된 n개의 1-D 분자 나선 세트로 설명될 수 있다.헬리컬 변환은 2가지 범주로 분류된다. 1차원 및 2차원 헬리컬 [23]시스템이다.전체 나선 구조를 만드는 것은 단백질 데이터 [23]뱅크에서 코드화된 일련의 변환 및 회전 행렬에 의존합니다.나선대칭은 공식 P = μ x θ로 나타내며, μ는 나선의 회전당 구조 단위 수이고, μ는 단위당 축방향 상승, P는 나선의 피치이다.구조물은 [24]나선의 길이를 바꾸면 어떤 부피도 봉입할 수 있다는 특성 때문에 개방적이라고 한다.가장 잘 알려진 나선형 바이러스는 담배 모자이크 [22]바이러스이다.바이러스는 (+) 가닥 RNA의 단일 분자이며, 나선 내부의 각 피복 단백질은 RNA 게놈의 세 개의 뉴클레오티드를 결합합니다.인플루엔자 A 바이러스는 여러 개의 리보핵단백질을 구성함으로써 달라지며, 바이러스 NP 단백질은 RNA를 나선 구조로 조직한다.담배 모자이크 바이러스는 나선 [22]회전당 16.33개의 단백질 서브유닛을 가지고 있는 반면, 인플루엔자 A 바이러스는 28개의 아미노산 꼬리 [25]루프를 가지고 있습니다.
기능들
capsid의 기능은 다음과 같습니다.
- 게놈을 보호하고
- 게놈을 전달하고
- 호스트와 상호 작용합니다.
이 바이러스는 치명적인 화학 물질과 물리적 물질로부터 게놈을 보호하기 위해 안정적이고 보호적인 단백질 껍데기를 조립해야 한다.여기에는 극단적인 pH 또는 온도, 단백질 분해 및 핵 분해 효소가 포함된다.비포장 바이러스의 경우 캡시드 자체는 숙주 세포상의 수용체와의 상호작용에 관여하여 숙주 세포막의 침투와 캡시드의 내부화를 초래할 수 있다.게놈의 송달은 캡시드의 후속적인 언코팅 또는 분해와 세포질로의 게놈 방출 또는 숙주 세포핵으로의 특수한 간문구조를 통한 게놈의 직접 방출에 의해 이루어진다.
기원과 진화
많은 바이러스 캡시드 단백질이 기능적으로 다양한 세포 [26]단백질로부터 여러 경우에 걸쳐 진화해왔다는 것이 제안되었다.세포 단백질의 모집은 진화의 다른 단계에서 일어난 것으로 보여서, 세포 유기체의 분리에 앞서 일부 세포 단백질이 포획되고 재관능화된 반면, 다른 세포 단백질은 비교적 최근에 납치되었다.그 결과, 일부 캡시드 단백질은 원거리 관련 유기체를 감염시키는 바이러스(예: 젤리 롤 접힌 캡시드 단백질)에 널리 분포하는 반면, 다른 것들은 특정 바이러스 그룹(예: 알파 바이러스의 [26][27]캡시드 단백질)에 제한된다.
컴퓨터 모델(2015)은 캡시드가 바이러스보다 먼저 생겨났을 수 있으며, 특정 유전자가 이러한 구조의 형성에 책임이 있는 경우 유전자 기생충의 수가 증가하면 생존할 수 없기 때문에 리플리케이터 군집 사이의 수평 전달 수단 역할을 했다는 것을 보여주었다.자급자족하는 [28]공동체의 생존에 기여했습니다세포 유기체 사이의 이러한 조상 유전자의 이동은 [27]진화 과정에서 새로운 바이러스의 출현에 유리할 수 있다.
「 」를 참조해 주세요.
레퍼런스
- ^ Asensio MA, Morella NM, Jakobson CM, Hartman EC, Glasgow JE, Sankaran B, et al. (September 2016). "A Selection for Assembly Reveals That a Single Amino Acid Mutant of the Bacteriophage MS2 Coat Protein Forms a Smaller Virus-like Particle". Nano Letters. 16 (9): 5944–50. Bibcode:2016NanoL..16.5944A. doi:10.1021/acs.nanolett.6b02948. OSTI 1532201. PMID 27549001.
- ^ Lidmar J, Mirny L, Nelson DR (November 2003). "Virus shapes and buckling transitions in spherical shells". Physical Review E. 68 (5 Pt 1): 051910. arXiv:cond-mat/0306741. Bibcode:2003PhRvE..68e1910L. doi:10.1103/PhysRevE.68.051910. PMID 14682823. S2CID 6023873.
- ^ Vernizzi G, Olvera de la Cruz M (November 2007). "Faceting ionic shells into icosahedra via electrostatics". Proceedings of the National Academy of Sciences of the United States of America. 104 (47): 18382–6. Bibcode:2007PNAS..10418382V. doi:10.1073/pnas.0703431104. PMC 2141786. PMID 18003933.
- ^ Vernizzi G, Sknepnek R, Olvera de la Cruz M (March 2011). "Platonic and Archimedean geometries in multicomponent elastic membranes". Proceedings of the National Academy of Sciences of the United States of America. 108 (11): 4292–6. Bibcode:2011PNAS..108.4292V. doi:10.1073/pnas.1012872108. PMC 3060260. PMID 21368184.
- ^ Branden C, Tooze J (1991). Introduction to Protein Structure. New York: Garland. pp. 161–162. ISBN 978-0-8153-0270-4.
- ^ "Virus Structure (web-books.com)".
- ^ Alberts B, Bray D, Lewis J, Raff M, Roberts K, Watson JD (1994). Molecular Biology of the Cell (4th ed.). p. 280.
- ^ Newcomb WW, Homa FL, Brown JC (August 2005). "Involvement of the portal at an early step in herpes simplex virus capsid assembly". Journal of Virology. 79 (16): 10540–6. doi:10.1128/JVI.79.16.10540-10546.2005. PMC 1182615. PMID 16051846.
- ^ Krupovic M, Bamford DH (December 2008). "Virus evolution: how far does the double beta-barrel viral lineage extend?". Nature Reviews. Microbiology. 6 (12): 941–8. doi:10.1038/nrmicro2033. PMID 19008892. S2CID 31542714.
- ^ Forterre P (March 2006). "Three RNA cells for ribosomal lineages and three DNA viruses to replicate their genomes: a hypothesis for the origin of cellular domain". Proceedings of the National Academy of Sciences of the United States of America. 103 (10): 3669–74. Bibcode:2006PNAS..103.3669F. doi:10.1073/pnas.0510333103. PMC 1450140. PMID 16505372.
- ^ Khayat R, Tang L, Larson ET, Lawrence CM, Young M, Johnson JE (December 2005). "Structure of an archaeal virus capsid protein reveals a common ancestry to eukaryotic and bacterial viruses". Proceedings of the National Academy of Sciences of the United States of America. 102 (52): 18944–9. doi:10.1073/pnas.0506383102. PMC 1323162. PMID 16357204.
- ^ Laurinmäki PA, Huiskonen JT, Bamford DH, Butcher SJ (December 2005). "Membrane proteins modulate the bilayer curvature in the bacterial virus Bam35". Structure. 13 (12): 1819–28. doi:10.1016/j.str.2005.08.020. PMID 16338410.
- ^ Caspar DL, Klug A (1962). "Physical principles in the construction of regular viruses". Cold Spring Harbor Symposia on Quantitative Biology. 27: 1–24. doi:10.1101/sqb.1962.027.001.005. PMID 14019094.
- ^ Carrillo-Tripp M, Shepherd CM, Borelli IA, Venkataraman S, Lander G, Natarajan P, et al. (January 2009). "VIPERdb2: an enhanced and web API enabled relational database for structural virology". Nucleic Acids Research. 37 (Database issue): D436-42. doi:10.1093/nar/gkn840. PMC 2686430. PMID 18981051. Archived from the original on 2018-02-11. Retrieved 2011-03-18.
- ^ Johnson JE, Speir JA (2009). Desk Encyclopedia of General Virology. Boston: Academic Press. pp. 115–123. ISBN 978-0-12-375146-1.
- ^ Mannige RV, Brooks CL (March 2010). "Periodic table of virus capsids: implications for natural selection and design". PLOS ONE. 5 (3): e9423. Bibcode:2010PLoSO...5.9423M. doi:10.1371/journal.pone.0009423. PMC 2831995. PMID 20209096.
- ^ Sgro J. "Virusworld". Institute for Molecular Virology. University of Wisconsin-Madison.
- ^ Damodaran KV, Reddy VS, Johnson JE, Brooks CL (December 2002). "A general method to quantify quasi-equivalence in icosahedral viruses". Journal of Molecular Biology. 324 (4): 723–37. doi:10.1016/S0022-2836(02)01138-5. PMID 12460573.
- ^ Luque A, Reguera D (June 2010). "The structure of elongated viral capsids". Biophysical Journal. 98 (12): 2993–3003. Bibcode:2010BpJ....98.2993L. doi:10.1016/j.bpj.2010.02.051. PMC 2884239. PMID 20550912.
- ^ Casjens S (2009). Desk Encyclopedia of General Virology. Boston: Academic Press. pp. 167–174. ISBN 978-0-12-375146-1.
- ^ a b 마루시치 EI, 쿠로치키나 LP, 메시안지노프 VV박테리오파지 T4 어셈블리의 샤페론.생화학(모스크)1998;63(4):399-406
- ^ a b c Yamada S, Matsuzawa T, Yamada K, Yoshioka S, Ono S, Hishinuma T (December 1986). "Modified inversion recovery method for nuclear magnetic resonance imaging". The Science Reports of the Research Institutes, Tohoku University. Ser. C, Medicine. Tohoku Daigaku. 33 (1–4): 9–15. PMID 3629216.
- ^ a b c Aldrich RA (February 1987). "Children in cities--Seattle's KidsPlace program". Acta Paediatrica Japonica. 29 (1): 84–90. doi:10.1111/j.1442-200x.1987.tb00013.x. PMID 3144854. S2CID 33065417.
- ^ Racaniello VR, Enquist LW (2008). Principles of Virology, Vol. 1: Molecular Biology. Washington, D.C: ASM Press. ISBN 978-1-55581-479-3.
- ^ Ye Q, Guu TS, Mata DA, Kuo RL, Smith B, Krug RM, Tao YJ (26 December 2012). "Biochemical and structural evidence in support of a coherent model for the formation of the double-helical influenza A virus ribonucleoprotein". mBio. 4 (1): e00467–12. doi:10.1128/mBio.00467-12. PMC 3531806. PMID 23269829.
- ^ a b Krupovic M, Koonin EV (March 2017). "Multiple origins of viral capsid proteins from cellular ancestors". Proceedings of the National Academy of Sciences of the United States of America. 114 (12): E2401–E2410. doi:10.1073/pnas.1621061114. PMC 5373398. PMID 28265094.
- ^ a b Krupovic M, Dolja VV, Koonin EV (July 2019). "Origin of viruses: primordial replicators recruiting capsids from hosts" (PDF). Nature Reviews. Microbiology. 17 (7): 449–458. doi:10.1038/s41579-019-0205-6. PMID 31142823. S2CID 169035711.
- ^ Jalasvuori M, Mattila S, Hoikkala V (2015). "Chasing the Origin of Viruses: Capsid-Forming Genes as a Life-Saving Preadaptation within a Community of Early Replicators". PLOS ONE. 10 (5): e0126094. Bibcode:2015PLoSO..1026094J. doi:10.1371/journal.pone.0126094. PMC 4425637. PMID 25955384.
추가 정보
- Williams R (1 June 1979). The Geometrical Foundation of Natural Structure: A Source Book of Design. pp. 142–144, Figures 4-49, 50, 51: Custers of 12 spheres, 42 spheres, 92 spheres. ISBN 978-0-486-23729-9.
- Pugh A (1 September 1976). Polyhedra: A Visual Approach. Chapter 6. The Geodesic Polyhedra of R. Buckminster Fuller and Related Polyhedra. ISBN 978-0-520-02926-2.
- Almansour I, Alhagri M, Alfares R, Alshehri M, Bakhashwain R, Maarouf A (January 2019). "IRAM: virus capsid database and analysis resource". Database: The Journal of Biological Databases and Curation. 2019. doi:10.1093/database/baz079. PMC 6637973. PMID 31318422.








