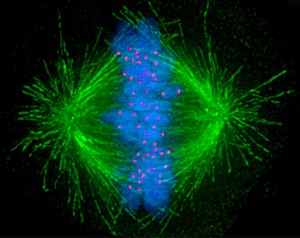
키네토코레
Kinetochore키네토코어(/kˈnɛtɔk,r/, /-ːniətɔk)r/)는 진핵세포 내의 중복염색체와 관련된 원반상 단백질 구조이며, 이 원반상 단백질 구조에서는 방추섬유가 세포 분열 중에 부착되어 자매염색체를 [1]떼어낸다.동원체는 동원체에 모여 유사분열과 감수분열 동안 유사분열 방추의 미세관 중합체와 염색체를 연결한다.키네토코어라는 용어는 1934년 Lester W[2]. Sharp에 의해 Cytology 책의 각주에 처음 사용되었고 [3]1936년에 일반적으로 받아들여졌다.샤프의 각주에는 다음과 같이 적혀 있다: "편리한 용어 kinetochore (= movement place)"는 1932년 [4]컬럼비아 대학교 1학년 때 입사한 존 알렉산더 무어를 지칭하는 것으로 보인다.
척추동물, 곰팡이, 그리고 대부분의 식물을 포함한 단일 중심 유기체는 각각의 염색체 위에 단일 국소적인 동심원을 조립하는 단일 중심 영역을 가지고 있습니다.선충과 몇몇 식물과 같은 홀로중심적인 유기체는 [5]염색체의 전체 길이를 따라 운동체를 형성한다.
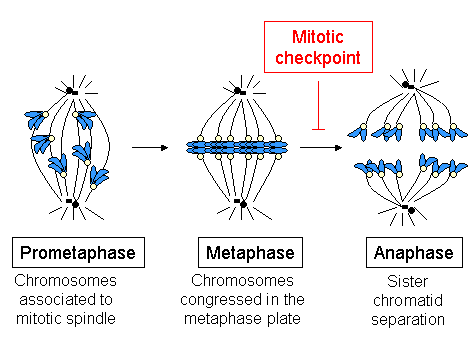
키네토코어는 세포 분열 동안 염색체의 두드러진 움직임을 시작하고, 통제하고, 감독한다.S단계에서 각 염색체에서 DNA의 양이 두 배로 늘어난 후 일어나는 유사분열 동안 (같은 수의 염색체를 유지하면서) 두 개의 자매 염색체가 동원체에 의해 결합된다.각 염색분체는 반대방향으로 마주보고 유사분열방추장치의 반대극에 부착하는 자체 키네토코어를 가지고 있다.중기에서 후기로의 이행에 따라 자매 염색체들은 서로 분리되고, 각 염색체 상의 개별 키네토코어는 두 개의 새로운 딸세포를 정의하는 방추극으로 그들의 움직임을 촉진합니다.그러므로 운동세포는 전형적으로 유사분열과 감수분열과 관련된 염색체 분리에 필수적이다.
키네토초레 구조
키네토코어에는 다음 2개의 영역이 있습니다.
- 동원체 DNA와 밀접하게 관련되어 세포 주기 내내 지속되는 염색질의 특수한 형태로 조립되는 내부 키네토코어
- 마이크로튜브와 상호작용하는 외부 키네토코어; 외부 키네토코어는 세포 분열 중에만 조립되고 기능하는 많은 동일한 구성 요소를 가진 매우 역동적인 구조입니다.
심지어 가장 단순한 키네토코어도 19개 이상의 다른 단백질로 구성되어 있다.이러한 단백질의 많은 부분이 진핵생물 종 사이에 보존되는데, 여기에는 운동세포가 DNA와 결합하는 것을 돕는 특별한 히스톤 H3 변종(CENP-A 또는 CenH3)이 포함된다.키네토코어의 다른 단백질은 그것을 유사분열 방추의 미소관(MTs)에 부착시킨다.다이나인과 키네신을 포함한 운동단백질도 있어 유사분열 중에 염색체를 움직이는 힘을 발생시킨다.Mad2와 같은 다른 단백질은 자매 키네토코어 사이의 장력뿐만 아니라 미소관 부착을 감시하고 스핀들 체크포인트를 활성화하여 이들 중 하나가 [6]없을 때 세포 사이클을 정지시킨다.키네토코어 기능에 필수적인 실제 유전자 세트는 종마다 다르다.[7][8]
키네토코어 기능에는 염색체의 스핀들 내 MT에 대한 정착, 정착 확인, 스핀들 체크포인트 활성화 및 세포 [9]분열 중 염색체 이동을 촉진하는 힘 생성 참여가 포함된다.한편, 마이크로튜브는 α-튜브린과 β-튜브린으로 이루어진 준안정성 고분자로, [10]동적 불안정성으로 알려진 현상인 성장기와 수축기를 번갈아 이동한다.MT는 염색체의 움직임과 분리를 제어하기 위해 키네토코어 기능과 통합된 매우 역동적인 구조이다.또한 유사분열과 감수분열 사이에 키네토코어 조직이 다르며 감수분열 특이사건인 감수분열, 자매키네토코어 모노 오리엔테이션, 중심체 응집체 보호, 방추극체 응집 및 [11][12]복제에 필수적이라는 보고가 있다.
동물 세포 내
키네토코어는 여러 층으로 구성되어 있으며, 처음에 전자현미경법의 [13][14]기존 고정 및 염색 방법에 의해 관찰되었다(C에 의해 검토됨).1982년[15] Rieder)와 최근에는 급속 냉동 및 [16]치환에 의한 것입니다.

키네토코어의 가장 깊은 층은 내판이며, 이는 특수한 히스톤(이 영역에서 히스톤 H3를 대체하는 CENP-A로 명명됨), 보조 단백질 및 DNA를 나타내는 뉴클레오솜을 포함하는 염색질 구조 위에 구성되어 있다. 센트로메어(위성 DNA)에서의 DNA 구성은 척추동물의 가장 잘 알려지지 않은 측면 중 하나이다.키네토코어내부 판은 세포 주기 내내 분리된 헤테로크로마틴 도메인처럼 보입니다.
안쪽 판의 바깥쪽은 대부분 단백질로 구성된 바깥쪽 판이다.이 구조는 핵 외피가 [13]분해된 후에만 염색체 표면에 조립된다.척추동물의 키네토코어의 외판은 MTs(+) 말단(Kinetochore MTs, kMTs, kinetochore MTs의 이름을 따서 kMTs라고 함)을 위한 약 20개의 고정 부위를 포함하고 있으며, 효모의 키네토코어의 외판(Sacharomyces cerevisiae)은 1개의 고정 부위만을 포함하고 있다.
키네토코어의 가장 바깥쪽 도메인은 섬유상 코로나를 형성하며, 이는 기존의 현미경 검사로 시각화할 수 있지만 MT가 없는 경우에만 시각화할 수 있다.이 코로나는 방추 검사점, 미세관 고정 및 염색체 거동 조절에 관여하는 상주 및 임시 단백질의 동적 네트워크에 의해 형성된다.
유사분열 동안, 완전한 염색체를 형성하는 각각의 자매 염색체들은 그들만의 키네토코어를 가지고 있다.배양된 포유동물 [17]세포에서 G2상 말기에 먼저 뚜렷한 자매 운동세포가 관찰될 수 있다.이 초기 키네토코어는 핵 외피가 붕괴되기 [18]전에 성숙한 층 구조를 보여준다.고등 진핵생물에서 키네토코어 조립을 위한 분자 경로는 생쥐와 배양된 닭 세포에서 유전자 녹아웃을 사용하여 연구되었으며, C. 엘레강스, 드로소필라 및 인간 세포에서 RNA 간섭(RNAi)을 사용하여 지금까지 [citation needed]얻은 데이터를 설명할 수 있는 간단한 선형 경로는 없다.
키네토코어에 조립되는 첫 번째 단백질은 CENP-A(사카로미세스 세레비시아의 Cse4)이다.이 단백질은 히스톤 H3의 [19]특수 아이소폼이다.CENP-A는 내부 키네토코어 단백질 CENP-C, CENP-H 및 CENP-I/MIS6의 [20][21][22][23][24]통합에 필요하다.CENP-A 의존 경로에서 이러한 단백질의 관계는 완전히 정의되지 않았다.예를 들어, CENP-C 국소화는 닭 세포에서 CENP-H를 요구하지만, 인간 세포에서는 CENP-I/MIS6와 독립적이다.C. elegans와 메타조아에서 외부 키네토코어에서 많은 단백질의 통합은 궁극적으로 CENP-A에 의존한다.
키네토코어 단백질은 유사분열 동안 키네토코어에서의 농도에 따라 분류될 수 있다: 일부 단백질은 세포 분열 내내 결합되어 있는 반면, 다른 단백질은 농도가 변화한다.또한 키네토코어의 결합 부위에서 천천히(안정적) 또는 빠르게(동적) 재활용할 수 있다.
- 전상부터 후기까지 레벨이 안정된 단백질은 Ndc80 복합체,[25][26] KNL/KBP 단백질(kinetochore-null/[27]KNL 결합단백질[27]), MIS 단백질 및 CENP-F와 [28][29]같은 내측 플레이트의 구성성분과 외측 키네토코어의 안정성분을 포함한다.이러한 단백질은 구성 성분과 함께 운동장에서 내부 및 외부 구조의 핵핵을 구성하는 것으로 보인다.
- 유사분열 시 키네토코어의 농도가 변화하는 동적 성분에는 분자 모터 CENP-E와 다이네인(및 이들의 표적 성분 ZW10과 ROD), 스핀들 체크포인트 단백질(Mad1, Mad2, BubR1, Cdc20 등)이 포함된다.이러한 단백질은 미소관이 없을 때 높은 농도로 키네토코어에 모여들지만, 키네토코어에 고정된 MT의 수가 많을수록 이들 단백질의 [30]농도는 낮아진다.중기에서 CENP-E, Bub3 및 Bub1은 유리 키네토코어와 비교하여 약 3~4배 감소하는 반면, 다이닌/다이낙틴, Mad1, Mad2 및 BubR1은 10~[30][31][32][33]100배 이상 감소한다.
- 외판에 존재하는 방추체크포인트 단백질 수치는 MT가 [33]고정될 때 감소하는 반면, EB1, APC 및 Ran 경로(RanGap1, RanBP2)의 단백질과 같은 다른 성분들은 MT가 [34][35][36][37]고정될 때만 키네토코어와 관련된다.이는 마이크로튜브의 플러스 엔드(+)를 인식하는 키네토코어의 메커니즘에 속할 수 있으며, 마이크로튜브가 올바르게 고정되고 고정된 상태로 있을 때의 동적 거동을 조절합니다.
2010년 연구는 키네토코어를 [38]포함한 척추동물 염색체의 단백질 구성을 분석하기 위해 복잡한 방법(MCCP)을 사용했다.이 연구는 키네토코어에 대한 생화학적 농도를 포함하지 않지만, 125개의 알려진 모든 중심색소 단백질로부터 펩타이드를 포함한 모든 중심색소 서브콤플렉스를 포함한다.이 연구에 따르면, 아직 알려지지 않은 약 100개의 키네토코어 단백질이 존재하는데, 이것은 가장 복잡한 세포 하부 구조 중 하나인 키네토코어를 확인하는 유사분열 동안 알려진 구조를 두 배로 증가시킨다.일관되게, 포괄적인 문헌 조사는 최소 196개의 [39]인간 단백질이 이미 실험적으로 키네토코어에서 국소화된 것으로 나타난 것을 보여주었다.
기능.
하나의 키네토코어에 부착된 미세관의 수는 다양합니다: 사카로미세스 세레비시아에 있는 MT는 각 키네토코어에 결합되어 있는 반면, 포유동물에는 각 키네토코어에 [40]결합되어 있는 MT가 15-35개 있을 수 있습니다.그러나 스핀들 내의 모든 MT가 하나의 kinetochore에 부착되는 것은 아닙니다.하나의 중심체에서 다른 중심체로 뻗어나가는 MT가 있고(스핀들 길이를 담당한다), 긴 MT 사이에 짧은 MT가 서로 맞물려 있다(B교수).니클라스(듀크대)는 레이저 광선을 이용해 MT-키네토코어 부착을 분해하면 염색분체가 더 이상 움직일 수 없어 염색체 [41]분포에 이상이 생긴다는 것을 보여줬다.이 실험들은 또한 키네토코어가 극성을 가지고 있다는 것을 보여주었고, 한쪽 또는 다른 쪽 중심체에서 나오는 MT에 대한 키네토코어 부착은 그 방향에 따라 결정된다는 것을 보여주었다.이 특이성은 오직 하나의 염색분체가 각 스핀들 쪽으로 이동함을 보장하며, 따라서 유전 물질의 정확한 분포를 보장합니다.따라서 Kinetochore의 기본 기능 중 하나는 스핀들에 대한 MT 부착이며, 이는 자매 염색체를 올바르게 분리하기 위해 필수적이다.고정 상태가 올바르지 않으면 오류가 발생하여 세포에 치명적인 결과를 초래할 수 있습니다.이를 방지하기 위해 (스핀들어셈블리 체크포인트로서) 에러 검출 및 정정 메커니즘이 있으며, 그 컴포넌트는 키네토코어에도 있습니다.하나의 염색체가 중심체를 향해 이동하는 것은 주로 키네토코어와의 결합 부위에서 MT 탈중합에 의해 발생한다.이러한 움직임은 또한 동력 발생을 필요로 하며, 마찬가지로 운동장에 위치한 분자 모터도 포함합니다.
유사분열 방추의 MT에 고정된 염색체
MT 캡처
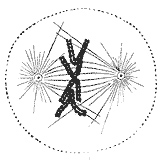
세포주기의 합성단계(S상)에서는 중심체가 복제되기 시작한다.유사분열이 시작될 때, 각 중심체의 양쪽 중심체는 최대 길이에 도달하고, 중심체는 추가 물질을 모집하며, 미세관에 대한 핵 형성 능력이 증가한다.유사분열이 진행됨에 따라 두 개의 중심체가 분리되어 유사분열 [42]방추체를 형성한다.이와 같이, 유사분열 셀의 스핀들은 미소관을 방출하는 2개의 극을 가진다.미세관은 비대칭 극단을 가진 긴 단백질 필라멘트, 중심체 옆에 비교적 안정적인 "마이너스" 끝, 그리고 세포의 중심을 탐색하는 성장-수축의 지속적 교대 단계인 "플러스" 끝이다.이 탐색 과정 동안 미소관은 키네토코어를 [43][44]통해 염색체를 만나 포착할 수 있다.키네토코어를 발견하여 부착하는 미세관은 안정화되지만, 자유 상태로 남아 있는 미세관은 빠르게 탈중합된다.[45]염색체가 백투백으로 연결된 두 개의 키네토코어를 가지고 있기 때문에(각 자매 염색체 위에 하나씩), 그들 중 하나가 세포 극 중 하나에 의해 생성된 미소관에 부착될 때 자매 염색체 위의 키네토코어는 반대 극에 노출된다; 이러한 이유로, 대부분의 경우, 두 번째 키네토코어는 마이크에 부착된다.염색체가 [47][48]현재 쌍방향으로 되어 있는 방식으로 세포가 분열할 때 두 염색체의 올바른 분리를 보장하는 하나의 기본 구성(양성체라고도 함)을 가지고 있는 반대쪽 [46]극에서 나오는 회전관
하나의 미소관이 하나의 동심원에 고정되면, 그것은 연관된 염색체의 빠른 움직임을 시작하여 그 미소관을 생성하는 극을 향해 갑니다.이러한 움직임은 아마도 MT에 [51]고정되지 않은 키네토코어에 매우 집중된 운동 단백질 세포질 다이네인의 [49][50]"마이너스" (-)에 대한 운동 활동에 의해 매개될 것이다.kinetochore가 kMT(kinetochore에 고정된 MT)를 획득하는 한 극을 향한 움직임은 느려지고 kMT 길이의 변화에 의해 방향화된다.다이닌은 kMT를[30] 획득함에 따라 키네토코어에서 방출되며, 배양된 포유동물 세포에서는 방추체크포인트 불활성화에 필요하지만, 방추적도에서의 염색체 조립, kMTs 획득 또는 [52]염색체 분리 중의 아나페이즈 A에는 필요하지 않다.고등 식물이나 효모에서 다이네인의 증거는 없지만, (-) 말단 쪽으로 향하는 다른 키네인은 다이네인의 부족을 보상할 수 있다.
MTs의 초기 포획에 관여하는 또 다른 운동 단백질은 CENP-E이다. 이것은 프로메타파아제에서 [53]아나기까지 포유류의 키네토코르의 섬유성 코로나와 관련된 고분자량 키네신이다.낮은 수준의 CENP-E를 가진 세포에서, 염색체는 종종 메타기 플레이트에서 조립하는 능력에 결함이 있는 그들의 키네토코어에서 이 단백질이 부족하다.이 경우, 대부분의 염색체가 중기의 [54]판에서 정확하게 결합할 수 있지만, 일부 염색체는 만성적으로 단일 지향(한 극에만 고정)을 유지할 수 있다.
kMTs 섬유(kinetochore에 결합된 미소관 다발)는 포유동물 [43]배양세포의 중심체와 방추극에서 중합된 MTs를 포획함으로써 유래한다는 것이 널리 인정되고 있다.그러나 키네토코어에서 직접 중합된 MT도 상당한 [55]기여를 할 수 있다.는centromeric 지역 또는 동원체 kMTs의 형성과 이 일이 일어나면 주파수를 시작하는 방식이 중요한 questions,[누구에 따르면?]기 때문에 이 메커니즘 kMTs의 초기 형성 뿐만 아니라 MTs의 정확한 결함이 있는 기점화 kinetochores는 방법으로 조절하다 기여할 수 있다. kMTs을 따라 운동.
Ndc80 콤플렉스의 역할
키네토코어와 관련된 MT는 특별한 특징을 보인다.자유 MT와 비교하여 kMT는 냉기에 의한 탈중합, 높은 정수압 또는 칼슘 [56]노출에 훨씬 더 강하다.또한 kMT는 자유단(+)을 가진 아스트랄 MT나 스핀들 MT보다 훨씬 더 느리게 재활용되며 레이저광을 이용하여 kMT를 키네토코어에서 방출하면 빠르게 탈중합된다.[41]
다이닌과 CENP-E가 kMT 형성에 필수적이지 않다는 것이 분명할 때, 다른 분자는 kMT 안정화를 담당해야 한다.효모의 선구자 유전자 연구는 kMTs [25][57][58][59]앵커링에서의 Ndc80 복합체의 관련성을 밝혀냈다.사카로미세스 세레비시아에서 Ndc80 복합체는 4가지 성분을 가지고 있다.Ndc80p, Nuf2p, Spc24p 및 Spc25p.이 복합체의 어떤 성분도 결여된 돌연변이는 키네토코어-마이크로튜브 연결의 손실을 나타내지만, 키네토코어 구조가 완전히 [25][57]상실된 것은 아니다.그러나 키네토코어 구조가 상실된 돌연변이(예를 들어 효모 중[60] Ndc10 돌연변이)는 미소관과의 접속 및 방추체크포인트를 활성화하는 능력이 결여되어 있는데, 아마도 키네토코어가 반응의 구성요소가 조립되는 플랫폼으로서 기능하기 때문일 것이다.
Ndc80 복합체는 보존성이 뛰어나며, S. pombe, C. elegans, Xenopus, 닭 및 [25][26][57][61][62][63][64]인간에서 확인되었습니다.Ndc80p의 인간 호몰로그인 Hec1(암세포 1에서 고도로 발현)에 대한 연구는 염색체 조립과 유사분열 진행에 중요하며 응집체와 응축소 [65]복합체의 구성요소와 상호작용한다는 것을 보여준다.
다른 실험실에서 Ndc80 복합체는 높은 진핵생물에서 [26][62][63][64]올바른 염색체 결합을 확립하는 데 수반되는 동원체-마이크로튜브 고정의 안정화에 필수적인 것으로 나타났다.Ndc80의 기능이 손상된 세포(RNAi, 유전자 녹아웃 또는 항체 미세주입 사용)는 비정상적으로 긴 스핀들, 자매 키네토코어 사이의 긴장 부족, 중합성 판에 모일 수 없는 염색체 및 관련된 kMT가 거의 없다.
Ndc80 복합체가 마이크로튜브와 직접 관련지어 kinetochore-microtuble [66]인터페이스의 핵심 보존 컴포넌트를 형성하는 기능에 대해서는 다양한 강력한 지원이 있습니다.그러나 강력한 키네토코어-마이크로튜브 상호작용의 형성은 추가적인 단백질의 기능을 필요로 할 수도 있다.효모에서 이 연결은 복합체 Dam1-DASH-DDD의 존재를 필요로 한다.이 복합체의 일부 구성원은 MT에 직접 결합하는 반면 다른 구성원은 Ndc80 [58][59][67]복합체에 결합한다.즉, 복잡한 Dam1-DASH-DD는 kinetochore와 마이크로튜브 사이의 필수 어댑터일 수 있습니다.그러나, 동물에 있어서, 동등한 콤플렉스는 확인되지 않았으며, 이 질문은 여전히 집중적인 조사를 받고 있다.
키네토코어 검증-MT 앵커
S상 동안, 세포는 DNA 복제라고 불리는 과정에서 염색체에 저장된 모든 유전 정보를 복제한다.이 과정의 마지막에, 각 염색체는 두 개의 완전하고 동일한 DNA 분자인 두 개의 자매 염색체를 포함한다.두 염색체 모두 염색체 분리가 일어나는 아나phase까지 응집체 복합체에 의해 연관되어 있다.염색체 분리가 올바르게 이루어지면, 각 딸 세포는 완전한 세트의 염색체를 받게 되고, 이를 위해 각 자매 염색체는 (대응하는 키네토코어를 통해) 유사분열 방추의 반대 극에서 생성된 MT에 고정되어야 한다.이 구성을 양성 또는 양방향이라고 합니다.
단, 앵커링 프로세스 중에는 다음과 같은 잘못된 설정이 표시될 수 있습니다.[68]

- 단색성: 염색분체 중 하나만 MT에 고정되고, 두 번째 키네토코어는 고정되지 않습니다. 이 상황에서는 중심 장력이 없으며, 스핀들 체크포인트가 활성화되어 아나페이즈 진입이 지연되고 셀이 오류를 수정하는 데 시간이 걸립니다.만약 수정되지 않는다면, 고정되지 않은 염색분체는 두 개의 딸 세포 중 어느 하나에서든 무작위로 끝나게 될 것이고, 이배수를 생성하게 될 것이다: 한 딸 세포는 과도한 염색체를 가질 것이고 다른 세포는 일부 염색체가 없을 것이다.
- Syntelic: 두 크로마토미드는 같은 극에서 방출되는 MT에 고정됩니다.이 상황도 구심성 장력을 발생시키지 않고 스핀들 체크포인트가 활성화됩니다.수정되지 않으면 두 염색분체가 같은 딸 세포에서 끝나면서 이배수를 발생시킵니다.
- 메로텔릭: 적어도 하나의 염색체가 양쪽 극에서 방출되는 MT에 동시에 고정된다.이 경우 중심 장력이 발생하므로 스핀들 체크포인트가 활성화되지 않습니다.수정되지 않으면, 양쪽 극에 결합되어 있는 염색체는 후행 염색체로 남아있을 것이고, 최종적으로 두 조각으로 분해되어 딸 세포 사이에 분산되어 이배수를 생성하게 될 것이다.
모노텔릭 및 신텔릭 구성 모두 중심 장력을 발생시키지 못하고 스핀들 체크 포인트에 의해 감지됩니다.이와는 대조적으로 이 제어 메커니즘에 의해 혈중 배치가 검출되지 않는다.다만, 이러한 에러의 대부분은, 셀이 아나페이즈가 [68]되기 전에 검출되어 수정됩니다.이러한 고정 오류를 수정하는 핵심 인자는 염색체 승객 복합체이며, 여기에는 키나아제 단백질 Aurora B와 그 표적 및 활성화 서브유닛 INCENP 및 두 개의 다른 서브유닛인 Survivin과 Borealin/Dasra B(2001년 애덤스와[69] 공동 연구자들에 의해 검토됨)가 포함된다.지배적인 음성 돌연변이, RNAi, 항체 미세 주입 또는 선택적 약물에 의해 이 복합체의 기능이 폐지된 세포는 염색체 고정에 오류를 축적한다.많은 연구에서 잘못된 고정 키네토코어-MT를 불안정하게 만들기 위해 Aurora B가 필요하며, 양성 연결의 생성을 선호합니다.효모(Ipl1p) 내 오로라 B 호몰로는 구성단백질 Ndc10p 및 Ndc80 [70]및 Dam1-DASH-DD 복합체 구성원과 같은 일부 키네토코어 단백질을 형광화한다.Ndc80 복합성분의 인산화로 kMTs 앵커링이 불안정해진다.Aurora B의 국재성은 그 기능에 있어서 중요한데, 그것은 중심체의 내부 영역(중심체 헤테로크로마틴 내)에 위치하고, 중심체의 장력이 자매의 키네토코어 별개로 확립될 때, Aurora B는 기질에 도달할 수 없기 때문에 kMT가 안정화된다는 것이 제안되었다.Aurora B는 여러 종류의 암에서 과압되는 경우가 많고, 현재 [71]항암제 개발의 표적이 되고 있다.
스핀들 체크포인트 활성화
스핀들 체크포인트 또는 SAC(스핀들어셈블리 체크포인트용)는 유사분열 체크포인트로도 알려져 있으며 다음을 검출하는 셀 메커니즘입니다.
- 유사분열 스핀들의 올바른 조립;
- 양극성 방식으로 모든 염색체를 유사분열 방추에 부착할 것.
- 중기의 모든 염색체의 집합.
(어떤 이유로든) 하나의 염색체만이 의회 기간 동안 지연된 상태로 남아있을 때, 방추체크포인트 기계는 세포 주기 진행의 지연을 발생시킨다: 세포는 정지되어, 검출된 문제를 해결하기 위한 복구 메커니즘의 시간을 허용한다.시간이 좀 지나도 문제가 해결되지 않으면 세포는 유기체에 극적인 결과를 초래하는 이배수의 발생을 피하기 위한 안전 메커니즘인 아포토시스(프로그램된 세포사망)의 표적이 될 것이다.
구조 중심체 단백질(CENP-B 등)은 유사분열 내내 안정적으로 국소화된 상태로 유지되는 반면(말단기 중 포함), 방추체크포인트 구성 요소는 미소관이 없는 상태에서 고농도로 키네토코어에 조립되며, 그 농도는 키네토코어에 부착된 미소관의 수가 증가함에 따라 감소한다.as.[30]
중기에서 CENP-E, Bub3 및 Bub1은 미부착 키네토코어 수치에 비해 3~4배 감소하며, 다이네인/다이낙틴, Mad1, Mad2 및 BubR1은 10~100배 [30][31][32][33]이상 감소한다.따라서 중기에서, 모든 염색체가 중기판에 정렬될 때, 모든 체크포인트 단백질은 키네토코어에서 방출된다.체크포인트 단백질이 운동장에서 사라진 것은 염색체가 중기에 도달해 양극성 긴장 상태에 있는 순간을 의미한다.이 때 Cdc20(Mad1-Mad2 및 BubR1)에 결합 및 저해하는 체크포인트 단백질은 Cdc20을 방출하여 APCCdc20/C를 결합 및 활성화하고, 이 복합체는 자매 염색체 분리를 유발하여 결과적으로 아나phase 진입을 유발한다.
여러 연구에 따르면 Ndc80 복합체는 Mad1-Mad2와 다이네인과 키네토코어의 [26][63][64]안정적인 연관성 조절에 관여한다.그러나 키네토코어 관련 단백질 CENP-A, CENP-C, CENP-E, CENP-H 및 BubR1은 Ndc80/Hec1과 독립적이다.Ndc80/Hec1이 낮은 세포에서 관찰된 프로메타phase의 장기 중단은 Mad2에 의존하지만, 이러한 세포는 키네토코어(미부착 키네토코어 대비 10~15%)에서 낮은 수준의 Mad1, Mad2, 다이네인을 보인다.그러나 Ndc80/Hec1과 Nuf2의 레벨이 모두 낮아지면 Mad1, Mad2는 키네토코어에서 완전히 사라져 스핀들 체크포인트가 [72]비활성화된다.
슈고신(Drosophila melanogaster의[73] Sgo1, MEI-S332)은 동원체에 결합되어 있는 응집체를 아나phase까지 유지하는 데 필수적인 동원체 단백질이다.인간의 호몰로그인 hsSgo1은 전기에 동원체와 결합하고 후기가 [74]시작되면 사라집니다.HeLa 세포에서 슈고신 수치가 RNAi에 의해 감소하면 유사분열 중에 응집체가 동원체에 남아있을 수 없고, 결과적으로 자매 염색체들이 아나피아가 시작되기 전에 동기적으로 분리되어 긴 유사분열 정지를 유발한다.
한편, Dasso와 공동 연구진은 Ran 사이클에 관여하는 단백질이 유사분열 중에 키네토코어에서 검출될 수 있다는 것을 발견했다.RanGAP1(Ran-GDP에서 Ran-GTP의 변환을 자극하는 GTPase 활성화 단백질)과 RanBP2/Nup358이라는 [75]Ran 결합 단백질.중간상 동안, 이러한 단백질은 핵공에 위치하고 핵 세포질 운반에 참여합니다.Ran-GTP의 수치를 증가시키는 일부 치료제가 Bub1, Bub3, Mad2, CENP-E의 [76]키네토코어 방출을 억제하기 때문에 이러한 단백질의 키네토코어 국부화는 기능적으로 중요한 것으로 보인다.
Orc2(S기 중에 원산지 인정을 단지에 속하는 단백질 -ORC- DNA복제 개시에 연루되어)또한 인간 세포에서 유사 분열 동안 kinetochores에;[77]이 지역화에 합의하여 일부 연구는 Orc2 효모 세포 안에서 여동생 chromatids 결합 속에서,에서 제거된 것을 보여 준다 지역이다. 그셀, 스핀들 체크 포인트 활성화가 [78]뒤따른다.일부 다른 ORC 성분(S. pombe의 orc5 등)[79]도 응집력에 관여하는 것으로 밝혀졌다.그러나, ORC 단백질은 응집 경로에 부가적인 분자 경로에 참여하는 것으로 보이며, 대부분 알려지지 않았다.
염색체 이동을 촉진하는 힘 생성
스핀들 극과 관련된 대부분의 염색체 이동은 kMT의 연장 및 단축과 관련이 있다.키네토코어의 특징 중 하나는 관련 kMT의 상태(약 20)를 탈중합 상태에서 중합 상태로 변경하는 능력이다.이를 통해 프로메타기의 세포에서 나오는 키네토코어는 각각 kMTs 탈중합과 중합이 번갈아 일어나는 극(극방향) 또는 반전(반극방향)으로 이동하는 지속적 위상 사이에서 변화하면서 "방향 불안정"[80]을 나타낼 수 있다.이 운동성 쌍안정성은 운동성 극과 운동성 극 사이의 기계적 연결을 잃지 않고 방추체의 적도에서 염색체를 정렬하는 메커니즘의 일부로 보입니다.kinetochore bi-stability는 kMTs(+)의 동적 불안정성에 기초하고 있으며, kinetochore에 존재하는 장력에 의해 부분적으로 제어된다.포유류의 배양 세포에서 키네토코어의 저장력은 kMTs 탈중합으로의 변화를 촉진하고, 고장력은 kMTs [81][82]중합으로의 변화를 촉진한다.
키네토코어 단백질과 MT(+) 말단에 결합하는 단백질은 kMT(+) 말단 역학 [83]조절을 통해 키네토코어 움직임을 조절한다.그러나, 키네토코어-마이크로튜브 계면은 매우 역동적이며, 이러한 단백질 중 일부는 두 구조의 진정한 성분인 것으로 보인다.단백질의 두 그룹은 특히 중요한 것으로 보인다: KinI 키네신과 같이 탈중합효소처럼 작용하는 키네신, 그리고 MT(+) 말단에 결합되어 있는 단백질, +TIP, 중합 촉진, 아마도 탈중합효소 [84]효과와 길항한다.
- KinI 키네신은 미세관인 튜불린 폴리머의 탈중합 촉진을 위해 ATP를 사용하는 내부 운동 영역을 나타내기 때문에 "I"라고 불립니다.척추동물에서 (+) 말단 어셈블리의 역학을 제어하는 가장 중요한 KinI 키네신은 MCAK이다.[85]하지만 다른 키네신도 관련된 것 같습니다.
- 키네토코어 기능을 가진 +TIP에는 2개의 그룹이 있습니다.
- 첫 번째는 단백질 선종성 용종성 대장균(APC)과 관련 단백질 EB1을 포함하며, 이들은 키네토코어에 위치하기 위해 MTs를 필요로 한다.두 단백질 모두 정확한 염색체 [86]분리에 필요하다.EB1은 중합 상태에서 MT에만 결합하므로 이 단계에서 kMT의 안정화를 촉진할 수 있습니다.
- +TIP의 두 번째 그룹은 MT가 없어도 키네토코어에 위치할 수 있는 단백질을 포함한다.이 그룹에는 CLIP-170과 관련 단백질 CLASS(CLIP 관련 단백질)의 두 가지 단백질이 널리 연구되고 있다.키네토코어에서 CLIP-170의 역할은 알려져 있지 않지만, 지배적인 음성 돌연변이의 발현은 염색체 정렬에서 활발한 역할을 한다는 것을 암시하는 프로메타페이즈 [87]지연을 생성한다.HLASPs 단백질은 Drosophila, 인간 및 [88][89]효모의 양극성 방추의 염색체 정렬과 유지를 위해 필요합니다.
레퍼런스
- ^ Santaguida S, Musacchio A (September 2009). "The life and miracles of kinetochores". The EMBO Journal. 28 (17): 2511–31. doi:10.1038/emboj.2009.173. PMC 2722247. PMID 19629042.
- ^ Sharp LW (1934). Introduction to cytology (3rd ed.). New York: McGraw-Hill Book Company, inc. doi:10.5962/bhl.title.6429.
- ^ Schrader F (1936-06-01). "The kinetochore or spindle fibre locus in amphiuma tridactylum". The Biological Bulletin. 70 (3): 484–498. doi:10.2307/1537304. ISSN 0006-3185. JSTOR 1537304.
- ^ Kops GJ, Saurin AT, Meraldi P (July 2010). "Finding the middle ground: how kinetochores power chromosome congression". Cellular and Molecular Life Sciences. 67 (13): 2145–61. doi:10.1007/s00018-010-0321-y. PMC 2883098. PMID 20232224.
- ^ Albertson DG, Thomson JN (May 1993). "Segregation of holocentric chromosomes at meiosis in the nematode, Caenorhabditis elegans". Chromosome Research. 1 (1): 15–26. doi:10.1007/BF00710603. PMID 8143084. S2CID 5644126.
- ^ 피터 드 울프, 윌리엄 C언쇼, 키네토코어: 분자 발견에서 암 치료로
- ^ van Hooff JJ, Tromer E, van Wijk LM, Snel B, Kops GJ (September 2017). "Evolutionary dynamics of the kinetochore network in eukaryotes as revealed by comparative genomics". EMBO Reports. 18 (9): 1559–1571. doi:10.15252/embr.201744102. PMC 5579357. PMID 28642229.
- ^ Vijay N (2020-09-29). "Loss of inner kinetochore genes is associated with the transition to an unconventional point centromere in budding yeast". PeerJ. 8: e10085. doi:10.7717/peerj.10085. PMC 7531349. PMID 33062452.
- ^ a b Maiato H, DeLuca J, Salmon ED, Earnshaw WC (November 2004). "The dynamic kinetochore-microtubule interface". Journal of Cell Science. 117 (Pt 23): 5461–77. doi:10.1242/jcs.01536. PMID 15509863.
- ^ Mitchison T, Kirschner M (1984). "Dynamic instability of microtubule growth" (PDF). Nature. 312 (5991): 237–42. Bibcode:1984Natur.312..237M. doi:10.1038/312237a0. PMID 6504138. S2CID 30079133. Archived from the original (PDF) on 2010-06-22. Retrieved 2010-08-23.
- ^ Mehta GD, Agarwal M, Ghosh SK (March 2014). "Functional characterization of kinetochore protein, Ctf19 in meiosis I: an implication of differential impact of Ctf19 on the assembly of mitotic and meiotic kinetochores in Saccharomyces cerevisiae". Molecular Microbiology. 91 (6): 1179–99. doi:10.1111/mmi.12527. PMID 24446862.
- ^ Agarwal M, Mehta G, Ghosh SK (March 2015). "Role of Ctf3 and COMA subcomplexes in meiosis: Implication in maintaining Cse4 at the centromere and numeric spindle poles". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1853 (3): 671–84. doi:10.1016/j.bbamcr.2014.12.032. PMID 25562757.
- ^ a b Brinkley BR, Stubblefield E (1966). "The fine structure of the kinetochore of a mammalian cell in vitro". Chromosoma. 19 (1): 28–43. doi:10.1007/BF00332792. PMID 5912064. S2CID 43314146.
- ^ Jokelainen PT (July 1967). "The ultrastructure and spatial organization of the metaphase kinetochore in mitotic rat cells". Journal of Ultrastructure Research. 19 (1): 19–44. doi:10.1016/S0022-5320(67)80058-3. PMID 5339062.
- ^ Rieder CL (1982). The formation, structure, and composition of the mammalian kinetochore and kinetochore fiber. International Review of Cytology. Vol. 79. pp. 1–58. doi:10.1016/S0074-7696(08)61672-1. ISBN 978-0-12-364479-4. PMID 6185450.
- ^ McEwen BF, Hsieh CE, Mattheyses AL, Rieder CL (December 1998). "A new look at kinetochore structure in vertebrate somatic cells using high-pressure freezing and freeze substitution". Chromosoma. 107 (6–7): 366–75. doi:10.1007/s004120050320. PMC 2905855. PMID 9914368.
- ^ Brenner S, Pepper D, Berns MW, Tan E, Brinkley BR (October 1981). "Kinetochore structure, duplication, and distribution in mammalian cells: analysis by human autoantibodies from scleroderma patients". The Journal of Cell Biology. 91 (1): 95–102. doi:10.1083/jcb.91.1.95. PMC 2111947. PMID 7298727.
- ^ Pluta AF, Mackay AM, Ainsztein AM, Goldberg IG, Earnshaw WC (December 1995). "The centromere: hub of chromosomal activities". Science. 270 (5242): 1591–4. Bibcode:1995Sci...270.1591P. doi:10.1126/science.270.5242.1591. PMID 7502067. S2CID 44632550.
- ^ Palmer DK, O'Day K, Trong HL, Charbonneau H, Margolis RL (May 1991). "Purification of the centromere-specific protein CENP-A and demonstration that it is a distinctive histone". Proceedings of the National Academy of Sciences of the United States of America. 88 (9): 3734–8. Bibcode:1991PNAS...88.3734P. doi:10.1073/pnas.88.9.3734. PMC 51527. PMID 2023923.
- ^ Howman EV, Fowler KJ, Newson AJ, Redward S, MacDonald AC, Kalitsis P, Choo KH (February 2000). "Early disruption of centromeric chromatin organization in centromere protein A (Cenpa) null mice". Proceedings of the National Academy of Sciences of the United States of America. 97 (3): 1148–53. Bibcode:2000PNAS...97.1148H. doi:10.1073/pnas.97.3.1148. PMC 15551. PMID 10655499.
- ^ Oegema K, Desai A, Rybina S, Kirkham M, Hyman AA (June 2001). "Functional analysis of kinetochore assembly in Caenorhabditis elegans". The Journal of Cell Biology. 153 (6): 1209–26. doi:10.1083/jcb.153.6.1209. PMC 2192036. PMID 11402065.
- ^ Van Hooser AA, Ouspenski II, Gregson HC, Starr DA, Yen TJ, Goldberg ML, et al. (October 2001). "Specification of kinetochore-forming chromatin by the histone H3 variant CENP-A". Journal of Cell Science. 114 (Pt 19): 3529–42. doi:10.1242/jcs.114.19.3529. PMID 11682612.
- ^ Fukagawa T, Mikami Y, Nishihashi A, Regnier V, Haraguchi T, Hiraoka Y, et al. (August 2001). "CENP-H, a constitutive centromere component, is required for centromere targeting of CENP-C in vertebrate cells". The EMBO Journal. 20 (16): 4603–17. doi:10.1093/emboj/20.16.4603. PMC 125570. PMID 11500386.
- ^ Goshima G, Kiyomitsu T, Yoda K, Yanagida M (January 2003). "Human centromere chromatin protein hMis12, essential for equal segregation, is independent of CENP-A loading pathway". The Journal of Cell Biology. 160 (1): 25–39. doi:10.1083/jcb.200210005. PMC 2172742. PMID 12515822.
- ^ a b c d Wigge PA, Kilmartin JV (January 2001). "The Ndc80p complex from Saccharomyces cerevisiae contains conserved centromere components and has a function in chromosome segregation". The Journal of Cell Biology. 152 (2): 349–60. doi:10.1083/jcb.152.2.349. PMC 2199619. PMID 11266451.
- ^ a b c d DeLuca JG, Moree B, Hickey JM, Kilmartin JV, Salmon ED (November 2002). "hNuf2 inhibition blocks stable kinetochore-microtubule attachment and induces mitotic cell death in HeLa cells". The Journal of Cell Biology. 159 (4): 549–55. doi:10.1083/jcb.200208159. PMC 2173110. PMID 12438418.
- ^ a b Cheeseman IM, Niessen S, Anderson S, Hyndman F, Yates JR, Oegema K, Desai A (September 2004). "A conserved protein network controls assembly of the outer kinetochore and its ability to sustain tension". Genes & Development. 18 (18): 2255–68. doi:10.1101/gad.1234104. PMC 517519. PMID 15371340.
- ^ Rattner JB, Rao A, Fritzler MJ, Valencia DW, Yen TJ (1993). "CENP-F is a .ca 400 kDa kinetochore protein that exhibits a cell-cycle dependent localization". Cell Motility and the Cytoskeleton. 26 (3): 214–26. doi:10.1002/cm.970260305. PMID 7904902.
- ^ Liao H, Winkfein RJ, Mack G, Rattner JB, Yen TJ (August 1995). "CENP-F is a protein of the nuclear matrix that assembles onto kinetochores at late G2 and is rapidly degraded after mitosis". The Journal of Cell Biology. 130 (3): 507–18. doi:10.1083/jcb.130.3.507. PMC 2120529. PMID 7542657.
- ^ a b c d e Hoffman DB, Pearson CG, Yen TJ, Howell BJ, Salmon ED (July 2001). "Microtubule-dependent changes in assembly of microtubule motor proteins and mitotic spindle checkpoint proteins at PtK1 kinetochores". Molecular Biology of the Cell. 12 (7): 1995–2009. doi:10.1091/mbc.12.7.1995. PMC 55648. PMID 11451998.
- ^ a b King SM (March 2000). "The dynein microtubule motor". Biochimica et Biophysica Acta (BBA) - Molecular Cell Research. 1496 (1): 60–75. doi:10.1016/S0167-4889(00)00009-4. PMID 10722877.
- ^ a b Howell BJ, Moree B, Farrar EM, Stewart S, Fang G, Salmon ED (June 2004). "Spindle checkpoint protein dynamics at kinetochores in living cells". Current Biology. 14 (11): 953–64. doi:10.1016/j.cub.2004.05.053. PMID 15182668.
- ^ a b c Shah JV, Botvinick E, Bonday Z, Furnari F, Berns M, Cleveland DW (June 2004). "Dynamics of centromere and kinetochore proteins; implications for checkpoint signaling and silencing". Current Biology. 14 (11): 942–52. doi:10.1016/j.cub.2004.05.046. PMID 15182667.
- ^ Tirnauer JS, Canman JC, Salmon ED, Mitchison TJ (December 2002). "EB1 targets to kinetochores with attached, polymerizing microtubules". Molecular Biology of the Cell. 13 (12): 4308–16. doi:10.1091/mbc.E02-04-0236. PMC 138635. PMID 12475954.
- ^ Kaplan KB, Burds AA, Swedlow JR, Bekir SS, Sorger PK, Näthke IS (April 2001). "A role for the Adenomatous Polyposis Coli protein in chromosome segregation". Nature Cell Biology. 3 (4): 429–32. doi:10.1038/35070123. PMID 11283619. S2CID 12645435.
- ^ Joseph J, Liu ST, Jablonski SA, Yen TJ, Dasso M (April 2004). "The RanGAP1-RanBP2 complex is essential for microtubule-kinetochore interactions in vivo". Current Biology. 14 (7): 611–7. doi:10.1016/j.cub.2004.03.031. PMID 15062103.
- ^ Salina D, Enarson P, Rattner JB, Burke B (September 2003). "Nup358 integrates nuclear envelope breakdown with kinetochore assembly". The Journal of Cell Biology. 162 (6): 991–1001. doi:10.1083/jcb.200304080. PMC 2172838. PMID 12963708.
- ^ Ohta S, Bukowski-Wills JC, Sanchez-Pulido L, Alves F, Wood L, Chen ZA, et al. (September 2010). "The protein composition of mitotic chromosomes determined using multiclassifier combinatorial proteomics". Cell. 142 (5): 810–21. doi:10.1016/j.cell.2010.07.047. PMC 2982257. PMID 20813266.
- ^ Tipton AR, Wang K, Oladimeji P, Sufi S, Gu Z, Liu ST (June 2012). "Identification of novel mitosis regulators through data mining with human centromere/kinetochore proteins as group queries". BMC Cell Biology. 13: 15. doi:10.1186/1471-2121-13-15. PMC 3419070. PMID 22712476.
- ^ McEwen BF, Heagle AB, Cassels GO, Buttle KF, Rieder CL (June 1997). "Kinetochore fiber maturation in PtK1 cells and its implications for the mechanisms of chromosome congression and anaphase onset". The Journal of Cell Biology. 137 (7): 1567–80. doi:10.1083/jcb.137.7.1567. PMC 2137823. PMID 9199171.
- ^ a b Nicklas RB, Kubai DF (1985). "Microtubules, chromosome movement, and reorientation after chromosomes are detached from the spindle by micromanipulation". Chromosoma. 92 (4): 313–24. doi:10.1007/BF00329815. PMID 4042772. S2CID 24739460.
- ^ Mayor T, Meraldi P, Stierhof YD, Nigg EA, Fry AM (June 1999). "Protein kinases in control of the centrosome cycle". FEBS Letters. 452 (1–2): 92–5. doi:10.1016/S0014-5793(99)00534-7. PMID 10376685. S2CID 22671038.
- ^ a b Kirschner M, Mitchison T (May 1986). "Beyond self-assembly: from microtubules to morphogenesis". Cell. 45 (3): 329–42. doi:10.1016/0092-8674(86)90318-1. PMID 3516413. S2CID 36994346.
- ^ Holy TE, Leibler S (June 1994). "Dynamic instability of microtubules as an efficient way to search in space". Proceedings of the National Academy of Sciences of the United States of America. 91 (12): 5682–5. Bibcode:1994PNAS...91.5682H. doi:10.1073/pnas.91.12.5682. PMC 44060. PMID 8202548.
- ^ Hayden JH, Bowser SS, Rieder CL (September 1990). "Kinetochores capture astral microtubules during chromosome attachment to the mitotic spindle: direct visualization in live newt lung cells". The Journal of Cell Biology. 111 (3): 1039–45. doi:10.1083/jcb.111.3.1039. PMC 2116290. PMID 2391359.
- ^ Nicklas RB (January 1997). "How cells get the right chromosomes". Science. 275 (5300): 632–7. doi:10.1126/science.275.5300.632. PMID 9005842. S2CID 30090031.
- ^ Loncarek J, Kisurina-Evgenieva O, Vinogradova T, Hergert P, La Terra S, Kapoor TM, Khodjakov A (November 2007). "The centromere geometry essential for keeping mitosis error free is controlled by spindle forces". Nature. 450 (7170): 745–9. Bibcode:2007Natur.450..745L. doi:10.1038/nature06344. PMC 2586812. PMID 18046416.
- ^ Dewar H, Tanaka K, Nasmyth K, Tanaka TU (March 2004). "Tension between two kinetochores suffices for their bi-orientation on the mitotic spindle". Nature. 428 (6978): 93–7. Bibcode:2004Natur.428...93D. doi:10.1038/nature02328. PMID 14961024. S2CID 4418232.
- ^ Echeverri CJ, Paschal BM, Vaughan KT, Vallee RB (February 1996). "Molecular characterization of the 50-kD subunit of dynactin reveals function for the complex in chromosome alignment and spindle organization during mitosis". The Journal of Cell Biology. 132 (4): 617–33. doi:10.1083/jcb.132.4.617. PMC 2199864. PMID 8647893.
- ^ Sharp DJ, Rogers GC, Scholey JM (December 2000). "Cytoplasmic dynein is required for poleward chromosome movement during mitosis in Drosophila embryos". Nature Cell Biology. 2 (12): 922–30. doi:10.1038/35046574. PMID 11146657. S2CID 11753626.
- ^ Banks JD, Heald R (February 2001). "Chromosome movement: dynein-out at the kinetochore". Current Biology. 11 (4): R128-31. doi:10.1016/S0960-9822(01)00059-8. PMID 11250166.
- ^ Howell BJ, McEwen BF, Canman JC, Hoffman DB, Farrar EM, Rieder CL, Salmon ED (December 2001). "Cytoplasmic dynein/dynactin drives kinetochore protein transport to the spindle poles and has a role in mitotic spindle checkpoint inactivation". The Journal of Cell Biology. 155 (7): 1159–72. doi:10.1083/jcb.200105093. PMC 2199338. PMID 11756470.
- ^ Cooke CA, Schaar B, Yen TJ, Earnshaw WC (December 1997). "Localization of CENP-E in the fibrous corona and outer plate of mammalian kinetochores from prometaphase through anaphase". Chromosoma. 106 (7): 446–55. doi:10.1007/s004120050266. PMID 9391217. S2CID 18884489.
- ^ Weaver BA, Bonday ZQ, Putkey FR, Kops GJ, Silk AD, Cleveland DW (August 2003). "Centromere-associated protein-E is essential for the mammalian mitotic checkpoint to prevent aneuploidy due to single chromosome loss". The Journal of Cell Biology. 162 (4): 551–63. doi:10.1083/jcb.200303167. PMC 2173788. PMID 12925705.
- ^ a b Maiato H, Rieder CL, Khodjakov A (December 2004). "Kinetochore-driven formation of kinetochore fibers contributes to spindle assembly during animal mitosis". The Journal of Cell Biology. 167 (5): 831–40. doi:10.1083/jcb.200407090. PMC 2172442. PMID 15569709.
- ^ Mitchison TJ (1988). "Microtubule dynamics and kinetochore function in mitosis". Annual Review of Cell Biology. 4 (1): 527–49. doi:10.1146/annurev.cb.04.110188.002523. PMID 3058165.
- ^ a b c He X, Rines DR, Espelin CW, Sorger PK (July 2001). "Molecular analysis of kinetochore-microtubule attachment in budding yeast". Cell. 106 (2): 195–206. doi:10.1016/S0092-8674(01)00438-X. PMID 11511347.
- ^ a b Westermann S, Cheeseman IM, Anderson S, Yates JR, Drubin DG, Barnes G (October 2003). "Architecture of the budding yeast kinetochore reveals a conserved molecular core". The Journal of Cell Biology. 163 (2): 215–22. doi:10.1083/jcb.200305100. PMC 2173538. PMID 14581449.
- ^ a b De Wulf P, McAinsh AD, Sorger PK (December 2003). "Hierarchical assembly of the budding yeast kinetochore from multiple subcomplexes". Genes & Development. 17 (23): 2902–21. doi:10.1101/gad.1144403. PMC 289150. PMID 14633972.
- ^ Goh PY, Kilmartin JV (May 1993). "NDC10: a gene involved in chromosome segregation in Saccharomyces cerevisiae". The Journal of Cell Biology. 121 (3): 503–12. doi:10.1083/jcb.121.3.503. PMC 2119568. PMID 8486732.
- ^ Nabetani A, Koujin T, Tsutsumi C, Haraguchi T, Hiraoka Y (September 2001). "A conserved protein, Nuf2, is implicated in connecting the centromere to the spindle during chromosome segregation: a link between the kinetochore function and the spindle checkpoint". Chromosoma. 110 (5): 322–34. doi:10.1007/s004120100153. PMID 11685532. S2CID 22443613.
- ^ a b Howe M, McDonald KL, Albertson DG, Meyer BJ (June 2001). "HIM-10 is required for kinetochore structure and function on Caenorhabditis elegans holocentric chromosomes". The Journal of Cell Biology. 153 (6): 1227–38. doi:10.1083/jcb.153.6.1227. PMC 2192032. PMID 11402066.
- ^ a b c Martin-Lluesma S, Stucke VM, Nigg EA (September 2002). "Role of Hec1 in spindle checkpoint signaling and kinetochore recruitment of Mad1/Mad2". Science. 297 (5590): 2267–70. Bibcode:2002Sci...297.2267M. doi:10.1126/science.1075596. PMID 12351790. S2CID 7879023.
- ^ a b c McCleland ML, Gardner RD, Kallio MJ, Daum JR, Gorbsky GJ, Burke DJ, Stukenberg PT (January 2003). "The highly conserved Ndc80 complex is required for kinetochore assembly, chromosome congression, and spindle checkpoint activity". Genes & Development. 17 (1): 101–14. doi:10.1101/gad.1040903. PMC 195965. PMID 12514103.
- ^ Zheng L, Chen Y, Lee WH (August 1999). "Hec1p, an evolutionarily conserved coiled-coil protein, modulates chromosome segregation through interaction with SMC proteins". Molecular and Cellular Biology. 19 (8): 5417–28. doi:10.1128/mcb.19.8.5417. PMC 84384. PMID 10409732.
- ^ Wei RR, Al-Bassam J, Harrison SC (January 2007). "The Ndc80/HEC1 complex is a contact point for kinetochore-microtubule attachment". Nature Structural & Molecular Biology. 14 (1): 54–9. doi:10.1038/nsmb1186. PMID 17195848. S2CID 5991912.
- ^ Courtwright AM, He X (November 2002). "Dam1 is the right one: phosphoregulation of kinetochore biorientation". Developmental Cell. 3 (5): 610–1. doi:10.1016/S1534-5807(02)00332-5. PMID 12431367.
- ^ a b Cimini D, Moree B, Canman JC, Salmon ED (October 2003). "Merotelic kinetochore orientation occurs frequently during early mitosis in mammalian tissue cells and error correction is achieved by two different mechanisms". Journal of Cell Science. 116 (Pt 20): 4213–25. doi:10.1242/jcs.00716. PMID 12953065.
- ^ Adams RR, Carmena M, Earnshaw WC (February 2001). "Chromosomal passengers and the (aurora) ABCs of mitosis". Trends in Cell Biology. 11 (2): 49–54. doi:10.1016/S0962-8924(00)01880-8. PMID 11166196.
- ^ Cheeseman IM, Anderson S, Jwa M, Green EM, Kang J, Yates JR, et al. (October 2002). "Phospho-regulation of kinetochore-microtubule attachments by the Aurora kinase Ipl1p". Cell. 111 (2): 163–72. doi:10.1016/S0092-8674(02)00973-X. PMID 12408861.
- ^ Gautschi O, Heighway J, Mack PC, Purnell PR, Lara PN, Gandara DR (March 2008). "Aurora kinases as anticancer drug targets". Clinical Cancer Research. 14 (6): 1639–48. doi:10.1158/1078-0432.CCR-07-2179. PMID 18347165.
- ^ Meraldi P, Draviam VM, Sorger PK (July 2004). "Timing and checkpoints in the regulation of mitotic progression". Developmental Cell. 7 (1): 45–60. doi:10.1016/j.devcel.2004.06.006. PMID 15239953.
- ^ Tang TT, Bickel SE, Young LM, Orr-Weaver TL (December 1998). "Maintenance of sister-chromatid cohesion at the centromere by the Drosophila MEI-S332 protein". Genes & Development. 12 (24): 3843–56. doi:10.1101/gad.12.24.3843. PMC 317262. PMID 9869638.
- ^ McGuinness BE, Hirota T, Kudo NR, Peters JM, Nasmyth K (March 2005). "Shugoshin prevents dissociation of cohesin from centromeres during mitosis in vertebrate cells". PLOS Biology. 3 (3): e86. doi:10.1371/journal.pbio.0030086. PMC 1054882. PMID 15737064.
- ^ Joseph J, Tan SH, Karpova TS, McNally JG, Dasso M (February 2002). "SUMO-1 targets RanGAP1 to kinetochores and mitotic spindles". The Journal of Cell Biology. 156 (4): 595–602. doi:10.1083/jcb.200110109. PMC 2174074. PMID 11854305.
- ^ Arnaoutov A, Dasso M (July 2003). "The Ran GTPase regulates kinetochore function". Developmental Cell. 5 (1): 99–111. doi:10.1016/S1534-5807(03)00194-1. PMID 12852855.
- ^ Prasanth SG, Prasanth KV, Siddiqui K, Spector DL, Stillman B (July 2004). "Human Orc2 localizes to centrosomes, centromeres and heterochromatin during chromosome inheritance". The EMBO Journal. 23 (13): 2651–63. doi:10.1038/sj.emboj.7600255. PMC 449767. PMID 15215892.
- ^ Shimada K, Gasser SM (January 2007). "The origin recognition complex functions in sister-chromatid cohesion in Saccharomyces cerevisiae". Cell. 128 (1): 85–99. doi:10.1016/j.cell.2006.11.045. PMID 17218257.
- ^ Kato H, Matsunaga F, Miyazaki S, Yin L, D'Urso G, Tanaka K, Murakami Y (April 2008). "Schizosaccharomyces pombe Orc5 plays multiple roles in the maintenance of genome stability throughout the cell cycle". Cell Cycle. 7 (8): 1085–96. doi:10.4161/cc.7.8.5710. PMID 18414064.
- ^ Skibbens RV, Skeen VP, Salmon ED (August 1993). "Directional instability of kinetochore motility during chromosome congression and segregation in mitotic newt lung cells: a push-pull mechanism". The Journal of Cell Biology. 122 (4): 859–75. doi:10.1083/jcb.122.4.859. PMC 2119582. PMID 8349735.
- ^ Rieder CL, Salmon ED (February 1994). "Motile kinetochores and polar ejection forces dictate chromosome position on the vertebrate mitotic spindle". The Journal of Cell Biology. 124 (3): 223–33. doi:10.1083/jcb.124.3.223. PMC 2119939. PMID 8294508.
- ^ Skibbens RV, Rieder CL, Salmon ED (July 1995). "Kinetochore motility after severing between sister centromeres using laser microsurgery: evidence that kinetochore directional instability and position is regulated by tension". Journal of Cell Science. 108 ( Pt 7) (7): 2537–48. doi:10.1242/jcs.108.7.2537. PMID 7593295.
- ^ Askham JM, Vaughan KT, Goodson HV, Morrison EE (October 2002). "Evidence that an interaction between EB1 and p150(Glued) is required for the formation and maintenance of a radial microtubule array anchored at the centrosome". Molecular Biology of the Cell. 13 (10): 3627–45. doi:10.1091/mbc.E02-01-0061. PMC 129971. PMID 12388762.
- ^ Schuyler SC, Pellman D (May 2001). "Microtubule "plus-end-tracking proteins": The end is just the beginning". Cell. 105 (4): 421–4. doi:10.1016/S0092-8674(01)00364-6. PMID 11371339.
- ^ Howard J, Hyman AA (April 2003). "Dynamics and mechanics of the microtubule plus end". Nature. 422 (6933): 753–8. Bibcode:2003Natur.422..753H. doi:10.1038/nature01600. PMID 12700769. S2CID 4427406.
- ^ Green RA, Wollman R, Kaplan KB (October 2005). "APC and EB1 function together in mitosis to regulate spindle dynamics and chromosome alignment". Molecular Biology of the Cell. 16 (10): 4609–22. doi:10.1091/mbc.E05-03-0259. PMC 1237068. PMID 16030254.
- ^ Dujardin D, Wacker UI, Moreau A, Schroer TA, Rickard JE, De Mey JR (May 1998). "Evidence for a role of CLIP-170 in the establishment of metaphase chromosome alignment". The Journal of Cell Biology. 141 (4): 849–62. doi:10.1083/jcb.141.4.849. PMC 2132766. PMID 9585405.
- ^ Maiato H, Khodjakov A, Rieder CL (January 2005). "Drosophila CLASP is required for the incorporation of microtubule subunits into fluxing kinetochore fibres". Nature Cell Biology. 7 (1): 42–7. doi:10.1038/ncb1207. PMC 2596653. PMID 15592460.
- ^ Maiato H, Fairley EA, Rieder CL, Swedlow JR, Sunkel CE, Earnshaw WC (June 2003). "Human CLASP1 is an outer kinetochore component that regulates spindle microtubule dynamics". Cell. 113 (7): 891–904. doi:10.1016/S0092-8674(03)00465-3. hdl:10216/53832. PMID 12837247. S2CID 13936836.