miR-155
miR-155| 신기루 이전의 | |
|---|---|
 미러링 전 2차 구조 및 시퀀스 보존ard | |
| 식별자 | |
| 기호 | miR-155 |
| Rfam | RF00731 |
| miRBase 계열 | MIPF0000157 |
| 기타자료 | |
| RNA형 | 마이크로RNA |
| 도메인 | 에우카리오타; |
| PDB 구조 | PDBe |
| MIR155 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||||||||||||||||||
| 별칭 | MIR155, MIRN155, miRNA155, MIR-155, miR-155, microRNA 155 | ||||||||||||||||||||||||
| 외부 ID | OMIM: 609337 GeneCard: MIR155 | ||||||||||||||||||||||||
| |||||||||||||||||||||||||
| 직교체 | |||||||||||||||||||||||||
| 종 | 인간 | 마우스 | |||||||||||||||||||||||
| 엔트레스 |
| ||||||||||||||||||||||||
| 앙상블 |
| ||||||||||||||||||||||||
| 유니프로트 |
| ||||||||||||||||||||||||
| RefSeq(mRNA) |
|
| |||||||||||||||||||||||
| RefSeq(단백질) |
|
| |||||||||||||||||||||||
| 위치(UCSC) | Cr 21: 25.57 – 25.57Mb | n/a | |||||||||||||||||||||||
| PubMed 검색 | [2] | n/a | |||||||||||||||||||||||
| 위키다타 | |||||||||||||||||||||||||
| |||||||||||||||||||||||||
MiR-155는 인간에서 MIR155 호스트 유전자 또는 MIR155HG에 의해 암호화되는 마이크로RNA이다.[3]MiR-155는 다양한 생리학적, 병리학적 과정에 역할을 한다.[4][5][6][7][8][9]miR-155 발현 체외 분자 조절은 악성 성장, [10][11]바이러스 감염을 억제하고 심혈관 질환의 진행을 강화시킬 수 있다.[12][13]
디스커버리
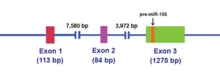
MIR155HG는 초기에는 B세포 림프종의 공통 레트로바이러스 통합현장에서 촉진자 삽입에 의해 전사적으로 활성화된 유전자로 확인되었고, 이전에는 BIC(B-세포 통합클러스터)라고 불렸다.MIR155HG는 RNA 중합효소 II에 의해 전사되며, 그 결과 약 1,500개의 뉴클레오티드 RNA가 캡을 씌우고 폴리아데닐화된다.exon 3에 수록된 23개의 뉴클레오티드 단일 가닥 miR-155는 이후 모체 RNA 분자에서 처리된다.[14]
바이오제네시스
MIR155HG RNA 대본은 긴 개방형 판독 프레임(ORF)을 포함하지 않지만, 여러 종에 걸쳐 보존되는 불완전한 기저부식 스템 루프를 포함하고 있다.[15]이 비코딩 RNA(ncRNA)는 이제 1차 miRNA(miRNA 이전)로 정의된다.[15]Once miR-155 pri-miRNA is transcribed, this transcript is cleaved by the nuclear microprocessor complex, of which the core components are the RNase III type endonuclease Drosha and the DiGeorge critical region 8 (DGCR8) protein,[16][17] to produce a 65 nucleotide stem-loop precursor miRNA (pre-mir-155) (see Figure 2).
exportin-5에 의한 핵으로부터의 수출에 이어, mir-155 이전의 분자는 Dicer에 의해 단자 루프 근처에서 분해되어 RNA 듀플렉스 ~22nucleotide가 된다.[16][17]디케르 클라베이지에 이어 아르고누테(Ago) 단백질이 짧은 RNA 듀플렉스(RISC)에 결합해 RNA 유도 음소거 콤플렉스(Risc)라고 불리는 다단위 콤플렉스의 핵심을 형성한다.[18]siRNA 듀플렉스(duplex)와 유사한 방식으로, 두 가닥 중 하나인 "조수석 miRNA"(miR-155*)가 해제되고 분해되는 반면, "가이드 스트랜드" 또는 "자연의 miRNA"(miR-155)로 지정된 다른 가닥은 RISC 내에 유지된다.[18]
최근 자료에 따르면, miRNA 헤어핀의 양쪽 팔은 성숙한 miRNA를 일으킬 수 있다.[19][20]Due to the increasing number of examples where two functional mature miRNAs are processed from opposite arms of the same pre-miRNA, pre-mir-155 products are now denoted with the suffix -5p (from the 5′ arm) (e.g. miR-155-5p) and -3p (from the 3′ arm) (e.g. miR-155-3p) following their name (see Figure 3).[21]
Once miR-155-5p/-3p is assembled into the RISC, these molecules subsequently recognize their target messenger RNA (mRNA) by base pairing interactions between nucleotides 2 and 8 of miR-155-5p/-3p (the seed region) and complementary nucleotides predominantly in the 3′-untranslated region (3′-UTR) of mRNAs (see Figure 4 and 5 below).[22]마지막으로, miR-155-5p/-3p가 RISC의 어댑터로 작용하면서, 복잡한 바인딩 mRNA는 변환적 억제(즉, 변환 시작의 억제) 및/또는 데데닐레이션에 따른 저하(deadenylation)를 받는다.[18]
진화보존
초기 유전체 분석 결과, miR-155와 miR-155-5p의 염기서열이 인간, 생쥐, 닭 사이에 보존되어 있는 것으로 나타났다.[15]최근 주석 처리된 염기서열 분석 자료에 따르면 포유류, 양서류, 조류, 파충류, 미더덕, 바다 등 22개 생물체가 보존된 miR-155-5p를 표현하고 있다.[1] 현재 miR-155-3p에 관한 시퀀스 데이터가 훨씬 더 적기 때문에, 이 miRNA가 종에 걸쳐 얼마나 보존되어 있는지 명확하지 않다.[2]
조직 분포
북방 블롯 분석 결과 miR-155 pri-miRNA는 인간의 비장과 흉선에 풍부하게 표현되었고 간, 폐,[15] 신장에서 검출이 가능했다.그 후 중합효소 연쇄반응(PCR) 실험을 통해 miR-155-5p가 조사된 모든 인체 조직에서 검출될 수 있음을 입증했다.[23]Sequence analysis of small RNA clone libraries comparing miRNA expression to all other organ systems examined established that miR-155-5p was one of five miRNAs (i.e. miR-142, miR-144, miR-150, miR-155, and miR-223) that was specific for hematopoietic cells including B-cells, T-cells, monocytes and granulocytes.[24]이러한 결과는 miR-155-5p가 많은 조직과 세포 유형으로 표현되므로 조혈증을[4][5][6] 포함한 다양한 생물학적 과정에 중요한 역할을 할 수 있음을 시사한다.
miR-155-3p의 발현 수준을 조사한 연구는 거의 없지만, 랜드그라프 외 연구진은 이 miRNA의 발현 수준이 조혈세포에서 매우 낮다는 것을 규명했다.[24]또한 PCR 분석 결과 miR-155-3p는 다수의 인체 조직에서 검출할 수 있었지만 miR-155-5p 수준과 비교할 때 이 miRNA의 표현 수준은 20~200배 낮았다.[25]miR-155-3p의 기능은 대체로 무시되어 왔음에도 불구하고, 현재 여러 연구에서 일부의 경우(아스트로시테스 및 플라스마시토이드 덴드리트리트 세포)에서는 miR-155-5p와 -3p 모두 miR-155 이전부터 기능적으로 성숙될 수 있다고 제시하고 있다.[26][27]
대상
TargetScan 6.2를 사용한 생체정보 분석[3]을 통해 최소 4,174개의 putative human miR-155-5p mRNA 표적이 존재한다고 밝혀 총 918개의 보존 사이트(즉, 마우스와 사람 사이)와 4,249개의 저보존 사이트(즉, 인간만 해당)가 확인되었다.[22][28]TargetScan 6.2 알고리즘을 사용하여 miR-155-3p putive target을 결정할 수는 없지만, 이 miRNA가 수천 mRNA 대상의 표현도 잠재적으로 규제할 수 있다고 추측할 수 있다.
miR-155-5p에 의한 내생성 성적서 규제 시연과 리포터 검사를 통한 miR-155-5p 시드 시퀀스의 유효성 검사에 의해 실험적으로 인증된 miR-155-5p/mRNA 대상의 종합 목록이 최근에 수집되었다.[29]This list included 140 genes and included regulatory proteins for myelopoiesis and leukemogenesis (e.g. SHIP-1, AICDA, ETS1, JARID2, SPI1, etc.), inflammation (e.g. BACH1, FADD, IKBKE, INPP5D, MYD88, RIPK1, SPI1, SOCS, etc.) and known tumor suppressors (e.g. CEBPβ, IL17RB, PCCD4, TCF12, ZNF652, etc.).[29]SPI1 mRNA에 저장되어 있는 검증된 miR-155-5p 바인딩 사이트와 IRAK3 mRNA에 저장되어 있는 검증된 miR-155-3p 바인딩 사이트는 각각 그림 4와 5에 나타나 있다.
생리적 역할
조혈증
조혈증은 혈액 세포의 형성과 발달로 정의되는데, 모두 조혈모세포-조혈모세포(HSPC)에서 유래한 것이다.[31]HSPC는 자가 재생이 가능한 원시 세포로, 처음에는 공통 골수성 증식기(CMP) 또는 공통 림프성 증식기(CLP) 세포로 분화한다.[31]CMP는 골수성 혈통이 된 세포군을 나타내며 골수성 혈통이 시작되는 지점이다.[31]골수성 근막염 중에는 혈전증, 적혈구증, 과립구증, 모노소포성 근막염 등 세포 분화가 추가로 일어난다.[31]CLP는 이후 지정된 림프포피증 공정에서 B세포와 T세포로 구분된다.[31]miR-155-5p가 조혈세포로[24] 표현된다는 점을 고려할 때, 이 miRNA가 이러한 세포 분화 과정에 중요한 역할을 한다는 가설이 제기되었다.In support of this premise, miR-155-5p was found to be expressed in CD34(+) human HSPCs, and it was speculated that this miRNA may hold these cells at an early stem-progenitor stage, inhibiting their differentiation into a more mature cell (i.e. megakaryocytic/erythroid/granulocytic/monocytic/B-lymphoid/T-lymphoid).[32]이 가설은 mir-155 변환 전 HSPC가 골수성 군집을 5배 감소시키고 적혈구 군집을 3배 감소시켰을 때 입증되었다.[32]또한 Hu 등에서는 [33]homeobox 단백질인 HOXA9가 MIR155를 규제한다는 것을 증명하였다.골수세포에서 HG표현과 이 miRNA가 조혈증에 기능적 역할을 했다는 것이다.이 조사관들은 골수 세포에서 miR-155-5p의 강제 발현으로 인해 전사인자 및 골수 세포의 조절기인 [33]SPI1(즉, PU.1)이 약 50% 감소하고 [34]이 miRNA의 검증된 대상이 된다는 것을 발견했다.[30]또한 정화된 인간 적혈구 세포의 체외 분화로 성숙한 적혈구에서 miR-155-5p 발현이 점진적으로 감소하는 결과를 초래한다는 것이 확립되었다.[35]또한 mir-155 이전이 부족한 생쥐는 림프구 발달과 생체 내 B-세포 및 T-세포 반응 생성에 뚜렷한 결함을 보였다.[30][36][37]마지막으로, 규제 T-셀(Tregs) 개발에는 miR-155-5p가 필요하며, 이 miRNA는 IL-2 신호의 음성 조절기인 SOCS1을 직접 겨냥하여 Treg홈ostasis와 전반적인 생존에 역할을 하는 것으로 나타났다.[38][39]이러한 결과를 종합하면 miR-155-5p가 골수, 적혈구, 림프구 등 조혈증의 여러 측면을 제어하는 데 필수적인 분자임을 강력히 시사한다.
면역계
선천적 면역체계는 병원균 침입에 대한 방어 1선을 구성하며 염증반응의 주요 개시자로 간주된다.[40]세포성분은 주로 단세포/대형세포, 과립세포, 덴드리트 세포(DC)를 포함하며, 톨러라이스 수용체(TLR)와 같은 패턴인식 수용체에 의해 보존된 병원체 구조(PAMP)를 감지하면 활성화된다.[41]MIR155HG(즉, miR-155-5p) 표현은 대식세포와 덴드리트 세포의 TLR 작용제 자극에 의해 크게 강화된다.[42][43][44][45][46][47]미생물 리포폴리사당체(TLR4의 작용제)는 NF-κB와 AP-1 전사 인자의 자극으로 이어지는 일련의 사건을 활성화하기 때문에,[41] MIR155의 엔도톡신 활성화가 가정되었다.HG는 이러한 전사 인자에 의해 매개될 수 있다.[42]과연 MIR155HG 표현은 NF-164B 매개 메커니즘에 의해 처리된 황색 대식세포(즉, Raw264.7)에서 활성화된 것으로 확인되었다.[43]또한 일차적 황색 골수에서 유래된 대식세포의 H. 필로리 감염은 MIR155HG의 NF-118B 의존적 상향 조정으로 귀결되었다.[48]바이러스 감염의 맥락에서, 뮤린 복막 대식세포의 VSV(vesicular stopatitis virus) 도전은 레티노산 유도 유전자 I/JNK/NF-NF-B 의존 경로를 통해 miR-155-5p 과표출을 유발하는 것으로 보고되었다.[49]MIR155에서 AP-1 역할 지원HG 활성화는 TLR3 리간드 폴리(I:C) 또는 인터페론 베타(IFN-β)와 같은 바이러스 감염과 관련된 자극을 사용한 연구에서 비롯된다.[44]그러한 자극의 다운스트림 AP-1은 MIR155에서 중요한 역할을 하는 것으로 보인다.HG 활성화.[44][50][51][52]
예를 들어, 활성화를 통한 개시시점.병원체 자극에 의한 TLRs miR-155-5p는 선천적인 면역 신호 경로의 변환 후 조절기 역할을 한다.중요한 것은 miR-155-5p는 주요 친염증 표식기 mRNA와 병원체 자극(예: TLR4 작용제 LPS)에 대한 반응성이 유사하다는 점이다.[53]miR-155-5p가 활성화되면 염증의 음성 조절기를 억제한다.여기에는 이노시톨 폴리인산염-5인산염효소(INPP5D도 SHIP1)와 사이토카인 신호 1 억제기(SOCS1)가 포함되며, 억제기는 세포 생존, 성장, 이동, 항병원성 반응을 촉진한다.[49][54][55][56]방어 경로 miR-155-5p의 활성화를 지원하는 것 외에도 염증 단계마다 miR-155의 다양한 기능을 제안하면서 결과로 발생하는 NF-156B 종속 염증 반응의 강도를 제한할 수 있다.[53]
이러한 관찰 결과를 종합하면 MIR155의 활성화가AP-1과 NF-medB 매개 메커니즘이 모두 이 유전자의 발현을 조절한다는 점에서 HG는 상황에 따라 달라질 수 있다.이러한 연구들은 또한 광범위한 바이러스 및 세균성 염증 매개체가 miR-155-5p의 발현을 자극할 수 있으며 염증, 선천성 면역, MIR155 사이에 친밀한 관계가 있음을 나타낸다.HG 표현.
활동성 및 표현형
miR-155가 심혈관질환과 고혈압과 관련된 폭포(cascades)에 참여한다는 증거가 있으며, 면역, 유전적 불안정성, 세포분화, 염증, 바이러스 관련 감염, 암, 당뇨병 명예에도 관여하는 것으로 밝혀졌다.[57]
miR-155의 보호 역할은 유전자의 음소거에 대한 작용에 대응하여 그들의 발현 시간을 조절하고, miR-155 대상 사이트의 돌연변이는 유전자 음소거 발생에 필요한 최적의 접근을 거부하여, 예를 들어, miR-155의 보호제 역할로서 악성적일 수 있는 비행활동이 과도하게 많아지게 된다.B세포 관련 악성종양에 대한 게인스트 성향은 활성화 유도 시티딘 디아미나제(AID) 효소의 균형을 유지함으로써 강조된다.MiR-155는 면역학적 단서들에 따른 AID 풍부과 표현 시간의 조절을 중재하지만, AID mRNA의 표적에서 돌연변이는 miR-155 침묵에 대한 반응이 없고 단백질의 조절되지 않은 발현이 발생하여 야생의 미성숙 B-림프세포 서지와 AID 매개 염색체 변환을 유발한다.[5][6]
임상적 유의성
심혈관의
miR-155를 인간의 1차 폐섬유화합물로 전이하면 안지오텐신 II 수용체 AT1R 단백질의 내생적 발현이 감소한다.또한 AT1R은 혈관신 II 관련 혈압 상승을 매개하고 심부전의 병원생성에 기여한다.AT1R(miR-155 대상 사이트)의 3' UTR의 시스 규제 사이트가 AT1R 자체의 SNP 다형성으로 인해 영향을 받은 경우 결함 있는 miR-155 기능이 고혈압 및 심혈관 질환에 관련될 수 있다.이 돌연변이는 miR-155 표적을 파괴하여 AT1R 표현 하한을 방지한다.[5]저혈압에서 miR-155의 과표현은 AT1R 활동의 손상과 관련이 있다.[4]
면역
miR-155는 예를 들어, miR-155 결핍 마우스에서 면역기억이 손상되어 동일한 병원체(Rodriz et al. 2007), 성숙 및 miR-155-피질 B-lymphociy의 반복적인 침입의 희생물이 된다.es는 공정이 그것의 3′ UTR 끝에서 miR-155 표적을 가진 AID 효소에 의존하기 때문에 손상되었다.[5][6]생쥐의 miR-155 결핍과 관련된 표현형 결과는 말년에 동물들이 폐와 장에 병변이 생기는 것을 보여준다.[4]
활성화된 B세포와 T세포는 miR-155 발현이 증가하며, 면역체계의 대식세포와 덴드리트세포도 마찬가지다.MiR-155는 적절한 림프구 발달과 성숙을 위해 중요하다.miR-155 수준의 다양한 징후와 최적의 면역 반응을 확인하는 활동에 대한 자세한 내용은 많은 연구의 대상이 되었다.
IgG1의 감소
결함이 있는 T 세포와 B 세포 그리고 현저하게 감소된 IgG1 반응이 miR-155 결핍 마우스에서 관찰되었다. IgG1은 감소하는 반면 IgM 면역글로불린의 표현은 이러한 마우스에서 정상으로 유지된다.IgG1 수준의 변화는 B세포에서 miR-155의 표적이라는 사실, 전사조절기 Pu.1-단백질을 위한 단백질 인코딩 mRNA, Pu.1 단백질의 상승은 IgG1 생산의 결함을 유발한다.Pu.1 외에도, miR-155 부족한 B세포에는 60개의 다른 미분증상 유전자가 있으며, 추가 검사 결과 이들 유전자의 3 3 UTR 부위에서 miR-155 표적 부위가 있을 가능성이 있는 것으로 밝혀졌다.[6]
림프구 악성종
성숙한 수용체 친화력과 병원성 물질에 대한 림프구의 특수성은 적절한 면역 반응을 기초로 하며, 정상 B 림프구의 제조, 고선호도 항체의 생산 및 BCR 신호 전달의 균형을 위해 최적의 miR-155 조율이 필요하다.miR-155는 백혈구 세포에서 건강한 B세포로 갭접합을 통해 전이될 수 있으며, 종양유전세포로 변환을 촉진할 수 있다는 것이 입증되었다.
유능한 B세포의 선택은 생식 중심에서 이루어지는데, 거기서 체세포와 외국 항원을 구별하기 위한 훈련을 받고 항원인식을 위해 경쟁하며 T세포의 도움을 위해 이러한 선택적 압력의 방식에서 고선호감 수용체를 입증한 B세포와 T세포와의 협력(적정성숙성)이 재현된다.골수나 기억력 B 세포에 배치되어, 경쟁에서 탈락한 B 세포들에게 세포종식이 일어난다.Bcl-2 단백질 수치의 상승으로 인한 미숙한 B세포 탈구현상, B세포 악성종양에 관여하고 miR-155에 의해 통제되는 것으로 밝혀진 단백질.[6]
염증
TNF-α와 같은 트리거에 대한 염증 반응은 miR-155를 포함하는 성분의 대식세포와 아토피 피부염에서 과다압축되며 CTLA-4의 다운규제를 통해 T(H)세포의 증식반응을 증가시켜 만성피부염에 기여한다.[59]류마티스 관절염과 같은 자가면역 장애에서 miR-155는 환자의 조직과 시노브 섬유질에서 더 높은 발현을 보였다.[4]다발성 경화증에서 mir-155의 표현 증가도 혈액 단세포 순환과 활성 미세 글리아를 포함한 주변 세포와 CNS 거주 골수 세포에서 측정되었다.[60]mir-155가 염증에 연루돼 있다는 사실도 밝혀졌다.mir-155의 과도한 압박은 인간에게 만성적인 염증 상태를 초래할 것이다.[61]
DNA 바이러스
그non-oncogenic 상대 MDV-2, 이 implica을 제안하지 않는다 DNA바이러스에서 miRNAs 실험적으로 증명되었다, 바이러스에 miRNAs 같은 바이러스의 dsDNAs,[5]예에 의하여 암호화 됩니다 Humans-Epstein-Barr 바이러스(EBV) 같은 herpesviruses과 다른 바이러스 닭에서miR-155-like miRNA을 표현하는 adenoviruses,[4]은 종양 형성의 MDV-1을 포함한다.tion 제일의 것이다fmiR-155 [5]림프구 내.바이러스 정도에 그들이 예를 들어 바이러스 복제에 인코딩하는 데 호스트 miRNAs을 사용합니다. Kaposi's-sarcoma-associated Herpesvirus에 miR-K12-11 그 miR-155의에 대상을 특이성 지역 orthologous이 있습니다, 그것으로 대상을 공유하는 것miR-155[62]의 행동을 흉내 내므로 그것은 miR-155 accessib을 억제하기 위하여 생각될 수 있호스트 miRNAs 활용할 수 있다.ili경쟁과 이것은 사실상 miR-155에 의한 규제를 무시하는 방식으로 세포 성장과 세포 사멸에서 역할을 하는 유전자의 발현을 규제한다.[4]EBV는 호스트 miR-155 표현을 변조하는데, 이는 EBV에 감염된 B 세포의 성장에 필수적이다.[63]EBV에 감염된 세포는 miR-155의 발현을 증가시켜 그 세포에서 전사를 조절하는 유전자의 발현 평형을 방해했다.[4][5]
암
miR-155에 의한 과도한 음소거로 인해 사포체 저항으로 시작되는 발작성 폭포성 종양 단백질-53 유도 핵-단백질1(TP53INP1)이 miR-155에 의해 침묵되고 miR-155의 과대 발현으로 인해 TP53 수준이 감소할 수 있다.췌장 덕트 아데노카르시노마스와 TP53이 있는 다른 상피암에서 INP1INP1 활동이 상실되어 사멸 회피와 통제되지 않은 성장 덩어리가 된다.[5]
Inactivation of DNA Mismatch Repair (MMR) as identified by elevation of mutation rates is the cause of Lynch Syndrome (LS), also known as hereditary nonpolyposis colorectal cancer (HNPCC), down-regulation of MMR controlling protein is carried out by over-expression of miR-155, MMR is controlled by a group of conserved proteins, reduced activity of이 단백질들은 이런 종류의 암을 발전시키기 위한 진보를 촉발하는 표현형에서 높은 수준의 돌연변이를 초래한다.[64]
miR-155 과표현이 보고된 다른 유형의 종양에는 갑상선암, 유방암, 대장암, 자궁경부암, 폐암 등이 포함되며, 여기서 구별되는 miR-155 표현 프로파일 정량화가 잠재적으로 종양 검출 및 예후 결과의 평가의 신호 역할을 할 수 있다.[4]miR-155 표현이 삼중 음성 유방암에서 생존과 연관돼 있다는 분석이 나온다.[65]
메모들
이 글의 2012년 버전은 이중 출판 모델에 따라 외부 전문가에 의해 갱신되었다.해당 학술적 동료 검토 기사는 진에 게재되었으며, 다음과 같이 인용할 수 있다. Terry S Elton; Helina Selemon; Shane M Elton; Narasimham L Parinandi (14 December 2012). "Regulation of the MIR155 host gene in physiological and pathological processes". Gene. 532 (1): 1–12. doi:10.1016/J.GENE.2012.12.009. ISSN 0378-1119. PMID 23246696. Wikidata Q34318174. |
참고 항목
참조
- ^ a b c GRCh38: 앙상블 릴리스 89: ENSG00000283904 - 앙상블, 2017년 5월
- ^ "Human PubMed Reference:". National Center for Biotechnology Information, U.S. National Library of Medicine.
- ^ "Entrez Gene: MIR155HG".
- ^ a b c d e f g h i Faraoni I, Antonetti FR, Cardone J, Bonmassar E (Jun 2009). "miR-155 gene: a typical multifunctional microRNA" (PDF). Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease. 1792 (6): 497–505. doi:10.1016/j.bbadis.2009.02.013. PMID 19268705.
- ^ a b c d e f g h i Teng G, Papavasiliou FN (Mar 2009). "Shhh! Silencing by microRNA-155". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 364 (1517): 631–637. doi:10.1098/rstb.2008.0209. PMC 2660923. PMID 19008191.
- ^ a b c d e f Calame K (Dec 2007). "MicroRNA-155 function in B Cells". Immunity. 27 (6): 825–827. doi:10.1016/j.immuni.2007.11.010. PMID 18093533.
- ^ Tili E, Croce CM, Michaille JJ (2009). "miR-155: on the crosstalk between inflammation and cancer". International Reviews of Immunology. 28 (5): 264–284. doi:10.1080/08830180903093796. PMID 19811312. S2CID 205589961.
- ^ O'Connell RM, Rao DS, Baltimore D (2012). "microRNA regulation of inflammatory responses". Annual Review of Immunology. 30: 295–312. doi:10.1146/annurev-immunol-020711-075013. PMID 22224773.
- ^ Elton TS, Selemon H, Elton SM, Parinandi NL (Dec 2013). "Regulation of the MIR155 host gene in physiological and pathological processes". Gene. 532 (1): 1–12. doi:10.1016/j.gene.2012.12.009. PMID 23246696.
- ^ Mattiske S, Suetani RJ, Neilsen PM, Callen DF (Aug 2012). "The oncogenic role of miR-155 in breast cancer". Cancer Epidemiology, Biomarkers & Prevention. 21 (8): 1236–1243. doi:10.1158/1055-9965.EPI-12-0173. PMID 22736789.
- ^ Babar IA, Cheng CJ, Booth CJ, Liang X, Weidhaas JB, Saltzman WM, Slack FJ (Jun 2012). "Nanoparticle-based therapy in an in vivo microRNA-155 (miR-155)-dependent mouse model of lymphoma". Proceedings of the National Academy of Sciences of the United States of America. 109 (26): E1695–1704. doi:10.1073/pnas.1201516109. PMC 3387084. PMID 22685206.
- ^ Wang L, Toomey NL, Diaz LA, Walker G, Ramos JC, Barber GN, Ning S (Aug 2011). "Oncogenic IRFs provide a survival advantage for Epstein-Barr virus- or human T-cell leukemia virus type 1-transformed cells through induction of BIC expression". Journal of Virology. 85 (16): 8328–8337. doi:10.1128/JVI.00570-11. PMC 3147954. PMID 21680528.
- ^ Corsten MF, Papageorgiou A, Verhesen W, Carai P, Lindow M, Obad S, Summer G, Coort SL, Hazebroek M, van Leeuwen R, Gijbels MJ, Wijnands E, Biessen EA, De Winther MP, Stassen FR, Carmeliet P, Kauppinen S, Schroen B, Heymans S (Aug 2012). "MicroRNA profiling identifies microRNA-155 as an adverse mediator of cardiac injury and dysfunction during acute viral myocarditis". Circulation Research. 111 (4): 415–425. doi:10.1161/CIRCRESAHA.112.267443. PMID 22715471.
- ^ Eis PS, Tam W, Sun L, Chadburn A, Li Z, Gomez MF, Lund E, Dahlberg JE (Mar 2005). "Accumulation of miR-155 and BIC RNA in human B cell lymphomas". Proceedings of the National Academy of Sciences of the United States of America. 102 (10): 3627–3632. Bibcode:2005PNAS..102.3627E. doi:10.1073/pnas.0500613102. PMC 552785. PMID 15738415.
- ^ a b c d Tam W (Aug 2001). "Identification and characterization of human BIC, a gene on chromosome 21 that encodes a noncoding RNA". Gene. 274 (1–2): 157–167. doi:10.1016/S0378-1119(01)00612-6. PMID 11675008.
- ^ a b Kim VN, Han J, Siomi MC (Feb 2009). "Biogenesis of small RNAs in animals". Nature Reviews Molecular Cell Biology. 10 (2): 126–139. doi:10.1038/nrm2632. PMID 19165215. S2CID 8360619.
- ^ a b Krol J, Loedige I, Filipowicz W (Sep 2010). "The widespread regulation of microRNA biogenesis, function and decay". Nature Reviews Genetics. 11 (9): 597–610. doi:10.1038/nrg2843. PMID 20661255. S2CID 2619579.
- ^ a b c Fabian MR, Sonenberg N (Jun 2012). "The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC". Nature Structural & Molecular Biology. 19 (6): 586–593. doi:10.1038/nsmb.2296. PMID 22664986. S2CID 13176647.
- ^ Bushati N, Cohen SM (2007). "microRNA functions". Annual Review of Cell and Developmental Biology. 23: 175–205. doi:10.1146/annurev.cellbio.23.090506.123406. PMID 17506695.
- ^ Filipowicz W, Bhattacharyya SN, Sonenberg N (Feb 2008). "Mechanisms of post-transcriptional regulation by microRNAs: are the answers in sight?". Nature Reviews Genetics. 9 (2): 102–114. doi:10.1038/nrg2290. PMID 18197166. S2CID 11824239.
- ^ Griffiths-Jones S (Jan 2004). "The microRNA Registry". Nucleic Acids Research. 32 (Database issue): D109–11. doi:10.1093/nar/gkh023. PMC 308757. PMID 14681370.
- ^ a b Friedman RC, Farh KK, Burge CB, Bartel DP (Jan 2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Martin MM, Lee EJ, Buckenberger JA, Schmittgen TD, Elton TS (Jul 2006). "MicroRNA-155 regulates human angiotensin II type 1 receptor expression in fibroblasts". The Journal of Biological Chemistry. 281 (27): 18277–18284. doi:10.1074/jbc.M601496200. PMID 16675453.
- ^ a b c Landgraf P, Rusu M, Sheridan R, Sewer A, Iovino N, Aravin A, Pfeffer S, Rice A, Kamphorst AO, Landthaler M, Lin C, Socci ND, Hermida L, Fulci V, Chiaretti S, Foà R, Schliwka J, Fuchs U, Novosel A, Müller RU, Schermer B, Bissels U, Inman J, Phan Q, Chien M, Weir DB, Choksi R, De Vita G, Frezzetti D, Trompeter HI, Hornung V, Teng G, Hartmann G, Palkovits M, Di Lauro R, Wernet P, Macino G, Rogler CE, Nagle JW, Ju J, Papavasiliou FN, Benzing T, Lichter P, Tam W, Brownstein MJ, Bosio A, Borkhardt A, Russo JJ, Sander C, Zavolan M, Tuschl T (Jun 2007). "A mammalian microRNA expression atlas based on small RNA library sequencing". Cell. 129 (7): 1401–1414. doi:10.1016/j.cell.2007.04.040. PMC 2681231. PMID 17604727.
- ^ Elton TS, Sansom SE, Martin MM (2010). "Trisomy-21 gene dosage over-expression of miRNAs results in the haploinsufficiency of specific target proteins". RNA Biology. 7 (5): 540–547. doi:10.4161/rna.7.5.12685. PMC 3073250. PMID 21081842.
- ^ Tarassishin L, Loudig O, Bauman A, Shafit-Zagardo B, Suh HS, Lee SC (Dec 2011). "Interferon regulatory factor 3 inhibits astrocyte inflammatory gene expression through suppression of the proinflammatory miR-155 and miR-155*". Glia. 59 (12): 1911–1922. doi:10.1002/glia.21233. PMC 3241213. PMID 22170100.
- ^ a b Zhou H, Huang X, Cui H, Luo X, Tang Y, Chen S, Wu L, Shen N (Dec 2010). "miR-155 and its star-form partner miR-155* cooperatively regulate type I interferon production by human plasmacytoid dendritic cells". Blood. 116 (26): 5885–5894. doi:10.1182/blood-2010-04-280156. PMID 20852130.
- ^ Lewis BP, Burge CB, Bartel DP (Jan 2005). "Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets". Cell. 120 (1): 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477. S2CID 17316349.
- ^ a b Neilsen PM, Noll JE, Mattiske S, Bracken CP, Gregory PA, Schulz RB, Lim SP, Kumar R, Suetani RJ, Goodall GJ, Callen DF (Jun 2013). "Mutant p53 drives invasion in breast tumors through up-regulation of miR-155". Oncogene. 32 (24): 2992–3000. doi:10.1038/onc.2012.305. PMID 22797073.
- ^ a b c Vigorito E, Perks KL, Abreu-Goodger C, Bunting S, Xiang Z, Kohlhaas S, Das PP, Miska EA, Rodriguez A, Bradley A, Smith KG, Rada C, Enright AJ, Toellner KM, Maclennan IC, Turner M (Dec 2007). "microRNA-155 regulates the generation of immunoglobulin class-switched plasma cells". Immunity. 27 (6): 847–859. doi:10.1016/j.immuni.2007.10.009. PMC 4135426. PMID 18055230.
- ^ a b c d e Mayani H (Mar 2010). "Biological differences between neonatal and adult human hematopoietic stem/progenitor cells". Stem Cells and Development. 19 (3): 285–298. doi:10.1089/scd.2009.0327. PMID 19778207.
- ^ a b Georgantas RW, Hildreth R, Morisot S, Alder J, Liu CG, Heimfeld S, Calin GA, Croce CM, Civin CI (Feb 2007). "CD34+ hematopoietic stem-progenitor cell microRNA expression and function: a circuit diagram of differentiation control". Proceedings of the National Academy of Sciences of the United States of America. 104 (8): 2750–2755. Bibcode:2007PNAS..104.2750G. doi:10.1073/pnas.0610983104. PMC 1796783. PMID 17293455.
- ^ a b Hu YL, Fong S, Largman C, Shen WF (Sep 2010). "HOXA9 regulates miR-155 in hematopoietic cells". Nucleic Acids Research. 38 (16): 5472–5478. doi:10.1093/nar/gkq337. PMC 2938212. PMID 20444872.
- ^ Kastner P, Chan S (2008). "PU.1: a crucial and versatile player in hematopoiesis and leukemia". The International Journal of Biochemistry & Cell Biology. 40 (1): 22–27. doi:10.1016/j.biocel.2007.01.026. PMID 17374502.
- ^ Masaki S, Ohtsuka R, Abe Y, Muta K, Umemura T (Dec 2007). "Expression patterns of microRNAs 155 and 451 during normal human erythropoiesis". Biochemical and Biophysical Research Communications. 364 (3): 509–514. doi:10.1016/j.bbrc.2007.10.077. PMID 17964546.
- ^ Rodriguez A, Vigorito E, Clare S, Warren MV, Couttet P, Soond DR, van Dongen S, Grocock RJ, Das PP, Miska EA, Vetrie D, Okkenhaug K, Enright AJ, Dougan G, Turner M, Bradley A (Apr 2007). "Requirement of bic/microRNA-155 for normal immune function". Science. 316 (5824): 608–611. Bibcode:2007Sci...316..608R. doi:10.1126/science.1139253. PMC 2610435. PMID 17463290.
- ^ Thai TH, Calado DP, Casola S, Ansel KM, Xiao C, Xue Y, Murphy A, Frendewey D, Valenzuela D, Kutok JL, Schmidt-Supprian M, Rajewsky N, Yancopoulos G, Rao A, Rajewsky K (Apr 2007). "Regulation of the germinal center response by microRNA-155". Science. 316 (5824): 604–608. Bibcode:2007Sci...316..604T. doi:10.1126/science.1141229. PMID 17463289. S2CID 8174458.
- ^ Kohlhaas S, Garden OA, Scudamore C, Turner M, Okkenhaug K, Vigorito E (Mar 2009). "Cutting edge: the Foxp3 target miR-155 contributes to the development of regulatory T cells". Journal of Immunology. 182 (5): 2578–2582. doi:10.4049/jimmunol.0803162. PMID 19234151.
- ^ Lu LF, Thai TH, Calado DP, Chaudhry A, Kubo M, Tanaka K, Loeb GB, Lee H, Yoshimura A, Rajewsky K, Rudensky AY (Jan 2009). "Foxp3-dependent microRNA155 confers competitive fitness to regulatory T cells by targeting SOCS1 protein". Immunity. 30 (1): 80–91. doi:10.1016/j.immuni.2008.11.010. PMC 2654249. PMID 19144316.
- ^ Medzhitov R (Mar 2010). "Inflammation 2010: new adventures of an old flame". Cell. 140 (6): 771–776. doi:10.1016/j.cell.2010.03.006. PMID 20303867. S2CID 10297400.
- ^ a b Takeda K, Akira S (Jan 2005). "Toll-like receptors in innate immunity". International Immunology. 17 (1): 1–14. doi:10.1093/intimm/dxh186. PMID 15585605.
- ^ a b Taganov KD, Boldin MP, Chang KJ, Baltimore D (Aug 2006). "NF-kappaB-dependent induction of microRNA miR-146, an inhibitor targeted to signaling proteins of innate immune responses". Proceedings of the National Academy of Sciences of the United States of America. 103 (33): 12481–12486. Bibcode:2006PNAS..10312481T. doi:10.1073/pnas.0605298103. PMC 1567904. PMID 16885212.
- ^ a b Tili E, Michaille JJ, Cimino A, Costinean S, Dumitru CD, Adair B, Fabbri M, Alder H, Liu CG, Calin GA, Croce CM (Oct 2007). "Modulation of miR-155 and miR-125b levels following lipopolysaccharide/TNF-alpha stimulation and their possible roles in regulating the response to endotoxin shock". Journal of Immunology. 179 (8): 5082–5089. doi:10.4049/jimmunol.179.8.5082. PMID 17911593.
- ^ a b c O'Connell RM, Taganov KD, Boldin MP, Cheng G, Baltimore D (Jan 2007). "MicroRNA-155 is induced during the macrophage inflammatory response". Proceedings of the National Academy of Sciences of the United States of America. 104 (5): 1604–1609. Bibcode:2007PNAS..104.1604O. doi:10.1073/pnas.0610731104. PMC 1780072. PMID 17242365.
- ^ Ceppi M, Pereira PM, Dunand-Sauthier I, Barras E, Reith W, Santos MA, Pierre P (Feb 2009). "MicroRNA-155 modulates the interleukin-1 signaling pathway in activated human monocyte-derived dendritic cells". Proceedings of the National Academy of Sciences of the United States of America. 106 (8): 2735–2740. Bibcode:2009PNAS..106.2735C. doi:10.1073/pnas.0811073106. PMC 2650335. PMID 19193853.
- ^ Cremer TJ, Ravneberg DH, Clay CD, Piper-Hunter MG, Marsh CB, Elton TS, Gunn JS, Amer A, Kanneganti TD, Schlesinger LS, Butchar JP, Tridandapani S (2009). "MiR-155 induction by F. novicida but not the virulent F. tularensis results in SHIP down-regulation and enhanced pro-inflammatory cytokine response". PLOS ONE. 4 (12): e8508. Bibcode:2009PLoSO...4.8508C. doi:10.1371/journal.pone.0008508. PMC 2794384. PMID 20041145.
- ^ Mao CP, He L, Tsai YC, Peng S, Kang TH, Pang X, Monie A, Hung CF, Wu TC (2011). "In vivo microRNA-155 expression influences antigen-specific T cell-mediated immune responses generated by DNA vaccination". Cell & Bioscience. 1 (1): 3. doi:10.1186/2045-3701-1-3. PMC 3116247. PMID 21711593.
- ^ Koch M, Mollenkopf HJ, Klemm U, Meyer TF (May 2012). "Induction of microRNA-155 is TLR- and type IV secretion system-dependent in macrophages and inhibits DNA-damage induced apoptosis". Proceedings of the National Academy of Sciences of the United States of America. 109 (19): E1153–1162. doi:10.1073/pnas.1116125109. PMC 3358876. PMID 22509021.
- ^ a b Wang P, Hou J, Lin L, Wang C, Liu X, Li D, Ma F, Wang Z, Cao X (Nov 2010). "Inducible microRNA-155 feedback promotes type I IFN signaling in antiviral innate immunity by targeting suppressor of cytokine signaling 1". Journal of Immunology. 185 (10): 6226–6233. doi:10.4049/jimmunol.1000491. PMID 20937844.
- ^ Tili E, Michaille JJ, Adair B, Alder H, Limagne E, Taccioli C, Ferracin M, Delmas D, Latruffe N, Croce CM (Sep 2010). "Resveratrol decreases the levels of miR-155 by upregulating miR-663, a microRNA targeting JunB and JunD". Carcinogenesis. 31 (9): 1561–1566. doi:10.1093/carcin/bgq143. PMC 4647642. PMID 20622002.
- ^ McCoy CE, Sheedy FJ, Qualls JE, Doyle SL, Quinn SR, Murray PJ, O'Neill LA (Jul 2010). "IL-10 inhibits miR-155 induction by toll-like receptors". The Journal of Biological Chemistry. 285 (27): 20492–20498. doi:10.1074/jbc.M110.102111. PMC 2898307. PMID 20435894.
- ^ Ruggiero T, Trabucchi M, De Santa F, Zupo S, Harfe BD, McManus MT, Rosenfeld MG, Briata P, Gherzi R (Sep 2009). "LPS induces KH-type splicing regulatory protein-dependent processing of microRNA-155 precursors in macrophages". FASEB Journal. 23 (9): 2898–2908. doi:10.1096/fj.09-131342. PMID 19423639. S2CID 588328.
- ^ a b Schulte LN, Westermann AJ, Vogel J (Jan 2013). "Differential activation and functional specialization of miR-146 and miR-155 in innate immune sensing". Nucleic Acids Research. 41 (1): 542–553. doi:10.1093/nar/gks1030. PMC 3592429. PMID 23143100.
- ^ Androulidaki A, Iliopoulos D, Arranz A, Doxaki C, Schworer S, Zacharioudaki V, Margioris AN, Tsichlis PN, Tsatsanis C (Aug 2009). "The kinase Akt1 controls macrophage response to lipopolysaccharide by regulating microRNAs". Immunity. 31 (2): 220–231. doi:10.1016/j.immuni.2009.06.024. PMC 2865583. PMID 19699171.
- ^ O'Connell RM, Chaudhuri AA, Rao DS, Baltimore D (Apr 2009). "Inositol phosphatase SHIP1 is a primary target of miR-155". Proceedings of the National Academy of Sciences of the United States of America. 106 (17): 7113–7118. Bibcode:2009PNAS..106.7113O. doi:10.1073/pnas.0902636106. PMC 2678424. PMID 19359473.
- ^ Costinean S, Sandhu SK, Pedersen IM, Tili E, Trotta R, Perrotti D, Ciarlariello D, Neviani P, Harb J, Kauffman LR, Shidham A, Croce CM (Aug 2009). "Src homology 2 domain-containing inositol-5-phosphatase and CCAAT enhancer-binding protein beta are targeted by miR-155 in B cells of Emicro-MiR-155 transgenic mice". Blood. 114 (7): 1374–1382. doi:10.1182/blood-2009-05-220814. PMC 2727407. PMID 19520806.
- ^ Jankauskas SS, Gambardella J, Sardu C, Lombardi A, Santulli G (July 2021). "Functional Role of miR-155 in the Pathogenesis of Diabetes Mellitus and Its Complications". Non-Coding RNA. 7 (3): 39. doi:10.3390/ncrna7030039. PMC 8293470. PMID 34287359.
- ^ Nesmiyanov P, Strygin A, Tolkachev B, Kaplanov K, Dotsenko A, Strygina A (2016). "mIRNA-155 shuttling through gap junctions facilitates CLL progression". The 14th CIMT Annual Meeting Mechanisms of Efficacy in Cancer Immunotherapy, Mainz, Germany, 2016.
- ^ Sonkoly E, Janson P, Majuri ML, Savinko T, Fyhrquist N, Eidsmo L, Xu N, Meisgen F, Wei T, Bradley M, Stenvang J, Kauppinen S, Alenius H, Lauerma A, Homey B, Winqvist O, Ståhle M, Pivarcsi A (Sep 2010). "MiR-155 is overexpressed in patients with atopic dermatitis and modulates T-cell proliferative responses by targeting cytotoxic T lymphocyte-associated antigen 4". The Journal of Allergy and Clinical Immunology. 126 (3): 581–589.e1–20. doi:10.1016/j.jaci.2010.05.045. PMID 20673989.
- ^ Moore CS, Rao VT, Durafourt BA, Bedell BJ, Ludwin SK, Bar-Or A, Antel JP (Nov 2013). "miR-155 as a multiple sclerosis-relevant regulator of myeloid cell polarization". Annals of Neurology. 74 (5): 709–720. doi:10.1002/ana.23967. PMID 23818336. S2CID 205344718.
- ^ O'Connell RM, Rao DS, Baltimore D (2012). "microRNA regulation of inflammatory responses". Annual Review of Immunology. 30: 295–312. doi:10.1146/annurev-immunol-020711-075013. PMID 22224773.
- ^ Skalsky RL, Samols MA, Plaisance KB, Boss IW, Riva A, Lopez MC, Baker HV, Renne R (Dec 2007). "Kaposi's sarcoma-associated herpesvirus encodes an ortholog of miR-155". Journal of Virology. 81 (23): 12836–12845. doi:10.1128/JVI.01804-07. PMC 2169101. PMID 17881434.
- ^ Linnstaedt SD, Gottwein E, Skalsky RL, Luftig MA, Cullen BR (Nov 2010). "Virally induced cellular microRNA miR-155 plays a key role in B-cell immortalization by Epstein-Barr virus". Journal of Virology. 84 (22): 11670–11678. doi:10.1128/JVI.01248-10. PMC 2977875. PMID 20844043.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (Apr 2010). "Modulation of mismatch repair and genomic stability by miR-155". Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–6987. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ Lánczky, András; Nagy, Ádám; Bottai, Giulia; Munkácsy, Gyöngyi; Szabó, András; Santarpia, Libero; Győrffy, Balázs (2016-12-01). "miRpower: a web-tool to validate survival-associated miRNAs utilizing expression data from 2178 breast cancer patients". Breast Cancer Research and Treatment. 160 (3): 439–446. doi:10.1007/s10549-016-4013-7. ISSN 1573-7217. PMID 27744485. S2CID 11165696.
추가 읽기
- Bentwich I, Avniel A, Karov Y, Aharonov R, Gilad S, Barad O, Barzilai A, Einat P, Einav U, Meiri E, Sharon E, Spector Y, Bentwich Z (Jul 2005). "Identification of hundreds of conserved and nonconserved human microRNAs". Nature Genetics. 37 (7): 766–770. doi:10.1038/ng1590. PMID 15965474. S2CID 7746954.
- Huang RS, Hu GQ, Lin B, Lin ZY, Sun CC (Dec 2010). "MicroRNA-155 silencing enhances inflammatory response and lipid uptake in oxidized low-density lipoprotein-stimulated human THP-1 macrophages". Journal of Investigative Medicine. 58 (8): 961–967. doi:10.2310/jim.0b013e3181ff46d7. PMID 21030878. S2CID 83123221.
- Ceolotto G, Papparella I, Bortoluzzi A, Strapazzon G, Ragazzo F, Bratti P, Fabricio AS, Squarcina E, Gion M, Palatini P, Semplicini A (Feb 2011). "Interplay between miR-155, AT1R A1166C polymorphism, and AT1R expression in young untreated hypertensives". American Journal of Hypertension. 24 (2): 241–246. doi:10.1038/ajh.2010.211. PMID 20966899.
- Wang G, Tam LS, Li EK, Kwan BC, Chow KM, Luk CC, Li PK, Szeto CC (Dec 2010). "Serum and urinary cell-free MiR-146a and MiR-155 in patients with systemic lupus erythematosus". The Journal of Rheumatology. 37 (12): 2516–2522. doi:10.3899/jrheum.100308. PMID 20952466. S2CID 23208771.
- Kutty RK, Nagineni CN, Samuel W, Vijayasarathy C, Hooks JJ, Redmond TM (Nov 2010). "Inflammatory cytokines regulate microRNA-155 expression in human retinal pigment epithelial cells by activating JAK/STAT pathway". Biochemical and Biophysical Research Communications. 402 (2): 390–395. doi:10.1016/j.bbrc.2010.10.042. PMC 2992362. PMID 20950585.
- Imaizumi T, Tanaka H, Tajima A, Yokono Y, Matsumiya T, Yoshida H, Tsuruga K, Aizawa-Yashiro T, Hayakari R, Inoue I, Ito E, Satoh K (2010). "IFN-γ and TNF-α synergistically induce microRNA-155 which regulates TAB2/IP-10 expression in human mesangial cells". American Journal of Nephrology. 32 (5): 462–468. doi:10.1159/000321365. PMID 20948191. S2CID 12859943.
- Thompson RC, Herscovitch M, Zhao I, Ford TJ, Gilmore TD (Jan 2011). "NF-kappaB down-regulates expression of the B-lymphoma marker CD10 through a miR-155/PU.1 pathway". The Journal of Biological Chemistry. 286 (3): 1675–1682. doi:10.1074/jbc.M110.177063. PMC 3023462. PMID 20947507.
- Wang P, Hou J, Lin L, Wang C, Liu X, Li D, Ma F, Wang Z, Cao X (Nov 2010). "Inducible microRNA-155 feedback promotes type I IFN signaling in antiviral innate immunity by targeting suppressor of cytokine signaling 1". Journal of Immunology. 185 (10): 6226–6233. doi:10.4049/jimmunol.1000491. PMID 20937844.
- O'Connell RM, Kahn D, Gibson WS, Round JL, Scholz RL, Chaudhuri AA, Kahn ME, Rao DS, Baltimore D (Oct 2010). "MicroRNA-155 promotes autoimmune inflammation by enhancing inflammatory T cell development". Immunity. 33 (4): 607–619. doi:10.1016/j.immuni.2010.09.009. PMC 2966521. PMID 20888269.
- Xia QS, Ishigaki Y, Zhao X, Shimasaki T, Nakajima H, Nakagawa H, Takegami T, Chen ZH, Motoo Y (Jan 2011). "Human SMG-1 is involved in gemcitabine-induced primary microRNA-155/BIC up-regulation in human pancreatic cancer PANC-1 cells". Pancreas. 40 (1): 55–60. doi:10.1097/MPA.0b013e3181e89f74. PMID 20871480. S2CID 39789659.
- Zhou H, Huang X, Cui H, Luo X, Tang Y, Chen S, Wu L, Shen N (Dec 2010). "miR-155 and its star-form partner miR-155* cooperatively regulate type I interferon production by human plasmacytoid dendritic cells". Blood. 116 (26): 5885–5894. doi:10.1182/blood-2010-04-280156. PMID 20852130.
- Linnstaedt SD, Gottwein E, Skalsky RL, Luftig MA, Cullen BR (Nov 2010). "Virally induced cellular microRNA miR-155 plays a key role in B-cell immortalization by Epstein-Barr virus". Journal of Virology. 84 (22): 11670–11678. doi:10.1128/JVI.01248-10. PMC 2977875. PMID 20844043.
- Zheng L, Xu CC, Chen WD, Shen WL, Ruan CC, Zhu LM, Zhu DL, Gao PJ (Oct 2010). "MicroRNA-155 regulates angiotensin II type 1 receptor expression and phenotypic differentiation in vascular adventitial fibroblasts". Biochemical and Biophysical Research Communications. 400 (4): 483–488. doi:10.1016/j.bbrc.2010.08.067. PMID 20735984.
- Tili E, Michaille JJ, Adair B, Alder H, Limagne E, Taccioli C, Ferracin M, Delmas D, Latruffe N, Croce CM (Sep 2010). "Resveratrol decreases the levels of miR-155 by upregulating miR-663, a microRNA targeting JunB and JunD". Carcinogenesis. 31 (9): 1561–1566. doi:10.1093/carcin/bgq143. PMC 4647642. PMID 20622002.
- Boesch-Saadatmandi C, Loboda A, Wagner AE, Stachurska A, Jozkowicz A, Dulak J, Döring F, Wolffram S, Rimbach G (Mar 2011). "Effect of quercetin and its metabolites isorhamnetin and quercetin-3-glucuronide on inflammatory gene expression: role of miR-155". The Journal of Nutritional Biochemistry. 22 (3): 293–299. doi:10.1016/j.jnutbio.2010.02.008. PMID 20579867.
- Liu J, van Mil A, Vrijsen K, Zhao J, Gao L, Metz CH, Goumans MJ, Doevendans PA, Sluijter JP (Jul 2011). "MicroRNA-155 prevents necrotic cell death in human cardiomyocyte progenitor cells via targeting RIP1". Journal of Cellular and Molecular Medicine. 15 (7): 1474–1482. doi:10.1111/j.1582-4934.2010.01104.x. PMC 3823192. PMID 20550618.
- Pogribny IP, Starlard-Davenport A, Tryndyak VP, Han T, Ross SA, Rusyn I, Beland FA (Oct 2010). "Difference in expression of hepatic microRNAs miR-29c, miR-34a, miR-155, and miR-200b is associated with strain-specific susceptibility to dietary nonalcoholic steatohepatitis in mice". Laboratory Investigation. 90 (10): 1437–1446. doi:10.1038/labinvest.2010.113. PMC 4281935. PMID 20548288.
- Zhang Y, Diao Z, Su L, Sun H, Li R, Cui H, Hu Y (May 2010). "MicroRNA-155 contributes to preeclampsia by down-regulating CYR61". American Journal of Obstetrics and Gynecology. 202 (5): 466.e1–7. doi:10.1016/j.ajog.2010.01.057. PMID 20452491.
- Hu YL, Fong S, Largman C, Shen WF (Sep 2010). "HOXA9 regulates miR-155 in hematopoietic cells". Nucleic Acids Research. 38 (16): 5472–5478. doi:10.1093/nar/gkq337. PMC 2938212. PMID 20444872.
- McCoy CE, Sheedy FJ, Qualls JE, Doyle SL, Quinn SR, Murray PJ, O'Neill LA (Jul 2010). "IL-10 inhibits miR-155 induction by toll-like receptors". The Journal of Biological Chemistry. 285 (27): 20492–20498. doi:10.1074/jbc.M110.102111. PMC 2898307. PMID 20435894.
- Yin Q, Wang X, Fewell C, Cameron J, Zhu H, Baddoo M, Lin Z, Flemington EK (Jul 2010). "MicroRNA miR-155 inhibits bone morphogenetic protein (BMP) signaling and BMP-mediated Epstein-Barr virus reactivation". Journal of Virology. 84 (13): 6318–6327. doi:10.1128/JVI.00635-10. PMC 2903268. PMID 20427544.
- Zhu J, Hu XQ, Guo GL, Zhang Y, Wang OC, You J, Huang QD, Zhang XH (Feb 2010). "[Expression and its clinical significance of miR-155 in human primary breast cancer]". Zhonghua Wai Ke Za Zhi [Chinese Journal of Surgery]. 48 (3): 205–208. PMID 20388420.
- Kong W, He L, Coppola M, Guo J, Esposito NN, Coppola D, Cheng JQ (Jun 2010). "MicroRNA-155 regulates cell survival, growth, and chemosensitivity by targeting FOXO3a in breast cancer". The Journal of Biological Chemistry. 285 (23): 17869–17879. doi:10.1074/jbc.M110.101055. PMC 2878550. PMID 20371610.
- Xia QS, Ishigaki Y, Sun L, Chen R, Motoo Y (Jan 2010). "[Effect of anti-cancer drugs on the expression of BIC/miR-155 in human pancreatic cancer PANC-1 cells]". Zhonghua Yi Xue Za Zhi. 90 (2): 123–127. PMID 20356498.
- Jiang S, Zhang HW, Lu MH, He XH, Li Y, Gu H, Liu MF, Wang ED (Apr 2010). "MicroRNA-155 functions as an OncomiR in breast cancer by targeting the suppressor of cytokine signaling 1 gene". Cancer Research. 70 (8): 3119–3127. doi:10.1158/0008-5472.CAN-09-4250. PMID 20354188.
- Ryu JK, Hong SM, Karikari CA, Hruban RH, Goggins MG, Maitra A (2010). "Aberrant MicroRNA-155 expression is an early event in the multistep progression of pancreatic adenocarcinoma". Pancreatology. 10 (1): 66–73. doi:10.1159/000231984. PMC 2865485. PMID 20332664.
- Fabani MM, Abreu-Goodger C, Williams D, Lyons PA, Torres AG, Smith KG, Enright AJ, Gait MJ, Vigorito E (Jul 2010). "Efficient inhibition of miR-155 function in vivo by peptide nucleic acids". Nucleic Acids Research. 38 (13): 4466–4475. doi:10.1093/nar/gkq160. PMC 2910044. PMID 20223773.
- Tang B, Xiao B, Liu Z, Li N, Zhu ED, Li BS, Xie QH, Zhuang Y, Zou QM, Mao XH (Apr 2010). "Identification of MyD88 as a novel target of miR-155, involved in negative regulation of Helicobacter pylori-induced inflammation". FEBS Letters. 584 (8): 1481–1486. doi:10.1016/j.febslet.2010.02.063. PMID 20219467. S2CID 41984991.
- Fassi Fehri L, Koch M, Belogolova E, Khalil H, Bolz C, Kalali B, Mollenkopf HJ, Beigier-Bompadre M, Karlas A, Schneider T, Churin Y, Gerhard M, Meyer TF (2010). "Helicobacter pylori induces miR-155 in T cells in a cAMP-Foxp3-dependent manner". PLOS ONE. 5 (3): e9500. Bibcode:2010PLoSO...5.9500F. doi:10.1371/journal.pone.0009500. PMC 2830477. PMID 20209161.
- Sidorkiewicz M, Grek M, Jozwiak B, Majda-Stanislawska E, Piekarska A, Bartkowiak J (2010). "Expression of microRNA-155 precursor in peripheral blood mononuclear cells from Hepatitis C patients after antiviral treatment". Acta Virologica. 54 (1): 75–78. doi:10.4149/av_2010_01_75. PMID 20201617.
- Gueta K, Molotski N, Gerchikov N, Mor E, Savion S, Fein A, Toder V, Shomron N, Torchinsky A (2010). "Teratogen-induced alterations in microRNA-34, microRNA-125b and microRNA-155 expression: correlation with embryonic p53 genotype and limb phenotype". BMC Developmental Biology. 10: 20. doi:10.1186/1471-213X-10-20. PMC 2841584. PMID 20170545.
- Rai D, Kim SW, McKeller MR, Dahia PL, Aguiar RC (Feb 2010). "Targeting of SMAD5 links microRNA-155 to the TGF-beta pathway and lymphomagenesis". Proceedings of the National Academy of Sciences of the United States of America. 107 (7): 3111–3116. Bibcode:2010PNAS..107.3111R. doi:10.1073/pnas.0910667107. PMC 2840369. PMID 20133617.
- Cremer TJ, Ravneberg DH, Clay CD, Piper-Hunter MG, Marsh CB, Elton TS, Gunn JS, Amer A, Kanneganti TD, Schlesinger LS, Butchar JP, Tridandapani S (2009). "MiR-155 induction by F. novicida but not the virulent F. tularensis results in SHIP down-regulation and enhanced pro-inflammatory cytokine response". PLOS ONE. 4 (12): e8508. Bibcode:2009PLoSO...4.8508C. doi:10.1371/journal.pone.0008508. PMC 2794384. PMID 20041145.
- Forrest AR, Kanamori-Katayama M, Tomaru Y, Lassmann T, Ninomiya N, Takahashi Y, de Hoon MJ, Kubosaki A, Kaiho A, Suzuki M, Yasuda J, Kawai J, Hayashizaki Y, Hume DA, Suzuki H (Feb 2010). "Induction of microRNAs, mir-155, mir-222, mir-424 and mir-503, promotes monocytic differentiation through combinatorial regulation". Leukemia. 24 (2): 460–466. arXiv:1007.2689. doi:10.1038/leu.2009.246. PMID 19956200. S2CID 2333658.
- Pedersen IM, Otero D, Kao E, Miletic AV, Hother C, Ralfkiaer E, Rickert RC, Gronbaek K, David M (Aug 2009). "Onco-miR-155 targets SHIP1 to promote TNFalpha-dependent growth of B cell lymphomas". EMBO Molecular Medicine. 1 (5): 288–295. doi:10.1002/emmm.200900028. PMC 2771872. PMID 19890474.
- He M, Xu Z, Ding T, Kuang DM, Zheng L (Oct 2009). "MicroRNA-155 regulates inflammatory cytokine production in tumor-associated macrophages via targeting C/EBPbeta". Cellular & Molecular Immunology. 6 (5): 343–352. doi:10.1038/cmi.2009.45. PMC 4003217. PMID 19887047.
- Tili E, Croce CM, Michaille JJ (2009). "miR-155: on the crosstalk between inflammation and cancer". International Reviews of Immunology. 28 (5): 264–284. doi:10.1080/08830180903093796. PMID 19811312. S2CID 205589961.
- Stahl HF, Fauti T, Ullrich N, Bopp T, Kubach J, Rust W, Labhart P, Alexiadis V, Becker C, Hafner M, Weith A, Lenter MC, Jonuleit H, Schmitt E, Mennerich D (2009). "miR-155 inhibition sensitizes CD4+ Th cells for TREG mediated suppression". PLOS ONE. 4 (9): e7158. Bibcode:2009PLoSO...4.7158S. doi:10.1371/journal.pone.0007158. PMC 2743997. PMID 19777054.
- Bolisetty MT, Dy G, Tam W, Beemon KL (Dec 2009). "Reticuloendotheliosis virus strain T induces miR-155, which targets JARID2 and promotes cell survival". Journal of Virology. 83 (23): 12009–12017. doi:10.1128/JVI.01182-09. PMC 2786729. PMID 19759154.
- Wang B, Majumder S, Nuovo G, Kutay H, Volinia S, Patel T, Schmittgen TD, Croce C, Ghoshal K, Jacob ST (Oct 2009). "Role of microRNA-155 at early stages of hepatocarcinogenesis induced by choline-deficient and amino acid-defined diet in C57BL/6 mice". Hepatology. 50 (4): 1152–1161. doi:10.1002/hep.23100. PMC 2757532. PMID 19711427.
- Pottier N, Maurin T, Chevalier B, Puisségur MP, Lebrigand K, Robbe-Sermesant K, Bertero T, Lino Cardenas CL, Courcot E, Rios G, Fourre S, Lo-Guidice JM, Marcet B, Cardinaud B, Barbry P, Mari B (2009). "Identification of keratinocyte growth factor as a target of microRNA-155 in lung fibroblasts: implication in epithelial-mesenchymal interactions". PLOS ONE. 4 (8): e6718. Bibcode:2009PLoSO...4.6718P. doi:10.1371/journal.pone.0006718. PMC 2726943. PMID 19701459.
- Xiao B, Liu Z, Li BS, Tang B, Li W, Guo G, Shi Y, Wang F, Wu Y, Tong WD, Guo H, Mao XH, Zou QM (Sep 2009). "Induction of microRNA-155 during Helicobacter pylori infection and its negative regulatory role in the inflammatory response". The Journal of Infectious Diseases. 200 (6): 916–925. doi:10.1086/605443. PMID 19650740.
- Worm J, Stenvang J, Petri A, Frederiksen KS, Obad S, Elmén J, Hedtjärn M, Straarup EM, Hansen JB, Kauppinen S (Sep 2009). "Silencing of microRNA-155 in mice during acute inflammatory response leads to derepression of c/ebp Beta and down-regulation of G-CSF". Nucleic Acids Research. 37 (17): 5784–5792. doi:10.1093/nar/gkp577. PMC 2761263. PMID 19596814.
- Costinean S, Sandhu SK, Pedersen IM, Tili E, Trotta R, Perrotti D, Ciarlariello D, Neviani P, Harb J, Kauffman LR, Shidham A, Croce CM (Aug 2009). "Src homology 2 domain-containing inositol-5-phosphatase and CCAAT enhancer-binding protein beta are targeted by miR-155 in B cells of Emicro-MiR-155 transgenic mice". Blood. 114 (7): 1374–1382. doi:10.1182/blood-2009-05-220814. PMC 2727407. PMID 19520806.
- Ruggiero T, Trabucchi M, De Santa F, Zupo S, Harfe BD, McManus MT, Rosenfeld MG, Briata P, Gherzi R (Sep 2009). "LPS induces KH-type splicing regulatory protein-dependent processing of microRNA-155 precursors in macrophages". FASEB Journal. 23 (9): 2898–2908. doi:10.1096/fj.09-131342. PMID 19423639. S2CID 588328.
- Martinez-Nunez RT, Louafi F, Friedmann PS, Sanchez-Elsner T (Jun 2009). "MicroRNA-155 modulates the pathogen binding ability of dendritic cells (DCs) by down-regulation of DC-specific intercellular adhesion molecule-3 grabbing non-integrin (DC-SIGN)". The Journal of Biological Chemistry. 284 (24): 16334–16342. doi:10.1074/jbc.M109.011601. PMC 2713543. PMID 19386588.
- O'Connell RM, Chaudhuri AA, Rao DS, Baltimore D (Apr 2009). "Inositol phosphatase SHIP1 is a primary target of miR-155". Proceedings of the National Academy of Sciences of the United States of America. 106 (17): 7113–7118. Bibcode:2009PNAS..106.7113O. doi:10.1073/pnas.0902636106. PMC 2678424. PMID 19359473.
- Kohlhaas S, Garden OA, Scudamore C, Turner M, Okkenhaug K, Vigorito E (Mar 2009). "Cutting edge: the Foxp3 target miR-155 contributes to the development of regulatory T cells". Journal of Immunology. 182 (5): 2578–2582. doi:10.4049/jimmunol.0803162. PMID 19234151.
- Ceppi M, Pereira PM, Dunand-Sauthier I, Barras E, Reith W, Santos MA, Pierre P (Feb 2009). "MicroRNA-155 modulates the interleukin-1 signaling pathway in activated human monocyte-derived dendritic cells". Proceedings of the National Academy of Sciences of the United States of America. 106 (8): 2735–2740. Bibcode:2009PNAS..106.2735C. doi:10.1073/pnas.0811073106. PMC 2650335. PMID 19193853.
- Habbe N, Koorstra JB, Mendell JT, Offerhaus GJ, Ryu JK, Feldmann G, Mullendore ME, Goggins MG, Hong SM, Maitra A (Feb 2009). "MicroRNA miR-155 is a biomarker of early pancreatic neoplasia". Cancer Biology & Therapy. 8 (4): 340–346. doi:10.4161/cbt.8.4.7338. PMC 2692997. PMID 19106647.
- Jung I, Aguiar RC (Jan 2009). "MicroRNA-155 expression and outcome in diffuse large B-cell lymphoma". British Journal of Haematology. 144 (1): 138–140. doi:10.1111/j.1365-2141.2008.07424.x. PMC 4654117. PMID 19016736.
- Romania P, Lulli V, Pelosi E, Biffoni M, Peschle C, Marziali G (Nov 2008). "MicroRNA 155 modulates megakaryopoiesis at progenitor and precursor level by targeting Ets-1 and Meis1 transcription factors". British Journal of Haematology. 143 (4): 570–580. doi:10.1111/j.1365-2141.2008.07382.x. PMID 18950466. S2CID 9025761.
- Zhao Y, Yao Y, Xu H, Lambeth L, Smith LP, Kgosana L, Wang X, Nair V (Jan 2009). "A functional MicroRNA-155 ortholog encoded by the oncogenic Marek's disease virus". Journal of Virology. 83 (1): 489–492. doi:10.1128/JVI.01166-08. PMC 2612317. PMID 18945769.
- Gatto G, Rossi A, Rossi D, Kroening S, Bonatti S, Mallardo M (Nov 2008). "Epstein-Barr virus latent membrane protein 1 trans-activates miR-155 transcription through the NF-kappaB pathway". Nucleic Acids Research. 36 (20): 6608–6619. doi:10.1093/nar/gkn666. PMC 2582607. PMID 18940871.
- Rahadiani N, Takakuwa T, Tresnasari K, Morii E, Aozasa K (Dec 2008). "Latent membrane protein-1 of Epstein-Barr virus induces the expression of B-cell integration cluster, a precursor form of microRNA-155, in B lymphoma cell lines". Biochemical and Biophysical Research Communications. 377 (2): 579–583. doi:10.1016/j.bbrc.2008.10.007. PMID 18926796.
- Kong W, Yang H, He L, Zhao JJ, Coppola D, Dalton WS, Cheng JQ (Nov 2008). "MicroRNA-155 is regulated by the transforming growth factor beta/Smad pathway and contributes to epithelial cell plasticity by targeting RhoA". Molecular and Cellular Biology. 28 (22): 6773–6784. doi:10.1128/MCB.00941-08. PMC 2573297. PMID 18794355.
- Lu F, Weidmer A, Liu CG, Volinia S, Croce CM, Lieberman PM (Nov 2008). "Epstein-Barr virus-induced miR-155 attenuates NF-kappaB signaling and stabilizes latent virus persistence". Journal of Virology. 82 (21): 10436–10443. doi:10.1128/JVI.00752-08. PMC 2573162. PMID 18753206.
- Dorsett Y, McBride KM, Jankovic M, Gazumyan A, Thai TH, Robbiani DF, Di Virgilio M, Reina San-Martin B, Heidkamp G, Schwickert TA, Eisenreich T, Rajewsky K, Nussenzweig MC (May 2008). "MicroRNA-155 suppresses activation-induced cytidine deaminase-mediated Myc-Igh translocation". Immunity. 28 (5): 630–638. doi:10.1016/j.immuni.2008.04.002. PMC 2713656. PMID 18455451.
- Teng G, Hakimpour P, Landgraf P, Rice A, Tuschl T, Casellas R, Papavasiliou FN (May 2008). "MicroRNA-155 is a negative regulator of activation-induced cytidine deaminase". Immunity. 28 (5): 621–629. doi:10.1016/j.immuni.2008.03.015. PMC 2430982. PMID 18450484.
- Yin Q, McBride J, Fewell C, Lacey M, Wang X, Lin Z, Cameron J, Flemington EK (Jun 2008). "MicroRNA-155 is an Epstein-Barr virus-induced gene that modulates Epstein-Barr virus-regulated gene expression pathways". Journal of Virology. 82 (11): 5295–5306. doi:10.1128/JVI.02380-07. PMC 2395216. PMID 18367535.
- Zhang T, Nie K, Tam W (Sep 2008). "BIC is processed efficiently to microRNA-155 in Burkitt lymphoma cells". Leukemia. 22 (9): 1795–1797. doi:10.1038/leu.2008.62. PMID 18354490.
- Wang M, Tan LP, Dijkstra MK, van Lom K, Robertus JL, Harms G, Blokzijl T, Kooistra K, van T'veer MB, Rosati S, Visser L, Jongen-Lavrencic M, Kluin PM, van den Berg A (May 2008). "miRNA analysis in B-cell chronic lymphocytic leukaemia: proliferation centres characterized by low miR-150 and high BIC/miR-155 expression". The Journal of Pathology. 215 (1): 13–20. doi:10.1002/path.2333. PMID 18348159. S2CID 29470560.
- O'Connell RM, Rao DS, Chaudhuri AA, Boldin MP, Taganov KD, Nicoll J, Paquette RL, Baltimore D (Mar 2008). "Sustained expression of microRNA-155 in hematopoietic stem cells causes a myeloproliferative disorder". The Journal of Experimental Medicine. 205 (3): 585–594. doi:10.1084/jem.20072108. PMC 2275382. PMID 18299402.
- Rai D, Karanti S, Jung I, Dahia PL, Aguiar RC (Feb 2008). "Coordinated expression of microRNA-155 and predicted target genes in diffuse large B-cell lymphoma". Cancer Genetics and Cytogenetics. 181 (1): 8–15. doi:10.1016/j.cancergencyto.2007.10.008. PMC 2276854. PMID 18262046.
- Gottwein E, Mukherjee N, Sachse C, Frenzel C, Majoros WH, Chi JT, Braich R, Manoharan M, Soutschek J, Ohler U, Cullen BR (Dec 2007). "A viral microRNA functions as an orthologue of cellular miR-155". Nature. 450 (7172): 1096–1099. Bibcode:2007Natur.450.1096G. doi:10.1038/nature05992. PMC 2614920. PMID 18075594.
- Vigorito E, Perks KL, Abreu-Goodger C, Bunting S, Xiang Z, Kohlhaas S, Das PP, Miska EA, Rodriguez A, Bradley A, Smith KG, Rada C, Enright AJ, Toellner KM, Maclennan IC, Turner M (Dec 2007). "microRNA-155 regulates the generation of immunoglobulin class-switched plasma cells". Immunity. 27 (6): 847–859. doi:10.1016/j.immuni.2007.10.009. PMC 4135426. PMID 18055230.
- Yin Q, Wang X, McBride J, Fewell C, Flemington E (Feb 2008). "B-cell receptor activation induces BIC/miR-155 expression through a conserved AP-1 element". The Journal of Biological Chemistry. 283 (5): 2654–2662. doi:10.1074/jbc.M708218200. PMC 2810639. PMID 18048365.
- Tili E, Michaille JJ, Cimino A, Costinean S, Dumitru CD, Adair B, Fabbri M, Alder H, Liu CG, Calin GA, Croce CM (Oct 2007). "Modulation of miR-155 and miR-125b levels following lipopolysaccharide/TNF-alpha stimulation and their possible roles in regulating the response to endotoxin shock". Journal of Immunology. 179 (8): 5082–5089. doi:10.4049/jimmunol.179.8.5082. PMID 17911593.
- Gironella M, Seux M, Xie MJ, Cano C, Tomasini R, Gommeaux J, Garcia S, Nowak J, Yeung ML, Jeang KT, Chaix A, Fazli L, Motoo Y, Wang Q, Rocchi P, Russo A, Gleave M, Dagorn JC, Iovanna JL, Carrier A, Pébusque MJ, Dusetti NJ (Oct 2007). "Tumor protein 53-induced nuclear protein 1 expression is repressed by miR-155, and its restoration inhibits pancreatic tumor development". Proceedings of the National Academy of Sciences of the United States of America. 104 (41): 16170–16175. Bibcode:2007PNAS..10416170G. doi:10.1073/pnas.0703942104. PMC 2042180. PMID 17911264.
- Skalsky RL, Samols MA, Plaisance KB, Boss IW, Riva A, Lopez MC, Baker HV, Renne R (Dec 2007). "Kaposi's sarcoma-associated herpesvirus encodes an ortholog of miR-155". Journal of Virology. 81 (23): 12836–12845. doi:10.1128/JVI.01804-07. PMC 2169101. PMID 17881434.
- Sethupathy P, Borel C, Gagnebin M, Grant GR, Deutsch S, Elton TS, Hatzigeorgiou AG, Antonarakis SE (Aug 2007). "Human microRNA-155 on chromosome 21 differentially interacts with its polymorphic target in the AGTR1 3′ untranslated region: a mechanism for functional single-nucleotide polymorphisms related to phenotypes". American Journal of Human Genetics. 81 (2): 405–413. doi:10.1086/519979. PMC 1950808. PMID 17668390.
- Martin MM, Buckenberger JA, Jiang J, Malana GE, Nuovo GJ, Chotani M, Feldman DS, Schmittgen TD, Elton TS (Aug 2007). "The human angiotensin II type 1 receptor +1166 A/C polymorphism attenuates microRNA-155 binding". The Journal of Biological Chemistry. 282 (33): 24262–24269. doi:10.1074/jbc.M701050200. PMC 2413065. PMID 17588946.
- Rodriguez A, Vigorito E, Clare S, Warren MV, Couttet P, Soond DR, van Dongen S, Grocock RJ, Das PP, Miska EA, Vetrie D, Okkenhaug K, Enright AJ, Dougan G, Turner M, Bradley A (Apr 2007). "Requirement of bic/microRNA-155 for normal immune function". Science. 316 (5824): 608–611. Bibcode:2007Sci...316..608R. doi:10.1126/science.1139253. PMC 2610435. PMID 17463290.
- Thai TH, Calado DP, Casola S, Ansel KM, Xiao C, Xue Y, Murphy A, Frendewey D, Valenzuela D, Kutok JL, Schmidt-Supprian M, Rajewsky N, Yancopoulos G, Rao A, Rajewsky K (Apr 2007). "Regulation of the germinal center response by microRNA-155". Science. 316 (5824): 604–608. Bibcode:2007Sci...316..604T. doi:10.1126/science.1141229. PMID 17463289. S2CID 8174458.
- O'Connell RM, Taganov KD, Boldin MP, Cheng G, Baltimore D (Jan 2007). "MicroRNA-155 is induced during the macrophage inflammatory response". Proceedings of the National Academy of Sciences of the United States of America. 104 (5): 1604–1609. Bibcode:2007PNAS..104.1604O. doi:10.1073/pnas.0610731104. PMC 1780072. PMID 17242365.
- Martin MM, Lee EJ, Buckenberger JA, Schmittgen TD, Elton TS (Jul 2006). "MicroRNA-155 regulates human angiotensin II type 1 receptor expression in fibroblasts". The Journal of Biological Chemistry. 281 (27): 18277–18284. doi:10.1074/jbc.M601496200. PMID 16675453.
- Chung KH, Hart CC, Al-Bassam S, Avery A, Taylor J, Patel PD, Vojtek AB, Turner DL (2006). "Polycistronic RNA polymerase II expression vectors for RNA interference based on BIC/miR-155". Nucleic Acids Research. 34 (7): e53. doi:10.1093/nar/gkl143. PMC 1435982. PMID 16614444.
- Tam W, Dahlberg JE (Feb 2006). "miR-155/BIC as an oncogenic microRNA". Genes, Chromosomes & Cancer. 45 (2): 211–212. doi:10.1002/gcc.20282. PMID 16252262. S2CID 8513721.
- Kluiver J, Haralambieva E, de Jong D, Blokzijl T, Jacobs S, Kroesen BJ, Poppema S, van den Berg A (Feb 2006). "Lack of BIC and microRNA miR-155 expression in primary cases of Burkitt lymphoma". Genes, Chromosomes & Cancer. 45 (2): 147–153. doi:10.1002/gcc.20273. PMID 16235244. S2CID 16897980.
- Jiang J, Lee EJ, Schmittgen TD (Jan 2006). "Increased expression of microRNA-155 in Epstein-Barr virus transformed lymphoblastoid cell lines". Genes, Chromosomes & Cancer. 45 (1): 103–106. doi:10.1002/gcc.20264. PMID 16175574. S2CID 19138209.
- Kluiver J, Poppema S, de Jong D, Blokzijl T, Harms G, Jacobs S, Kroesen BJ, van den Berg A (Oct 2005). "BIC and miR-155 are highly expressed in Hodgkin, primary mediastinal and diffuse large B cell lymphomas". The Journal of Pathology. 207 (2): 243–249. doi:10.1002/path.1825. PMID 16041695. S2CID 14329069.
- Eis PS, Tam W, Sun L, Chadburn A, Li Z, Gomez MF, Lund E, Dahlberg JE (Mar 2005). "Accumulation of miR-155 and BIC RNA in human B cell lymphomas". Proceedings of the National Academy of Sciences of the United States of America. 102 (10): 3627–3632. Bibcode:2005PNAS..102.3627E. doi:10.1073/pnas.0500613102. PMC 552785. PMID 15738415.
- Metzler M, Wilda M, Busch K, Viehmann S, Borkhardt A (Feb 2004). "High expression of precursor microRNA-155/BIC RNA in children with Burkitt lymphoma". Genes, Chromosomes & Cancer. 39 (2): 167–169. doi:10.1002/gcc.10316. PMID 14695998. S2CID 10009892.
- Xu, Yanan; Wu, Junhua; Yuan, Xiaoqi; Liu, Wenyuan; Pan, Jiewen; Xu, Binbin (2021). "MicroRNA-155 contributes to host immunity against Toxoplasma gondii". Parasite. 28: 83. doi:10.1051/parasite/2021082. PMC 8672677. PMID 34907898.

외부 링크