I형 topoisomerase
Type I topoisomerase이 기사는 실제 촉매영역에 대한 pfam 박스에 대한 정보가 누락되었다.; BRE(Break-rejining 효소) 슈퍼 패밀리의 멤버; BRE와 ECOD당 AraC homeobox 사이의 링크.(2021년 2월) |
| DNA Topoisomerase I, N-단자(비촉매), 바이러스 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
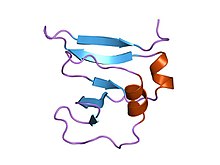 아미노 단자 9kda의 바이러스 dna topoisomerase i 잔류물 1-77, 잔류물에 대한 실험 전자 밀도 1-77 | |||||||||
| 식별자 | |||||||||
| 기호 | VirDNA-topo-I_N | ||||||||
| Pfam | PF09266 | ||||||||
| 인터프로 | IPR015346 | ||||||||
| SCOP2 | 1vcc / SCOPe / SUPFAM | ||||||||
| |||||||||
분자생물학 Type I topoisomeras는 두 가닥의 이중 가닥 DNA 중 하나를 자르고 가닥을 이완시키고 가닥을 다시 새로 만드는 효소다.그들은 구조적으로 그리고 기계적으로 구별되는 두 가지 토포아세라아제, 즉 유형 IA와 유형 IB로 더 세분화된다.
- IA topoisomerases 타입 IA topoisomeras는 원형 DNA 가닥의 연결 수를 엄격히 1의 단위로 변경한다.
- IB형 topoisomeras는 1 (n)의 배수로 연결 번호를 변경한다.
역사적으로 IA형 토포아세미아제는 원핵산 토포아세아제 I형, IB형 토포아세아제는 진핵산 토포아세아제라고 한다.그러나 이러한 구분은 IA형 및 IB형 토포아세머라제가 모든 생명체 영역에 존재하기 때문에 더 이상 적용되지 않는다.
기능적으로, 이러한 하위 클래스들은 매우 전문화된 기능을 수행한다.프로카리오틱 토포아세머레이스 I(topo IA)는 음성의 슈퍼코일 DNA만 완화할 수 있고, 진핵 토포아세머레이스 I(topo IB)는 양성 슈퍼코일을 도입해 DNA 복제 후 딸 염색체의 DNA를 분리하고 DNA를 완화할 수 있다.
함수
이 효소들은 여러 가지 기능을 가지고 있다: 전사 및 DNA 복제 중에 DNA 슈퍼코일을 제거하는 것, 재결합 중에 가닥이 깨지는 것, 염색체 결로증, 유사시 얽힌 DNA를 분해하는 것.[1][2]
구조
이 영역은 베타(2)-알파-베타-알파-베타(2) 접힌 것으로 가정하며, 가닥 베타2와 베타3 사이에 왼손 교차한다.네 개의 알파헬리크로 둘러싸인 네 개의 십자형 베타 가닥이 있으며, 로스만 접이식으로[3] 배열되어 있다.
메커니즘
제1형 토포아세라제는 ATP 독립 효소(역방향 자이스 제외)로, 유형 IA(박테리아 및 고고학적 토포아세라제 IA, 토포아세라제 III 및 역방향 자이스)와 IB(eukaryotoosomerase I, 토포아세라제 V) 등 구조와 반응 메커니즘에 따라 세분할 수 있다.이러한 효소는 DNA에 양의 슈퍼코일을 도입할 수 있는 역방향 자레이즈를 제외하고 양성 및/또는 부정적으로 코팅된 DNA를 이완시키는 데 주로 책임이 있다.
DNA topoisomerases는 두 DNA 가닥 사이의 위상학적 연결 수(즉, 초신성 회전 수 변경)를 조절하여 일시적인 단일 가닥 또는 이중 가닥을 강직하게 하고 가닥을 서로 교차시킨 다음 그 틈을 다시 봉합한다.[4]
반
DNA topoisomeras는 두 종류로 나뉜다: 타입 I 효소(EC; topoisomeras I, III, V)는 단일 가닥 DNA를, 타입 II 효소(EC; topoisomeras II, IV, VI)는 이중 가닥 DNA를 깨뜨린다.[5]
유형 IA 토포아세머라제
소개
역사적으로 원핵생물에서 발견되는 것으로 알려진 IA형 토포아세머라아제는 DNA에 단일 파단을 일으키고 그 파동을 통해 두 번째 가닥이나 이중 가닥을 통과시킨다.이 스트랜드 패스 메커니즘은 IIA topoisomerases 유형과 몇 가지 특징을 공유한다.둘 다 5'인산염 중간을 형성하며, 그 작업을 수행하기 위해 분절 금속 이온이 필요하다.타입 II topoisomeras와는 달리, 타입 IA topoisomeras는 그 작업을 하기 위해 에너지를 사용하지 않는다(역방향 자이스(역방향 자이스)
구조
유형 IA topoisomeras에는 여러 도메인이 있으며, 종종 Domain 1-4로 번호가 매겨진다.도메인 I에는 Toprim 도메인(마그네슘 이온을 조정하는 것으로 알려진 로스만 접이식), 도메인 IV 및 도메인 III가 각각 나선-회전-헬릭스(HTH) 도메인으로 구성되며, 촉매 타이로신은 도메인 III의 HTH에 존재한다.도메인 II는 도메인 III와 IV 사이의 유연한 브리지다.IA topoisomerase 유형의 구조는 잠금장치와 유사하며, 구조물의 하단에 도메인 I, III, IV가 놓여 있다.[6]단일 가닥 DNA[7](pdb id = 1I7D)에 바인딩된 topo III(아래 참조)의 구조는 DNA에 대해 HTH와 Toprime 도메인이 어떻게 조정되는지를 보여준다.
IA topoisomerase 변형 유형
IA topoisomerase에는 여러 가지 변형이 있는데, 주 코어에 부착된 부속물("topo-fold"라고도 함)에 따라 다르다.이 하위 클래스의 멤버는 topo I, topo III(추가 아연 바인딩 모티브 포함) 및 역방향 자레이즈를 포함한다.역자이스는 특히 Rho 전사 인자의 헬리카제 유사 도메인을 닮은 ATPase 도메인이 부착되어 있어 흥미롭다(역자이스 구조는 로드리게스와 스톡, EMBO J 2002가 해결했다).이 효소는 ATP의 가수분해를 이용하여 양성 슈퍼코일과 오버인드 DNA를 도입하는데, 이는 역방향 자이가 존재하는 것으로 알려진 초열성 물질에서 매력적인 특징이다.로드리게스와 스톡은 양성 슈퍼코일의 도입에 ATP의 가수분해를 전달하는 데 관여하는 "래치"를 식별하기 위한 추가 작업을 했다.
Topo III 변종도 마찬가지로 매우 흥미롭다. 왜냐하면 그것은 단일 가닥의 DNA를 결합하는 것으로 생각되는 아연 결합 모티브를 가지고 있기 때문이다.Topo III는 재조합 시 BLM(Bloom Syndrome용) 헬리코아제와 관련된 것으로 확인되었다.
메커니즘
타입 IA topoisomerases는 단일 게이트를 사용하여 Strand-passage 메커니즘을 통해 작동한다(타입 II topoisomeras와는 대조적으로).첫째, 단일 가닥의 DNA는 도메인 III와 I를 결합한다.촉매 타이로신은 DNA 백본을 분해하여 과도성 5'인산염 중간을 만든다.그런 다음 도메인 II를 힌지로 사용하여 단절된 부분을 분리하고, 두 번째 이중 또는 가닥의 DNA를 통과한다.도메인 3과 내가 닫으면 DNA가 다시 제거된다.
IB형 토포아세메라제
소개
IA형 토포아세메라아제와는 대조적으로 1B형 토포아세메라제(Topoisomerase)는 방해를 받는 회전 메커니즘을 통해 오버워드와 언트라우드(양극 또는 음극 슈퍼코일링이라고도 함) DNA의 문제를 해결한다.수정 구조, 생화학, 단일 분자 실험은 일반적인 메커니즘에 기여했다.이 효소는 먼저 DNA를 감싸고 3'의 인산염 중간을 만든다.그런 다음 5' 끝은 자유롭게 회전하여 다른 가닥을 중심으로 비틀어 토포아세머레이즈가 끊어진 가닥을 재결합할 때까지 DNA를 이완시킨다.
구조
DNA에 바인딩된 topo IB의 구조가 해결되었다(pdb id = 1A36).Topo IB는 NTD, 캡팅 로브, 촉매 로브, C-단자 영역으로 구성되어 있다.캡팅 로브와 촉매 로브는 DNA를 감싸고 있다.
메커니즘
이완은 활성 과정이 아니며 (ATP 형태의) 에너지가 닉싱 또는 레깅 단계 동안 소비되지 않는다. 이는 효소의 활성 부위에서 인산염 DNA 백본과 티로신 잔여물 사이의 반응이 단지 한 인산염화제 결합을 다른 인산염화제 결합으로 대체하기 때문이다.Topoisomerase는 또한 DNA의 코팅이 해제되는 동안 ATP를 사용하지 않는다. 오히려, DNA에 존재하는 토크는 코팅되지 않은 것을 구동하고 평균적으로 활력 있게 내리막길을 따라간다.최근의 단일 분자 실험은 앞서 제안했던 벌크-플라스미드 이완 실험이 어떤 것을 확인시켜 주었는데, 이것은 DNA의 언코일링이 토크로 구동되어 종교화가 일어날 때까지 진행된다는 것이다.어떤 데이터도 Topo IB가 특정 수의 슈퍼코일을 제거한 후 종교화를 유발하는 메커니즘을 가지고 있는 한 회전하는 것을 "제어"한다고 제안하지 않는다.반대로 단분자 실험은 종교화가 무작위 과정이며 회전하는 5'-OH 끝이 효소 연계 3'-end의 부착 부위와 근접하게 다가올 때마다 어느 정도 발생할 확률을 가지고 있음을 시사한다.
타입 IB topoisomeras는 원래 진핵생물과 바이러스에서 확인되었다.바이러스성 topo I는 DNA를 시퀀스별 방식으로 결합하기 때문에 독특하다.
이 잘 학습된 유형 1B topoisomerase에 대한 자세한 내용은 TOP1 기사를 참조하십시오.
유형 IC 토포아세머라제
세 번째 유형의 토포아소메라아제인 Topoo V가 고고학자인 Methanophyrus 칸들리에서 발견되었다.Topo V는 창립 멤버로, 지금까지 IC topoisomerase 타입의 유일한 멤버로, 일부 저자들은 그것이 바이러스 기원을 가지고 있을 수 있다고 주장하지만.[8]탑오브이의 결정구조는 해결되었다.[9]Type IC topoisomerases는 Type IB topoisomerases[10](pdb ID = 2CSB 및 2CSD)와 마찬가지로 제어된 회전 메커니즘을 통해 작동하지만 접히는 부분이 독특하다.
중간자
모든 토포아세머라이즈는 효소의 촉매 타이로신과 DNA 백본의 가위 인광 사이에서 인광성 중간체를 형성한다.
- 타입 IA topoisomerases는 촉매 타이로신과 5'-인산염 사이에 공밸런트 연결을 형성한다.
- IB형 효소는 공발효소 3'-인산염 중간을 형성한다.
- IC형 토포아세머라아제는 공밸런트 3'-인스포티로신 중간을 형성한다.
이 중간은 이소제네제틱으로, 전방 갈라짐 반응과 후방 종교 반응이 모두 정력적으로 동일하다는 것을 의미한다.이와 같이, 이러한 반응을 실시하기 위해 외부 에너지원은 필요하지 않다.
억제
토포이아세라제는 DNA에 균열을 일으키기 때문에 효소를 억제하는 소분자 억제제의 표적이다.타입 1 topoisomerase는 이리노테칸, topotecan, hexylresorcinol, 캠토테신 등에 의해 억제된다.
인간 Topoisomerase형 IB 효소는 공밸런트 3'-인스포티로신 중간, Topoisomerase 1-cleavage-complex (Top1cc)를 형성한다.활성 이리노테칸 대사물인 SN-38은 Top1cc의 부분집합물인 구아닌 +1을 DNA 서열로 가둬서 작용한다.[11]이리노테리아에서 파생된 SN-38 분자는 Topoisomerase 유도 갈라짐 부위와 함께 있는 염기쌍에 쌓이고 Topoisomerase 1 효소를 독(불활성화)한다.[11]
세균 숙주인 대장균의 박테리오파지(페이지) T4 감염에 따라, 페이지 게놈은 박테리아 토포아세머레이스 I를 억제하는 유전자 제품(gp55.2)을 지정한다.[12]Gp55.2는 DNA를 결합하고 특히 topoisomerase I에 의해 부정적으로 슈퍼코팅된 DNA의 이완을 차단한다.이러한 억제는 최적의 페이징 수율을 보장하기 위해 감염 중 호스트 topoisomerase I 활동을 미묘하게 조절하기 위한 적응으로 보인다.
합성 치사율
합성 치사율은 두 개 이상의 유전자의 발현에 결함의 조합이 세포사망으로 이어지는 반면, 이들 유전자 중 단 한 개의 결핍은 그렇지 않을 때 발생한다.이러한 결함은 돌연변이, 후생적 변화 또는 유전자 발현 억제에 의해 발생할 수 있다.
Topoisomerase 1 억제제는 특정 DNA 수리 유전자의 발현이 부족하여 종합적으로 치명적이다.인간의 환자에서 부족한 DNA 수리 유전자는 WRN과[13] MRE11을 포함한다.[14]암과 관련된 임상 전 연구에서 부족한 DNA 수리 유전자는 ATM과[15] NDRG1을 포함한다.[16][17]
오토앤티비즈
I형 topoisomerase를 대상으로 하는 자동항체들은 scl-70 항체라고 불리며, sclorderma와 70 kD 추출 가능한 면역항체 활성 파편들이 항체의 다른 큰 (100-105 kD) 표적 topoisomerase 항원 (SCL-70 항원이라 함)[18]에서 얻을 수 있다.
참조
- ^ Wang JC (June 2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nat. Rev. Mol. Cell Biol. 3 (6): 430–40. doi:10.1038/nrm831. PMID 12042765. S2CID 205496065.
- ^ Champoux JJ (2001). "DNA topoisomerases: structure, function, and mechanism". Annu. Rev. Biochem. 70: 369–413. doi:10.1146/annurev.biochem.70.1.369. PMID 11395412.
- ^ Sharma A; Hanai R; Mondragón A (August 1994). "Crystal structure of the amino-terminal fragment of vaccinia virus DNA topoisomerase I at 1.6 A resolution". Structure. 2 (8): 767–77. doi:10.1016/s0969-2126(94)00077-8. PMID 7994576.
- ^ Roca J (April 1995). "The mechanisms of DNA topoisomerases". Trends Biochem. Sci. 20 (4): 156–60. doi:10.1016/s0968-0004(00)88993-8. PMID 7770916.
- ^ Gadelle D, Filée J, Buhler C, Forterre P (March 2003). "Phylogenomics of type II DNA topoisomerases". BioEssays. 25 (3): 232–42. doi:10.1002/bies.10245. PMID 12596227. S2CID 4642743.
- ^ Lima, C. D.; Wang, J. C.; Mondragón, A (1994). "Three-dimensional structure of the 67K N-terminal fragment of E. Coli DNA topoisomerase I". Nature. 367 (6459): 138–46. Bibcode:1994Natur.367..138L. doi:10.1038/367138a0. PMID 8114910. S2CID 4314431.
- ^ Changela, A; Digate, R. J.; Mondragón, A (2001). "Crystal structure of a complex of a type IA DNA topoisomerase with a single-stranded DNA molecule". Nature. 411 (6841): 1077–81. Bibcode:2001Natur.411.1077C. doi:10.1038/35082615. PMID 11429611. S2CID 4426078.
- ^ Forterre P (June 2006). "DNA topoisomerase V: a new fold of mysterious origin". Trends Biotechnol. 24 (6): 245–7. doi:10.1016/j.tibtech.2006.04.006. PMID 16650908.
- ^ Taneja B, Patel A, Slesarev A, Mondragón A (January 2006). "Structure of the N-terminal fragment of topoisomerase V reveals a new family of topoisomerases". EMBO J. 25 (2): 398–408. doi:10.1038/sj.emboj.7600922. PMC 1383508. PMID 16395333.
- ^ Taneja, B; Schnurr, B; Slesarev, A; Marko, J. F.; Mondragón, A (2007). "Topoisomerase V relaxes supercoiled DNA by a constrained swiveling mechanism". Proceedings of the National Academy of Sciences. 104 (37): 14670–5. Bibcode:2007PNAS..10414670T. doi:10.1073/pnas.0701989104. PMC 1976220. PMID 17804808.
- ^ a b Pommier Y (2013). "Drugging topoisomerases: lessons and challenges". ACS Chem. Biol. 8 (1): 82–95. doi:10.1021/cb300648v. PMC 3549721. PMID 23259582.
- ^ Mattenberger Y, Silva F, Belin D. 55.2, 페이지는 T4 ORFAN 유전자로 대장균 토포아세머레이스 I의 억제제를 암호화하고 페이지의 체력을 높인다.PLoS One. 2015년 4월 14일 (4):e0124309. doi: 10.1371/journal.pone.0124309.PMID: 25875362; PMCID: PMC4396842
- ^ Agrelo R, Cheng WH, Setien F, Ropero S, Espada J, Fraga MF, Herranz M, Paz MF, Sanchez-Cespedes M, Artiga MJ, Guerrero D, Castells A, von Kobbe C, Bohr VA, Esteller M (2006). "Epigenetic inactivation of the premature aging Werner syndrome gene in human cancer". Proc. Natl. Acad. Sci. U.S.A. 103 (23): 8822–7. Bibcode:2006PNAS..103.8822A. doi:10.1073/pnas.0600645103. PMC 1466544. PMID 16723399.
- ^ Pavelitz T, Renfro L, Foster NR, Caracol A, Welsch P, Lao VV, Grady WB, Niedzwiecki D, Saltz LB, Bertagnolli MM, Goldberg RM, Rabinovitch PS, Emond M, Monnat RJ, Maizels N (2014). "MRE11-deficiency associated with improved long-term disease free survival and overall survival in a subset of stage III colon cancer patients in randomized CALGB 89803 trial". PLOS ONE. 9 (10): e108483. Bibcode:2014PLoSO...9j8483P. doi:10.1371/journal.pone.0108483. PMC 4195600. PMID 25310185.
- ^ Subhash VV, Tan SH, Yeo MS, Yan FL, Peethala PC, Liem N, Krishnan V, Yong WP (2016). "ATM expression predicts Veliparib and Irinotecan sensitivity in gastric cancer by mediating P53 independent regulation of cell cycle and apoptosis". Mol. Cancer Ther. 15 (12): 3087–3096. doi:10.1158/1535-7163.MCT-15-1002. PMID 27638859.
- ^ Wissing MD, Mendonca J, Kim E, Kim E, Shim JS, Kaelber NS, Kant H, Hammers H, Commes T, Van Diest PJ, Liu JO, Kachhap SK (2013). "Identification of cetrimonium bromide and irinotecan as compounds with synthetic lethality against NDRG1 deficient prostate cancer cells". Cancer Biol. Ther. 14 (5): 401–10. doi:10.4161/cbt.23759. PMC 3672184. PMID 23377825.
- ^ Dominick G, Bowman J, Li X, Miller RA, Garcia GG (2016). "mTOR regulates the expression of DNA damage response enzymes in long-lived Snell dwarf, GHRKO, and PAPPA-KO mice". Aging Cell. 16 (1): 52–60. doi:10.1111/acel.12525. PMC 5242303. PMID 27618784.
- ^ 제품 이름: SCL-70 항원 ImmunoVision.com의 웨이백 머신에 2006-03-19 보관, 2011년 4월 검색
외부 링크
| 위키미디어 커먼스는 Topoisomerase 제1종과 관련된 미디어를 보유하고 있다. |
- DNA+Topoisomerase,+Type+미국 국립 의학 도서관 제목(MesH)에 있음




