소핵소 RNA
Small nucleolar RNA분자생물학에서, 작은 핵 RNA는 주로 리보솜 RNA, 전달 RNA 및 작은 핵 RNA와 같은 다른 RNA의 화학적 변화를 이끄는 작은 RNA 분자의 한 종류이다.snoRNA에는 메틸화와 관련된 C/D박스 snoRNA와 의사유리지화와 관련된H/ACA 박스 snoRNA의 2가지 주요 클래스가 있습니다.SnoRNA는 일반적으로 가이드 RNA라고 하지만 트리파노솜에서 RNA 편집을 지시하는 가이드 RNA와 혼동해서는 안 됩니다.
snoRNA 유도 변경
전사 후 초기 rRNA 분자(사전 rRNA로 명명)는 성숙한 rRNA 분자를 생성하기 위한 일련의 처리 단계를 거친다.엑소핵산 분해효소 및 엔도핵산 분해효소에 의한 분해 전에, 사전 rRNA는 뉴클레오시드의 복잡한 변형 패턴을 거친다.여기에는 snoRNA에 의해 유도되는 메틸화 및 의사유도 포함된다.
- 메틸화는 다양한 기질에 메틸기를 부착 또는 치환하는 것이다.인간의 rRNA는 약 115개의 메틸기 변형을 포함한다.이들 중 대부분은 2µO-리보스-메틸화(메틸기가 리보스기에 [1]부착되어 있음)이다.
- 유사우리딜화는 뉴클레오시드 우리딘이 다른 이성질 형태인 유사우리딘(δ)으로 변환(이성화)되는 것이다.이러한 변형은 RNA 골격의 리보스에 대한 글리코실 결합을 중심으로 우리딘 염기가 180° 회전하는 것으로 구성됩니다.이 회전 후 질소염기는 일반적인 질소원자 대신 탄소원자를 글리코실 결합에 기여시킨다.이 수정의 유익한 측면은 베이스에서 사용할 수 있는 추가적인 수소 결합 공여체이다.우리딘은 왓슨-크릭 염기쌍, 아데닌, 슈도우리딘과 2개의 수소 결합을 만드는 반면, 3개의 수소 결합을 만들 수 있다.슈도우리딘이 아데닌과 염기쌍을 이루면 또 다른 수소 결합을 만들어 성숙한 rRNA 구조의 복잡성을 형성할 수 있다.활성 수소 결합 공여자는 종종 자신으로부터 멀리 떨어진 염기와 결합을 형성하여 rRNA가 기능적이어야 하는 3차 구조를 만듭니다.성숙한 인간의 rRNA는 약 95Ω의 [1]수정을 포함한다.
각 snoRNA 분자는 표적 [2]RNA에서 한 개(또는 두 개)의 개별 수정에 대한 지침 역할을 합니다.수식을 실시하기 위해 각 snoRNA는 소핵 리보핵단백질 입자(snoRNP)[3]로 불리는 RNA/단백질 복합체 내의 적어도 4개의 코어 단백질과 관련짓는다.각 RNA와 관련된 단백질은 snoRNA 분자의 유형에 따라 달라집니다(아래 snoRNA 가이드 패밀리 참조).snoRNA 분자는 반센스 요소(10~20개의 뉴클레오티드의 연장선)를 포함하고 있으며, 이는 사전 RNA 분자의 변형을 목표로 하는 염기(뉴클레오티드)를 둘러싼 염기서열을 보완한다.이를 통해 snoRNP가 표적 RNA를 인식하고 결합할 수 있습니다.일단 snoRNP가 표적 부위에 결합되면, 관련 단백질은 표적 [4]염기의 화학적 변형을 촉매하기 위해 올바른 물리적 위치에 놓인다.
snoRNA 가이드 패밀리
두 가지 다른 유형의 rRNA 수정(메틸화 및 의사유도릴화)은 두 개의 서로 다른 snoRNA 패밀리에 의해 지시됩니다.이러한 snoRNA 패밀리는 snoRNA 내의 보존된 시퀀스모티브의 존재에 근거하여 안티센스 C/D 박스 및 H/ACA 박스 snoRNA라고 불립니다.예외는 있지만 일반적으로 C/D 박스 구성원은 메틸화를, H/ACA 구성원은 의사 전달을 각각 안내합니다.각 패밀리의 구성원은 생물 발생, 구조 및 기능에 따라 다를 수 있지만, 각 패밀리는 다음과 같은 일반화된 특성에 따라 분류됩니다.자세한 것은,[5] 리뷰를 참조해 주세요.SnoRNA는 MeSH에서 작은 핵 RNA로 분류된다.HGNC는 snoRNABase 및 이 분야 전문가들과 협력하여 snoRNA를 [6]코드하는 인간 유전자의 고유 이름을 승인했습니다.
C/D 박스
C/D 박스 snoRNA는 각각 snoRNA의 5' 및 3' 끝 부근에 위치한 2개의 짧은 보존 배열 모티브인 C(RUGAUGA)와 D(CUGA)를 포함한다.C박스 상류와 D박스 하류에 위치한 짧은 영역(~ 5 뉴클레오티드)은 일반적으로 베이스 상보적이며, C박스 및 D박스 모티브를 근접하게 하는 줄기세포 구조를 형성한다.이 줄기세포 구조는 올바른 snoRNA 합성과 핵자 [7]국재화에 필수적인 것으로 나타났다.많은 C/D 박스 snoRNA는 또한 snoRNA 분자의 중앙 부분에 위치한 C 및 D 모티브(C' 및 D'로 지칭)의 보존 상태가 덜한 복사본을 추가로 포함합니다.D박스 상류의 10~21뉴클레오티드의 보존영역은 표적 RNA의 메틸화 부위와 상보적이며,[8] snoRNA가 RNA와 이중 RNA를 형성할 수 있도록 한다.표적 RNA에서 수정되는 뉴클레오티드는 보통 D박스(또는 D'박스)[9][10]에서 상류의 5번째 위치에 위치한다.C/D 박스 SnoRNA는 핵심 C/D 박스 SnoRNP를 [5]구성하는 4개의 진화 보존 필수 단백질인 피브릴라린(Nop1p), NOP56, NOP58 및 SNU13(진핵생물에서는 15.5-kD 단백질, 고고 상동물은 L7ae)과 관련된다.
2μ-O-메틸화를 유도하는 것으로 나타나지 않은 진핵생물 C/D 박스 snoRNA(snoRNA U3)가 존재한다.대신, 사전 RNA 분할을 지시함으로써 rRNA 처리에서 기능합니다.
H/ACA 박스
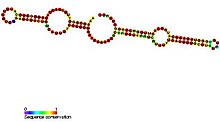
H/ACA 박스 snoRNA는 헤어핀-힌지-헤어핀-테일 [5]구조라고 불리는 2개의 단일가닥 영역으로 이루어진 공통 2차 구조를 가진다.H/ACA snoRNA에는 H 박스(컨센서스 ANANA) 및 ACA 박스(ACA)로 알려진 보존된 시퀀스 모티브도 포함되어 있습니다.두 모티브는 보통 2차 구조의 단일 가닥 영역에 위치한다.H 모티브는 힌지에 있고 ACA 모티브는 테일 영역에 있습니다. 즉,[11] 시퀀스의 3' 말단에서 3개의 뉴클레오티드가 나옵니다.헤어핀 영역에는 안티센스 가이드 시퀀스(타깃 시퀀스를 보완하는 베이스)가 위치한 인식 루프라고 하는 내부 팽대부가 있습니다.이러한 가이드 배열은 기본적으로 수정될 표적 rRNA에서 우리딘의 위치를 표시합니다.H/ACA 박스 snoRNA는 HAC 코어의 핵심을 구성하는 네 가지 진화적 보존 필수 단백질인 Dyskerin(Cbf5p), GAR1, NHP2, NOP10과 관련된다.디스케린은 보존된 여러 의사우리딘 합성효소 서열을 가지고 있기 때문에 리보핵단백질(RNP) 복합체의 촉매 성분일 가능성이 높고, tRNA에서 우리딘을 수정하는 의사우리딘 합성효소와 밀접한 관련이 있다.트리파노솜과 같은 하층 진핵세포에서는 유사한 RNA가 단일 머리핀 구조와 RNA의 3⁄[12] 말단에 ACA 박스 대신 AGA 박스의 형태로 존재한다.단일 헤어핀 snoRNA에 대한 이중 헤어핀 H/ACA box snoRNA의 처리가 있었던 것으로 보고되었으나, 트리파노솜과 달리 [19]꼬리에 규칙적인 ACA 모티브가 있다.
인간 텔로머라아제(hTERC)의 RNA 성분은 텔로머라아제 [13]RNP 자체의 사전 RNP 형성과 핵소재화를 위한 H/ACA 도메인을 포함한다.H/ACA snoRNP는 인간 텔로머라아제와의 관련성 때문에 희귀 유전병인 선천성각화장애(DKC)에 관여하고 있다.H/ACA snoRNP의 단백질 성분 돌연변이는 생리적 TERC 수준의 감소를 초래한다.이는 주로 텔로미어 유지 부실한 질병으로 보이는 DKC의 이면에 있는 병리와 강한 상관관계가 있다.
컴포지트 H/ACA 및 C/D 박스
snRNA(snRNA) U5의 2-O-리보스 메틸화 및 의사유도릴화 모두에서 기능하는 특이 가이드 snoRNA U85가 확인되었다.[14]이 복합 snoRNA는 C/D 및 H/ACA 박스 도메인을 모두 포함하고 있으며, snoRNA의 각 등급에 고유한 단백질(각각 피브릴라린 및 Gar1p)과 관련이 있다.보다 복합적인 snoRNA가 [15]특징지어졌습니다.
이러한 복합 snoRNA는 카잘 바디라고 불리는 핵소기관 내에 축적되는 것으로 발견되었고 작은 카잘 바디 특이 RNA라고 불립니다.이는 대부분의 C/D 박스 또는 H/ACA 박스 snoRNA가 핵에 위치하는 것과 대조적이다.이러한 카잘체 특이 RNA는 스플리세오솜 RNA U1, U2, U4, U5 및 U12 [15]전사된 RNA 중합효소 II의 개조에 관여하는 것으로 제안되었다.Cajal 본체에 현지화된 모든 snoRNA가 복합 C/D 및 H/ACA 박스 snoRNA인 것은 아닙니다.
고립된 snoRNA
새롭게 식별된 snoRNA의 표적은 추정 대상 RNA와 snoRNA 배열의 안티센스 요소 또는 인식 루프 사이의 배열 상보성에 기초하여 예측된다.그러나 알려진 RNA 타겟이 없는 'orphan' 가이드의 수가 증가하고 있으며, 이는 rRNA에 이전보다 더 많은 단백질 또는 전사물이 관여할 수 있으며/또는 일부 snoRNA가 [16][17]rRNA와 관련되지 않은 다른 기능을 가지고 있음을 시사한다.이러한 고아 snoRNA 중 일부는 교대로 스플라이스된 [18]트랜스크립트를 조절한다는 증거가 있다.예를 들어, C/D 박스 snoRNA SNORD115는 상호보완성 [19][20]보존 영역을 통해 세로토닌 2C 수용체 mRNA의 대체 스플라이싱을 조절하는 것으로 보인다.SNORD115와 동일한 클러스터에 존재하는 또 다른 C/D 박스 snoRNA, SNORD116은 생물정보학적 접근법을 사용하여 단백질 코딩 유전자 내에 23개의 가능한 표적을 가질 것으로 예측되었다.이들 중 많은 부분이 대체 스플라이싱으로 확인되었으며, 이는 대체 스플라이싱 [21]조절에서 SNORD116의 역할을 시사한다.
대상 수정
성숙한 RNA의 기능에 대한 메틸화 및 의사유리기변성의 정확한 영향은 아직 알려지지 않았다.이러한 변형은 필수적인 것으로 보이지는 않지만 RNA 접힘과 리보솜 단백질과의 상호작용을 미묘하게 강화하는 것으로 알려져 있다.이들의 중요성을 뒷받침하기 위해 표적 부위 수정은 성숙한 RNA의 보존되고 기능적으로 중요한 영역 내에 배타적으로 위치하며 먼 진핵생물들 [5]사이에서 일반적으로 보존된다.
- 2'-O-메틸화 리보스는 3'-엔도 배열을 증가시킨다.
- Pseudouridine(psi/Ω)은 H 결합을 위한 다른 옵션을 추가합니다.
- 고도로 메틸화된 RNA는 가수분해로부터 보호되며, rRNA는 자체 가수분해와 스플라이싱을 촉매하여 리보자임 역할을 한다.
게놈 조직
SnoRNA는 게놈에 다양하게 존재한다.대부분의 척추동물 snoRNA 유전자는 리보솜 합성 또는 번역에 관여하는 단백질을 코드하는 유전자의 인트론 내에 부호화되어 RNA 중합효소 II에 의해 합성된다.SnoRNA는 또한 유전자간 영역, 단백질을 코드하는 유전자의 ORF 및 UTR에 [22]위치하는 것으로 나타났다.SnoRNA는 또한 RNA 중합효소 II 또는 III에 의해 자체 촉진제로부터 전사될 수 있다.
임프린트 위치
인간 게놈에는 C/D박스 snoRNA가 임프린트된 위치 내에서 연속적으로 발견되는 적어도 2개의 예가 있다.이 두 개의 위치(염색체 14의 14q32와 15개의 15q11q13)는 광범위하게 특징지어졌으며, 두 영역 모두에서 밀접하게 관련된 복사본의 클러스터에서 여러 snoRNA가 인트라온 내에 위치했다.
15q11q13에서는 5개의 다른 snoRNA가 식별되었습니다(SNORD64, SNORD107, SNORD108, SNORD109(2부), SNORD116(29부) 및 SNORD115(48부)).이 지역에서 나온 29개의 SNORD116(HBII-85) 사본의 분실은 프래더-윌리 증후군의[23][24][25][26] 원인으로 확인되었으며, SNORD115의 추가 사본의 분실은 [27][28][29]자폐증과 관련이 있다.
영역 14q32에는 조직 고유의 ncRNA 트랜스크립트(MEG8)의 인트론 내에 2개의 snoRNAs SNORD113(9복사) 및 SNORD114(31복사)의 반복이 포함되어 있습니다.14q32 도메인은 임프린트된 15q11-q13 loci와 공통 게놈 특징을 공유하는 것으로 나타났으며, 임프린트된 loci의 진화 또는 메커니즘에서 C/D 박스 snoRNA의 탠덤 반복에 대한 가능한 역할이 [30][31]제안되었다.
기타 기능
snoRNA는 miRNA로서 기능할 수 있습니다.인간 ACA45는 RNAse III 패밀리 엔도리보핵산가수분해효소 다이커에 [32]의해 21-뉴클레오티드 길이의 성숙한 miRNA로 처리될 수 있는 진정한 의미의 snoRNA인 것으로 나타났다.이 snoRNA 생성물은 이전에 mmu-miR-1839로 식별되었으며 다른 miRNA 생성 엔도리보핵산가수분해효소 드로사와 [33]독립적으로 처리되는 것으로 나타났다.생물정보학적 분석에 따르면 추정적으로 snoRNA에서 파생된 miRNA와 유사한 조각이 [34]다른 유기체에서 발생한다는 것이 밝혀졌다.
최근에는 snoRNA가 rRNA와 관련이 없는 기능을 가질 수 있다는 사실이 밝혀졌습니다.그러한 기능 중 하나는 SNORD115로도 알려진 snoRNA HBII-52에 의해 이루어지는 트랜스 유전자 전사체의 대체 스플라이싱 조절이다.[19]
2012년 11월 슈버트 외 연구진은 특정 RNA가 드로소필라 [35]세포의 염색질 압축과 접근성을 조절한다는 사실을 밝혔다.
레퍼런스
- ^ a b Maden BE, Hughes JM (June 1997). "Eukaryotic ribosomal RNA: the recent excitement in the nucleotide modification problem". Chromosoma. 105 (7–8): 391–400. doi:10.1007/BF02510475. PMID 9211966. S2CID 846233.
- ^ Gjerde, Douglas T. (2009). RNA purification and analysis : sample preparation, extraction, chromatography. Weinheim: Wiley-VCH. pp. 25–26. ISBN 978-3-527-62720-2. Retrieved 28 September 2020.
- ^ Bertrand, Edouard; Fournier, Maurille J. (2013). "The snoRNPs and Related Machines: Ancient Devices That Mediate Maturation of rRNA and Other RNAs". Landes Bioscience. Retrieved 28 September 2020.
- ^ Lykke-Andersen, Søren; Ardal, Britt Kidmose; Hollensen, Anne Kruse; Damgaard, Christian Kroun; Jensen, Torben Heick (October 2018). "Box C/D snoRNP Autoregulation by a cis-Acting snoRNA in the NOP56 Pre-mRNA". Molecular Cell. 72 (1): 99–111.e5. doi:10.1016/j.molcel.2018.08.017. PMID 30220559.
- ^ a b c d e Bachellerie JP, Cavaillé J, Hüttenhofer A (August 2002). "The expanding snoRNA world". Biochimie. 84 (8): 775–790. doi:10.1016/S0300-9084(02)01402-5. PMID 12457565.
- ^ Wright MW, Bruford EA (January 2011). "Naming 'junk': human non-protein coding RNA (ncRNA) gene nomenclature". Human Genomics. 5 (2): 90–98. doi:10.1186/1479-7364-5-2-90. PMC 3051107. PMID 21296742.
- ^ Samarsky DA, Fournier MJ, Singer RH, Bertrand E (July 1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". The EMBO Journal. 17 (13): 3747–3757. doi:10.1093/emboj/17.13.3747. PMC 1170710. PMID 9649444.
- ^ Kiss-László Z, Henry Y, Kiss T (February 1998). "Sequence and structural elements of methylation guide snoRNAs essential for site-specific ribose methylation of pre-rRNA". The EMBO Journal. 17 (3): 797–807. doi:10.1093/emboj/17.3.797. PMC 1170428. PMID 9451004.
- ^ Cavaillé J, Nicoloso M, Bachellerie JP (October 1996). "Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides". Nature. 383 (6602): 732–735. Bibcode:1996Natur.383..732C. doi:10.1038/383732a0. PMID 8878486. S2CID 4334683.
- ^ Kiss-László Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M, Kiss T (June 1996). "Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs". Cell. 85 (7): 1077–1088. doi:10.1016/S0092-8674(00)81308-2. PMID 8674114. S2CID 10418885.
- ^ Ganot P, Caizergues-Ferrer M, Kiss T (April 1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes & Development. 11 (7): 941–956. doi:10.1101/gad.11.7.941. PMID 9106664.
- ^ Liang XH, Liu L, Michaeli S (October 2001). "Identification of the first trypanosome H/ACA RNA that guides pseudouridine formation on rRNA". The Journal of Biological Chemistry. 276 (43): 40313–40318. doi:10.1074/jbc.M104488200. PMID 11483606.
- ^ Trahan C, Dragon F (February 2009). "Dyskeratosis congenita mutations in the H/ACA domain of human telomerase RNA affect its assembly into a pre-RNP". RNA. 15 (2): 235–243. doi:10.1261/rna.1354009. PMC 2648702. PMID 19095616.
- ^ Jády BE, Kiss T (February 2001). "A small nucleolar guide RNA functions both in 2′-O-ribose methylation and pseudouridylation of the U5 spliceosomal RNA". The EMBO Journal. 20 (3): 541–551. doi:10.1093/emboj/20.3.541. PMC 133463. PMID 11157760.
- ^ a b Darzacq X, Jády BE, Verheggen C, Kiss AM, Bertrand E, Kiss T (June 2002). "Cajal body-specific small nuclear RNAs: a novel class of 2′-O-methylation and pseudouridylation guide RNAs". The EMBO Journal. 21 (11): 2746–2756. doi:10.1093/emboj/21.11.2746. PMC 126017. PMID 12032087.
- ^ Jády BE, Kiss T (March 2000). "Characterisation of the U83 and U84 small nucleolar RNAs: two novel 2′-O-ribose methylation guide RNAs that lack complementarities to ribosomal RNAs" (Free full text). Nucleic Acids Research. 28 (6): 1348–1354. doi:10.1093/nar/28.6.1348. PMC 111033. PMID 10684929.
- ^ Li SG, Zhou H, Luo YP, Zhang P, Qu LH (April 2005). "Identification and functional analysis of 20 Box H/ACA small nucleolar RNAs (snoRNAs) from Schizosaccharomyces pombe". The Journal of Biological Chemistry. 280 (16): 16446–16455. doi:10.1074/jbc.M500326200. PMID 15716270.
- ^ Kishore S, Stamm S (2006). "Regulation of alternative splicing by snoRNAs". Cold Spring Harbor Symposia on Quantitative Biology. 71: 329–334. doi:10.1101/sqb.2006.71.024. PMID 17381313.
- ^ a b Kishore S, Stamm S (January 2006). "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C". Science. 311 (5758): 230–232. Bibcode:2006Sci...311..230K. doi:10.1126/science.1118265. PMID 16357227. S2CID 44527461.
- ^ Doe CM, Relkovic D, Garfield AS, Dalley JW, Theobald DE, Humby T, Wilkinson LS, Isles AR (June 2009). "Loss of the imprinted snoRNA mbii-52 leads to increased 5htr2c pre-RNA editing and altered 5HT2CR-mediated behaviour". Human Molecular Genetics. 18 (12): 2140–2148. doi:10.1093/hmg/ddp137. PMC 2685753. PMID 19304781.
- ^ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A (January 2008). "snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions". Gene. 408 (1–2): 172–179. doi:10.1016/j.gene.2007.10.037. PMC 6800007. PMID 18160232.
- ^ Kaur D, Gupta AK, Kumari V, Sharma R, Bhattacharya A, Bhattacharya S (14 August 2012). "Computational prediction and validation of C/D, H/ACA and Eh_U3 snoRNAs of Entamoeba histolytica". BMC Genomics. 13: 390. doi:10.1186/1471-2164-13-390. PMC 3542256. PMID 22892049.
- ^ Skryabin BV, Gubar LV, Seeger B, Pfeiffer J, Handel S, Robeck T, Karpova E, Rozhdestvensky TS, Brosius J (December 2007). "Deletion of the MBII-85 snoRNA gene cluster in mice results in postnatal growth retardation". PLOS Genetics. 3 (12): e235. doi:10.1371/journal.pgen.0030235. PMC 2323313. PMID 18166085.
- ^ Sahoo T, del Gaudio D, German JR, Shinawi M, Peters SU, Person RE, Garnica A, Cheung SW, Beaudet AL (June 2008). "Prader-Willi phenotype caused by paternal deficiency for the HBII-85 C/D box small nucleolar RNA cluster". Nature Genetics. 40 (6): 719–721. doi:10.1038/ng.158. PMC 2705197. PMID 18500341.
- ^ Ding F, Li HH, Zhang S, Solomon NM, Camper SA, Cohen P, Francke U (March 2008). Akbarian S (ed.). "SnoRNA Snord116 (Pwcr1/MBII-85) deletion causes growth deficiency and hyperphagia in mice". PLOS ONE. 3 (3): e1709. Bibcode:2008PLoSO...3.1709D. doi:10.1371/journal.pone.0001709. PMC 2248623. PMID 18320030.
- ^ Ding F, Prints Y, Dhar MS, Johnson DK, Garnacho-Montero C, Nicholls RD, Francke U (June 2005). "Lack of Pwcr1/MBII-85 snoRNA is critical for neonatal lethality in Prader-Willi syndrome mouse models". Mammalian Genome. 16 (6): 424–431. doi:10.1007/s00335-005-2460-2. PMID 16075369. S2CID 12256515.
- ^ Nakatani J, Tamada K, Hatanaka F, Ise S, Ohta H, Inoue K, Tomonaga S, Watanabe Y, Chung YJ, Banerjee R, Iwamoto K, Kato T, Okazawa M, Yamauchi K, Tanda K, Takao K, Miyakawa T, Bradley A, Takumi T (June 2009). "Abnormal behavior in a chromosome-engineered mouse model for human 15q11–13 duplication seen in autism". Cell. 137 (7): 1235–1246. doi:10.1016/j.cell.2009.04.024. PMC 3710970. PMID 19563756.
- ^ Bolton PF, Veltman MW, Weisblatt E, Holmes JR, Thomas NS, Youings SA, Thompson RJ, Roberts SE, Dennis NR, Browne CE, Goodson S, Moore V, Brown J (September 2004). "Chromosome 15q11–13 abnormalities and other medical conditions in individuals with autism spectrum disorders". Psychiatric Genetics. 14 (3): 131–137. doi:10.1097/00041444-200409000-00002. PMID 15318025. S2CID 37344935.
- ^ Cook EH, Scherer SW (October 2008). "Copy-number variations associated with neuropsychiatric conditions". Nature. 455 (7215): 919–923. Bibcode:2008Natur.455..919C. doi:10.1038/nature07458. PMID 18923514. S2CID 4377899.
- ^ Cavaillé J, Seitz H, Paulsen M, Ferguson-Smith AC, Bachellerie JP (June 2002). "Identification of tandemly-repeated C/D snoRNA genes at the imprinted human 14q32 domain reminiscent of those at the Prader-Willi/Angelman syndrome region". Human Molecular Genetics. 11 (13): 1527–1538. doi:10.1093/hmg/11.13.1527. PMID 12045206.
- ^ Labialle S, Cavaillé J (August 2011). "Do repeated arrays of regulatory small-RNA genes elicit genomic imprinting?: Concurrent emergence of large clusters of small non-coding RNAs and genomic imprinting at four evolutionarily distinct eutherian chromosomal loci". BioEssays. 33 (8): 565–573. doi:10.1002/bies.201100032. PMID 21618561. S2CID 10408004.
- ^ Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N, Meister G (November 2008). "A human snoRNA with microRNA-like functions". Molecular Cell. 32 (4): 519–528. doi:10.1016/j.molcel.2008.10.017. PMID 19026782.
- ^ Babiarz JE, Ruby JG, Wang Y, Bartel DP, Blelloch R (October 2008). "Mouse ES cells express endogenous shRNAs, siRNAs, and other Microprocessor-independent, Dicer-dependent small RNAs". Genes & Development. 22 (20): 2773–2785. doi:10.1101/gad.1705308. PMC 2569885. PMID 18923076.
- ^ Taft RJ, Glazov EA, Lassmann T, Hayashizaki Y, Carninci P, Mattick JS (July 2009). "Small RNAs derived from snoRNAs". RNA. 15 (7): 1233–1240. doi:10.1261/rna.1528909. PMC 2704076. PMID 19474147.
- ^ Schubert T, Pusch MC, Diermeier S, Benes V, Kremmer E, Imhof A, Längst G (November 2012). "Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin". Molecular Cell. 48 (3): 434–444. doi:10.1016/j.molcel.2012.08.021. PMID 23022379.



