피루브산염, 인산디키나아제
Pyruvate, phosphate dikinase| 피루브산염, 인산디키나아제 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| 식별자 | |||||||||
| EC 번호 | 2.7.9.1 | ||||||||
| CAS 번호 | 9027-40-1 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
| 피루브산염, 인산디키나아제 | |
|---|---|
 | |
| 식별자 | |
| 기호. | PDK |
| 인터프로 | IPR010121 |
| 아래 도메인을 참조하십시오. | |
피루브산, 인산디키나아제 또는 PPDK(EC 2.7.9.1)는 전달효소 계열의 효소로서 화학반응을 촉매한다.
- ATP + 피루브산염 + 인산염δ(\+ 이인산염)
이 효소는 주로 식물에서 연구되어 왔지만 일부 박테리아에서도 연구되어 왔다.[1]당합성 및 광합성의 핵심 효소로서 엠벤-마이어호프-파르나스 당분해에서 피루브산 키나제에 의해 수행되는 반응을 역전시키는 역할을 한다.피루브산염, 물디키나아제 등과 혼동해서는 안 된다.
포스포트랜스포트랜스포트랜스포트랜스포트레이스(phosphotransferase)와 쌍으로 구성된 수용체(다이키나아제)를 전달하는 전달효소군에 속한다.이 효소는 피루브산 대사 및 탄소 고정에 관여합니다.
명명법
이 효소 클래스의 계통명은 ATP:피루브산염, 인산인산전달효소이다.일반적으로 사용되는 다른 명칭으로는 피루브산염, 오르토인산디키나아제, 피루브산염디키나아제, 피루브산염디키나제, 피루브산염리가아제, 피루브산염디키나아제, 피루브산염디키나아제 및 PPDK가 있다.
반응 메커니즘
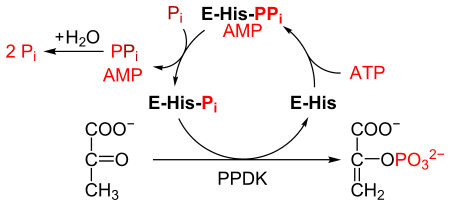
PPDK는 피루브산의 포스포에놀피루브산(PEP)으로의 전환을 촉매하여 1분자의 ATP를 소비하고 그 과정에서 1분자의 AMP를 생성한다.이 메커니즘은 다음 3가지 [2]가역 반응으로 구성됩니다.
- PPDK 효소는 ATP와 결합하여 AMP와 디포스포릴화 PPDK를 생성한다.
- 디포스포릴화 PPDK는 무기인산에 결합하여 디포스포인산 및 (모노)인산 PPDK를 생성한다.
- 인산화된 PPDK는 피루브산에 결합하여 포스포에놀피루브산을 생성하고 PPDK를 재생한다.
이 반응은 피루브산 키네이스에 의해 촉매되는 반응과 유사하며, 피루브산 키네이스는 피루브산을 [3]PEP로 전환하기도 한다.그러나 피루브산 키나제는 되돌릴 수 없는 반응을 촉매하며 ATP를 소비하지 않는다.반면, PPDK는 가역 반응을 촉매하고 변환된 피루브산 분자당 1분자의 ATP를 소비한다.
현재 각 기계적 단계의 세부 사항은 알려지지[3] 않았습니다.
구조.
활성 형태에서 PPDK는 약 95kDa의[4] 서브유닛을 가진 호모테트라머이다.
약 45 앵스트롬 간격으로 서로 다른 기질이 [5]결합하는 두 개의 다른 반응 센터가 있습니다.뉴클레오티드(ATP) 결합 부위는 N 말단에 있고 240개의 아미노산과 특징적인 ATP-grasp를 가지고 있습니다.피루브산/PEP 결합 부위는 C 말단에 있으며, 340개의 아미노산과 α/β-배럴 접힘을 가지고 있다.촉매 작용을 담당하는 1차 잔류물인 His455를 포함하는 중심 도메인도 있습니다.His455는 포스포릴 수용체 또는 공여 [3]잔기이다.효소의 구조는 His455 암이 두 반응 [6]중심 사이에서 포스포릴기를 셔틀링하기 위해 회전 운동을 한다는 것을 암시합니다.이 스위블링 중에 중앙 도메인은 92도 이상 회전하며 0.5 앵스트롬을 [7]변환합니다.
PPDK의 결정 구조에 대한 연구는 중심 도메인이 [7]효소의 소스에 따라 다른 두 도메인에 서로 다른 근접 위치에 있음을 보여준다.옥수수에서는 C 말단에 가깝고 클로스트리듐 심비오섬에서는 N 말단에 가깝다.
연구에 따르면 PPDK 결합 메커니즘은 D-Ala-D-Ala 연결효소 및 피루브산 [5]키나제의 결합 메커니즘과 유사하다.특히, PPDK는 피루브산 키네이스와 매우 유사하며, 피루브산의 포스포에놀피루브산으로의 전환을 촉매하지만, 포스포에놀피루브산 인산화효소 [3]중간체가 없다.아미노산 배열은 다르지만 촉매 작용에 중요한 잔류물은 두 효소 모두에서 보존됩니다.점 돌연변이 유발 실험에서는 촉매 잔류물이 Arg561, Arg617, Glu745, Asn768 및 Cys831(C, 심비오섬 단백질, PDB: 1KBL, 1KC7)[3]을 포함하는 것으로 나타났습니다.
|
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
생물학적 기능과 진화
PPDK는 이산화탄소 [8]고정의 효율성을 개선하기 위해 C4 경로에서 사용됩니다.빛이 많은 환경에서, 식물의 광합성 속도는 이산화탄소 흡수2 속도에 의해 제한된다.이것은 일련의 화학 반응을 사용하여 CO를 중엽 세포에서 다발 피복 세포로 운반함으로써2 개선될 수 있다.PPDK는 피루브산을 PEP로 변환하고, PEP는 CO와2 반응하여 옥살아세트산을 생성한다.다발 피복 세포에서 CO가 방출되면2 피루브산은 재생되고 주기는 [8]계속된다.
PPDK에 의해 촉매되는 반응은 가역적이지만 생물학적 조건에서는 PEP가 제품으로서 바람직하다.이것은 반응이 일어나는 스트로마 내의 기본 pH와 아데닐산 키나아제 및 피로포스파타아제의 고농도 때문이다.이 두 효소는 각각 AMP와 이인산을 수반하는 발기성 반응을 촉매하기 때문에 PPDK-카탈리시스 반응을 [9]촉진한다.PPDK는 ATP를 소비하기 때문에, C4 경로는 많은 [8]양의 ATP를 생산할 수 없기 때문에 빛에 거의 접근하지 않는 환경에 있는 식물에게 불리하다.
PPDK는 총 단백질의 [10]최대 10%를 차지하는 C4 잎에 매우 풍부하다.연구에 따르면 효소는 여러 종의 식물에서 약 96%가 동일하다.잡종 실험은 유전적 차이가 식물이 C4 경로를 수행하는 범위와 관련이 있다는 것을 밝혀냈다. 드문 배열은 C3 [11]특성을 보이는 식물에도 존재한다.PPDK는 C3 발전소에서도 소량 검출된다.진화 역사는 유사한 피루브산 키나아제처럼 당분해에서 역할을 했고, 결국 C4 [10]경로로 진화했음을 시사한다.
PPDK는 식물 외에 기생 아메오바 엔타메바 히스토리티카(P37213) 및 클로스트리디움 심비오섬(P22983; 및 기타 세균)[12]에서도 발견된다.이러한 두 유기체에서 PPDK는 피루브산 키네이스와 유사하게(때로는 대체) 작용하여 해당과정의 일부로 ATP 생성 방향의 반응을 촉매한다.엔타메바 PPDK의 억제제는 이 [13]유기체에 대한 아메바 살인으로 제안되었다.
규정
식물 PPDK는 피루브산염 디키나아제 조절 단백질(PDRP)[4]에 의해 조절된다.광량이 높을 때 PDRP는 AMP를 사용하여 PDK의 Thr456을 탈인산염시켜 [10]효소를 활성화시킨다.PDRP는 동일한 트레오닌 잔기를 이인산을 사용하여 인산화함으로써 PDK를 비활성화한다.PDRP는 두 가지 [10]다른 메커니즘을 통해 PPDK의 활성화와 비활성화를 모두 촉매하기 때문에 고유한 조절기입니다.
옥수수 PPDK에 대한 연구는 인트론, 터미네이터 배열, 그리고 아마도 다른 강화제 배열이 기능적이고 안정적인 mRNA의 수준을 증가시키기 위해 함께 작용함을 시사한다. PPDK cDNA는 유의한 [14]발현을 보이는 온전한 DNA에 비해 트랜스제닉 쌀에서 약간만 발현되었다.
구조 연구
2018년 초 현재 이 등급의 효소에 대해 14개의 구조가 해결되었으며, PDB 가입 코드 1DIK, 1GO, 1H6Z, 1JDE, 1KBL, 1KC7, 1VBG, 1VBH, 2DIK, 2FM4, 5J.
레퍼런스
- ^ Pocalyko DJ, Carroll LJ, Martin BM, Babbitt PC, Dunaway-Mariano D (December 1990). "Analysis of sequence homologies in plant and bacterial pyruvate phosphate dikinase, enzyme I of the bacterial phosphoenolpyruvate: sugar phosphotransferase system and other PEP-utilizing enzymes. Identification of potential catalytic and regulatory motifs". Biochemistry. 29 (48): 10757–65. doi:10.1021/bi00500a006. PMID 2176881.
- ^ Evans HJ, Wood HG (December 1968). "The mechanism of the pyruvate, phosphate dikinase reaction". Proceedings of the National Academy of Sciences of the United States of America. 61 (4): 1448–53. Bibcode:1968PNAS...61.1448E. doi:10.1073/pnas.61.4.1448. PMC 225276. PMID 4303480.
- ^ a b c d e Herzberg O, Chen CC, Liu S, Tempczyk A, Howard A, Wei M, et al. (January 2002). "Pyruvate site of pyruvate phosphate dikinase: crystal structure of the enzyme-phosphonopyruvate complex, and mutant analysis". Biochemistry. 41 (3): 780–7. doi:10.1021/bi011799+. PMID 11790099.
- ^ a b Chastain CJ, Failing CJ, Manandhar L, Zimmerman MA, Lakner MM, Nguyen TH (May 2011). "Functional evolution of C(4) pyruvate, orthophosphate dikinase". Journal of Experimental Botany. 62 (9): 3083–91. doi:10.1093/jxb/err058. PMID 21414960.
- ^ a b Herzberg O, Chen CC, Kapadia G, McGuire M, Carroll LJ, Noh SJ, Dunaway-Mariano D (April 1996). "Swiveling-domain mechanism for enzymatic phosphotransfer between remote reaction sites". Proceedings of the National Academy of Sciences of the United States of America. 93 (7): 2652–7. Bibcode:1996PNAS...93.2652H. doi:10.1073/pnas.93.7.2652. PMC 39685. PMID 8610096.
- ^ Lim K, Read RJ, Chen CC, Tempczyk A, Wei M, Ye D, et al. (December 2007). "Swiveling domain mechanism in pyruvate phosphate dikinase". Biochemistry. 46 (51): 14845–53. CiteSeerX 10.1.1.421.2653. doi:10.1021/bi701848w. PMID 18052212.
- ^ a b Nakanishi T, Nakatsu T, Matsuoka M, Sakata K, Kato H (February 2005). "Crystal structures of pyruvate phosphate dikinase from maize revealed an alternative conformation in the swiveling-domain motion". Biochemistry. 44 (4): 1136–44. doi:10.1021/bi0484522. PMID 15667207.
- ^ a b c Berg J, Tymoczko J, Stryer L (2012). "The Calvin Cycle and the Pentose Phosphate Pathway". Biochemistry (7th ed.). New York: W.H Freeman. pp. 599–600. ISBN 9780716787242.
- ^ Chastain C (2010). "Structure, Function, and Post-Translational Regulation of C4 Pyruvate Orthophosphate Dikinase". In Raghavendra A (ed.). C4 Photosynthesis and Related CO2 Concentrating Mechanisms. pp. 301–305. ISBN 9789048194063.
- ^ a b c d Chastain CJ, Fries JP, Vogel JA, Randklev CL, Vossen AP, Dittmer SK, et al. (April 2002). "Pyruvate,orthophosphate dikinase in leaves and chloroplasts of C(3) plants undergoes light-/dark-induced reversible phosphorylation". Plant Physiology. 128 (4): 1368–78. doi:10.1104/pp.010806. PMC 154264. PMID 11950985.
- ^ Rosche E, Streubel M, Westhoff P (October 1994). "Primary structure of the photosynthetic pyruvate orthophosphate dikinase of the C3 plant Flaveria pringlei and expression analysis of pyruvate orthophosphate dikinase sequences in C3, C3-C4 and C4 Flaveria species". Plant Molecular Biology. 26 (2): 763–9. doi:10.1007/bf00013761. PMID 7948930. S2CID 23276817.
- ^ UniProt 50%~90% 클러스터: Clostridium PPDK에서 제공
- ^ Stephen P, Vijayan R, Bhat A, Subbarao N, Bamezai RN (September 2008). "Molecular modeling on pyruvate phosphate dikinase of Entamoeba histolytica and in silico virtual screening for novel inhibitors". Journal of Computer-Aided Molecular Design. 22 (9): 647–60. Bibcode:2008JCAMD..22..647S. doi:10.1007/s10822-007-9130-2. PMID 17710553. S2CID 25026913.
- ^ Fukayama H, Tsuchida H, Agarie S, Nomura M, Onodera H, Ono K, et al. (November 2001). "Significant accumulation of C(4)-specific pyruvate, orthophosphate dikinase in a C(3) plant, rice". Plant Physiology. 127 (3): 1136–46. doi:10.1104/pp.010641. PMC 129282. PMID 11706193.
추가 정보
- Hatch MD, Slack CR (January 1968). "A new enzyme for the interconversion of pyruvate and phosphopyruvate and its role in the C4 dicarboxylic acid pathway of photosynthesis". The Biochemical Journal. 106 (1): 141–6. doi:10.1042/bj1060141. PMC 1198479. PMID 4305612.
- Reeves RE (June 1968). "A new enzyme with the glycolytic function of pyruvate kinase". The Journal of Biological Chemistry. 243 (11): 3202–4. doi:10.1016/S0021-9258(18)93395-8. PMID 4297474.
- Reeves RE (November 1971). "Pyruvate,phosphate dikinase from Bacteroides symbiosus". The Biochemical Journal. 125 (2): 531–9. doi:10.1042/bj1250531. PMC 1178089. PMID 5144757.
- Reeves RE, Menzies RA, Hsu DS (October 1968). "The pyruvate-phosphate dikinase reaction. The fate of phosphate and the equilibrium". The Journal of Biological Chemistry. 243 (20): 5486–91. doi:10.1016/S0021-9258(18)91972-1. PMID 4302788.