DNA중합효소II
DNA polymerase II| DNA중합효소II | |||||||
|---|---|---|---|---|---|---|---|
 DNA pol II의 결정구조(PDB 엔트리 3K5M 기준) | |||||||
| 식별자 | |||||||
| 유기체 | |||||||
| 기호. | 폴비 | ||||||
| 엔트레즈 | 944779 | ||||||
| PDB | 3K5M | ||||||
| RefSeq(프로트) | NP_414602.1 | ||||||
| 유니프로트 | P2189 | ||||||
| 기타 데이터 | |||||||
| EC 번호 | 2.7.7.7 | ||||||
| 염색체 | 게놈: 0.06~0.07 Mb | ||||||
| |||||||
DNA 중합효소 II(DNA Pol II 또는 Pol II라고도 함)는 PolB [1]유전자에 의해 암호화된 원핵 DNA 의존성 DNA 중합효소이다.
DNA 중합효소 II는 89.9kDa 단백질로 B 계열의 DNA 중합효소 중 하나입니다.1970년 토마스 콘버그에 의해 처음 분리되었고, 이후 몇 [2][3][4]년 동안 특징지어졌습니다.Pol II의 생체내 기능성은 논의 중이지만, 합의된 바에 따르면 Pol II는 원핵생물 DNA 복제에 주로 백업 효소로 관여한다.효소는 5μ→3μ DNA 합성 능력과 3μ→5μ 핵산가수분해효소 교정 활성을 가지고 있다.DNA Pol II는 DNA Pol III와 공통의 여러 결합 파트너와 상호작용하여 충실도와 처리 능력을 [1]향상시킵니다.
검출
DNA 중합효소 I은 [5]대장균에서 분리된 최초의 DNA 유도 DNA 중합효소였다.이 분리된 효소와 관련된 여러 연구에 따르면 DNA pol I은 복구 복제에 관여했을 가능성이 가장 높으며 주요 복제 [6]중합효소가 아니었다.DNA pol I의 생체내 역할을 더 잘 이해하기 위해 1969년 De Lucia와 Cairns에 의해 [7]이 효소에 결핍된 대장균 돌연변이(Termed Pol A1−)가 생성되었다.특징적으로, 이 새로운 돌연변이 변종은 자외선에 더 민감하여 DNA 폴리 I이 복구 복제에 관여했다는 가설을 뒷받침합니다.돌연변이는 야생형과 같은 속도로 성장해 DNA 복제를 담당하는 또 다른 효소의 존재를 나타냈다.여러 [2][3][4]연구소가 실시한 병행 연구에서 반보수적 DNA 복제에 관련된 이 새로운 중합효소의 분리 및 특성화가 뒤따랐다.새로운 중합효소는 DNA 중합효소 II로 불리며 한동안 [8]대장균의 주요 복제 효소로 여겨졌다.DNA pol II는 앤더슨 [9]등에 의해 1994년에 처음 결정화 되었다.
구조.
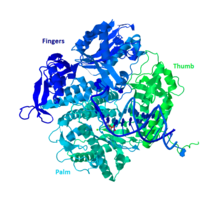
DNA Pol II는 783개의 아미노산으로 구성된 89.9 kD 단백질로, polB(dinA) 유전자에 의해 암호화된다.구상 단백질인 DNA Pol II는 단량체로 기능하는 반면, 다른 많은 중합효소는 복합체를 형성합니다.이 단량체에는 손바닥, 손가락, 엄지손가락으로 불리는 세 가지 주요 섹션이 있습니다.이 "손"은 DNA 가닥을 감싼다.복합체의 팜에는 2가의 금속 이온과 조정하여 기능하는 3개의 촉매 잔류물이 포함되어 있습니다.DNA Pol II는 세포 내에서 약 30-50개의 많은 양의 복사를 가지고 있는 반면, 세포 내 DNA Pol III의 수치는 5배 더 적다.
다른 B군 중합효소와의 유사성
대부분의 중합효소는 유사한 구조와 기능을 바탕으로 군으로 분류되었다.DNA Pol II는 인간 DNA Polα, β, β, β와 함께 B군에 속한다.이것들은 모두 RB69, 9°N-7 및 Tgo의 상동어입니다.그룹 B의 다른 구성원들은 DNA Pol II를 [10]독특하게 만드는 적어도 하나의 다른 서브유닛을 가지고 있다.
기능.
공정.
중합효소는 모두 DNA 복제에 어느 정도 관여하며 핵산의 사슬을 합성한다.DNA 복제는 세포 증식의 중요한 측면이다.DNA를 복제하지 않으면 세포는 분열하여 자손에게 유전 정보를 공유할 수 없다.대장균과 같은 원핵생물에서, DNA Pol III는 DNA 복제와 관련된 주요 중합효소이다.DNA Pol II는 염색체 복제의 주요 인자는 아니지만, 채워야 할 다른 역할이 있다.
DNA Pol II는 DNA 복제에 참여합니다.DNA Pol III만큼 빠르지 않을 수도 있지만, 효과적인 효소를 만드는 몇 가지 능력이 있습니다.이 효소는 프리마아제 활성과 함께 3µ→5µ 엑소핵산가수분해효소 활성을 가진다.DNA Pol II는 치환오차율이 2×10−6 이하, 프레임시프트오차율이 1×10−6 이하인 고충실성 효소이다.DNA Pol II는 Pol III로 인한 불일치를 교정하고 처리할 수 있습니다.바나흐-올로프스카 외 연구진은 DNA Pol II가 복제에 관여하지만 가닥에 의존하며 지연 가닥을 우선적으로 복제한다는 것을 보여주었다.제안된 메커니즘에 따르면 DNA Pol III가 정지하거나 기능하지 않게 되면 DNA Pol II가 복제 지점으로 특이하게 모집되어 [1]복제를 계속할 수 있다.
UV 손상에서 산화까지 DNA가 손상되는 방법은 다양하기 때문에 이러한 손상을 고치기 위해 다양한 종류의 중합효소가 있는 것은 당연하다.DNA Pol II의 한 가지 중요한 역할은 가닥간 교차 고리를 복구하기 위한 주요 중합효소이다.스트랜드 간 교잡은 세포독성 병변을 만드는 질소 머스터드 및 솔라렌과 같은 화학물질에 의해 발생합니다.화학 물질에 의해 양쪽 DNA 가닥이 손상되어 양쪽 가닥의 유전 정보가 잘못되었기 때문에 이러한 병변을 복구하는 것은 어렵다.이러한 스트랜드 간 크로스 링크가 어떻게 고정되는지에 대한 정확한 메커니즘은 아직 연구되고 있지만, Pol II가 매우 [10]관여하고 있는 것으로 알려져 있습니다.
활동
DNA Pol II는 가장 많이 연구된 중합효소가 아니기 때문에 이 효소의 기능은 모두 기능할 가능성이 높지만 궁극적으로 [1]확인되지 않은 여러 가지 기능이 있습니다.
- 자외선 조사에 의해 손상된 DNA 복구
- 자외선 조사 대장균 복제 재개
- 적응 돌연변이 유발
- 장기 생존
메커니즘
DNA 복제 중 염기쌍은 순차적으로 손상될 수 있습니다.손상된 DNA 배열로 인해 [11]복제가 중단될 수 있습니다.DNA Pol II는 염기서열의 오류를 수정하기 위해 뉴클레오티드 염기쌍의 수복을 촉매한다.시험관내 연구에 따르면 Pol II는 때때로 Pol III 부속 단백질(β-clamp 및 클램프 부하 복합체)과 상호작용하여 성장하는 초기 [1][12][13][14]가닥에 대한 Pol II의 접근을 제공한다.DNA 복제 중 DNA Pol II의 기능에 관해, 이것은 폴 III가 생성하는 실수가 보수적인 가닥이 아닌 성장하는 가닥에 있을 것이기 때문에 이치에 맞는다.DNA Pol II의 N 말단 도메인은 DNA 가닥을 촉매 서브유닛으로 연결 및 분리하는 역할을 합니다.DNA Pol II의 N 말단 도메인에는 단일 가닥 DNA를 인식하는 두 개의 사이트가 있을 가능성이 높습니다.한 사이트는 DNA Pol II에 단일 가닥 DNA를 모집하는 역할을 하고 다른 사이트는 DNA Pol [15]II에서 단일 가닥 DNA 해리를 담당한다.
기질 결합 시, DNA Pol II는 DNA의 수소 결합 구조를 유지하기 위해 뉴클레오시드 트리포스페이트와 결합한다.그런 다음 올바른 dNTP가 결합되고 효소 복합체는 하위 도메인과 아미노산 잔기의 입체구조 변화를 겪는다.이러한 구조 변경으로 수리 합성의 속도가 [16]빨라집니다.활성 부위에는 촉매 아스파르트산 D419 및 D547에 [17]의해 안정화되는 두 개의2+ Mg 이온이 포함되어 있습니다.마그네슘 이온은 개방 상태에서 dNTP와 함께 DNA에 결합하고 촉매 작용을 하기 위해 활성 부위 아미노산 잔류물의 구조 변화를 조정한다(폐쇄 상태).마그네슘 이온이 방출되면 효소는 열린 [18]상태로 돌아갑니다.
종 분포
원핵생물
DNA 중합효소 II는 중합효소 B족에 속하며, 3' 말단에서 5' [19]말단으로 이동하는 DNA 복제에서 중합효소 III를 지지한다.복제 오류 중에 중합효소 III가 멈춘 경우, 중합효소 II는 불일치 염기를 중단하고 제거할 수 있다.중합효소 II는 중합효소 III보다 충실도 인자가 훨씬 높기 때문에 오쌍을 만들 가능성이 훨씬 낮습니다.중합효소 II의 교정 단계가 없다면, 중합효소 III는 잘못된 페어링을 확장하여 [1]돌연변이를 일으킬 것이다.
중합효소 III에 의해 야기될 수 있는 돌연변이로부터 보호하는 것 외에, 중합효소 II는 중합효소 IV에 의해 야기되는 돌연변이로부터 보호하는 기능을 합니다.중합효소 IV는 중합효소 II보다 오류 발생 가능성이 훨씬 높지만 3' 말단부터 짝이 맞지 않는 염기쌍을 복구하는 기능도 한다.중합효소 II는 중합효소 IV로부터 3µ 말단을 보호하여 작용하지 못하게 한다.이 보호는 중합효소 II가 정상적으로 작동하는 동안 돌연변이가 생기는 것을 방지합니다.중합효소 II가 돌연변이에 의해 녹아웃되거나 다른 요인에 의해 비활성화된 경우, 중합효소 IV가 짝을 이루지 못한 [1]염기를 고정하는 역할을 합니다.
진핵생물
중합효소 II는 B 계열의 진핵생물들과 함께 자연적으로 기능하지는 않지만, 유사한 구조 및 기능 모티브를 공유합니다.B족에는 중합효소α, α, β, β 및 β가 포함된다.이들 중합효소는 모두 새로 합성된 DNA를 3µ→5µ 방향으로 교정하는 기능을 한다.이러한 중합효소는 선행 및 지연된 가닥 모두에서 DNA를 합성할 수 있습니다.이 중합효소의 등급은 DNA [19]합성 중에 발생하는 모든 오쌍을 교정할 수 있게 해주는 매우 정확한 경향이 있다.
규정
DNA 중합효소 II는 자연적으로 세포에 풍부하며, 이것은 보통 중합효소 III의 양보다 5배나 더 많다.이렇게 함량이 커지면 폴리머라아제 II가 잘못 페어링된 경우 폴리머라아제 III를 제압할 수 있습니다.이 양은 폴리머라아제 II의 양이 약 7배 증가하도록 polB 유전자를 상향 조정하는 SOS 반응 유도 시 증가할 수 있습니다.중합효소 II는 두 가닥 모두에서 작동할 수 있지만, 선행 [1]가닥보다 지연 가닥을 선호하는 것으로 나타났습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ a b c d e f g h Banach-Orlowska M, Fijalkowska IJ, Schaaper RM, Jonczyk P (October 2005). "DNA polymerase II as a fidelity factor in chromosomal DNA synthesis in Escherichia coli". Molecular Microbiology. 58 (1): 61–70. doi:10.1111/j.1365-2958.2005.04805.x. PMID 16164549.
- ^ a b Kornberg T, Gefter ML (September 1970). "DNA synthesis in cell-free extracts of a DNA polymerase-defective mutant". Biochemical and Biophysical Research Communications. 40 (6): 1348–55. doi:10.1016/0006-291X(70)90014-8. PMID 4933688.
- ^ a b Moses RE, Richardson CC (December 1970). "A new DNA polymerase activity of Escherichia coli. I. Purification and properties of the activity present in E. coli polA1". Biochemical and Biophysical Research Communications. 41 (6): 1557–64. doi:10.1016/0006-291X(70)90565-6. PMID 4922636.
- ^ a b Knippers R (December 1970). "DNA polymerase II". Nature. 228 (5276): 1050–3. Bibcode:1970Natur.228.1050K. doi:10.1038/2281050a0. PMID 4921664. S2CID 4211529.
- ^ Lehman IR, Bessman MJ, Simms ES, Kornberg A (July 1958). "Enzymatic synthesis of deoxyribonucleic acid. I. Preparation of substrates and partial purification of an enzyme from Escherichia coli". The Journal of Biological Chemistry. 233 (1): 163–70. doi:10.1016/S0021-9258(19)68048-8. PMID 13563462.
- ^ Smith DW, Schaller HE, Bonhoeffer FJ (May 1970). "DNA synthesis in vitro". Nature. 226 (5247): 711–3. Bibcode:1970Natur.226..711S. doi:10.1038/226711a0. PMID 4910150. S2CID 1505496.
- ^ De Lucia P, Cairns J (December 1969). "Isolation of an E. coli strain with a mutation affecting DNA polymerase". Nature. 224 (5225): 1164–6. Bibcode:1969Natur.224.1164D. doi:10.1038/2241164a0. PMID 4902142. S2CID 4182917.
- ^ Kornberg T, Gefter ML (April 1971). "Purification and DNA synthesis in cell-free extracts: properties of DNA polymerase II". Proceedings of the National Academy of Sciences of the United States of America. 68 (4): 761–4. Bibcode:1971PNAS...68..761K. doi:10.1073/pnas.68.4.761. PMC 389037. PMID 4927672.
- ^ Anderson WF, Prince DB, Yu H, McEntee K, Goodman MF (April 1994). "Crystallization of DNA polymerase II from Escherichia coli". Journal of Molecular Biology. 238 (1): 120–2. doi:10.1006/jmbi.1994.1765. PMID 8145251.
- ^ a b Bebenek K, Kunkel TA (2004). "Functions of DNA polymerases". Advances in Protein Chemistry. 69: 137–65. doi:10.1016/S0065-3233(04)69005-X. ISBN 9780120342693. PMID 15588842.
- ^ Becherel OJ, Fuchs RP (July 2001). "Mechanism of DNA polymerase II-mediated frameshift mutagenesis". Proceedings of the National Academy of Sciences of the United States of America. 98 (15): 8566–71. Bibcode:2001PNAS...98.8566B. doi:10.1073/pnas.141113398. PMC 37476. PMID 11447256.
- ^ Wickner S, Hurwitz J (October 1974). "Conversion of phiX174 viral DNA to double-stranded form by purified Escherichia coli proteins". Proceedings of the National Academy of Sciences of the United States of America. 71 (10): 4120–4. Bibcode:1974PNAS...71.4120W. doi:10.1073/pnas.71.10.4120. PMC 434340. PMID 4610569.
- ^ Hughes AJ, Bryan SK, Chen H, Moses RE, McHenry CS (March 1991). "Escherichia coli DNA polymerase II is stimulated by DNA polymerase III holoenzyme auxiliary subunits". The Journal of Biological Chemistry. 266 (7): 4568–73. doi:10.1016/S0021-9258(20)64360-5. PMID 1999435.
- ^ Bonner CA, Stukenberg PT, Rajagopalan M, Eritja R, O'Donnell M, McEntee K, et al. (June 1992). "Processive DNA synthesis by DNA polymerase II mediated by DNA polymerase III accessory proteins". The Journal of Biological Chemistry. 267 (16): 11431–8. doi:10.1016/S0021-9258(19)49928-6. PMID 1534562.
- ^ Maki S, Hashimoto K, Ohara T, Sugino A (August 1998). "DNA polymerase II (epsilon) of Saccharomyces cerevisiae dissociates from the DNA template by sensing single-stranded DNA". The Journal of Biological Chemistry. 273 (33): 21332–41. doi:10.1074/jbc.273.33.21332. PMID 9694894.
- ^ Beard WA, Wilson SH (May 2014). "Structure and mechanism of DNA polymerase β". Biochemistry. 53 (17): 2768–80. doi:10.1021/bi500139h. PMC 4018062. PMID 24717170.
- ^ Wang F, Yang W (December 2009). "Structural insight into translesion synthesis by DNA Pol II". Cell. 139 (7): 1279–89. doi:10.1016/j.cell.2009.11.043. PMC 3480344. PMID 20064374.
- ^ Yang L, Arora K, Beard WA, Wilson SH, Schlick T (July 2004). "Critical role of magnesium ions in DNA polymerase beta's closing and active site assembly". Journal of the American Chemical Society. 126 (27): 8441–53. doi:10.1021/ja049412o. PMID 15238001.
- ^ a b Mandal A (26 February 2019). "Prokaryotic DNA Polymerases". News Medical.