올리고사카릴전달효소
Oligosaccharyltransferase| STT3 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
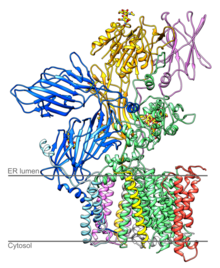 효모 OST의 구조 | |||||||||
| 식별자 | |||||||||
| 기호 | STT3 | ||||||||
| Pfam | PF02516 | ||||||||
| Pfam 씨 | CL0111 | ||||||||
| 인터프로 | IPR003674 | ||||||||
| OPM 슈퍼 패밀리 | 242 | ||||||||
| OPM단백질 | 3rce | ||||||||
| CAZY | GT66 | ||||||||
| 멤브라노메 | 275 | ||||||||
| |||||||||
올리고샤릴전달효소 또는 OST(EC 2.4.1.119)는 14당류의 올리고당류를 돌리콜에서 초기 단백질로 전달하는 막단 단백질 복합체다.그것은 글리코실전달효소의 일종이다.설탕 GlcManGlcNAc392(여기서 Glc=Glucose, Man=Mannose, GlcNAc=N-acetyllucosamine)는 Asn-X-Ser 또는 Asn-X-Thr의 순서로 아스파라긴(Asn-X-Thr) 잔류물에 부착된다.이 염기서열을 글리코실화 세컨이라고 한다.OST에 의해 촉매되는 반응은 N-연계 글리코실레이션 경로의 중심 단계다.null
위치

OST는 소포체 망막(ER)에 있는 반투명체의 성분이다.지질연계 코어-올리고사카라이드는 내포체성 망막에서 조립되어 올리고자릴전달효소 콤플렉스에 의해 초기 폴리펩타이드 체인의 선택된 아스파라긴 잔류물로 전달된다.[3]OST의 활성 부위는 ER 막의 내강면에서 약 4nm 떨어져 있다.[4]null
이것은 보통 초기 단백질이 ER로 들어오기 때문에 번역하는 동안 작용하지만, 그럼에도 불구하고 이 동사성 글리코실레이션을 변환 후 수정이라고 부른다.번역이 완료된 후 OST 활동에 대한 몇 가지 예가 발견되었다.[5][6]단백질이 잘 접히지 않거나 천천히 접히면 변환 후 활동이 일어날 수 있다는 것이 현재의 의견이다.[6]null
구조 및 기능
효모 OST는 3개의 서브콤플렉스에 8개의 서로 다른 막간 신장 단백질로 구성되어 있다(그 중 하나가 OST4).[7][8]이 8진법은 고차적 과점체를 형성하지 않으며, 8개의 단백질 중 3개가 글리코실화된다.[7]포유류의 OST는 구성이 비슷한 것으로 알려져 있다.[9][10]null
OST는 다음과 같은 이유로 많은 서브유닛이 필요한 것으로 생각된다.[11]
- 반투명 모공 근처에 위치한다.
- 올리고사카릴돌리콜을 인식하고 묶어라.
- 세컨을 인식하고 바인딩하기 위해 초기 단백질을 스캔하십시오.
- 이 두 개의 큰 기판을 적절한 위치와 순응으로 옮기십시오.
- 실제 올리고당 전달을 위해 Asn amide 질소 원자를 활성화한다.
- 기판을 풀어줘
OST의 촉매 활성 서브 유닛을 STT3라고 한다.eukaryotes에는 STT3A와 STT3B라고 불리는 두 개의 파라로그가 존재한다. STT3A는 내포체성 망막의 루멘에 들어갈 때 초기의 폴리펩타이드의 등변성 글리코실화를 주로 담당한다. 반면 STT3B는 또한 후전성 글리코실화를 중재할 수 있다.[12]STT3의 원핵동어인 PglB의 구조가 해결되었다.[13]원핵과 진핵성 STT3의 높은 염기서열 유사성은 이들의 구조가 유사함을 시사한다.null
임상적 유의성
CDG 신드롬은 글리코실화 경로의 유전적 장애다.결함이 있는 유전자가 GlcManGlcNAc-dolichol392 전구체의 조립이나 전달에 관여하는 효소를 위한 것이라면 "유형 I"로 표기한다.N 연계 글리코실레이션 경로에서 OST의 작용 후 결함 단계가 발생하거나 O 연계 글리코실레이션이 수반되는 경우 "타입 II"로 라벨을 표시한다.[14]null
참고 항목
참조
- ^ Wild R, Kowal J, Eyring J, Ngwa EM, Aebi M, Locher KP (2018). "Structure of the yeast oligosaccharyltransferase complex gives insight into eukaryotic N-glycosylation". Science. 359 (6375): 545–550. Bibcode:2018Sci...359..545W. doi:10.1126/science.aar5140. PMID 29301962.
- ^ Pfeffer S, Dudek J, Gogala M, Schorr S, Linxweiler J, Lang S, Becker T, Beckmann R, Zimmermann R, Förster F (2014). "Structure of the mammalian oligosaccharyl-transferase complex in the native ER protein translocon". Nat. Commun. 5 (5): 3072. Bibcode:2014NatCo...5.3072P. doi:10.1038/ncomms4072. PMID 24407213.
- ^ Zufferey R, Knauer R, Burda P, Stagljar I, te Heesen S, Lehle L, Aebi M (October 1995). "STT3, a highly conserved protein required for yeast oligosaccharyl transferase activity in vivo". EMBO J. 14 (20): 4949–60. doi:10.1002/j.1460-2075.1995.tb00178.x. PMC 394598. PMID 7588624.
- ^ Nilsson IM, von Heijne G (March 1993). "Determination of the distance between the oligosaccharyltransferase active site and the endoplasmic reticulum membrane". J. Biol. Chem. 268 (8): 5798–801. doi:10.1016/S0021-9258(18)53389-5. PMID 8449946.
- ^ Pless DD, Lennarz WJ; Lennarz (January 1977). "Enzymatic conversion of proteins to glycoproteins". Proc. Natl. Acad. Sci. U.S.A. 74 (1): 134–8. Bibcode:1977PNAS...74..134P. doi:10.1073/pnas.74.1.134. PMC 393212. PMID 264667.
- ^ a b Duvet S, Op De Beeck A, Cocquerel L, Wychowski C, Cacan R, Dubuisson J (February 2002). "Glycosylation of the hepatitis C virus envelope protein E1 occurs posttranslationally in a mannosylphosphoryldolichol-deficient CHO mutant cell line". Glycobiology. 12 (2): 95–101. doi:10.1093/glycob/12.2.95. PMID 11886842.
- ^ a b Knauer R, Lehle L (January 1999). "The oligosaccharyltransferase complex from yeast". Biochim. Biophys. Acta. 1426 (2): 259–73. doi:10.1016/S0304-4165(98)00128-7. PMID 9878773.
- ^ Dempski RE, Imperiali B (December 2002). "Oligosaccharyl transferase: gatekeeper to the secretory pathway". Curr Opin Chem Biol. 6 (6): 844–50. doi:10.1016/S1367-5931(02)00390-3. PMID 12470740.
- ^ Nilsson I, Kelleher DJ, Miao Y, Shao Y, Kreibich G, Gilmore R, von Heijne G, Johnson AE (May 2003). "Photocross-linking of nascent chains to the STT3 subunit of the oligosaccharyltransferase complex". J. Cell Biol. 161 (4): 715–25. doi:10.1083/jcb.200301043. PMC 2199356. PMID 12756234.
- ^ Karaoglu D, Kelleher DJ, Gilmore R (October 2001). "Allosteric regulation provides a molecular mechanism for preferential utilization of the fully assembled dolichol-linked oligosaccharide by the yeast oligosaccharyltransferase". Biochemistry. 40 (40): 12193–206. doi:10.1021/bi0111911. PMID 11580295.
- ^ Imperiali B (November 1997). "Protein Glycosylation: The Clash of the Titans". Accounts of Chemical Research. 30 (11): 452–459. doi:10.1021/ar950226k.
- ^ Ruiz-Canada C, Kelleher DJ, Gilmore R (January 2009). "Cotranslational and posttranslational N-glycosylation of polypeptides by distinct mammalian OST isoforms". Cell. 136 (2): 272–83. doi:10.1016/j.cell.2008.11.047. PMC 2859625. PMID 19167329.
- ^ Lizak C, Gerber S, Numao S, Aebi M, Locher KP (June 2011). "X-ray structure of a bacterial oligosaccharyltransferase". Nature. 474 (7351): 350–5. doi:10.1038/nature10151. PMID 21677752. S2CID 205225231.
- ^ Marquardt T, Denecke J (June 2003). "Congenital disorders of glycosylation: review of their molecular bases, clinical presentations and specific therapies". Eur. J. Pediatr. 162 (6): 359–79. doi:10.1007/s00431-002-1136-0. PMID 12756558. S2CID 3196550.
외부 링크
- 미국 국립 의학 도서관의 올리고차릴트랜스퍼레이즈(MesH(MeSH)

