내인성 레트로바이러스
Endogenous retrovirus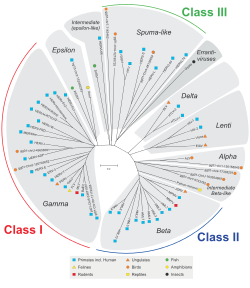
내인성 레트로바이러스(ERV)는 레트로바이러스와 매우 흡사하고 레트로바이러스에서 파생될 수 있는 게놈 내인성 바이러스 요소이다.그것들은 턱이 있는 척추동물의 게놈에 풍부하며, 인간 게놈의 5~8%를 차지한다(낮은 추정치는 약 1%).[1][2]
EERV는 수직으로 유전되는 프로바이러스 배열이며 트랜스포존이라고 불리는 유전자의 하위 등급으로, 유전자 발현과 [3][4]조절에 중요한 역할을 하기 위해 일반적으로 게놈 내에서 포장되고 이동할 수 있습니다.그러나 EERV는 대부분의 트랜스포존 기능이 부족하고, 일반적으로 감염되지 않으며, 종종 레트로바이러스 [5][6]복제 주기의 결함이 있는 게놈 잔재이다.그것들은 숙주 세포의 핵 게놈에 통합 및 역전사되기 때문에 생식선 프로바이러스의 역원소로 구별된다.
연구원들은 레트로바이러스가 클래스 I [7]요소인 레트로트랜스포존이라고 불리는 트랜스포존으로부터 진화했다고 제안했다; 이 유전자들은 돌연변이를 일으킬 수 있고 게놈의 다른 위치로 이동하는 대신에 그들은 외인성 또는 병원성이 될 수 있다.이는 모든 EERV가 레트로바이러스에 의한 삽입으로 시작된 것이 아니라,[8] 일부 EERV가 레트로바이러스와 유사한 레트로바이러스의 유전자 정보의 원천일 수 있다는 것을 의미한다.바이러스 DNA의 통합이 배아줄에서 일어날 때, 그것은 나중에 숙주 [1][9]집단의 유전자 풀에 고정될 수 있는 EERV를 발생시킬 수 있다.
형성
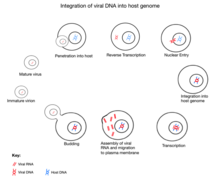
레트로 바이러스의 복제 사이클은 바이러스 게놈의 DNA 복사본을 숙주 세포의 핵 게놈에 삽입("통합")하는 것을 수반합니다.대부분의 레트로바이러스는 체세포를 감염시키지만, 간혹 생식세포(난자와 정자를 생산하는 세포)의 감염도 발생할 수 있다.드물게, 레트로바이러스 통합은 생존 가능한 유기체로 발전하는 생식세포에서 일어날 수 있다.이 유기체는 삽입된 레트로바이러스 게놈을 자신의 게놈의 필수적인 부분인 "내생" 레트로바이러스(ERV)를 가지고 다니며, 새로운 대립 유전자로 자손에 의해 유전될 수 있다.많은 ERV가 수백만 년 동안 숙주의 게놈을 유지해왔다.그러나 이들 대부분은 숙주 DNA 복제 중에 불활성화 돌연변이를 획득하여 더 이상 바이러스를 생성할 수 없습니다.EERVs는 또한 새롭게 통합된 레트로바이러스의 측면에 있는 동일한 배열 사이의 재조합이 바이러스 게놈의 내부 단백질 코드 영역을 결실시키는 재조합 결실이라고 알려진 과정에 의해 게놈에서 부분적으로 제거될 수 있다.
일반적인 레트로바이러스 게놈은 바이러스 게놈의 침입, 복제, 탈출, 확산에 필수적인 세 개의 유전자로 구성되어 있다.이 세 가지 유전자는 gag (바이러스 코어의 구조 단백질 코드), pol (역전사효소, 인테그라아제, 프로테아제 코드), env (바이러스 외부를 위한 외피 단백질 코드)입니다.이 바이러스 단백질은 폴리프로틴으로 암호화되어 있다.레트로바이러스는 그들의 라이프 사이클을 수행하기 위해 숙주 세포의 기계에 크게 의존한다.단백질 분해효소는 바이러스성 폴리단백질의 펩타이드 결합을 분해하여 분리된 단백질을 기능시킨다.역전사효소는 숙주 세포의 세포질에 있는 바이러스 RNA가 핵으로 들어가기 전에 바이러스 DNA를 합성하는 기능을 한다.Integase는 바이러스 DNA를 숙주 [9][10]게놈으로 통합하는 것을 안내합니다.
시간이 지남에 따라 EERV의 게놈은 포인트 돌연변이를 획득할 뿐만 아니라 다른 EERV와 [11]섞이고 재결합한다.환경용 감쇠 시퀀스를 가진 EERV가 전파될 가능성이 [12]높아집니다.
게놈 진화에서의 역할
내인성 레트로바이러스는 게놈 형성에 적극적인 역할을 할 수 있다.이 지역의 대부분의 연구는 인간과 고등 영장류의 게놈에 초점을 맞췄지만, 생쥐와 양과 같은 다른 척추 동물들도 깊이 [13][14][15][16]있게 연구되었다.ERV 게놈 옆에 있는 긴 말단 반복(LTR) 배열은 종종 대체 촉진제 및 증강제로 작용하며, 종종 조직 특이적 변형을 만들어냄으로써 트랜스크립텀에 기여한다.또한, 레트로바이러스 단백질 자체는 특히 생식 및 발달에서 새로운 숙주 기능을 제공하기 위해 함께 사용되어 왔다.상동성 레트로바이러스 배열 사이의 재조합은 또한 유전자 교환과 유전자 변이의 발생에 기여했다.또한 레트로바이러스 배열의 잠재적인 길항작용의 경우 억제유전자가 이들을 퇴치하기 위해 함께 진화했다.
내인성 레트로바이러스의 약 90%는 모든 ORF(Open Reading Frame)가 없는 단독 LTR입니다.완전한 레트로바이러스 배열과 관련된 단독 LTR과 LTR은 숙주 유전자에 전사 요소로서 작용하는 것으로 나타났다.이들의 작용 범위는 주로 단백질을 코드하는 5' UTR에 삽입하는 것이지만, 70-100kb 떨어진 [13][17][18][19]유전자에 작용하는 것으로 알려져 있다.이들 요소의 대부분은 대응하는 유전자에 대한 감각 방향으로 삽입되지만, LTR이 반감지 방향으로 작용하고 인접 [21][22]유전자에 대한 양방향 프로모터로 작용한다는 증거가[20] 있다.몇몇 경우에, LTR은 유전자의 주요 프로모터로서 기능한다.
예를 들어 인간에서 AMY1C는 프로모터 영역에 완전한 EERV 배열을 가지며, 관련된 LTR은 소화효소 아밀라아제의 [23]침 특이적 발현을 제공한다.또한 담즙대사에 필수적인 효소를 코드하는 담즙산-CoA:아미노산N-아실전달효소(BAAT)의 1차 프로모터가 LTR [18][24]유래이다.
로 렙틴 호르몬 수용체, 또는의 표현에 의해 솔로 ERV-9 내연 관계의 삽입,(IRGM)[25] 보기의 경우 삽입도 유전자에 직접 적분에 의해 대체 접속 사이트들을 일으키는 것으로 나타났다 인간의 면책 특권의 부활 GTP가수 분해 효소 유전자 관련을 불러 일으키고 있는 기능성 열려 독서 프레임 생산해 냈을 것. 한 upstream LTR, 포스포리파아제 A-2와 같은 단백질.[26]
그러나 대부분의 경우 LTR은 많은 대체 프로모터 중 하나로 기능하며, 종종 생식 및 발육과 관련된 조직 고유의 표현을 전달합니다.실제로 알려진 LTR 촉진 전사 변이체의 64%는 생식 [27]조직에서 발현된다.예를 들어, 유전자 CYP19는 에스트로겐 합성에 중요한 효소인 방향화효소 P450을 코드하는데, 이것은 보통 대부분의 [18]포유동물의 뇌와 생식기관에서 발현된다.그러나 영장류에서 LTR에 의해 촉진되는 전사 변종은 태반에 발현을 부여하고 임신 [18]중 에스트로겐 수치를 조절하는 역할을 한다.또한 통상 널리 분포하는 신경아포토시스억제단백질(NAIP)은 고환 및 [28]전립선에 발현을 부여하는 촉진제로서 작용하는 HERV-P족 LTR을 가진다.일산화질소합성효소3(NOS3), 인터류킨2수용체B(IL2RB) 및 에스트로겐 합성의 다른 매개체인 HSD17B1과 같은 다른 단백질도 태반발현을 부여하는 LTR에 의해 대체적으로 조절되지만 이들의 특이적 기능은 [24][29]아직 알려지지 않았다.높은 수준의 생식 발현은 그들이 내생화된 방법의 후유증으로 생각되지만, 이것은 또한 생식선 조직의 [24]DNA 메틸화의 부족 때문일 수도 있다.
태반 단백질 발현의 가장 잘 특징지어지는 예는 대체적으로 촉진되는 숙주 유전자가 아니라 레트로바이러스 단백질의 완전한 공동 선택에서 비롯된다.바이러스 바이러스가 숙주 세포에 침입하는 역할을 하는 레트로바이러스 융해성 환경 단백질은 포유류의 태반 발달에 중요한 영향을 미쳤다.포유동물에서, 신시틴이라고 불리는 온전한 환경 단백질은 신시토포아세포의 [15]형성과 기능을 담당합니다.이 다핵 세포들은 주로 영양소 교환을 유지하고 태아를 엄마의 면역 [15]체계로부터 분리하는 데 책임이 있다.이 기능을 위한 이러한 단백질의 선택과 고정은 태생 [30]진화에 중요한 역할을 했다고 제안되어 왔다.
또한 ERV와 각각의 LTR의 삽입은 염색체 간 궤적에서의 바이러스 배열 간의 재조합에 의한 염색체 재배치를 유도할 가능성이 있다.이러한 재배열은 유전자의 가소성에 크게 기여하고 유전자 기능의 역학을 극적으로 변화시키는 유전자 [31]복제와 결실을 유도하는 것으로 나타났다.또한, 레트로 요소는 일반적으로 스트레스 및 [18]외부 자극에 대한 반응과 크게 관련이 있는 기능이 빠르게 진화하는 포유류 특이 유전자 패밀리에서 널리 퍼져 있다.특히 인간 I급과 II급 MHC 유전자는 모두 다른 다로커스 [26]유전자에 비해 HERV 원소의 밀도가 높다.HERV는 HLA 클래스 1 [32]유전자군을 구성하는 광범위하게 중복된 중복된 블록 형성에 기여하는 것으로 나타났다.보다 구체적으로 HERV는 주로 이들 블록 사이의 중단점 내 및 그 사이의 영역을 차지하고 있으며, 이는 일반적으로 불균등한 크로스오버와 관련된 상당한 복제 및 삭제 이벤트가 이들 [33]블록의 형성을 촉진했음을 시사합니다.면역하플로타입으로 유전되는 이러한 블록의 생성은 인간에게 다른 [32]영장류보다 이점을 줄 수 있는 광범위한 항원에 대한 보호 다형성 역할을 합니다.
다른 종들 사이에서 매우 진화적으로 구별되는 기관인 태반의 특징은 ERV 강화제의 공동 선택에서 비롯된다고 제안되어 왔다.호르몬과 성장 인자를 코드하는 유전자의 돌연변이 대신, 조절 돌연변이는 특히 호르몬과 성장인자 유전자의 대부분이 태반 발달이 아닌 임신에 반응하여 발현되기 때문에 태반 형태학의 알려진 진화를 지지한다.연구자들은 서로 밀접한 관련이 있는 두 종인 쥐와 쥐 사이의 태반 발달의 조절 경관을 연구했다.이는 쥐 영양아세포(TSC)의 모든 조절 요소를 매핑하고 마우스 TSC의 각 조절 요소와 비교함으로써 이루어졌다.TSC는 태아 태반에서 발달하는 초기 세포를 반영하기 때문에 관찰되었다.그들의 명백한 유사점과 상관없이, 증강제와 억압된 지역은 대부분 특정 종에 특유했다.그러나 대부분의 프로모터 배열은 생쥐와 쥐 사이에 보존되었다.연구의 결론으로, 연구원들은 EERV가 태반 성장, 면역 억제, 그리고 세포 [34]융합의 중재를 통해 종별 태반 진화에 영향을 미쳤다고 제안했다.
세포 메커니즘을 이용하는 EERV의 또 다른 예는 종양억제유전자(TSG)인 p53이다.DNA 손상과 세포 스트레스는 p53 경로를 유도하여 세포자멸을 초래한다.염기서열 분석과 함께 크로마틴 면역 침강을 사용하여, 모든 p53 결합 부위의 30%가 몇몇 영장류 특이적 ERV 패밀리의 복사본 내에 위치했다.한 연구는 p53의 메커니즘이 숙주 [7]세포에서 바이러스 RNA가 빠져나가는 빠른 전사의 유도를 제공하기 때문에 이것이 레트로바이러스에 도움이 된다고 제안했다.
마지막으로, EERVs 또는 EERV 요소를 숙주 DNA의 유전자 영역에 삽입하거나 그들의 전사 변이체의 과잉 발현은 양성보다 유해한 효과를 발생시킬 가능성이 훨씬 높다.그들의 게놈으로의 출현은 억제제 유전자의 복제와 확장을 증식시키는 숙주-기생충 공진화 역학을 만들어냈다.이것의 가장 명확한 예는 포유류의 게놈에서 탠덤 아연 손가락 유전자의 빠른 복제와 증식을 포함한다.아연 손가락 유전자, 특히 KRAB 도메인을 포함하는 유전자는 척추동물의 게놈에 높은 복사수로 존재하며, 그 기능 범위는 전사적 [35]역할로 제한된다.그러나 포유동물에서 이러한 유전자의 다양화는 새로운 레트로바이러스 배열이나 그들의 [19]전사를 억제하기 위한 내인성 복제에 반응하여 여러 번 복제되고 고정된 사건들에 기인한 것으로 나타났다.
질병에서의 역할
척추동물의 게놈에서 발생하는 EERV의 대부분은 고대이고 돌연변이에 의해 활성화되지 않으며 그들의 숙주 종에서 유전적 고정에 도달했다.이러한 이유로 인해 비정상적인 상황을 제외하고 호스트에 부정적인 영향을 미칠 가능성은 매우 낮습니다.그럼에도 불구하고, 새와 쥐, 고양이, 코알라를 포함한 비인간 포유동물 종에 대한 연구에서 더 어린 (즉, 더 최근에 통합된) ERV가 [36]질병과 연관될 수 있다는 것이 명백하다.포유류의 게놈에서 활성 EERV의 수는 [37]암 발생을 통한 페토의 역설에 기여하는 신체 크기와 부정적인 관련이 있다.이것은 비록 결정적인 증거가 [38][39][40][41]부족하지만, 연구자들이 인간 암과 자가면역 질환의 여러 형태에서 ERV의 역할을 제안하도록 이끌었다.
신경 장애
사람의 경우, ERV는 다발성 경화증(MS)에 관여하는 것으로 제안되고 있다.MS와 [42][43]ERV 삽입에서 파생된 ERVWE1, 즉 "syncytin" 유전자 사이의 특정 연관성은 MS 관련 레트로바이러스(MSRV)의 존재와 함께 보고되었다.인간 ERV(Human ERVs)는 또한 ALS와 [45][46][47]중독에 관련되어[44] 있다.
2004년에는 HERV에 대한 항체가 정신분열증 환자의 혈청에서 더 자주 발견되었다고 보고되었다.또한 최근 정신분열증 발병자의 뇌척수액에는 레트로바이러스 마커, 역전사효소 수치가 대조군 [48]피험자보다 4배 높았다.연구자들은 HERVs와 정신분열증 사이의 연관성을 계속 관찰하고 있으며, [49]정신분열증을 유발하는 감염의 추가적인 가능성에도 주목하고 있다.
면역
ERV는 질병을 일으키는 관계뿐만 아니라 면역력을 통해서도 질병과 관련이 있는 것으로 밝혀졌다.장기 말단 반복(LTR)에서 EERV의 빈도는 바이러스 전사와 복제를 촉진하는 면역 신호 경로를 이용하기 위한 바이러스 적응과 관련이 있을 수 있다.2016년 실시된 연구는 선천성 [50]면역의 한 분야인 간섭자에 의해 유도되는 유전자 조절 네트워크를 통해 숙주에 통합된 고대 바이러스 DNA의 이점을 조사했다.이들 사이토카인은 바이러스 감염에 반응하는 첫 번째 물질이며 악성세포에 [51]대한 면역 감시에도 중요하다.EERV는 시스 조절 요소로 작용할 것으로 예상되지만, 특정 생리 기능에 대한 적응 결과의 대부분은 아직 알려지지 않았다.인간 간섭 반응, 특히 간섭 감마(IFNG)에 대한 조절에서 EERV의 일반적인 역할을 뒷받침하는 데이터가 있다.예를 들어 간섭자 자극 유전자는 CD14+[1] 대식세포에서 신호 변환기와 전사 1(STAT1) 및/또는 간섭자 조절인자(IRF1)의 활성제에 의해 결합된 EERV로 크게 농축된 것으로 밝혀졌다.
HERV는 또한 인간 선천적 면역 반응을 형성하는 다양한 역할을 하며, 일부 시퀀스는 시스템을 활성화하고 다른 시퀀스는 이를 억제한다.그들은 또한 외인성 레트로바이러스 감염으로부터 보호할 수 있다: 바이러스 유사 성분은 패턴 인식 수용체를 활성화 시킬 수 있고, 단백질은 활성 레트로바이러스를 방해할 수 있다.HERV-K(HML2)로부터의 개그 단백질이 HIV Gag와 혼합되어 결과적으로 [52]HIV 캡시드 형성을 저해하는 것으로 나타났다.
유전자 조절
제안된 또 다른 아이디어는 같은 계열의 EERV가 동일한 규제 네트워크로 여러 유전자를 모집하는 역할을 했다는 것이다.MER41 요소는 STAT1 결합 [1]부위 근처에 위치한 유전자에 추가적인 중복 조절 강화를 제공하는 것으로 밝혀졌다.
의학의 역할
돼지내인성 레트로바이러스
인간의 경우, 돼지 내인성 레트로바이러스(PERV)는 이종 이식에 돼지 조직과 장기를 사용할 때, 한 종의 유기체에서 다른 종의 유기체로 살아있는 세포, 조직 및 장기를 이식할 때 우려를 제기한다.비록 돼지는 일반적으로 가장 적합한 기증자 인간의 장기 질병을 치료하는 것은 실용적, 재정 안전 때문에, 윤리적 reasons,[50]PERVs 이전에 돼지로부터, 그들의 바이러스성 능력이 호스트의 게놈 속에와 자식에 통과될 통합하기 때문에, 올해 2017년, 실험실, CRISPR-Cas9을 사용하여 제거했다까지 제거할 수 없다. 모든.돼지 [53]게놈에서 62개의 레트로바이러스가 나왔어요이종 간 전염의 결과는 아직 연구되지 않은 채 위험한 [54]가능성을 가지고 있다.
연구자들은 특히 면역력이 저하된 개인에게서 PERV에 의한 인체 조직의 감염이 매우 가능성이 있다고 지적했다.면역 억제 상태는 잠재적으로 바이러스 DNA의 더 빠르고 끈질긴 복제를 가능하게 할 수 있고, 나중에 인간 대 인간 전염에 적응하는 데 어려움을 덜 수 있을 것이다.공여 장기/조직에 존재하는 알려진 감염성 병원균은 무병원체 군집을 번식시킴으로써 제거할 수 있지만, 공여체에는 알려지지 않은 레트로바이러스가 존재할 수 있다.이러한 레트로바이러스는 종종 기증자에게 잠재되어 증상이 없지만, 기증자에게서 활성화 될 수 있습니다.인간 세포에서 감염되고 증식할 수 있는 내생 바이러스의 예로는 개코원숭이, 고양이, 그리고 [50]쥐가 있다.
PERV에는 PERV-A, PERV-B 및 PERV-C의 세 가지 다른 클래스가 있다. PERV-A와 PERV-B는 다방성이며 체외에서 인체 세포를 감염시킬 수 있는 반면 PERV-C는 생태성이 있어 인체 세포에 복제되지 않는다.등급 간의 주요 차이점은 각 등급의 복제에 영향을 미치는 환경 단백질의 수용체 결합 도메인과 긴 말단 반복(LTRs)이다.PERV-A 및 PERV-B는 U3 영역에서 반복되는 LTR을 표시합니다.단, PERV-A 및 PERV-C에서는 반복 없는 LTR이 표시됩니다.연구원들은 배양 내 PERV가 숙주 세포가 수행할 수 있는 최고의 복제 성능을 맞추기 위해 LTR의 반복 구조에 능동적으로 적응한다는 것을 발견했습니다.연구의 마지막에, 연구원들은 반복 없는 PERV LTR이 반복 하버링 LTR에서 진화했다고 결론지었다.이는 삽입 돌연변이에 의해 발생했을 가능성이 높으며 LTR 및 환경/Env의 데이터 사용을 통해 입증되었다.반복 없는 LTR의 생성은 바이러스의 적응 과정을 반영하여 외인성 [55]생활 방식에서 내인성 생활 방식으로 변화할 수 있다고 생각된다.
1999년에 수행된 임상시험 연구는 다른 살아있는 돼지 조직으로 치료된 160명의 환자를 표본으로 추출했으며, PERV 시퀀스의 증폭을 위해 충분한 양의 DNA를 사용할 수 있는 환자의 97%에서 지속적인 PERV 감염의 증거를 관찰하지 못했다.그러나 이 연구는 소급 연구가 감염의 실제 발생률 또는 관련 임상 증상을 찾는 데 제한적이라고 밝혔다.이는 가능한 이종 간 PERV 전염에 대한 보다 완전하고 상세한 평가와 [56]PERV의 비교를 제공하는 면밀하게 모니터링되는 사전 시험을 사용할 것을 제안했다.
인간 내인성 레트로바이러스
인간 내인성 레트로바이러스(HERV)는 인간 게놈의 중요한 부분을 차지하며, 약 98,000개의 ERV 요소와 단편들이 5~8%[1]를 차지한다.2005년에 발표된 연구에 따르면, 복제 가능한 HERV는 확인되지 않았다.; 모두 중대한 결손이나 무의미한 돌연변이를 포함한 결함이 있는 것으로 보인다(HERV-K에는 해당되지 않는다).이것은 대부분의 HERV가 수백만 년 전에 처음으로 통합된 원래의 바이러스의 흔적에 불과하기 때문입니다.HERV 통합 분석은 10만 게놈 [57]프로젝트의 일환으로 진행 중입니다.
인간 내인성 레트로바이러스는 원래 인간 [1]게놈 라이브러리가 동물 레트로바이러스의 프로브를 사용하거나 바이러스 배열과 유사한 올리고뉴클레오티드를 사용하여 낮은 끈 조건에서 스크리닝되었을 때 발견되었다.
분류
HERV는 동물 레트로바이러스에 대한 상동성을 바탕으로 분류된다.Class I에 속하는 과는 포유동물 Gammaretrovirus(타입 C) 및 Epilonretrovirus(타입 E)와 순서가 유사하다.Class II에 속하는 과는 포유류 베타레트로바이러스(타입 B)와 델라레트로바이러스(타입 D)에 대한 호몰로지를 나타낸다.Class III에 속하는 패밀리는 거품 바이러스와 유사하다.모든 계층에서, 만약 동질성이 개그, 폴리, 그리고 환경 유전자에 잘 보존된 것으로 보인다면, 그들은 슈퍼 패밀리로 분류된다.존재하는 것으로 알려진 Class I 패밀리가 더 있습니다.[1][11]외인성 레트로바이러스, 프라이밍 tRNA(HERV-W, K), 또는 인접 유전자(HERV-ADP), 클론 번호(HERV-S71) 또는 제안된 아미노산 모티프(HERV-FRD)를 기반으로 한 명칭의 혼합과 함께 패밀리 자체는 덜 통일된 방식으로 명명된다.
기원.
인간의 진화 중에, HERV의 외생적인 조상들은 배아줄 세포에 자신들을 삽입하고 숙주의 세포 메커니즘을 사용하고 이용하는 숙주의 유전자와 함께 복제되었다.그들의 독특한 게놈 구조 때문에, HERVs는 여러 차례의 증폭과 전이를 겪었고, 이는 레트로바이러스 DNA의 [1]더 광범위한 분포로 이어졌다.
그럼에도 불구하고 인간과 침팬지의 다양성 이후 한 과의 바이러스가 활발하게 활동하고 있다.HERV-K(HML2)라고 불리는 이 계열은 HERV 요소의 1% 미만을 구성하지만 가장 많이 연구되고 있는 계열 중 하나이다.지난 수십만 년 동안 HML2가 활성화되었다는 징후가 있습니다. 예를 들어, 일부 개인은 다른 [59]개인보다 더 많은 HML2 복사본을 가지고 있습니다.전통적으로 HERV의 연령 추정은 HERV의 5'와 3' LTR을 비교하여 수행되지만, 이 방법은 전장 HERV에만 관련이 있다. 단면 [60]연대로 불리는 최근 방법은 단일 LTR 내의 변화를 사용하여 HERV 삽입 연령을 추정한다.이 방법은 HERV 경과시간을 보다 정확하게 추정할 수 있으며 모든 HERV 삽입에 사용할 수 있습니다.단면연대는 HERV-K(HML2)의 두 구성원인 HERV-K106과 HERV-K116이 지난 80만 년 동안 활동했고 HERV-K106이 15만 년 전에 [61]현생인류를 감염시켰을 수 있다는 것을 암시하기 위해 사용되어 왔다.그러나 HERV-K(HML2) 패밀리의 알려진 감염성 구성원의 부재와 공개된 인간 게놈 배열 내에서 완전한 코딩 잠재력을 가진 요소들의 부족은 일부 사람들에게 현재 패밀리가 덜 활성화될 가능성을 시사한다.2006년과 2007년에 프랑스와 미국에서 독립적으로 일하는 연구원들은 HERV-K(HML2)[62][63]의 기능 버전을 재현했다.
HERV 단백질 발현
HERV의 생물학적 활성 계열인 HERV-K의 발현은 태반에서 발견되는 단백질을 생산한다.또한 HERV-W(HERVW-1) 및 HERV-FRD(HERV-FRD-1)의 외피유전자의 발현에 의해 세포융합을 [64]유도함으로써 태반형성 시 신시토포아세포층 생성에 중요한 신시틴이 생성된다.Hugo Gene Nomenclature Committee(HGNC)는 전사된 인간 ERV에 [65]대한 유전자 기호를 승인합니다.
기능에 미치는 영향
MER41.AIM2는 외래 세포질 DNA 센서를 암호화하는 AIM2(Melanoma 2)의 전사를 조절하는 HERV입니다.이는 AIM2의 결합부위로서 작용하며, 이는 AIM2의 전사에 필요하다는 것을 의미한다.연구진은 MER41을 삭제함으로써 이를 증명했다.CRISPR/Cas9를 사용하는 Hela 세포에서 AIM2를 검출할 수 없는 전사 레벨로 합니다.MER41이 여전히 들어있던 제어 셀.AIM2 EERV는 정상적인 양의 AIM2 트랜스크립트에서 관찰되었습니다.면역성의 측면에서, 연구자들은 MER41로 결론지었다.AIM2는 [66]감염에 대한 염증 반응을 위해 필요하다.
HERV에 대한 면역 반응
면역학 연구는 HIV에 감염된 사람들의 [67]HERV에 대한 T세포 면역 반응에 대한 몇 가지 증거를 보여 주었다.HIV가 HIV에 감염된 세포에서 HERV 발현을 유도한다는 가설은 HERV 항원을 대상으로 한 백신이 HIV에 감염된 세포를 구체적으로 제거할 수 있다는 제안을 이끌어냈다.이 새로운 접근법의 잠재적인 장점은 HERV 항원을 HIV에 감염된 세포의 대리표지로서 사용함으로써, 악명높은 다양성과 빠른 돌연변이를 가진 HIV [67]항원을 직접 겨냥하는 데 내재된 어려움을 피할 수 있다는 것이다.
EERV의 특성화 기술
전체 게놈 배열 분석
예: 중국 태생의 돼지 ERV(PERV) 미니피그 분리체인 PERV-A-BM은 유전적 변이와 진화를 이해하기 위해 다른 품종 및 세포주와 함께 완전히 배열되었습니다.관찰된 뉴클레오티드 치환 횟수와 다양한 게놈 배열 중 연구자들이 PERV-A-BM이 유럽 태생의 돼지 분리체보다 [54]진화 연령이 빠른 숙주 게놈에 통합된 추정 연령을 결정하는 데 도움을 주었다.
염기서열을 포함한 염색질 면역침강(ChIP-seq)
이 기술은 DNA 단백질의 결합 부위인 촉진제 및 인핸서, 억제된 영역 및 [34]트리메틸화를 나타내는 히스톤 마크를 찾기 위해 사용된다.DNA 메틸화는 쥐의 체세포에서 ERV의 사일런싱을 유지하는 데 필수적인 것으로 나타났으며, 히스톤 마크는 배아줄기세포(ESC)와 초기 배아 [7]발생에서 동일한 목적을 위해 필수적이다.
적용들
계통 발생의 구축
대부분의 HERV는 기능이 없고 선택적으로 중성이며 영장류 게놈이 풍부하기 때문에 연계 분석을 위한 계통 발생학적 지표 역할을 쉽게 한다.그것들은 통합부위 다형성 또는 진화하는 프로바이러스성 뉴클레오티드 서열을 비교함으로써 이용될 수 있다.언제 통합이 일어났는지 추정하기 위해, 연구원들은 각각의 특정한 궤적에서 분자 진화의 속도를 찾기 위해 각 계통수로부터의 거리를 사용했다.또한 ERV는 다양한 계통 발생학적 [9]질문에 대한 해답을 위해 사용될 수 있기 때문에 많은 종 게놈(식물, 곤충, 연체동물, 물고기, 설치류, 가축)이 풍부하다는 점도 유용하다.
프로바이러스의 나이와 종분리 발생 시점 지정
이것은 다른 진화기간의 다른 HERV를 비교함으로써 달성된다.예를 들어, 이 연구는 인간에서 유인원, 그리고 원숭이까지 다양한 인간들을 대상으로 이루어졌다.이는 [55]PERV에서는 큰 다양성이 존재하기 때문에 하기 어렵습니다.
추가 조사
후생성 변이
연구자들은 후생유전학적 방출과 인간 질병과의 잠재적 연관성을 통해 휴면 트랜스포저블 요소의 재활성화와 유전자 조절 [7]네트워크의 특이성을 연구하기 위해 개별 후생유전자와 트랜스포텀을 분석할 수 있었다.
이종 이식의 면역학적 문제
기증자 상피에서 갈락토실-알파1-3갈라토실(alpha-Gal)[50] 항원을 인식하는 이종반응성 항체에 의해 개시되는 보체의 활성화에 이은 초급성 거부(HAR)를 극복하는 효과적인 방법에 대해서는 거의 알려져 있지 않다.
유전자 치료에서 HERV의 위험인자
레트로바이러스는 서로 그리고 다른 내인성 DNA 배열과 재조합할 수 있기 때문에, HERV가 야기할 수 있는 잠재적 위험을 탐구하는 것이 유전자 치료에 유익할 것이다.또한 HERV의 재조합 능력은 레트로바이러스 벡터에 [1]HERV 배열을 포함시킴으로써 부위 지향적 통합을 위해 조작할 수 있다.
HERV유전자발현
연구자들은 HERV 유전자에 의해 암호화된 RNA와 단백질은 세포 생리학 및 병리학적 조건에서의 추정 기능을 위해 계속 연구되어야 한다고 믿는다.이것은 [1]합성된 단백질의 생물학적 중요성을 더 깊게 정의하기 위해 검토하는 것이 이치에 맞을 것이다.
「 」를 참조해 주세요.
- 조류육종백혈증바이러스(ASLV)
- 내인성 바이러스 원소
- ERV3
- HERV-FRD
- 수평 유전자 이동
- 자크시크테양 레트로바이러스(JSRV)
- 코알라 레트로바이러스(KoRV)
- 마우스유방종양바이러스(MMTV)
- 쥐백혈병 바이러스(MLV) 및 이방성 쥐백혈병 바이러스 관련 바이러스(XMRV)
- 고생물학
레퍼런스
- ^ a b c d e f g h i j Belshaw R, Pereira V, Katzourakis A, Talbot G, Paces J, Burt A, Tristem M (April 2004). "Long-term reinfection of the human genome by endogenous retroviruses". Proc Natl Acad Sci USA. 101 (14): 4894–99. Bibcode:2004PNAS..101.4894B. doi:10.1073/pnas.0307800101. PMC 387345. PMID 15044706.
- ^ Nelson PN, Hooley P, Roden D, Davari Ejtehadi H, Rylance P, Warren P, et al. (October 2004). "Human endogenous retroviruses: transposable elements with potential?". Clinical and Experimental Immunology. 138 (1): 1–9. doi:10.1111/j.1365-2249.2004.02592.x. PMC 1809191. PMID 15373898.
- ^ Khodosevich K, Lebedev Y, Sverdlov E (October 2002). "Endogenous retroviruses and human evolution". Comparative and Functional Genomics. 3 (6): 494–98. doi:10.1002/cfg.216. PMC 2448423. PMID 18629260.
- ^ Kim FJ, Battini JL, Manel N, Sitbon M (January 2004). "Emergence of vertebrate retroviruses and envelope capture". Virology. 318 (1): 183–91. doi:10.1016/j.virol.2003.09.026. PMID 14972546.
- ^ Stoye JP, Boeke JD (1997). "Retrotransposons, Endogenous Retroviruses, and the Evolution of Retroelements". Retroviruses. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. p. 343. ISBN 9780879695712. PMID 21433351. NBK19468. Retrieved 2021-02-22.
- ^ Gifford RJ, Blomberg J, Coffin JM, Fan H, Heidmann T, Mayer J, Stoye J, Tristem M, Johnson WE (2018-09-28). "Nomenclature for endogenous retrovirus (ERV) loci". Retrovirology. 15 (1): 59. doi:10.1186/s12977-018-0442-1. PMC 6114882. PMID 30153831.
- ^ a b c d Rebollo R, Romanish MT, Mager DL (2012-01-01). "Transposable elements: an abundant and natural source of regulatory sequences for host genes". Annual Review of Genetics. 46 (1): 21–42. doi:10.1146/annurev-genet-110711-155621. PMID 22905872.
- ^ Cotton, J. (2001). "Retroviruses from retrotransposons". Genome Biology. 2 (2): 6. doi:10.1186/gb-2001-2-2-reports0006.
It appears that the transition from nonviral retrotransposon to retrovirus has occurred independently at least eight times, and the source of the envelope gene responsible for infectious ability can now be traced to a virus in at least four of these instances. This suggests that potentially, any LTR retrotransposon can become a virus through the acquisition of existing viral genes.
- ^ a b c Johnson WE, Coffin JM (August 1999). "Constructing primate phylogenies from ancient retrovirus sequences". Proceedings of the National Academy of Sciences of the United States of America. 96 (18): 10254–60. Bibcode:1999PNAS...9610254J. doi:10.1073/pnas.96.18.10254. PMC 17875. PMID 10468595.
- ^ Fujiwara T, Mizuuchi K (August 1988). "Retroviral DNA integration: structure of an integration intermediate". Cell. 54 (4): 497–504. doi:10.1016/0092-8674(88)90071-2. PMID 3401925. S2CID 37801365.
- ^ a b Vargiu L, Rodriguez-Tomé P, Sperber GO, Cadeddu M, Grandi N, Blikstad V, et al. (January 2016). "Classification and characterization of human endogenous retroviruses; mosaic forms are common". Retrovirology. 13: 7. doi:10.1186/s12977-015-0232-y. PMC 4724089. PMID 26800882.
- ^ Magiorkinis G, Gifford RJ, Katzourakis A, De Ranter J, Belshaw R (May 2012). "Env-less endogenous retroviruses are genomic superspreaders". Proceedings of the National Academy of Sciences of the United States of America. 109 (19): 7385–7390. doi:10.1073/pnas.1200913109. PMC 3358877. PMID 22529376.
- ^ a b Li J, Akagi K, Hu Y, Trivett AL, Hlynialuk CJ, Swing DA, Volfovsky N, Morgan TC, Golubeva Y, Stephens RM, Smith DE, Symer DE (March 2012). "Mouse endogenous retroviruses can trigger premature transcriptional termination at a distance". Genome Res. 22 (5): 870–84. doi:10.1101/gr.130740.111. PMC 3337433. PMID 22367191.
- ^ Spencer TE, Palmarini M (2012). "Endogenous retroviruses of sheep: a model system for understanding physiological adaptation to an evolving ruminant genome". J Reprod Dev. 58 (1): 33–37. doi:10.1262/jrd.2011-026. PMID 22450282.
- ^ a b c Black SG, Arnaud F, Palmarini M, Spencer TE (2010). "Endogenous Retroviruses in Trophoblast Differentiation and Placental Development". American Journal of Reproductive Immunology. 64 (4): 255–64. doi:10.1111/j.1600-0897.2010.00860.x. PMC 4198168. PMID 20528833.
- ^ Ryan FP (December 2004). "Human endogenous retroviruses in health and disease: a symbiotic perspective". Journal of the Royal Society of Medicine. 97 (12): 560–65. doi:10.1177/014107680409701202. PMC 1079666. PMID 15574851.
- ^ Pi W, Zhu X, Wu M, Wang Y, Fulzele S, Eroglu A, Ling J, Tuan D (July 2010). "Long-range function of an intergenic retrotransposon". PNAS. 107 (29): 12992–97. Bibcode:2010PNAS..10712992P. doi:10.1073/pnas.1004139107. PMC 2919959. PMID 20615953.
- ^ a b c d e van de Lagemaat LN, Landry JR, Mager DL, Medstrand P (October 2003). "Transposable elements in mammals promote regulatory variation and diversification of genes with specialized functions". Trends Genet. 19 (10): 530–36. doi:10.1016/j.tig.2003.08.004. PMID 14550626.
- ^ a b Kovalskaya E, Buzdin A, Gogvadze E, Vinogradova T, Sverdlov E (2006). "Functional human endogenous retroviral LTR transcription start sites are located between the R and U5 regions". Virology. 346 (2): 373–78. doi:10.1016/j.virol.2005.11.007. PMID 16337666.
- ^ Hata K, Okano M, Lei H, Li E (2002). "Dnmt3L cooperates with the Dnmt3 family of de novo DNA methyltransferases to establish maternal imprints in mice". Development. 129 (8): 1983–1993. doi:10.1242/dev.129.8.1983. PMID 11934864.
- ^ Dunn CA, Romanish MT, Gutierrez LE, van de Lagemaat LN, Mager DL (2006). "Transcription of two human genes from a bidirectional endogenous retrovirus promoter". Gene. 366 (2): 335–42. doi:10.1016/j.gene.2005.09.003. PMID 16288839.
- ^ Gogvadze E, Stukacheva E, Buzdin A, Sverdlov E (2009). "Human-specific modulation of transcriptional activity provided by endogenous retroviral insertions". J Virol. 83 (12): 6098–105. doi:10.1128/JVI.00123-09. PMC 2687385. PMID 19339349.
- ^ Ting CN, Rosenberg MP, Snow CM, Samuelson LC, Meisler MH (1992). "Endogenous retroviral sequences are required for tissue-specific expression of a human salivary amylase gene". Genes Dev. 6 (8): 1457–65. doi:10.1101/gad.6.8.1457. PMID 1379564.
- ^ a b c Cohen CJ, Lock WM, Mager DL (2009). "Endogenous retroviral LTRs as promoters for human genes: a critical assessment". Gene. 448 (2): 105–14. doi:10.1016/j.gene.2009.06.020. PMID 19577618.
- ^ Bekpen C, Marques-Bonet T, Alkan C, Antonacci F, Leogrande MB, Ventura M, Kidd JM, Siswara P, Howard JC, Eichler EE (2009). "Death and resurrection of the human IRGM gene". PLOS Genet. 5 (3): e1000403. doi:10.1371/journal.pgen.1000403. PMC 2644816. PMID 19266026.
- ^ a b Jern P, Coffin JM (2008). "Effects of Retroviruses on Host Genome Function". Annu Rev Genet. 42: 709–32. doi:10.1146/annurev.genet.42.110807.091501. PMID 18694346.
- ^ Oliver KR, Greene WK (2011). "Mobile DNA and the TE-Thrust hypothesis: supporting evidence from the primates". Mob DNA. 2 (1): 8. doi:10.1186/1759-8753-2-8. PMC 3123540. PMID 21627776.
- ^ Romanish MT, Lock WM, van de Lagemaat LN, Dunn CA, Mager DL (2007). "Repeated recruitment of LTR retrotransposons as promoters by the anti- apoptotic locus NAIP during mammalian evolution". PLOS Genet. 3 (1): e10. doi:10.1371/journal.pgen.0030010. PMC 1781489. PMID 17222062.
- ^ Huh JW, Ha HS, Kim DS, Kim HS (2008). "Placenta-restricted expression of LTR- derived NOS3". Placenta. 29 (7): 602–08. doi:10.1016/j.placenta.2008.04.002. PMID 18474398.
- ^ Villarreal LP, Villareal LP (February 1997). "On viruses, sex, and motherhood". J. Virol. 71 (2): 859–65. doi:10.1128/JVI.71.2.859-865.1997. PMC 191132. PMID 8995601.
- ^ Hughes JF, Coffin JM (December 2001). "Evidence for genomic rearrangements mediated by human endogenous retroviruses during primate evolution". Nature Genetics. 29 (4): 487–89. doi:10.1038/ng775. PMID 11704760. S2CID 20262212.
- ^ a b Dawkins R, Leelayuwat C, Gaudieri S, Tay G, Hui J, Cattley S, et al. (February 1999). "Genomics of the major histocompatibility complex: haplotypes, duplication, retroviruses and disease". Immunological Reviews. 167: 275–304. doi:10.1111/j.1600-065X.1999.tb01399.x. PMID 10319268. S2CID 9924684.
- ^ Doxiadis GG, de Groot N, Bontrop RE (July 2008). "Impact of endogenous intronic retroviruses on major histocompatibility complex class II diversity and stability". Journal of Virology. 82 (13): 6667–77. doi:10.1128/JVI.00097-08. PMC 2447082. PMID 18448532.
- ^ a b Chuong EB, Rumi MA, Soares MJ, Baker JC (March 2013). "Endogenous retroviruses function as species-specific enhancer elements in the placenta". Nature Genetics. 45 (3): 325–29. doi:10.1038/ng.2553. PMC 3789077. PMID 23396136.
- ^ Thomas JH, Schneider S (2011). "Coevolution of retroelements and tandem zinc finger genes". Genome Res. 21 (11): 1800–12. doi:10.1101/gr.121749.111. PMC 3205565. PMID 21784874.
- ^ McEwen GK, Alquezar-Planas DE, Dayaram A, Gillett A, Tarlinton R, Mongan N, et al. (February 2021). "Retroviral integrations contribute to elevated host cancer rates during germline invasion". Nature Communications. 12 (1): 1316. Bibcode:2021NatCo..12.1316M. doi:10.1038/s41467-021-21612-7. PMC 7910482. PMID 33637755.
- ^ Katzourakis A, Magiorkinis G, Lim AG, Gupta S, Belshaw R, Gifford R (July 2014). "Larger mammalian body size leads to lower retroviral activity". PLOS Pathogens. 10 (7): e1004214. doi:10.1371/journal.ppat.1004214. PMC 4102558. PMID 25033295.
- ^ Bannert N, Kurth R (October 2004). "Retroelements and the human genome: new perspectives on an old relation". Proceedings of the National Academy of Sciences of the United States of America. 101 (Suppl 2): 14572–79. Bibcode:2004PNAS..10114572B. doi:10.1073/pnas.0404838101. PMC 521986. PMID 15310846.
- ^ Nelson PN, Carnegie PR, Martin J, Davari Ejtehadi H, Hooley P, Roden D, et al. (February 2003). "Demystified. Human endogenous retroviruses". Molecular Pathology. 56 (1): 11–18. doi:10.1136/mp.56.1.11. PMC 1187282. PMID 12560456.
- ^ Singh SK (June 2007). "Endogenous retroviruses: suspects in the disease world". Future Microbiology. 2 (3): 269–75. doi:10.2217/17460913.2.3.269. PMID 17661701.
- ^ Magiorkinis G, Belshaw R, Katzourakis A (September 2013). "'There and back again': revisiting the pathophysiological roles of human endogenous retroviruses in the post-genomic era". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 368 (1626): 20120504. doi:10.1098/rstb.2012.0504. PMC 3758188. PMID 23938753.
- ^ Mameli G, Astone V, Arru G, Marconi S, Lovato L, Serra C, et al. (January 2007). "Brains and peripheral blood mononuclear cells of multiple sclerosis (MS) patients hyperexpress MS-associated retrovirus/HERV-W endogenous retrovirus, but not Human herpesvirus 6". The Journal of General Virology. 88 (Pt 1): 264–74. doi:10.1099/vir.0.81890-0. PMID 17170460.
- ^ Serra C, Mameli G, Arru G, Sotgiu S, Rosati G, Dolei A (December 2003). "In vitro modulation of the multiple sclerosis (MS)-associated retrovirus by cytokines: implications for MS pathogenesis". Journal of Neurovirology. 9 (6): 637–43. doi:10.1080/714044485. PMID 14602576.
- ^ "Reactivated Virus May Contribute to ALS". 2016-01-23.
- ^ Karamitros T, Hurst T, Marchi E, Karamichali E, Georgopoulou U, Mentis A, et al. (October 2018). "RASGRF2 is associated with intravenous drug abuse and modulates transcription in a cell-line model". Proceedings of the National Academy of Sciences of the United States of America. 115 (41): 10434–39. doi:10.1073/pnas.1811940115. PMC 6187174. PMID 30249655.
- ^ Rob Picheta (25 September 2018). "Addiction may stem from ancient retrovirus, study says". CNN. Retrieved 2019-10-13.
- ^ "An ancient virus may promote addiction in modern people". The Economist. 2018-10-02. ISSN 0013-0613. Retrieved 2019-10-13.
- ^ Yolken R (June 2004). "Viruses and schizophrenia: a focus on herpes simplex virus". Herpes. 11 (Suppl 2): 83A–88A. PMID 15319094.
- ^ Fox D (2010). "The Insanity Virus". Discover. Retrieved 2011-02-17.
- ^ a b c d Patience C, Takeuchi Y, Weiss RA (March 1997). "Infection of human cells by an endogenous retrovirus of pigs". Nature Medicine. 3 (3): 282–86. doi:10.1038/nm0397-282. PMID 9055854. S2CID 33977939.
- ^ Platanias LC (May 2005). "Mechanisms of type-I- and type-II-interferon-mediated signalling". Nature Reviews. Immunology. 5 (5): 375–86. doi:10.1038/nri1604. PMID 15864272.
- ^ Grandi N, Tramontano E (2018). "Human Endogenous Retroviruses Are Ancient Acquired Elements Still Shaping Innate Immune Responses". Frontiers in Immunology. 9: 2039. doi:10.3389/fimmu.2018.02039. PMC 6139349. PMID 30250470.
- ^ Niu D, Wei HJ, Lin L, George H, Wang T, Lee IH, Zhao HY, Wang Y, Kan Y, Shrock E, Lesha E, Wang G, Luo Y, Qing Y, Jiao D, Zhao H, Zhou X, Wang S, Wei H, Güell M, Church GM, Yang L (September 2017). "Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9". Science. 357 (6357): 1303–07. Bibcode:2017Sci...357.1303N. doi:10.1126/science.aan4187. PMC 5813284. PMID 28798043.
- ^ a b Tang HB, Ouyang K, Rao GB, Ma L, Zhong H, Bai A, et al. (2016-01-01). "Characterization of Complete Genome Sequences of a Porcine Endogenous Retrovirus Isolated From China Bama Minipig Reveals an Evolutionary Time Earlier Than That of Isolates From European Minipigs". Transplantation Proceedings. 48 (1): 222–28. doi:10.1016/j.transproceed.2015.12.005. PMID 26915872.
- ^ a b Tönjes RR, Niebert M (November 2003). "Relative age of proviral porcine endogenous retrovirus sequences in Sus scrofa based on the molecular clock hypothesis". Journal of Virology. 77 (22): 12363–68. doi:10.1128/JVI.77.22.12363-12368.2003. PMC 254287. PMID 14581574.
- ^ Paradis K, Langford G, Long Z, Heneine W, Sandstrom P, Switzer WM, et al. (August 1999). "Search for cross-species transmission of porcine endogenous retrovirus in patients treated with living pig tissue. The XEN 111 Study Group". Science. 285 (5431): 1236–41. doi:10.1126/science.285.5431.1236. PMID 10455044.
- ^ "Genomics England › Integrated Pathogens and Mobile Elements GeCIP Domain". Retrieved 2019-10-13.
- ^ Gifford RJ, Blomberg J, Coffin JM, Fan H, Heidmann T, Mayer J, et al. (August 2018). "Nomenclature for endogenous retrovirus (ERV) loci". Retrovirology. 15 (1): 59. doi:10.1186/s12977-018-0442-1. PMC 6114882. PMID 30153831.
- ^ Belshaw R, Dawson AL, Woolven-Allen J, Redding J, Burt A, Tristem M (October 2005). "Genomewide Screening Reveals High Levels of Insertional Polymorphism in the Human Endogenous Retrovirus Family HERV-K(HML2): Implications for Present-Day Activity". J. Virol. 79 (19): 12507–14. doi:10.1128/JVI.79.19.12507-12514.2005. PMC 1211540. PMID 16160178.
- ^ Jha AR, Pillai SK, York VA, Sharp ER, Storm EC, Wachter DJ, Martin JN, Deeks SG, Rosenberg MG, Nixon DF, Garrison KE (August 2009). "Cross-Sectional Dating of Novel Haplotypes of HERV-K 113 and HERV-K 115 Indicate These Proviruses Originated in Africa before Homo sapiens". Mol Biol Evol. 26 (11): 2617–26. doi:10.1093/molbev/msp180. PMC 2760466. PMID 19666991.
- ^ Jha AR, Nixon DF, Rosenberg MG, Martin JN, Deeks SG, Hudson RR, Garrison KE, Pillai SK (May 2011). "Human Endogenous Retrovirus K106 (HERV-K106) Was Infectious after the Emergence of Anatomically Modern Humans". PLOS ONE. 6 (5): e20234. Bibcode:2011PLoSO...620234J. doi:10.1371/journal.pone.0020234. PMC 3102101. PMID 21633511.
- ^ Lee YN, Bieniasz PD (January 2007). "Reconstitution of an infectious human endogenous retrovirus". PLoS Pathogens. 3 (1): e10. doi:10.1371/journal.ppat.0030010. PMC 1781480. PMID 17257061.
- ^ Dewannieux M, Harper F, Richaud A, Letzelter C, Ribet D, Pierron G, Heidmann T (December 2006). "Identification of an infectious progenitor for the multiple-copy HERV-K human endogenous retroelements". Genome Research. 16 (12): 1548–1556. doi:10.1101/gr.5565706. PMC 1665638. PMID 17077319.
- ^ Morozov VA, Dao Thi VL, Denner J (2013). "The transmembrane protein of the human endogenous retrovirus – K (HERV-K) modulates cytokine release and gene expression". PLOS ONE. 8 (8): e70399. Bibcode:2013PLoSO...870399M. doi:10.1371/journal.pone.0070399. PMC 3737193. PMID 23950929.
- ^ Mayer J, Blomberg J, Seal RL (May 2011). "A revised nomenclature for transcribed human endogenous retroviral loci". Mobile DNA. 2 (1): 7. doi:10.1186/1759-8753-2-7. PMC 3113919. PMID 21542922.
- ^ Chuong EB, Elde NC, Feschotte C (March 2016). "Regulatory evolution of innate immunity through co-option of endogenous retroviruses". Science. 351 (6277): 1083–87. Bibcode:2016Sci...351.1083C. doi:10.1126/science.aad5497. PMC 4887275. PMID 26941318.
- ^ a b Garrison KE, Jones RB, Meiklejohn DA, Anwar N, Ndhlovu LC, Chapman JM, Erickson AL, Agrawal A, Spotts G, Hecht FM, Rakoff-Nahoum S, Lenz J, Ostrowski MA, Nixon DF (November 2007). "T cell responses to human endogenous retroviruses in HIV-1 infection". PLOS Pathog. 3 (11): e165. doi:10.1371/journal.ppat.0030165. PMC 2065876. PMID 17997601.
추가 정보
- Löwer R, Löwer J, Kurth R (May 1996). "The viruses in all of us: characteristics and biological significance of human endogenous retrovirus sequences". Proc Natl Acad Sci USA. 93 (11): 5177–84. Bibcode:1996PNAS...93.5177L. doi:10.1073/pnas.93.11.5177. PMC 39218. PMID 8643549.
- Molès JP, Tesniere A, Guilhou JJ (Jul 2005). "A new endogenous retroviral sequence is expressed in skin of patients with psoriasis". Br J Dermatol. 153 (1): 83–89. doi:10.1111/j.1365-2133.2005.06555.x. PMID 16029331. S2CID 6642536.
- Seifarth W, Frank O, Zeilfelder U, Spiess B, Greenwood AD, Hehlmann R, Leib-Mösch C (Jan 2005). "Comprehensive analysis of human endogenous retrovirus transcriptional activity in human tissues with a retrovirus-specific microarray". J. Virol. 79 (1): 341–52. doi:10.1128/JVI.79.1.341-352.2005. PMC 538696. PMID 15596828.
- Knerr I, Beinder E, Rascher W (Feb 2002). "Syncytin, a novel human endogenous retroviral gene in human placenta: evidence for its dysregulation in preeclampsia and HELLP syndrome". Am J Obstet Gynecol. 186 (2): 210–13. doi:10.1067/mob.2002.119636. PMID 11854637.
- Gifford R, Tristem M (May 2003). "The evolution, distribution and diversity of endogenous retroviruses" (PDF). Virus Genes. 26 (3): 291–315. doi:10.1023/A:1024455415443. PMID 12876457. S2CID 34639116. Archived from the original (PDF) on 2013-02-15. Retrieved 2008-01-16.
외부 링크
- 미국 국립 의학 도서관 의학 주제 제목(MeSH)의 내인성+레트로바이러스
- HERVd – 인간 내인성 레트로바이러스 데이터베이스