콜리플라워 모자이크바이러스
Cauliflower mosaic virus| 콜리플라워 모자이크바이러스 | |
|---|---|
| 바이러스 분류 | |
| (랭킹되지 않음): | 바이러스 |
| 영역: | 리보비리아 |
| 킹덤: | 파라나비레아과 |
| 망울: | 아르트베르비리코타 |
| 클래스: | 레브트라비리케테스 |
| 순서: | 오르테르비르목 |
| 패밀리: | 코리모비루스과 |
| 속: | 콜리모바이러스 |
| 종: | 콜리플라워 모자이크바이러스 |
콜리플라워 모자이크바이러스(CaMV)는 콜리모바이러스의 속성으로, 식물을 감염시키는 파라레트로바이러스로, 콜리모비르과의 6개 제네라 중 하나이다.[1] 파라레트로바이러스는 역전사(retrovirus)처럼 역전사를 통해 복제되지만, 바이러스 입자에는 RNA 대신 DNA가 들어 있다.[2]
정의
콜리플라워 모자이크 바이러스(CaMV)는 콜리모비르과의 일원이다. 이 집단은 오르터비랄레스의 순서로 벨파오비르과, 메타비르과, 필로비르과, 레트로비르과(대신 DNA 중간을 통해 RNA 게놈을 복제함)와 함께 그룹화되어 있으며, 헤파드나비르과는 RNA 중간(Colimovirae와 같이)을 통해 복제된 DNA 게놈을 가지고 있음에도 불구하고 보다 먼 관계가 있다.ng는 별도의 순서인 Blubervirales(두 명령 모두 동일한 클래스, Revtraviricetes에 속함)에 속함.
CaMV는 주로 브라시카과의 식물(콜리플라워, 순무 등)을 감염시키지만, 일부 CaMV 변종(D4, W260)도 다투라와 니코티아나의 솔라나과 종을 감염시킬 수 있다. CaMV는 모자이크, 잎 표면의 괴사성 병변, 성장 저해, 전체 식물 구조의 변형 등 다양한 전신 증상을 유도한다. 나타나는 증상은 바이러스성 변종, 숙주 에코타입, 환경 조건에 따라 다르다.[3]
CaMV는 Myzus persicae와 같은 진딧물 종에 의해 비순환적으로 전염된다.[4] 일단 식물 숙주 세포 안에 도입되면, 처녀자리들은 식물 세포의 핵 봉투로 이동한다.
구조
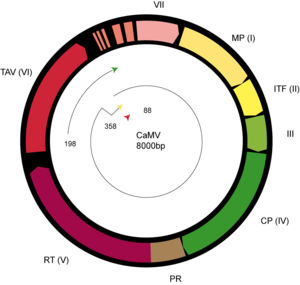
CaMV 입자는 삼각측량 T = 7로 배열된 420 capsid 단백질(CP) 서브유닛에서 직경이 52nm인 이코사헤드론(icosahedron)으로, 용매충전 중앙강을 둘러싸고 있다.[5][6]
CaMV는 약 8.0킬로바이트의 원형 이중 가닥 DNA 분자를 포함하고 있으며, 역전사 중 RNAse H의 작용으로 인해 발생하는 틈새에 의해 중단된다. 이 구멍들은 Met-tRNA에서 나온 것이고, 역전사에 사용되는 RNA 프라이머 2개에서 나온 것이다. 숙주 세포에 들어간 후, 바이러스 DNA에서 고립된 이들 단 하나의 "닉크"가 수리되어 히스톤에 결합하는 슈퍼코일 분자를 형성한다. 이 DNA는 전체 길이, 터미네이션, 35S RNA, 아유전자 19S RNA로 변환된다.
게놈
35S RNA의 발기인은 CaMV 게놈 전체의 전사를 담당하는 매우 강력한 구성 발기인이다. 식물 변형에 사용하는 것으로 잘 알려져 있다. 그것은 디코트 식물에서 높은 수준의 유전자 발현을 유발한다. 그러나 단색동물, 특히 시리얼에서는 덜 효과적이다. 행동의 차이는 아마도 규제 요인의 품질 및/또는 양상의 차이 때문일 것이다. 최근의 연구에 따르면, 사용된 촉진자 요소는 식물의 촉진자 요소와 다르지만, CaMV 35S 촉진자는 일부 동물 세포에서도 기능한다고 한다. 이 프로모터는 정식 동물 프로모터에 비해 활동이 적은 반면, 리포터 상품의 수준은 유의미했다. 이러한 관찰은 35S 프로모터가 동물에서 사용할 수 있는 잠재력을 가질 수 있음을 시사한다.[7]
이 프로모터에 의해 자연스럽게 표현이 추진되는 바이러스성 대본의 침전계수가 35S이기 때문에 프로모터 이름을 CaMV 35S 프로모터("35S 프로모터")라고 지었다. 그것은 가장 널리 사용되는 범용적 발기인 중 하나이다. 그것은 1980년대 초에 츄아와 록펠러 대학의 협력자들에 의해 발견되었다.
35S RNA는 특히 복잡하며, 6~8개의 짧은 읽기 프레임(ORF)을 가진 고도로 구조화된 600개의 뉴클레오티드 긴 리더 시퀀스를 포함하고 있다.[8][9][10]
이 리더는 모든 바이러스성 단백질을 인코딩하는 7개의 긴 ORF가 뒤따른다. 이러한 단백질의 표현 메커니즘은 ORF VI 단백질(19S RNA에 의해 인코딩됨)이 일반적으로 박테리아 mRNA에서만 일어나는 과정인 폴리시스트로닉 35S RNA에 대한 주요 개방형 판독 프레임의 변환 재조정을 제어한다는 점에서 독특하다. TAV 함수는 폴리솜 및 진핵 시작 인자 eIF3와의 연관성에 따라 달라진다.[11]
- ORF I – P1: 이동 단백질(P03545)
- ORF II – P2: 진딧물/곤충 전송 계수(P03548)
- ORF III – P3: 바이러스 관련 단백질(VAP, P03551): 구조 단백질, DNA 결합 기능
- ORF IV – P4: 캡시드 단백질(CP, P03542)
- ORF V – P5: 프로폴(P03554): 단백질 분해효소, 양방향 역분해효소 및 RNaseh
- ORF VI – P6: 트랜스액티브/바이로플라스민(P03559): 포함 차체 형성/트래핑, 기타 기능(문자 참조)
- ORF VII/VIII – 알 수 없음(감염에 필요한 참고 사항, Q83163, Q83164)
- tRNA-Met 바인딩 사이트 포함
P6는 변환 활성화와 포함체 형성에 관한 기능 외에도 P2와 P3와 같은 많은 다른 CaMV 단백질과 상호작용하는 것으로 나타나 바이러스 조립과 진딧물 매개 전달에도 어느 정도 기여할 수 있음을 시사했다. 또한, P6는 P7과 결합하는 것으로 보여졌다; 둘 사이의 상호작용을 조사하면 아직 알려지지 않은 P7의 기능을 해명하는 데 도움이 될 수 있다.[12]
P6의 또 다른 기능은 감염 과정 중에 호스트 비-엑스포러 OF PATOGENESIS Related 1 (NPR1)의 수정을 포함한다. NPR1은 살리실산(SA)과 자스몬산(JA) 의존 신호의 중요한 조절기로 둘 사이의 교차점(crossstalk)과 가장 밀접한 관련이 있다. NPR1의 수정은 SA 의존적 신호를 방지하여 식물 세포의 방어 반응을 억제하는 역할을 한다. 수정된 NPR1은 핵으로 적절하게 이동하며 PR-1 촉진자를 결합할 수 있지만 전사 작업을 시작할 수는 없다. SA 축적을 위해서는 적극적인 NPR1이 필요하기 때문에, 이는 SA의 고갈을 더욱 심화시킨다. P6 수정 NPR1에 의한 SA 종속 신호의 규제는 핵에 국부화되어 있는 반면에, JA 종속 신호의 규제는 본질적으로 세포질이며 COI1 경로를 포함한다. SA의 그것과 대조적으로, 수정된 NPR1이 있는 곳에서 JA 종속 신호는 증가한다.[13]
복제
CaMV 역전사 복제:
- 바이러스 입자는 식물 세포에 들어가 검증되지 않는다. 이 단계에서 바이러스 DNA는 3개의 파편, 즉 스트랜드(α)에 1개, + 스트랜드(β 및 γ)에 2개로 구성되며, 3개의 틈이나 불연속(D1, D2, D3)이 있는 원형 게놈으로 불완전하게 조립된다.
- 바이러스 DNA는 불연속부가 채워진 핵으로 들어간다. 이 시점에서 바이러스 DNA는 또한 호스트 히스톤과 연관되어 미니크로모솜(표시되지 않음)을 형성한다.
- 호스트 DNA 의존 RNA 중합효소 트랜지스터는 35S 프로모터의 바이러스 게놈을 중심으로 내내 35S 프로모터를 능가한다. (이를 통해 결과 RNA에서 35S 프로모터의 복사본 2개가 생성된다.) 또한 19S 프로모터(표시되지 않음)에서 전사가 시작된다.
- 바이러스성 RNA는 숙주 세포질 속으로 통과하며, 거기서 그들이 전사된다.
- tRNA의 3′ 끝은 35S RNA의 5′ 끝 근처에 있는 불연속성 1 (D1)에 해당하는 부지에 도달한다.
- tRNA primes 합성은, 새로운 α 가닥의 바이러스 역반사효소(ORF V에 의해 인코딩됨)에 의한 것이다.
- RNase H는 DNA-RNA 듀플렉스로부터 RNA를 제거하여 DNA를 남긴다.
- 이 새로운 DNA는 RNA 템플릿의 3′ 끝에서 35S 추진자를 묶고 DNA의 α 가닥의 합성이 계속되고 RNase H는 DNA에 복잡한 RNA를 계속 저하시킨다.
- α 가닥의 합성이 완료된다. RNase H 활성도는 dna DNA 가닥의 합성을 프리메이징하는 불연속성 3(D3) 위치에 푸린이 풍부한 부위를 노출한다.
- RNase H 활성도는 β DNA 가닥의 합성을 프리메이징하는 불연속성 2(D2) 위치에 푸린이 풍부한 부위를 노출한다. DNA의 새로운 dna 가닥이 새로운 α 가닥의 5′ 끝단에 도달하면 새로운 α 가닥의 5′ 끝부분으로 전환되어 불연속성 1(D1)을 재현한다. 새로운 γ 가닥의 DNA가 새로운 β 가닥의 5′ 끝단에 도달하면 프라이머와 새로 합성된 β 가닥의 일부를 대체하여 불연속성 2(D2)가 재생된다. DNA의 새로운 β 가닥이 새로운 β 가닥의 5㎛ 끝단에 도달하면 프라이머와 새로 합성된 β 가닥의 일부를 치환하여 불연속성 3(D3)이 재생된다.
이 시점에서 새로운 바이러스 게놈은 캡시드로 포장되어 세포에서 방출되거나 단백질 이동에 의해 인접하고 감염되지 않은 세포로 이동될 수 있다.[14]
콜리플라워 모자이크 바이러스 촉진제(CaMV 35S)는 대부분의 유전자 변형 작물에서 숙주공장에 인위적으로 삽입된 외래 유전자를 활성화하기 위해 사용된다. 그것은 자연 브라시카 식물 숙주에 있을 때 발견되는 것과 다른 형태로 유전자 변형 식물에 삽입된다. 이것은 그것이 그렇지 않으면 불가능할 광범위한 호스트-조직 환경에서 작동할 수 있게 한다.
CaMV는 약 8kb의 이중 스트랜드 DNA 게놈을 함유하고 있으며 구면 입자를 생성한다. CaMV 감염은 전신이며, 심지어 손상된 식물 표면에 접종하면 DNA도 감염된다. CaMV 게놈은 8개의 촘촘히 채워진 유전자를 가지고 있는데, 그 중 2개의 작은 유전자, 즉 유전자 II와 VII만이 비필수적이다. 그 결과 이 두 유전자만이 감염성의 손실 없이 교체/삭제될 수 있다. 또 자연 게놈 크기(8024bp)를 수백bp라도 초과하는 수정된 CaMV 게놈은 처녀자리(virion)로 포장되지 않는다. 이 두 가지 요인은 CaMV에서 클론블의 DNA 삽입 크기를 심각하게 제한한다. 박테리아 디하이드로폴리스 환원효소 DHFR 유전자는 유전자 II 대신 CaMV 게놈으로 성공적으로 복제되어 식물에서 성공적으로 발현되었다.
벡터 매개 CaMV 전송의 분자 메커니즘
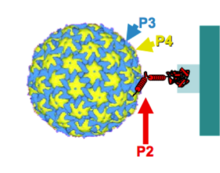
그 바이러스는 진딧물 벡터에 의해 먹이를 주는 동안 감염된 숙주로부터 획득된다. 발생하기 위해, 투과 가능한 콤플렉스는 벡터의 스타일에 위치한 처녀성과 단백질 P2로 구성된다. P2 N단자 영역은 스타일릿의 끝에 위치한 단백질 수용체를 인식하고, P2 C단자 영역은 P3 디코레이션 버전에 결합한다.[15]
벡터에 의한 획득 모드는 조직과 세포내 특이 P2 국소화에 의해 제어된다. 이 단백질은 표피 세포와 실질 조직 세포에서만 발견된다. 더욱이 이들 세포에서 P2는 단일 바이러스성 전자-루크 포함체(ELIB)로 국부화된다.[16] 숙주세포에서 바이러스 단백질 P2와 P3는 처음에는 수많은 바이러스 공장(전자감소포함체)에서 생산되며, 이후 수출되어 마이크로튜브와 공동 국산화한 후 ELIB에 집중된다. CaMV는 특히 마이크로튜브를 사용해 투과 가능한 본체를 형성해 벡터 전송이 가능하다.[17] 이 바이러스의 완전한 분자 특성화와 연구는 더 이상 진행되지 않았다.
플랜트 방어의 회피
콜리플라워 모자이크 바이러스는 숙주 식물 세포 방어에 대항할 수 있는 많은 메커니즘을 가지고 있다. 유전체전 35S RNA가 역분해효소에 의한 게놈 복제를 담당하지만, 바이러스 대방어에 관련된 인자의 생성을 위한 중요한 mRNA 역할을 하는 비코딩 600 베이스 쌍 리더 시퀀스도 포함하고 있다. 많은 CaMV 호스트들은 바이러스 감염을 제한하는 작은 RNA 기반의 바이러스 침묵 메커니즘을 가지고 있다. 전술한 600-bp 시퀀스의 생산물은 21, 22, 24개의 뉴클레오티드의 바이러스성 소형 RNA(vsRNA)로, 아르고네이트 1(AGO1)과 같은 호스트 음소거 기계의 디코이, 결합, 불활성화 효과제 역할을 한다. 원칙 증명으로서, 이러한 vsRNA에 대한 실험적인 과도한 압박은 감염된 식물의 바이러스 축적을 증가시킨다.[18]
유전자이전공장에서 CaMV 35S 프로모터 사용에 대한 우려
2010년대 초에는, 이 프로모터와 P6의 코딩 시퀀스 사이에 시퀀스가 중복되기 때문에, 유전자 변형 식물에서 표현에 CaMV 35S 프로모터를 사용하는 것에 대한 일부 우려가 제기되었다. 미국 출시를 위해 인증된 54개의 유전자 변형 이벤트는 ORF VI(P6의 C-단자 도메인 인코딩)의 최대 528bp를 포함한다.[19] P6는 전체 기능 범위를 알 수 없는 다기능 단백질인 만큼 하나 이상의 영역 표현이 유전자이전생물에 예기치 못한 결과를 가져올 수 있다는 우려도 있다. 최근의 연구들은 전체 프로모터 활동을 유지하면서 P6 도메인을 우발적으로 생산할 가능성이 가장 낮은 CaMV 35S 프로모터 길이를 결정하기 위해 시도했다. 예상할 수 있듯이, 짧은 프로모터 길이를 사용하면 포함된 P6 도메인의 수가 감소하고 원하지 않는 효과의 가능성도 감소한다.[19]
참조
- ^ Pringle, CR. (1999). "Virus taxonomy--1999. The universal system of virus taxonomy, updated to include the new proposals ratified by the International Committee on Taxonomy of Viruses during 1998". Arch Virol. 144 (2): 421–9. doi:10.1007/s007050050515. PMC 7086988. PMID 10470265.
- ^ Rothnie, HM.; Chapdelaine, Y.; Hohn, T. (1994). Pararetroviruses and retroviruses: a comparative review of viral structure and gene expression strategies. Adv Virus Res. Advances in Virus Research. Vol. 44. pp. 1–67. doi:10.1016/s0065-3527(08)60327-9. ISBN 9780120398447. PMID 7817872.
- ^ Khelifa, M.; Massé, D.; Blanc, S.; Drucker, M. (Jan 2010). "Evaluation of the minimal replication time of Cauliflower mosaic virus in different hosts". Virology. 396 (2): 238–45. doi:10.1016/j.virol.2009.09.032. PMID 19913268.
- ^ Brault, V.; Uzest, M.; Monsion, B.; Jacquot, E.; Blanc, S. (2010). "Aphids as transport devices for plant viruses". Comptes Rendus Biologies. 333 (6–7): 524–38. doi:10.1016/j.crvi.2010.04.001. PMID 20541164.
- ^ Cheng, RH.; Olson, NH.; Baker, TS. (Feb 1992). "Cauliflower mosaic virus: a 420 subunit (T = 7), multilayer structure". Virology. 186 (2): 655–68. doi:10.1016/0042-6822(92)90032-k. PMC 4167691. PMID 1733107.
- ^ Haas, M.; Bureau, M.; Geldreich, A.; Yot, P.; Keller, M. (Nov 2002). "Cauliflower mosaic virus: still in the news". Mol Plant Pathol. 3 (6): 419–29. doi:10.1046/j.1364-3703.2002.00136.x. PMID 20569349.
- ^ Tepfer, M.; Gaubert, S.; Leroux-Coyau, M.; Prince, S.; Houdebine, LM. (2004). "Transient expression in mammalian cells of transgenes transcribed from the Cauliflower mosaic virus 35S promoter" (PDF). Environ Biosafety Res. 3 (2): 91–7. doi:10.1051/ebr:2004010. PMID 15612506.
- ^ Fütterer, J.; Gordon, K.; Bonneville, JM.; Sanfaçon, H.; Pisan, B.; Penswick, J.; Hohn, T. (Sep 1988). "The leading sequence of caulimovirus large RNA can be folded into a large stem-loop structure". Nucleic Acids Res. 16 (17): 8377–90. doi:10.1093/nar/16.17.8377. PMC 338565. PMID 3419922.
- ^ Pooggin, MM.; Hohn, T.; Fütterer, J. (May 1998). "Forced evolution reveals the importance of short open reading frame A and secondary structure in the cauliflower mosaic virus 35S RNA leader". J Virol. 72 (5): 4157–69. doi:10.1128/JVI.72.5.4157-4169.1998. PMC 109645. PMID 9557705.
- ^ Hemmings-Mieszczak, M.; Steger, G.; Hohn, T. (Apr 1997). "Alternative structures of the cauliflower mosaic virus 35 S RNA leader: implications for viral expression and replication". J Mol Biol. 267 (5): 1075–88. doi:10.1006/jmbi.1997.0929. PMID 9150397.
- ^ Park, HS.; Himmelbach, A.; Browning, KS.; Hohn, T.; Ryabova, LA. (Sep 2001). "A plant viral reinitiation factor interacts with the host translational machinery". Cell. 106 (6): 723–33. doi:10.1016/S0092-8674(01)00487-1. PMID 11572778. S2CID 14384952.
- ^ Lutz, L.; Raikhy, G.; Leisner, SM. (Dec 2012). "Cauliflower mosaic virus major inclusion body protein interacts with the aphid transmission factor, the virion-associated protein, and gene VII product". Virus Res. 170 (1–2): 150–3. doi:10.1016/j.virusres.2012.08.017. PMC 4215633. PMID 22982205.
- ^ Love, AJ.; Geri, C.; Laird, J.; Carr, C.; Yun, BW.; Loake, GJ.; Tada, Y.; Sadanandom, A.; Milner, JJ. (2012). "Cauliflower mosaic virus protein P6 inhibits signaling responses to salicylic acid and regulates innate immunity". PLOS ONE. 7 (10): e47535. Bibcode:2012PLoSO...747535L. doi:10.1371/journal.pone.0047535. PMC 3469532. PMID 23071821.
- ^ Laliberté, JF.; Sanfaçon, H. (2010). "Cellular remodeling during plant virus infection". Annu Rev Phytopathol. 48: 69–91. doi:10.1146/annurev-phyto-073009-114239. PMID 20337516.
- ^ Hoh, F.; Uzest, M.; Drucker, M.; Plisson-Chastang, C.; Bron, P.; Blanc, S.; Dumas, C. (May 2010). "Structural insights into the molecular mechanisms of cauliflower mosaic virus transmission by its insect vector". J Virol. 84 (9): 4706–13. doi:10.1128/JVI.02662-09. PMC 2863735. PMID 20181714.
- ^ Martinière, A.; Zancarini, A.; Drucker, M. (Jun 2009). "Aphid transmission of cauliflower mosaic virus: the role of the host plant". Plant Signal Behav. 4 (6): 548–50. doi:10.4161/psb.4.6.8712. PMC 2688309. PMID 19816139.
- ^ Martinière, A.; Gargani, D.; Uzest, M.; Lautredou, N.; Blanc, S.; Drucker, M. (Apr 2009). "A role for plant microtubules in the formation of transmission-specific inclusion bodies of Cauliflower mosaic virus". Plant J. 58 (1): 135–46. doi:10.1111/j.1365-313X.2008.03768.x. PMC 2688309. PMID 19077170.
- ^ Blevins, T.; Rajeswaran, R.; Aregger, M.; Borah, BK.; Schepetilnikov, M.; Baerlocher, L.; Farinelli, L.; Meins, F.; et al. (Jul 2011). "Massive production of small RNAs from a non-coding region of Cauliflower mosaic virus in plant defense and viral counter-defense". Nucleic Acids Res. 39 (12): 5003–14. doi:10.1093/nar/gkr119. PMC 3130284. PMID 21378120.
- ^ a b Podevin, N.; du Jardin, P. (2012). "Possible consequences of the overlap between the CaMV 35S promoter regions in plant transformation vectors used and the viral gene VI in transgenic plants". GM Crops Food. 3 (4): 296–300. doi:10.4161/gmcr.21406. PMID 22892689.
외부 링크
- 콜리플라워 모자이크 바이러스
- "The CaMV 35S promoter". patentlens.net. Archived from the original on 2008-01-07.