식물 마이크로바이옴
Plant microbiome| 시리즈의 일부 |
| 마이크로바이옴 |
|---|
 |
식물 마이크로바이옴으로도 알려진 식물 마이크로바이옴은 식물의 건강과 생산성에 역할을 하고 있으며 최근 [1][2]몇 년 동안 상당한 관심을 받고 있다.마이크로바이옴은 "물리적 화학적 특성이 뚜렷한 상당히 잘 정의된 서식지를 차지하고 있는 특징적인 미생물 군집"으로 정의되어 왔다.따라서 이 용어는 관련된 미생물을 지칭할 뿐만 아니라 미생물의 활동 영역도 포괄한다."[3][4]
식물은 다양한 미생물 군집과 함께 산다.식물의 미생물이라고 불리는 이 미생물들은 식물 조직의 내부와 외부 둘 다 살고 [5]식물의 생태와 생리학에 중요한 역할을 합니다."핵심 식물 마이크로바이옴은 식물 적합성에 중요한 핵심 미생물 분류군을 구성하고 식물 홀로비온의 [6]적합성에 필수적인 기능 유전자를 포함하는 미생물 분류군의 선택과 농축의 진화적 메커니즘을 통해 확립된 것으로 생각됩니다."
식물 마이크로바이옴은 유전자형, 장기, 종, 건강상태와 같은 식물 자체와 관련된 요소뿐만 아니라 관리, 토지 사용 및 [7]기후와 같은 식물 환경과 관련된 요소들에 의해 형성된다.식물의 건강 상태는 마이크로바이옴에 의해 반영되거나 관련되는 것으로 [8][1][9][2]일부 연구에서 보고되었습니다.
개요

식물과 미생물의 연관성에 대한 연구는 동물과 인간의 마이크로바이옴, 특히 질소와 인의 흡수에 있어 미생물의 역할보다 앞선다.가장 주목할 만한 예는 식물의 뿌리-근육 균근(AM)과 콩뿌리-근육 공생체이며, 둘 다 뿌리가 토양에서 다양한 영양소를 흡수하는 능력에 큰 영향을 미친다.이 미생물들 중 일부는 미생물들에게 공간, 산소, 단백질, 탄수화물을 제공하는 식물 숙주가 없으면 살아남을 수 없습니다.AM 곰팡이와 식물의 관련성은 1842년부터 알려져 왔으며, 육지 식물의 80% 이상이 식물과 관련되어 [11]있습니다.AM 곰팡이는 식물의 [5]가축화에 도움을 준 것으로 생각됩니다.

전통적으로 식물과 미생물의 상호작용 연구는 배양 가능한 미생물에 국한되어 왔다.배양할 수 없었던 수많은 미생물들은 조사되지 않은 채로 남아 있기 때문에 그들의 역할에 대한 지식은 거의 [5]알려지지 않았다.이러한 식물-미생물 상호작용의 유형과 결과를 풀 수 있는 가능성은 생태학자, 진화생물학자,[12][13][1] 식물생물학자 및 농업학자들 사이에서 상당한 관심을 불러일으켰다.최근의 다체학 발전과 대규모 미생물 수집의 확립은 식물 마이크로바이옴의 구성과 다양성에 대한 지식을 극적으로 증가시켰다.메타제노믹스라고 불리는 미생물 군집 전체의 표지자 유전자의 배열은 식물의 미생물군의 계통학적 다양성을 밝혀준다.그것은 또한 식물 마이크로바이옴 군집 [13][5]형성에 책임이 있는 주요 생물 및 비생물 인자에 대한 지식을 더한다.
서로 다른 식물 종과 관련된 미생물 군집의 구성은 식물 종 사이의 계통 발생학적 거리와 관련이 있다. 즉, 가까운 관계가 있는 식물 종은 먼 [14]종보다 더 비슷한 미생물 군집을 갖는 경향이 있다.식물 마이크로바이옴 연구의 초점은 Arabidopsis Thaliana와 같은 모델 식물과 보리(Hordeum vulgare), 옥수수(Zea mays), 쌀(Oryza sativa), 콩(Glycine max), 밀(Tridicumivum)과 나무 미학 [15][2]종과 같은 중요한 경제 작물 종에 집중되었다.
식물 마이크로바이오타
시아노박테리아는 육지 [16][17][18][19]식물과 공생적으로 광범위하게 상호작용하는 미생물의 한 예이다.시아노박테리아는 기공을 통해 식물에 들어가 세포간 공간을 콜로니화해 고리 모양과 세포내 [20]코일을 형성할 수 있다.Anabaena spp.[21][22][23]는 밀과 목화 식물의 뿌리를 식민지화한다.칼로트릭스 SP밀 [22][23]뿌리계에서도 발견되었습니다.밀과 쌀과 같은 단일 식물들은 Nostoc spp.[24][25][26][27]에 의해 식민지화 되었다. 1991년 Ganther와 다른 사람들은 식물 뿌리와 토양에서 Nostoc, Anabaena, Cylindrospermum을 포함한 다양한 헤테로성 질소를 고정하는 시아노박테리아를 분리했다.밀 묘목 뿌리의 평가 결과 아나베나에 의한 뿌리 털의 느슨한 군집화와 [24][28]노스토크에 의한 제한 구역 내의 뿌리 표면의 엄격한 군집화의 두 가지 유형의 연관 패턴이 밝혀졌다.

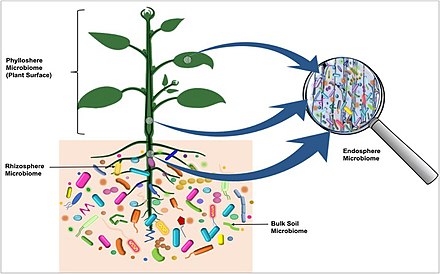
뿌리권 마이크로바이옴
뿌리권은 뿌리 삼출액, 점액 및 죽은 식물 세포의 [31]퇴적을 통해 식물의 영향을 받는 뿌리를 바로 둘러싼 1~10mm의 토양 구역으로 구성된다.박테리아, 곰팡이, 난균류, 선충류, 조류, 원생동물, 바이러스,[32] 고세균을 포함한 다양한 종류의 생물들이 뿌리권에 사는 것을 전문으로 한다.
"실험 증거는 식물의 건강에서 뿌리 마이크로바이옴의 중요성을 강조하고 있으며, 이 식물이 마이크로바이옴의 조성을 제어할 수 있다는 것이 점점 더 분명해지고 있습니다.그들의 생식 성공에 이로운 방법으로 마이크로바이옴을 관리하는 식물들이 진화적 선택 동안 선호될 것이라는 것은 당연하다.이러한 선택적 압력은 식물과 미생물 사이에 많은 특정한 상호작용을 가져온 것으로 보이며, 식물이 필요할 때 미생물의 도움을 필요로 한다는 증거가 축적되고 있습니다."
– Berendsen et al, 2012 [33]
균근균은 뿌리권 공동체의 풍부한 구성원으로 200,000종 이상의 식물에서 발견되었으며 전체 [35]식물의 80% 이상과 관련이 있는 것으로 추정됩니다.균근-뿌리 연합은 영양소와 탄소 순환을 조절함으로써 토지 생태계에서 깊은 역할을 합니다.균근은 질소와 인의 80%까지 필요하기 때문에 식물 건강에 필수적입니다.그 대가로, 곰팡이는 숙주 [36]식물로부터 탄수화물과 지질을 얻는다.염기서열분석 기술을 이용한 근근균에 대한 최근의 연구는 종간과 종내 다양성이 이전에 [37][5]알려진 것보다 더 크다는 것을 보여준다.
가장 자주 연구되는 유익한 뿌리권 유기체는 균류, 뿌리세균, 뿌리세균을 촉진하는 식물성장과 생체조절 미생물이다.토양 1g에 100만 개 이상의 서로 다른 박테리아 [38]게놈이 들어 있을 것으로 예상돼 감자 뿌리권 내에서 [39]5만 개 이상의 OTU가 발견됐다.뿌리권에 있는 원핵생물 중 가장 흔한 박테리아는 산성박테리오타, 유사모나도타, 플랜트균세토타, 방선균세토타, 박테로이드타,[40][41] 바실로타이다.일부 연구에서는 벌크 토양(식물 뿌리에 부착되지 않은 토양)과 뿌리권 [42][43]토양 사이의 미생물 군집 구성에서 유의미한 차이가 보고되지 않았다.특정 박테리아 그룹(예: Actinomycetota, Xanthomonadaceae)은 근처의 벌크 [40][5]토양보다 뿌리권에 덜 풍부합니다.
엽층 마이크로바이옴
식물의 공기 표면(줄기, 잎, 꽃, 과일)은 엽구라고 불리며 뿌리권과 내층권에 비해 상대적으로 영양분이 부족한 것으로 여겨진다.필로스피어의 환경은 뿌리권 및 내층 환경보다 더 역동적이다.미생물 콜로니저는 열, 습기 및 방사선의 주간 및 계절적 변동을 겪습니다.또한, 이러한 환경 요소는 식물 생리학(광합성, 호흡, 수분 흡수 등)에 영향을 미치고 마이크로바이옴 [5]구성에 간접적으로 영향을 미칩니다.비와 바람은 또한 엽층 마이크로바이옴에 [45]일시적인 변화를 일으킨다.
이 미생물들 중 많은 부분에서 식물과 그 관련 미생물들 사이의 상호작용은 숙주 식물의 건강, 기능, 그리고 [46]진화에 중추적인 역할을 할 수 있습니다.잎 표면, 즉 엽층은 박테리아, 곰팡이, 조류, 고세균,[47][48] 바이러스의 다양한 군집으로 이루어진 미생물군을 품고 있습니다.숙주 식물과 식물권 박테리아 사이의 상호작용은 숙주 식물 [49][50][51]생리의 다양한 측면을 촉진할 수 있는 잠재력을 가지고 있다.그러나 2020년 현재, 엽록권에서의 이러한 세균 결합에 대한 지식은 비교적 미미한 수준이며, 엽록권 마이크로바이옴 역학에 대한 [52][53]기초 지식을 발전시킬 필요가 있다.
전반적으로, 식물권 군집에는 높은 종의 풍부함이 남아있다.곰팡이 군집은 온대 지역의 엽록권에서 매우 다양하며 열대 [54]지역보다 더 다양합니다.식물의 잎 표면에는 평방 센티미터 당 107개의 미생물이 존재할 수 있으며, 전 세계적으로 엽구의 세균 개체 수는 [55]10개로 추정된다26.진균성 엽구의 개체 수는 더 [56]적을 것이다.
다른 식물에서 온 엽층 미생물은 높은 수준의 분류군에서는 다소 비슷한 것으로 보이지만, 낮은 수준의 분류군에서는 여전히 상당한 차이가 있다.이것은 미생물이 식물권 [54]환경에서 살아남기 위해 미세하게 조정된 대사 조절이 필요할 수 있다는 것을 나타냅니다.의사도모나도타가 지배적인 콜로나이저인 것으로 보이며, 박테로이드타와 방선균세토타도 또한 식물권에서 [57]우세하다.뿌리권과 토양 미생물 군집 사이에는 유사성이 있지만, 엽층 군집과 외기에 떠 있는 미생물(에어로플랭크톤)[58][5] 사이에는 유사성이 거의 발견되지 않았다.
잎 표면에서 착생 세균 군집으로 엄격하게 정의될 수 있는 엽구 마이크로바이옴의 집합은 주변 환경(예: 확률적 콜로니제이션)과 숙주 식물(예: 생물 선택)[47][59][53]에 존재하는 미생물 군집에 의해 형성될 수 있다.그러나, 잎 표면이 일반적으로 분리된 [60][61]미생물 서식지로 간주되지만, 엽구 마이크로바이옴을 가로지르는 군집결의 지배적인 동인에 대해서는 합의가 이루어지지 않는다.예를 들어, 숙주 특이적 박테리아 군집은 공생하는 식물 종의 엽구에서 보고되어 숙주 [61][62][63][53]선택의 지배적인 역할을 시사한다.
반대로 주변 환경의 마이크로바이옴은 엽층 [60][64][65][66]군집 구성의 주요 결정인자로 보고되었다.그 결과, 식물권 군집 집단을 움직이는 과정은 잘 이해되지 않지만 식물 종에 걸쳐 보편적일 가능성은 낮다.그러나 기존 증거는 숙주 특이적 연관성을 나타내는 엽층 마이크로바이옴이 주변 [49][67][68][69][53]환경에서 주로 모집된 것보다 숙주와 상호작용할 가능성이 더 높다는 것을 보여준다.
숙주와 연관된 미생물 군집에서의 핵심 마이크로바이옴의 탐색은 숙주와 그 [70][71]마이크로바이옴 사이에 일어날 수 있는 상호작용을 이해하기 위한 유용한 첫 번째 단계입니다.지배적인 핵심 마이크로바이옴 개념은 생태적 틈새의 공간적 경계를 가로지르는 분류군의 지속성이 그것이 점유하는 틈새 내에서 그것의 기능적 중요성을 직접적으로 반영한다는 개념에 기초한다. 따라서 그것은 지속적으로 연관된 기능적으로 중요한 미생물을 식별하기 위한 프레임워크를 제공한다.e는 숙주종이다.[70][72][73][53]
"핵심 마이크로바이옴"의 서로 다른 정의는 과학 문헌에서 나타났고, 연구자들은 "핵심 분류군"을 다른 숙주 마이크로바이타트와 심지어 다른 [63][67]종에 걸쳐 지속되는 것으로 다양하게 확인했습니다.로 지속적인 tissue-과 종의 호스트 microbiomes 내에 넓은 지리적 거리를 가로질러, 이 개념적인 framewor의 가장 생물학적으로 그리고 생태학적으로 적절한 응용 프로그램을 나타내는 미생물의 다른 숙주를 가로질러 기능적인 발산[63]과 microhabitats,[76]을 정의하는 핵심 TAXON의 복수 sensu stricto.k.[77][53]넓은 지리적 거리로 분리된 숙주 모집단의 조직 특이적 및 종 특이적 핵심 마이크로바이옴은 루넨이 [50][53]확립한 엄격한 정의를 사용하여 엽록권에 대해 널리 보고되지 않았다.
예:마누카 엽서
흔히 마누카로 알려진 꽃차나무는 뉴질랜드가 [78]원산지입니다.마누카 꽃의 과즙에서 생산되는 마누카 꿀은 비과산화물 항균성으로 [79][80]알려져 있다.이러한 비과산화물 항균 특성은 주로 성숙한 [81][82][83]꿀에서 메틸글리옥살(MGO)로 화학적 변환을 거치는 마누카 꽃의 과즙에 3탄당 디히드록시아세톤(DHA)의 축적과 관련이 있다.그러나 마누카 꽃의 과즙에 DHA의 농도는 변하기 쉬우며, 결과적으로 마누카 꿀의 항균 효과는 지역에 따라 그리고 [84][85][86]매년 다르다.광범위한 연구 노력에도 불구하고 DHA 생산과 기후,[87] 에다픽 [88]또는 숙주 유전자 [89][53]요인 사이에 신뢰할 수 있는 상관관계가 확인되지 않았다.
마누카 뿌리권과 [90][91][92]내층권에서는 미생물이 연구되어 왔다.초기 연구는 주로 곰팡이에 초점을 맞췄고, 2016년 연구는 지문 채취 기술을 사용하여 지리적으로나 환경적으로 구별되는 3개의 마누카 집단의 내생균 군집과 조직 고유의 핵심 내미량체를 [93][53]최초로 조사했다.2020년 연구에서는 모든 표본에서 지속적이었던 마누카 필로스피어에서 서식지 특이적이고 비교적 풍부한 핵심 마이크로바이옴을 확인했다.이와는 대조적으로, 비핵심 엽층 미생물은 환경 및 공간적 요인에 의해 강하게 추진된 개별 숙주 나무와 개체군에 걸쳐 유의한 변화를 보였다.그 결과 마누카의 [53]세포권 내에 지배적이고 유비쿼터스한 핵심 마이크로바이옴이 존재했다.
내층 마이크로바이옴
내생 식물과 같은 일부 미생물은 식물의 내부 조직을 침투하여 점유하여 내층 마이크로바이옴을 형성합니다.관절근균과 다른 내생균은 내층권의 [94]지배적인 콜로나이저이다.박테리아, 그리고 어느 정도 고세균은 내층 공동체의 중요한 구성원이다.이 내생 미생물들 중 일부는 숙주와 상호작용을 하고 [40][95][96]식물에게 명백한 이점을 제공한다.근층이나 근면과는 달리, 내층에는 매우 특정한 미생물 군집이 존재한다.근원 내생식물 군집은 인접한 토양 군집과 매우 다를 수 있다.일반적으로 내생식물 군집의 다양성은 [43]식물 외부의 미생물 군집의 다양성보다 낮다.식물 [97][94][5]내에서는 지상조직과 지하조직의 내생성 미생물군의 정체성과 다양성도 다를 수 있다.
종자 마이크로바이옴
각각의 씨앗은 높은 미생물 다양성을 가지고 있었고, 이는 심피보다 배아에서 더 높았다.식물의 씨앗은 질병에 대한 [98]저항력을 주는 유익한 내생식물의 수직 전달을 위한 자연적인 매개체 역할을 할 수 있습니다.씨앗에서 발육 중인 묘목으로의 미생물의 전달을 나타내는 증거는 실험적이고 자연적인 [99]조건에서 발생하는 것으로 밝혀졌다.또한 새로운 식물로의 전염은 특정 미생물이 씨앗에서 식물의 잎으로, 그리고 식물의 [100]뿌리로 이동하는 특정한 메커니즘을 통해 일어나는 것으로 보인다.
홀로비언트
4억 5천만 년 전 조상의 식물 계통에 의해 땅이 식민지화된 이후, 식물과 그 관련 미생물들은 종종 홀로비온트라고 불리는 종의 집단을 형성하면서 서로 상호작용을 해왔다.홀로비온트 성분에 작용하는 선택적 압력은 식물과 관련된 미생물 군집을 형성하고 식물 적합성에 영향을 미치는 숙주 적응 미생물에 선택될 수 있다.하지만, 미생물의 빠른 생성 시간과 그들의 숙주에 비해 더 오래된 기원과 함께 식물 조직에서 검출된 높은 미생물 밀도는 또한 미세-미생물 상호작용이 식물권, 뿌리권, 그리고 식물 내층권 비교에서 복잡한 미생물 집단을 조각하는 중요한 선택적 힘임을 암시합니다.tents를 클릭합니다.[101]
「 」를 참조해 주세요.
레퍼런스
- ^ a b c Turner, Thomas R.; James, Euan K.; Poole, Philip S. (2013). "The plant microbiome". Genome Biology. 14 (6): 209. doi:10.1186/gb-2013-14-6-209. PMC 3706808. PMID 23805896.
- ^ a b c Purahong, 위툰;Orrù, 루이지, 직경 약 35km., Irene은;Perpetuini, Giorgia;첼리니, 안토니오, Lamontanara, Antonella, Michelotti, Vania, Tacconi, 잔니;Spinelli, 프란체스코(2018년)."식물 미생물 군 유전체와 링크소 건강에:.호스트의 종, Organs과 슈도 모나스 syringae pv.Actinidiae 감염 Shaping 박테리아 Phyllosphere 경제 공동체. 키위 Plants".플랜트 과학에 국경.9:1563년. doi:10.3389/fpls.2018.01563.PMC 6234494.PMID 30464766..재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Whips J., Lewis K. and Cooke R.(1988) "미코파라시즘과 식물병해 통제"인: Burge M (Ed.) Burge M in Biological Control Systems, Manchester University Press, 161-187페이지.ISBN 9780719019791.
- ^ Berg, Gabriele; Daria Rybakova, Doren Fischer, Tomislav Cernava, Marie-Christine Champomier Vergés, Trevor Charles, Xiaoyulong Chen, Luca Cocolin, Kellye Eversole, Gema Herrerna, La Kazelene, Linda, La, Linda, Linda, Linda, La, Linda, Linda, Linda, Linda, Linda, La맥클린, 엠마뉴엘 마그인, 팀 모클라인, 라이언 맥클루어, 비르기트 미터, 매튜 라이언, 잉가 사란드, 하우케 스미트, 베티나 셸클, 휴고 루메, G. 세갈 키란, 조셉 셀빈, 라파엘 데 소레아레아Singh, Michael Wagner, Aaron Walsh, Angela Sessitch 및 Michael Schloter (2020) "마이크로바이오엠 정의 재방문: 오래된 개념과 새로운 도전"마이크로바이옴, 8(103) : 1~22.도이:10.1186/s40168-020-00875-0.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ a b c d e f g h i j Dastgeer, K.M., Tumpa, F.H., Sultana, A., A., Akter, M.A. 및 Chakraborty, A. (20) "식물 마이크로바이옴 – 공동체 구성과 다양성을 형성하는 요소의 설명"현재 식물생물학: 100161.doi:10.1016/j.cpb.2020.100161.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ Compant, S., Samad, A., Faist, H. 및 Sessitch, A.(2019) "식물 마이크로바이옴에 대한 검토:미생물 응용의 생태, 기능 및 새로운 트렌드.고급연구저널, 19: 29_37.doi:10.1016/j.jare.2019.03.004.
- ^ Bringel, Franã§Oise; Couã©e, Ivan (2015). "Pivotal roles of phyllosphere microorganisms at the interface between plant functioning and atmospheric trace gas dynamics". Frontiers in Microbiology. 06: 486. doi:10.3389/fmicb.2015.00486. PMC 4440916. PMID 26052316.
- ^ Berendsen, Roeland L.; Pieterse, Corné M.J.; Bakker, Peter A.H.M. (2012). "The rhizosphere microbiome and plant health". Trends in Plant Science. 17 (8): 478–486. doi:10.1016/j.tplants.2012.04.001. hdl:1874/255269. PMID 22564542.
- ^ Berg, Gabriele; Grube, M.; Schloter, M.; Smalla, K. (2014). "The plant microbiome and its importance for plant and human health". Frontiers in Microbiology. 5: 491. doi:10.3389/fmicb.2014.00491. PMC 4166366. PMID 25278934.
- ^ Shelake, R.M., Pramanik, D. and Kim, J.Y.(2019) "CRISPR 시대의 지속 가능한 농업을 위한 식물과 미생물 상호작용 연구"미생물, 7(8): 269. doi: 10.3390/microorganizations7080269.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ Koide, Roger T.; Mosse, Barbara (2004). "A history of research on arbuscular mycorrhiza". Mycorrhiza. 14 (3): 145–163. doi:10.1007/s00572-004-0307-4. PMID 15088135. S2CID 1809402.
- ^ Berendsen, Roeland L.; Pieterse, Corné M.J.; Bakker, Peter A.H.M. (2012). "The rhizosphere microbiome and plant health". Trends in Plant Science. 17 (8): 478–486. doi:10.1016/j.tplants.2012.04.001. hdl:1874/255269. PMID 22564542.
- ^ a b Bulgarelli, Davide; Schlaeppi, Klaus; Spaepen, Stijn; Van Themaat, Emiel Ver Loren; Schulze-Lefert, Paul (2013). "Structure and Functions of the Bacterial Microbiota of Plants". Annual Review of Plant Biology. 64: 807–838. doi:10.1146/annurev-arplant-050312-120106. PMID 23373698.
- ^ Abdelfattah, Ahmed; Tack, Ayco J. M.; Wasserman, Birgit; Liu, Jia; Berg, Gabriele; Norelli, John; Droby, Samir; Wisniewski, Michael (2021). "Evidence for host–microbiome co-evolution in apple". New Phytologist. 234 (6): 2088–2100. doi:10.1111/nph.17820. ISSN 1469-8137. PMID 34823272. S2CID 244661193.
- ^ Busby, Posy E.; Soman, Chinmay; Wagner, Maggie R.; Friesen, Maren L.; Kremer, James; Bennett, Alison; Morsy, Mustafa; Eisen, Jonathan A.; Leach, Jan E.; Dangl, Jeffery L. (2017). "Research priorities for harnessing plant microbiomes in sustainable agriculture". PLOS Biology. 15 (3): e2001793. doi:10.1371/journal.pbio.2001793. PMC 5370116. PMID 28350798. S2CID 6434145.
- ^ Gantar, M.; Elhai, J. (1999). "Colonization of wheatpara-nodules by the N2-fixing cyanobacterium Nostocsp. Strain 2S9B". New Phytologist. 141 (3): 373–379. doi:10.1046/j.1469-8137.1999.00352.x.
- ^ Gantar, M. (2000). "Mechanical damage of roots provides enhanced colonization of the wheat endorhizosphere by the dinitrogen-fixing cyanobacterium Nostoc sp. Strain 2S9B". Biology and Fertility of Soils. 32 (3): 250–255. doi:10.1007/s003740000243. S2CID 7590731.
- ^ Treves, Haim; Raanan, Hagai; Kedem, Isaac; Murik, Omer; Keren, Nir; Zer, Hagit; Berkowicz, Simon M.; Giordano, Mario; Norici, Alessandra; Shotland, Yoram; Ohad, Itzhak; Kaplan, Aaron (2016). "The mechanisms whereby the green alga Chlorella ohadii , isolated from desert soil crust, exhibits unparalleled photodamage resistance". New Phytologist. 210 (4): 1229–1243. doi:10.1111/nph.13870. PMID 26853530.
- ^ Zhu, Huan; Li, Shuyin; Hu, Zhengyu; Liu, Guoxiang (2018). "Molecular characterization of eukaryotic algal communities in the tropical phyllosphere based on real-time sequencing of the 18S rDNA gene". BMC Plant Biology. 18 (1): 365. doi:10.1186/s12870-018-1588-7. PMC 6299628. PMID 30563464.
- ^ Krings, Michael; Hass, Hagen; Kerp, Hans; Taylor, Thomas N.; Agerer, Reinhard; Dotzler, Nora (2009). "Endophytic cyanobacteria in a 400-million-yr-old land plant: A scenario for the origin of a symbiosis?". Review of Palaeobotany and Palynology. 153 (1–2): 62–69. doi:10.1016/j.revpalbo.2008.06.006.
- ^ Karthikeyan, N.; Prasanna, R.; Sood, A.; Jaiswal, P.; Nayak, S.; Kaushik, B. D. (2009). "Physiological characterization and electron microscopic investigation of cyanobacteria associated with wheat rhizosphere". Folia Microbiologica. 54 (1): 43–51. doi:10.1007/s12223-009-0007-8. PMID 19330544. S2CID 23420342.
- ^ a b Babu, Santosh; Prasanna, Radha; Bidyarani, Ngangom; Singh, Rajendra (2015). "Analysing the colonisation of inoculated cyanobacteria in wheat plants using biochemical and molecular tools". Journal of Applied Phycology. 27: 327–338. doi:10.1007/s10811-014-0322-6. S2CID 17353123.
- ^ a b Bidyarani, Ngangom; Prasanna, Radha; Chawla, Gautam; Babu, Santosh; Singh, Rajendra (2015). "Deciphering the factors associated with the colonization of rice plants by cyanobacteria". Journal of Basic Microbiology. 55 (4): 407–419. doi:10.1002/jobm.201400591. PMID 25515189. S2CID 5401526.
- ^ a b Gantar, M.; Kerby, N. W.; Rowell, P. (1991). "Colonization of wheat (Triticum vulgare L.) by N2-fixing cyanobacteria: II. An ultrastructural study". New Phytologist. 118 (3): 485–492. doi:10.1111/j.1469-8137.1991.tb00031.x.
- ^ Ahmed, Mehboob; Stal, Lucas J.; Hasnain, Shahida (2010). "Association of non-heterocystous cyanobacteria with crop plants". Plant and Soil. 336 (1–2): 363–375. doi:10.1007/s11104-010-0488-x. S2CID 21309970.
- ^ Hussain, Anwar; Hamayun, Muhammad; Shah, Syed Tariq (2013). "Root Colonization and Phytostimulation by Phytohormones Producing Entophytic Nostoc sp. AH-12". Current Microbiology. 67 (5): 624–630. doi:10.1007/s00284-013-0408-4. PMID 23794014. S2CID 14704537.
- ^ Hussain, Anwar; Shah, Syed T.; Rahman, Hazir; Irshad, Muhammad; Iqbal, Amjad (2015). "Effect of IAA on in vitro growth and colonization of Nostoc in plant roots". Frontiers in Plant Science. 6: 46. doi:10.3389/fpls.2015.00046. PMC 4318279. PMID 25699072.
- ^ a b c 리, Sang-Moo, 유 감독 Choong-Min(42월 2021년)."조류는 뉴 키즈 온 복지 공장 미생물 군 유전체에".플랜트 과학에 국경.둔 미디어 SA.12:599742.doi:10.3389/fpls.2021.599742.ISSN 1664-462X.PMC7889962.PMID 33613596.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ a b 초드리, Vasvi;룽게, 폴, Sengupta, Priyamedha, Doehlemann, 군터. 파커, 제인은 E;Kemen, 에릭(10일부터 9월 2020년).Kopriva, 스타니 슬라프(교육.)."leaf조성 미소 생물학:plant–microbe–microbe 상호 작용".실험 식물학 신문.옥스포드 대학 출판부(OUP). 72(1):36–56. doi:10.1093/jxb/eraa417.ISSN 0022-0957.PMC 8210630.PMID 32910810.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Vishwakarma, Kanchan, 쿠마르는 Nitin, Shandilya, Chitrakshi, Mohapatra, 스와트 인.;Bhayana, Sahil, Varma, 아지트(2020년)."Revisiting Plant–Microbe 상호 작용과 미생물 Consortia 적용 강화 지속 가능한 농업에 대한 리뷰".미생물학에 국경. 11:560406.doi:10.3389/fmicb.2020.560406.PMC7779480.PMID 33408698.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
- ^ Hinsinger, Philippe; Bengough, A. Glyn; Vetterlein, Doris; Young, Iain M. (2009). "Rhizosphere: Biophysics, biogeochemistry and ecological relevance". Plant and Soil. 321 (1–2): 117–152. doi:10.1007/s11104-008-9885-9. S2CID 8997382.
- ^ Bonkowski, Michael; Villenave, Cécile; Griffiths, Bryan (2009). "Rhizosphere fauna: The functional and structural diversity of intimate interactions of soil fauna with plant roots". Plant and Soil. 321 (1–2): 213–233. doi:10.1007/s11104-009-0013-2. S2CID 35701713.
- ^ Berendsen, R.L., Pieterse, C.M. 및 P.A. Bakker. (2012) "근층 마이크로바이옴과 식물 건강"식물과학 동향, 17(8): 478~486. doi:10.1016/j.tplants.2012.04.001.
- ^ Hassani, M.A., Duran, P. 및 Hacquard, S. (2018) "발전소 홀로비온트 내 미생물 상호작용"마이크로바이옴, 6 (1): 58.doi:10.118/s40168-0445-0.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ Van Der Heijden, Marcel G. A.; Martin, Francis M.; Selosse, Marc-André; Sanders, Ian R. (2015). "Mycorrhizal ecology and evolution: The past, the present, and the future". New Phytologist. 205 (4): 1406–1423. doi:10.1111/nph.13288. PMID 25639293.
- ^ Rich, Mélanie K.; Nouri, Eva; Courty, Pierre-Emmanuel; Reinhardt, Didier (2017). "Diet of Arbuscular Mycorrhizal Fungi: Bread and Butter?" (PDF). Trends in Plant Science. 22 (8): 652–660. doi:10.1016/j.tplants.2017.05.008. PMID 28622919.
- ^ Lee, Eun-Hwa; Eo, Ju-Kyeong; Ka, Kang-Hyeon; Eom, Ahn-Heum (2013). "Diversity of Arbuscular Mycorrhizal Fungi and Their Roles in Ecosystems". Mycobiology. 41 (3): 121–125. doi:10.5941/MYCO.2013.41.3.121. PMC 3817225. PMID 24198665.
- ^ Gans, J.; Wolinsky, M.; Dunbar, J. (2005). "Computational Improvements Reveal Great Bacterial Diversity and High Metal Toxicity in Soil". Science. 309 (5739): 1387–1390. Bibcode:2005Sci...309.1387G. doi:10.1126/science.1112665. PMID 16123304. S2CID 130269020.
- ^ i̇Nceoğlu, Özgül; Al-Soud, Waleed Abu; Salles, Joana Falcão; Semenov, Alexander V.; Van Elsas, Jan Dirk (2011). "Comparative Analysis of Bacterial Communities in a Potato Field as Determined by Pyrosequencing". PLOS ONE. 6 (8): e23321. Bibcode:2011PLoSO...623321I. doi:10.1371/journal.pone.0023321. PMC 3158761. PMID 21886785.
- ^ a b c Bulgarelli, Davide; Rott, Matthias; Schlaeppi, Klaus; Ver Loren Van Themaat, Emiel; Ahmadinejad, Nahal; Assenza, Federica; Rauf, Philipp; Huettel, Bruno; Reinhardt, Richard; Schmelzer, Elmon; Peplies, Joerg; Gloeckner, Frank Oliver; Amann, Rudolf; Eickhorst, Thilo; Schulze-Lefert, Paul (2012). "Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota". Nature. 488 (7409): 91–95. Bibcode:2012Natur.488...91B. doi:10.1038/nature11336. PMID 22859207. S2CID 4393146.
- ^ Uroz, Stéphane; Buée, Marc; Murat, Claude; Frey-Klett, Pascale; Martin, Francis (2010). "Pyrosequencing reveals a contrasted bacterial diversity between oak rhizosphere and surrounding soil". Environmental Microbiology Reports. 2 (2): 281–288. doi:10.1111/j.1758-2229.2009.00117.x. PMID 23766079.
- ^ Lundberg, Derek S.; Lebeis, Sarah L.; Paredes, Sur Herrera; Yourstone, Scott; Gehring, Jase; Malfatti, Stephanie; Tremblay, Julien; Engelbrektson, Anna; Kunin, Victor; Rio, Tijana Glavina del; Edgar, Robert C.; Eickhorst, Thilo; Ley, Ruth E.; Hugenholtz, Philip; Tringe, Susannah Green; Dangl, Jeffery L. (2012). "Defining the core Arabidopsis thaliana root microbiome". Nature. 488 (7409): 86–90. Bibcode:2012Natur.488...86L. doi:10.1038/nature11237. PMC 4074413. PMID 22859206.
- ^ a b Schlaeppi, K.; Dombrowski, N.; Oter, R. G.; Ver Loren Van Themaat, E.; Schulze-Lefert, P. (2014). "Quantitative divergence of the bacterial root microbiota in Arabidopsis thaliana relatives". Proceedings of the National Academy of Sciences. 111 (2): 585–592. Bibcode:2014PNAS..111..585S. doi:10.1073/pnas.1321597111. PMC 3896156. PMID 24379374. S2CID 13806811.
- ^ 그, Seng Yang (2020) 식물과 그 미생물이 일치하지 않을 때, 그 결과는 재앙적인 The Conversation, 2020년 8월 28일.
- ^ Lindow, Steven E. (1996). "Role of Immigration and Other Processes in Determining Epiphytic Bacterial Populations". Aerial Plant Surface Microbiology. pp. 155–168. doi:10.1007/978-0-585-34164-4_10. ISBN 978-0-306-45382-3.
- ^ Friesen, Maren L.; Porter, Stephanie S.; Stark, Scott C.; von Wettberg, Eric J.; Sachs, Joel L.; Martinez-Romero, Esperanza (2011). "Microbially Mediated Plant Functional Traits". Annual Review of Ecology, Evolution, and Systematics. 42: 23–46. doi:10.1146/annurev-ecolsys-102710-145039.
- ^ a b Leveau, Johan HJ (2019). "A brief from the leaf: Latest research to inform our understanding of the phyllosphere microbiome". Current Opinion in Microbiology. 49: 41–49. doi:10.1016/j.mib.2019.10.002. PMID 31707206. S2CID 207946690.
- ^ 루넨, J.(1956) "필로스피어에서의 베이제린키아종 발생"네이처, 177(4501) : 220 ~221 。
- ^ a b Vogel, Christine; Bodenhausen, Natacha; Gruissem, Wilhelm; Vorholt, Julia A. (2016). "The Arabidopsis leaf transcriptome reveals distinct but also overlapping responses to colonization by phyllosphere commensals and pathogen infection with impact on plant health" (PDF). New Phytologist. 212 (1): 192–207. doi:10.1111/nph.14036. hdl:20.500.11850/117578. PMID 27306148.
- ^ a b Cid, Fernanda P.; Maruyama, Fumito; Murase, Kazunori; Graether, Steffen P.; Larama, Giovanni; Bravo, Leon A.; Jorquera, Milko A. (2018). "Draft genome sequences of bacteria isolated from the Deschampsia antarctica phyllosphere". Extremophiles. 22 (3): 537–552. doi:10.1007/s00792-018-1015-x. PMID 29492666. S2CID 4320165.
- ^ Kumaravel, Sowmya; Thankappan, Sugitha; Raghupathi, Sridar; Uthandi, Sivakumar (2018). "Draft Genome Sequence of Plant Growth-Promoting and Drought-Tolerant Bacillus altitudinis FD48, Isolated from Rice Phylloplane". Genome Announcements. 6 (9). doi:10.1128/genomeA.00019-18. PMC 5834328. PMID 29496824.
- ^ Laforest‐Lapointe, Isabelle; Whitaker, Briana K. (2019). "Decrypting the phyllosphere microbiota: Progress and challenges". American Journal of Botany. 106 (2): 171–173. doi:10.1002/ajb2.1229. PMID 30726571.
- ^ a b c d e f g h i j k l 노블, Anya S.;노에, 스티비, 클리어 워터, 마이클 J., 대통령, 찰스 K.(2020년)."핵심 phyllosphere microbiome 수종의 먼 모집단에 대해 indigenous 뉴질랜드에 존재하".PLOS ONE.15세(8):e0237079.Bibcode:2020년PLoSO..1537079N. doi:10.1371/journal.pone.0237079.PMC 7425925.PMID 32790769.재료는 창조적 공용 귀인 4.0국제 라이센스 하에 가능하다 이 원본에서 복사되었다.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ a b Finkel, Omri M.; Burch, Adrien Y.; Lindow, Steven E.; Post, Anton F.; Belkin, Shimshon (2011). "Geographical Location Determines the Population Structure in Phyllosphere Microbial Communities of a Salt-Excreting Desert Tree". Applied and Environmental Microbiology. 77 (21): 7647–7655. Bibcode:2011ApEnM..77.7647F. doi:10.1128/AEM.05565-11. PMC 3209174. PMID 21926212.
- ^ Vorholt, Julia A. (2012). "Microbial life in the phyllosphere". Nature Reviews Microbiology. 10 (12): 828–840. doi:10.1038/nrmicro2910. hdl:20.500.11850/59727. PMID 23154261. S2CID 10447146.
- ^ Lindow, Steven E.; Brandl, Maria T. (2003). "Microbiology of the Phyllosphere". Applied and Environmental Microbiology. 69 (4): 1875–1883. Bibcode:2003ApEnM..69.1875L. doi:10.1128/AEM.69.4.1875-1883.2003. PMC 154815. PMID 12676659. S2CID 2304379.
- ^ Bodenhausen, Natacha; Horton, Matthew W.; Bergelson, Joy (2013). "Bacterial Communities Associated with the Leaves and the Roots of Arabidopsis thaliana". PLOS ONE. 8 (2): e56329. Bibcode:2013PLoSO...856329B. doi:10.1371/journal.pone.0056329. PMC 3574144. PMID 23457551.
- ^ Vokou, Despoina; Vareli, Katerina; Zarali, Ekaterini; Karamanoli, Katerina; Constantinidou, Helen-Isis A.; Monokrousos, Nikolaos; Halley, John M.; Sainis, Ioannis (2012). "Exploring Biodiversity in the Bacterial Community of the Mediterranean Phyllosphere and its Relationship with Airborne Bacteria". Microbial Ecology. 64 (3): 714–724. doi:10.1007/s00248-012-0053-7. PMID 22544345. S2CID 17291303.
- ^ Vorholt, Julia A. (2012). "Microbial life in the phyllosphere". Nature Reviews Microbiology. 10 (12): 828–840. doi:10.1038/nrmicro2910. hdl:20.500.11850/59727. PMID 23154261. S2CID 10447146.
- ^ a b Stone, Bram W. G.; Jackson, Colin R. (2016). "Biogeographic Patterns Between Bacterial Phyllosphere Communities of the Southern Magnolia (Magnolia grandiflora) in a Small Forest". Microbial Ecology. 71 (4): 954–961. doi:10.1007/s00248-016-0738-4. PMID 26883131. S2CID 17292307.
- ^ a b Redford, Amanda J.; Bowers, Robert M.; Knight, Rob; Linhart, Yan; Fierer, Noah (2010). "The ecology of the phyllosphere: Geographic and phylogenetic variability in the distribution of bacteria on tree leaves". Environmental Microbiology. 12 (11): 2885–2893. doi:10.1111/j.1462-2920.2010.02258.x. PMC 3156554. PMID 20545741.
- ^ Vokou, Despoina; Vareli, Katerina; Zarali, Ekaterini; Karamanoli, Katerina; Constantinidou, Helen-Isis A.; Monokrousos, Nikolaos; Halley, John M.; Sainis, Ioannis (2012). "Exploring Biodiversity in the Bacterial Community of the Mediterranean Phyllosphere and its Relationship with Airborne Bacteria". Microbial Ecology. 64 (3): 714–724. doi:10.1007/s00248-012-0053-7. PMID 22544345. S2CID 17291303.
- ^ a b c Laforest-Lapointe, Isabelle; Messier, Christian; Kembel, Steven W. (2016). "Host species identity, site and time drive temperate tree phyllosphere bacterial community structure". Microbiome. 4 (1): 27. doi:10.1186/s40168-016-0174-1. PMC 4912770. PMID 27316353.
- ^ Zarraonaindia, Iratxe; Owens, Sarah M.; Weisenhorn, Pamela; West, Kristin; Hampton-Marcell, Jarrad; Lax, Simon; Bokulich, Nicholas A.; Mills, David A.; Martin, Gilles; Taghavi, Safiyh; Van Der Lelie, Daniel; Gilbert, Jack A. (2015). "The Soil Microbiome Influences Grapevine-Associated Microbiota". mBio. 6 (2). doi:10.1128/mBio.02527-14. PMC 4453523. PMID 25805735.
- ^ Finkel, Omri M.; Burch, Adrien Y.; Lindow, Steven E.; Post, Anton F.; Belkin, Shimshon (2011). "Geographical Location Determines the Population Structure in Phyllosphere Microbial Communities of a Salt-Excreting Desert Tree". Applied and Environmental Microbiology. 77 (21): 7647–7655. Bibcode:2011ApEnM..77.7647F. doi:10.1128/AEM.05565-11. PMC 3209174. PMID 21926212.
- ^ Finkel, Omri M.; Burch, Adrien Y.; Elad, Tal; Huse, Susan M.; Lindow, Steven E.; Post, Anton F.; Belkin, Shimshon (2012). "Distance-Decay Relationships Partially Determine Diversity Patterns of Phyllosphere Bacteria on Tamrix Trees across the Sonoran Desert". Applied and Environmental Microbiology. 78 (17): 6187–6193. Bibcode:2012ApEnM..78.6187F. doi:10.1128/AEM.00888-12. PMC 3416633. PMID 22752165.
- ^ a b Kembel, S. W.; O'Connor, T. K.; Arnold, H. K.; Hubbell, S. P.; Wright, S. J.; Green, J. L. (2014). "Relationships between phyllosphere bacterial communities and plant functional traits in a neotropical forest". Proceedings of the National Academy of Sciences. 111 (38): 13715–13720. Bibcode:2014PNAS..11113715K. doi:10.1073/pnas.1216057111. PMC 4183302. PMID 25225376. S2CID 852584.
- ^ Innerebner, Gerd; Knief, Claudia; Vorholt, Julia A. (2011). "Protection of Arabidopsis thaliana against Leaf-Pathogenic Pseudomonas syringae by Sphingomonas Strains in a Controlled Model System". Applied and Environmental Microbiology. 77 (10): 3202–3210. Bibcode:2011ApEnM..77.3202I. doi:10.1128/AEM.00133-11. PMC 3126462. PMID 21421777.
- ^ Lajoie, Geneviève; Maglione, Rémi; Kembel, Steven W. (2020). "Adaptive matching between phyllosphere bacteria and their tree hosts in a neotropical forest". Microbiome. 8 (1): 70. doi:10.1186/s40168-020-00844-7. PMC 7243311. PMID 32438916.
- ^ a b Shade, Ashley; Handelsman, Jo (2012). "Beyond the Venn diagram: The hunt for a core microbiome". Environmental Microbiology. 14 (1): 4–12. doi:10.1111/j.1462-2920.2011.02585.x. PMID 22004523.
- ^ Berg, Gabriele; Rybakova, Daria; Fischer, Doreen; Cernava, Tomislav; Vergès, Marie-Christine Champomier; Charles, Trevor; Chen, Xiaoyulong; Cocolin, Luca; Eversole, Kellye; Corral, Gema Herrero; Kazou, Maria; Kinkel, Linda; Lange, Lene; Lima, Nelson; Loy, Alexander; MacKlin, James A.; Maguin, Emmanuelle; Mauchline, Tim; McClure, Ryan; Mitter, Birgit; Ryan, Matthew; Sarand, Inga; Smidt, Hauke; Schelkle, Bettina; Roume, Hugo; Kiran, G. Seghal; Selvin, Joseph; Souza, Rafael Soares Correa de; Van Overbeek, Leo; et al. (2020). "Microbiome definition re-visited: Old concepts and new challenges". Microbiome. 8 (1): 103. doi:10.1186/s40168-020-00875-0. PMC 7329523. PMID 32605663.
- ^ Turnbaugh, Peter J.; Hamady, Micah; Yatsunenko, Tanya; Cantarel, Brandi L.; Duncan, Alexis; Ley, Ruth E.; Sogin, Mitchell L.; Jones, William J.; Roe, Bruce A.; Affourtit, Jason P.; Egholm, Michael; Henrissat, Bernard; Heath, Andrew C.; Knight, Rob; Gordon, Jeffrey I. (2009). "A core gut microbiome in obese and lean twins". Nature. 457 (7228): 480–484. Bibcode:2009Natur.457..480T. doi:10.1038/nature07540. PMC 2677729. PMID 19043404.
- ^ Lundberg, Derek S.; Lebeis, Sarah L.; Paredes, Sur Herrera; Yourstone, Scott; Gehring, Jase; Malfatti, Stephanie; Tremblay, Julien; Engelbrektson, Anna; Kunin, Victor; Rio, Tijana Glavina del; Edgar, Robert C.; Eickhorst, Thilo; Ley, Ruth E.; Hugenholtz, Philip; Tringe, Susannah Green; Dangl, Jeffery L. (2012). "Defining the core Arabidopsis thaliana root microbiome". Nature. 488 (7409): 86–90. Bibcode:2012Natur.488...86L. doi:10.1038/nature11237. PMC 4074413. PMID 22859206.
- ^ Hamonts, Kelly; Trivedi, Pankaj; Garg, Anshu; Janitz, Caroline; Grinyer, Jasmine; Holford, Paul; Botha, Frederik C.; Anderson, Ian C.; Singh, Brajesh K. (2018). "Field study reveals core plant microbiota and relative importance of their drivers". Environmental Microbiology. 20 (1): 124–140. doi:10.1111/1462-2920.14031. PMID 29266641. S2CID 10650949.
- ^ Cernava, Tomislav; Erlacher, Armin; Soh, Jung; Sensen, Christoph W.; Grube, Martin; Berg, Gabriele (2019). "Enterobacteriaceae dominate the core microbiome and contribute to the resistome of arugula (Eruca sativa Mill.)". Microbiome. 7 (1): 13. doi:10.1186/s40168-019-0624-7. PMC 6352427. PMID 30696492.
- ^ Leff, Jonathan W.; Del Tredici, Peter; Friedman, William E.; Fierer, Noah (2015). "Spatial structuring of bacterial communities within individual Ginkgo bilobatrees". Environmental Microbiology. 17 (7): 2352–2361. doi:10.1111/1462-2920.12695. PMID 25367625.
- ^ Hernandez-Agreda, Alejandra; Gates, Ruth D.; Ainsworth, Tracy D. (2017). "Defining the Core Microbiome in Corals' Microbial Soup". Trends in Microbiology. 25 (2): 125–140. doi:10.1016/j.tim.2016.11.003. PMID 27919551.
- ^ Stephens, J. M. C.; Molan, P. C.; Clarkson, B. D. (2005). "A review of Leptospermum scoparium(Myrtaceae) in New Zealand". New Zealand Journal of Botany. 43 (2): 431–449. doi:10.1080/0028825X.2005.9512966. S2CID 53515334.
- ^ Cooper, R.A.; Molan, P.C.; Harding, K.G. (2002). "The sensitivity to honey of Gram-positive cocci of clinical significance isolated from wounds". Journal of Applied Microbiology. 93 (5): 857–863. doi:10.1046/j.1365-2672.2002.01761.x. PMID 12392533. S2CID 24517001.
- ^ Rabie, Erika; Serem, June Cheptoo; Oberholzer, Hester Magdalena; Gaspar, Anabella Regina Marques; Bester, Megan Jean (2016). "How methylglyoxal kills bacteria: An ultrastructural study". Ultrastructural Pathology. 40 (2): 107–111. doi:10.3109/01913123.2016.1154914. hdl:2263/52156. PMID 26986806. S2CID 13372064.
- ^ Adams, Christopher J.; Manley-Harris, Merilyn; Molan, Peter C. (2009). "The origin of methylglyoxal in New Zealand manuka (Leptospermum scoparium) honey". Carbohydrate Research. 344 (8): 1050–1053. doi:10.1016/j.carres.2009.03.020. PMID 19368902.
- ^ Atrott, Julia; Haberlau, Steffi; Henle, Thomas (2012). "Studies on the formation of methylglyoxal from dihydroxyacetone in Manuka (Leptospermum scoparium) honey". Carbohydrate Research. 361: 7–11. doi:10.1016/j.carres.2012.07.025. PMID 22960208.
- ^ Mavric, Elvira; Wittmann, Silvia; Barth, Gerold; Henle, Thomas (2008). "Identification and quantification of methylglyoxal as the dominant antibacterial constituent of Manuka (Leptospermum scoparium)honeys from New Zealand". Molecular Nutrition & Food Research. 52 (4): 483–489. doi:10.1002/mnfr.200700282. PMID 18210383.
- ^ Hamilton, G., Millner, J., Robertson, A. 및 Stephens, J. (2013년) "고유사 마누카 인자" 꿀 생산을 위한 마누카 산정의 평가"뉴질랜드 농업, 43:139~144.
- ^ Williams, Simon; King, Jessica; Revell, Maria; Manley-Harris, Merilyn; Balks, Megan; Janusch, Franziska; Kiefer, Michael; Clearwater, Michael; Brooks, Peter; Dawson, Murray (2014). "Regional, Annual, and Individual Variations in the Dihydroxyacetone Content of the Nectar of Ma̅nuka (Leptospermum scoparium) in New Zealand". Journal of Agricultural and Food Chemistry. 62 (42): 10332–10340. doi:10.1021/jf5045958. PMID 25277074.
- ^ Stephens, J.M.C. (2006) "마누카(렙토스페름 스코파륨) 꿀에서 UMF®의 다양한 수준에 영향을 미치는 요인", 와이카토 대학 박사 학위 논문.
- ^ Noe, Stevie; Manley-Harris, Merilyn; Clearwater, Michael J. (2019). "Floral nectar of wild mānuka (Leptospermum scoparium) varies more among plants than among sites". New Zealand Journal of Crop and Horticultural Science. 47 (4): 282–296. doi:10.1080/01140671.2019.1670681. S2CID 204143940.
- ^ Nickless, Elizabeth M.; Anderson, Christopher W. N.; Hamilton, Georgie; Stephens, Jonathan M.; Wargent, Jason (2017). "Soil influences on plant growth, floral density and nectar yield in three cultivars of mānuka (Leptospermum scoparium)". New Zealand Journal of Botany. 55 (2): 100–117. doi:10.1080/0028825X.2016.1247732. S2CID 88657399.
- ^ Clearwater, Michael J.; Revell, Maria; Noe, Stevie; Manley-Harris, Merilyn (2018). "Influence of genotype, floral stage, and water stress on floral nectar yield and composition of mānuka (Leptospermum scoparium)". Annals of Botany. 121 (3): 501–512. doi:10.1093/aob/mcx183. PMC 5838834. PMID 29300875.
- ^ Johnston, Peter R. (1998). "Leaf endophytes of manuka (Leptospermum scoparium)". Mycological Research. 102 (8): 1009–1016. doi:10.1017/S0953756297005765.
- ^ McKenzie, E. H. C.; Johnston, P. R.; Buchanan, P. K. (2006). "Checklist of fungi on teatree (Kunzeaand Leptospermumspecies) in New Zealand". New Zealand Journal of Botany. 44 (3): 293–335. doi:10.1080/0028825X.2006.9513025. S2CID 84538904.
- ^ Wicaksono, Wisnu Adi; Sansom, Catherine E.; Eirian Jones, E.; Perry, Nigel B.; Monk, Jana; Ridgway, Hayley J. (2018). "Arbuscular mycorrhizal fungi associated with Leptospermum scoparium (Mānuka): Effects on plant growth and essential oil content". Symbiosis. 75: 39–50. doi:10.1007/s13199-017-0506-3. S2CID 4819178.
- ^ Wicaksono, Wisnu Adi; Jones, E. Eirian; Monk, Jana; Ridgway, Hayley J. (2016). "The Bacterial Signature of Leptospermum scoparium (Mānuka) Reveals Core and Accessory Communities with Bioactive Properties". PLOS ONE. 11 (9): e0163717. Bibcode:2016PLoSO..1163717W. doi:10.1371/journal.pone.0163717. PMC 5038978. PMID 27676607.
- ^ a b Vokou, Despoina; Vareli, Katerina; Zarali, Ekaterini; Karamanoli, Katerina; Constantinidou, Helen-Isis A.; Monokrousos, Nikolaos; Halley, John M.; Sainis, Ioannis (2012). "Exploring Biodiversity in the Bacterial Community of the Mediterranean Phyllosphere and its Relationship with Airborne Bacteria". Microbial Ecology. 64 (3): 714–724. doi:10.1007/s00248-012-0053-7. PMID 22544345. S2CID 17291303.
- ^ Dastogeer, Khondoker M.G.; Li, Hua; Sivasithamparam, Krishnapillai; Jones, Michael G.K.; Du, Xin; Ren, Yonglin; Wylie, Stephen J. (2017). "Metabolic responses of endophytic Nicotiana benthamiana plants experiencing water stress". Environmental and Experimental Botany. 143: 59–71. doi:10.1016/j.envexpbot.2017.08.008.
- ^ Rodriguez, R. J.; White Jr, J. F.; Arnold, A. E.; Redman, R. S. (2009). "Fungal endophytes: Diversity and functional roles". New Phytologist. 182 (2): 314–330. doi:10.1111/j.1469-8137.2009.02773.x. PMID 19236579.
- ^ Abdelfattah, Ahmed; Wisniewski, Michael; Schena, Leonardo; Tack, Ayco J.M. (14 May 2020). "Experimental Evidence of Microbial Inheritance in Plants and Transmission Routes from Seed to Phyllosphere and Root". doi:10.21203/rs.3.rs-27656/v1.
{{cite journal}}:Cite 저널 요구 사항journal=(도움말) - ^ Matsumoto H, Fan X, Wang Y, Kusstatscher P, Duan J, Wu S, et al. (January 2021). "Bacterial seed endophyte shapes disease resistance in rice". Nature Plants. 7 (1): 60–72. doi:10.1038/s41477-020-00826-5. PMID 33398157. S2CID 230508404.
- ^ "Seeds transfer their microbes to the next generation". EurekAlert!. Retrieved 21 January 2021.
- ^ Abdelfattah, Ahmed; Wisniewski, Michael; Schena, Leonardo; Tack, Ayco J. M. (2021). "Experimental evidence of microbial inheritance in plants and transmission routes from seed to phyllosphere and root". Environmental Microbiology. 23 (4): 2199–2214. doi:10.1111/1462-2920.15392. ISSN 1462-2920. PMID 33427409.
- ^ Hassani MA, Durán P, Hacquard S (March 2018). "Microbial interactions within the plant holobiont". Microbiome. 6 (1): 58. doi:10.1186/s40168-018-0445-0. PMC 5870681. PMID 29587885.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
참고 도서
- Salem M (2015) 미생물 군집 생태: 기초와 어플리케이션의 원동력.ISBN 978319116655.
- 식물 건강과 질병에서 쿠마르 V, 프라사드 R, 쿠마르 M 및 초드하리 DK(2019) 마이크로바이옴: 도전과 기회의 원동력.ISBN 9789811384950.
- Kumar, Vivek; Prasad, Ram; Kumar, Manoj; Choudhary, Devendra K. (10 August 2019). Microbiome in Plant Health and Disease: Challenges and Opportunities. ISBN 9789811384950.
- Grube, Martin; Schloter, Michael; Smalla, Kornelia; Berg, Gabriele (22 January 2015). The plant microbiome and its importance for plant and human health. Frontiers in Microbiology. Vol. 5. p. 491. doi:10.3389/fmicb.2014.00491. ISBN 9782889193783. PMC 4166366. PMID 25278934.
- Egamberdieva, Dilfuza; Ahmad, Parvaiz (6 February 2018). Plant Microbiome: Stress Response. ISBN 9789811055140.
- Castiglione, Stefano; Cicatelli, Angela; Ferrol, Nuria; Rozpadek, Piotr (22 August 2019). Effects of Plant-Microbiome Interactions on Phyto- and Bio-Remediation Capacity. ISBN 9782889459322.
- Castiglione, Stefano; Cicatelli, Angela; Ferrol, Nuria; Rozpadek, Piotr (22 August 2019). Effects of Plant-Microbiome Interactions on Phyto- and Bio-Remediation Capacity. ISBN 9782889459322.
- Carvalhais, Lilia C.; Dennis, Paul G. (30 December 2020). The Plant Microbiome: Methods and Protocols. ISBN 9781071610398.
- Kumar, Vivek; Prasad, Ram; Kumar, Manoj; Choudhary, Devendra K. (10 August 2019). Microbiome in Plant Health and Disease: Challenges and Opportunities. ISBN 9789811384950.
- Solanki, Manoj Kumar; Kashyap, Prem Lal; Ansari, Rizwan Ali; Kumari, Baby (28 August 2020). Microbiomes and Plant Health: Panoply and Their Applications. ISBN 9780128226018.
- Yadav, Ajar (2020). Advances in plant microbiome and sustainable agriculture functional annotation and future challenges. Singapore: Springer. ISBN 978-981-15-3204-7. OCLC 1193118045.
- Varma, Ajit (28 August 2020). Plant Microbiome Paradigm. ISBN 9783030503956.
- Doty, Sharon Lafferty (21 September 2017). Functional Importance of the Plant Microbiome: Implications for Agriculture, Forestry and Bioenergy. ISBN 9783319658971.
- Antwis, Rachael E.; Harrison, Xavier A.; Cox, Michael J. (12 March 2020). Microbiomes of Soils, Plants and Animals: An Integrated Approach. ISBN 9781108473712.
- Varma, Ajit; Tripathi, Swati; Prasad, Ram (20 October 2020). Plant Microbiome Paradigm. ISBN 9783030503949.
- Rosenberg, Eugene; Zilber-Rosenberg, Ilana (31 January 2014). The Hologenome Concept: Human, Animal and Plant Microbiota. ISBN 9783319042411.
- Yadav, Ajar Nath; Singh, Joginder; Rastegari, Ali Asghar; Yadav, Neelam (6 March 2020). Plant Microbiomes for Sustainable Agriculture. ISBN 9783030384531.
| Wikimedia Commons에는 식물 마이크로바이옴과 관련된 미디어가 있습니다. |










