바이오파지
Virophage| 라비다바이러스과 | |
|---|---|
 | |
| 스푸트니크 생물영양체 | |
| 바이러스 분류 | |
| (순위 미지정): | 바이러스 |
| 영역: | 바리드나비리아속 |
| 왕국: | 밤포드비라과 |
| 문: | 프리플라스미비리코타 |
| 클래스: | 매버리비리케테스 |
| 주문: | 프리클라우소비랄레스 |
| 패밀리: | 라비다바이러스과 |
| 속과 종 | |

(나) 숙주세포가 거대 바이러스와 그 비오페라지(viropage)에 공동 감염되었을 때, 후자는 거대 바이러스 공장에 기생한다.이 거대 바이러스의 복제 효율을 떨어뜨리고 숙주 세포의 생존을 증가시킴으로써 거대 바이러스의 감염성에 심각한 영향을 미칠 수 있다.
(다) 거대 바이러스 게놈을 provirophage로 기생시키면 거대 바이러스 복제 중에 후자가 발현된다.거대 바이러스 공장에서 생성돼 거대 바이러스의 복제를 억제해 숙주 세포의 생존율을 높인다.

(B) 위성바이러스의 개념은 바이러스가 숙주세포기계를 이용하여 핵에서 게놈의 발현과 복제를 개시하고, 그 후 세포질로 가는 것을 의미한다.세포질에서 위성바이러스는 그 도우미 바이러스의 형태 형성 기구를 탈취하여 자손을 낳는다.
바이로파지는 다른 바이러스의 공동 감염을 필요로 하는 작은 이중 가닥 DNA 바이러스 페이지다.공감염 바이러스는 일반적으로 거대한 바이러스입니다.바이로파지들은 그들 자신의 복제를 위해 공동 감염 거대 바이러스의 바이러스 복제 공장에 의존한다.바이로파지의 특징 중 하나는 그들이 공감염 바이러스와 기생 관계에 있다는 것이다.복제를 위해 거대 바이러스에 의존하기 때문에 거대 바이러스가 비활성화되는 경우가 많습니다.생물영양은 숙주의 회복과 생존을 향상시킬 수 있다.
위성 바이러스와 달리, 바이러스영양체는 그들의 공동 감염 바이러스에 기생적인 영향을 미친다.바이러스영양은 거대한 바이러스를 비활성 상태로 만들어 숙주 유기체의 상태를 개선하는 것으로 관찰되어 왔다.
알려진 모든 생물영양체는 라비다바이러스과("대형 바이러스 의존 또는 관련" + - viridae)[2]로 분류됩니다.
검출
최초의 생물영양체는 2008년 프랑스 파리의 냉각탑에서 발견되었다.그것은 공동감염 거대 바이러스인 ACMV와 함께 발견되었다. castellanii mamavirus와 함께 발견되었다.이 생물의 이름은 스푸트니크(Sputnik)로, 그 복제는 ACMV와 그 세포질 복제 장치의 공동 감염에 전적으로 의존했다.스푸트니크는 또한 ACMV에 억제 효과가 있는 것으로 밝혀졌으며 숙주의 생존을 향상시켰다.다른 특징적인 생물영양으로는 스푸트니크 2, 스푸트니크 3, 자밀론,[3][4] 마이바이러스가 있다.
이 바이러스영양들의 대부분은 메타제노믹 데이터 세트를 분석함으로써 발견되고 있다.메타제노믹 분석에서 DNA 배열은 특정 중요한 패턴과 특성을 추출하는 여러 생체정보 알고리즘을 통해 실행된다.이들 데이터 세트에는 거대한 바이러스와 바이러스 생물이 포함되어 있습니다.이들은 이미 배열된 바이로파지와 유사한 17~20kbp 길이의 배열을 찾아 분리된다.이 생물영양들은 선형 또는 원형 이중사슬 DNA [5]게놈을 가질 수 있다.배양 중인 알려진 생물영양체에는 4080나노미터 [6]길이의 정십면체 캡시드 입자가 있으며 생물영양체 입자는 전자현미경을 통해 관찰해야 할 정도로 작다.메타게노믹 염기서열 기반 분석은 약 57개의 완전 및 부분 생체영양[7] 게놈을 예측하기 위해 사용되었으며, 2019년 12월에는 27개의 분류학적 [8]군락에서 인간의 내장, 식물 뿌리층, 지상 지하면 등 다양한 서식지에서 328개의 고품질(완전 또는 거의 완전한) 게놈을 식별하기 위해 사용되었다.
호스트 범위 및 레플리케이션
바이로파지들은 복제하기 위해 공동 감염 바이러스를 가지고 있어야 한다.생물영양체들은 스스로 복제하는데 필요한 효소를 가지고 있지 않다.바이로파지들은 거대한 바이러스 복제기구를 사용하여 자신의 게놈을 복제하고 그들의 존재를 지속시킨다.바이로파지의 숙주는 이중 가닥 DNA 게놈을 가진 거대 바이러스를 포함한다.바이로파지들은 숙주의 전사기계 대신 그들 자신의 복제를 위해 이 거대한 바이러스의 전사기계를 사용한다.예를 들어, Samba 바이러스와 관련된 바이러스의 발견으로 인해 바이러스의 생성이 거대 바이러스를 사용하여 복제되는 동안 호스트 내 바이러스의 농도가 감소했습니다.숙주 아메바는 또한 삼바 [5]바이러스에 의한 감염으로부터 부분적인 회복을 보였다.
게놈
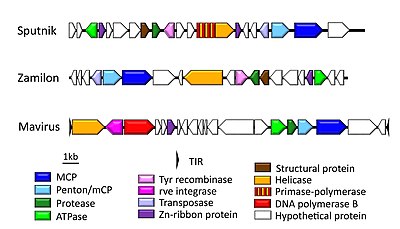
비오페이지는 원형 또는 선형 형태의 작은 이중 가닥 DNA 게놈을 가지고 있다.이 게놈의 크기는 그것이 감염시키는 거대한 바이러스에 따라 달라질 수 있다.대부분의 생물영양체는 약 17~30kbp의 [6][7]게놈을 가지고 있다.그들의 게놈은 길이가 [6]약 40-80 nm인 20면체 캡시드에 의해 보호된다.이와는 대조적으로, 그들의 공동 감염 거대 바이러스들은 1-2Mbp(메가베이스 쌍)[5]의 게놈을 가질 수 있다.가장 큰 바이러스 유전체 중 일부는 아데노 [6]바이러스의 유전체 크기와 유사하다.
| 게놈 크기(kbp) | 입자 크기 (표준, nm 단위) | |
|---|---|---|
| 바이러스: 폴리오바이러스 | 7 | 30 |
| 바이러스: 아데노바이러스과 | 26–48 | 90–100 |
| 바이로파지: 자밀론 바이로파지 | 17 | 50–60 |
| 비오피지:스푸트니크 바이오파지 | 18 | 74 |
| 거대 바이러스 : 카페테리아 로엔버그시스 바이러스 | 700 | 75 |
| 거대 바이러스:미미바이러스 | 1,181 | 400–800 |
지금까지 알려진 모든 생물영양체는 4개의 핵심 유전자를 가지고 있다.그것들은 비오피지 특이적 주요 및 부캡시드 단백질(MCP 및 mCP), PRO(시스테인 단백질 분해효소), DNA 포장 ATP 효소입니다.두 개의 캡시드는 거의 보편적으로 보존된 [8]블록에서 발견됩니다.MCP에는 Bamfordvirae의 전형적인 2개의 수직 젤리 롤 폴드 도메인이 있으며 mCP(펜톤)에는 규칙적인 젤리 롤 폴드 [9]도메인이 있습니다.
분류법
스푸트니크바이러스와 마이버스의 2개 속인 라비다바이러스과(Lavidaviridae)는 바이러스 분류를 위해 국제 바이러스 분류위원회에 의해 설립되었습니다.이 과는 프리클라우소비랄레스목(리투아니아 프리클라우소마목, "의존")에 속하는 유일한 과로, 다시 매버릭 트랜스포존목(Maverick transposons)[6][10]에 속하는 유일한 과이다.
- 라비다바이러스과
- 스푸트니크바이러스속
- 미미바이러스 의존성 바이러스 스푸트니크
- 미미바이러스 의존성 바이러스 자밀론
- 마이바이러스속
- 카페테리아 바이러스 의존형 mavirus
- 스푸트니크바이러스속
- 미지정속
또한 메타제놈에서 확인된 비로파지 게놈은 격리된 비로파지와 함께 일관된 게놈 길이, 유전자 함량, 서식지 분포를 [8]가진 27개의 다른 군으로 분류되었다.로키의 성 메타제놈에서 [11]일부 단편적인 생물영양학적 염기서열이 추가로 보고되었다.

레퍼런스
- ^ a b c d Mougari, S., Sahmi-Bounsiar, D., Levasseur, A., Colson, P. 및 La Scola, B.(2019) "거대 바이러스의 생물:11시에 갱신.바이러스, 11(8): 733.doi: 10.3390/v11080733.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다. - ^ Duponchel, S; Fischer, MG (March 2019). "Viva lavidaviruses! Five features of virophages that parasitize giant DNA viruses". PLOS Pathogens. 15 (3): e1007592. doi:10.1371/journal.ppat.1007592. PMC 6428243. PMID 30897185.
- ^ Fischer MG, Suttle CA (April 2011). "A virophage at the origin of large DNA transposons". Science. 332 (6026): 231–4. Bibcode:2011Sci...332..231F. doi:10.1126/science.1199412. PMID 21385722. S2CID 206530677.
- ^ Fischer MG, Hackl (December 2016). "Host genome integration and giant virus-induced reactivation of the virophage mavirus". Nature. 540 (7632): 288–91. Bibcode:2016Natur.540..288F. doi:10.1038/nature20593. PMID 27929021. S2CID 4458402.
- ^ a b c Katzourakis, Aris; Aswad, Amr (2014). "The origins of giant viruses, virophages and their relatives in host genomes". BMC Biology. 12: 2–3. doi:10.1186/s12915-014-0051-y. PMC 4096385. PMID 25184667.
- ^ a b c d e Krupovic, Mart; Kuhn, Jens; Fischer, Metthias (Fall 2015). "A classification system for virophages and satellite viruses" (PDF). Archives of Virology. 161 (1): 233–247. doi:10.1007/s00705-015-2622-9. PMID 26446887. S2CID 14196910 – via Springer.
- ^ a b Roux, Simon; Chan, Leong-Keat; Egan, Rob; Malmstrom, Rex R.; McMahon, Katherine D.; Sullivan, Matthew B. (2017-10-11). "Ecogenomics of virophages and their giant virus hosts assessed through time series metagenomics". Nature Communications. 8 (1): 858. Bibcode:2017NatCo...8..858R. doi:10.1038/s41467-017-01086-2. ISSN 2041-1723. PMC 5636890. PMID 29021524.
- ^ a b c Paez-Espino, David; Zhou, Jinglie; Roux, Simon; Nayfach, Stephen; Pavlopoulos, Georgios A.; Schulz, Frederik; McMahon, Katherine D.; Walsh, David; Woyke, Tanja; Ivanova, Natalia N.; Eloe-Fadrosh, Emiley A.; Tringe, Susannah G.; Kyrpides, Nikos C. (2019-12-10). "Diversity, evolution, and classification of virophages uncovered through global metagenomics". Microbiome. 7 (1): 157. doi:10.1186/s40168-019-0768-5. PMC 6905037. PMID 31823797.
- ^ Born, D; Reuter, L; Mersdorf, U; Mueller, M; Fischer, MG; Meinhart, A; Reinstein, J (10 July 2018). "Capsid protein structure, self-assembly, and processing reveal morphogenesis of the marine virophage mavirus". Proceedings of the National Academy of Sciences of the United States of America. 115 (28): 7332–7337. doi:10.1073/pnas.1805376115. PMC 6048507. PMID 29941605.
- ^ Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (October 2019). "Create a megataxonomic framework, filling all principal taxonomic ranks, for DNA viruses encoding vertical jelly roll-type major capsid proteins". ICTV Proposal (Taxoprop): 2019.003G. doi:10.13140/RG.2.2.14886.47684.
- ^ Bäckström D, Yutin N, Jørgensen SL, Dharamshi J, Homa F, Zaremba-Niedwiedzka K, Spang A, Wolf YI, Koonin EV, Ettema TJ (2019). "Virus genomes from deep sea sediments expand the ocean megavirome and support independent origins of viral gigantism". mBio. 10 (2): e02497-18. doi:10.1128/mBio.02497-18. PMC 6401483. PMID 30837339.PDF
- ^ 듀폰첼, S.와 피셔, M.G. (2019) "비바 라비다바이러스!거대 DNA 바이러스를 기생시키는 5가지 생물영양체.PLoS 병원체, 15(3)doi:10.1371/journal.ppat.1007592.
 자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.
자료는 Creative Commons Attribution 4.0 International License에 따라 제공되는 이 소스로부터 복사되었습니다.