오퍼론
Operon유전학에서 오퍼론은 단일 [1]프로모터에 의해 제어되는 유전자 클러스터를 포함하는 DNA의 기능 단위이다.유전자는 함께 mRNA 가닥으로 전사되고 세포질에서 함께 번역되거나, 또는 개별적으로 번역되는 단일istronic mRNA, 즉 각각 단일 유전자 생성물을 코드하는 여러 가닥의 mRNA를 만들기 위해 스플라이싱을 거친다.그 결과 오퍼론에 포함된 유전자가 함께 발현되거나 전혀 발현되지 않는다.오퍼론을 [2]정의하기 위해서는 여러 유전자가 함께 전사되어야 한다.
원래, 오퍼론은 원핵생물에만 존재하는 것으로 생각되었지만, 1990년대 [3][4]초 진핵생물에서 첫 오퍼론이 발견된 이후, 이전에 [5]추정되었던 것보다 더 흔하다는 것을 암시하는 더 많은 증거가 생겨났다.일반적으로, 원핵 오퍼론의 발현은 다핵성 mRNA의 생성으로 이어지는 반면, 진핵성 오퍼론은 단핵성 mRNA로 이어진다.
오퍼론은 박테리오파지와 [6][7]같은 바이러스에서도 발견된다.예를 들어 T7 페이지는 2개의 오퍼론이 있습니다.첫 번째 오퍼론은 두 번째 오퍼론과 결합하고 전사할 수 있는 특수 T7 RNA 중합효소를 포함하여 다양한 제품을 코드화합니다.두 번째 오퍼론은 숙주세포를 [8]폭발시키는 용융유전자를 포함한다.
역사
"[9]오페론"이라는 용어는 1960년 프랑스 과학 아카데미 회보에 실린 짧은 논문에서 처음 제안되었습니다.이 논문으로부터, 이른바 오퍼론의 일반 이론이 전개되었다.이 이론은 모든 경우에, 오퍼론 내의 유전자는 첫 번째 유전자 앞에 위치한 단일 오퍼레이터에서 작용하는 억제제에 의해 부정적으로 통제된다고 제안했다.나중에, 유전자는 적극적으로 조절될 수 있고 또한 전사가 시작되는 단계에서 조절될 수 있다는 것이 발견되었다.따라서 오퍼론마다 메커니즘이 다르기 때문에 일반적인 규제 메커니즘에 대해서는 언급할 수 없습니다.오늘날, 오퍼론은 단순히 단일 mRNA 분자로 변환된 유전자 클러스터로 정의된다.그럼에도 불구하고, 이 개념의 발전은 분자생물학 역사에서 획기적인 사건으로 여겨진다.첫 번째로 기술된 오퍼론은 [9]대장균의 라크 오퍼론이었다.1965년 노벨 생리의학상은 오퍼론과 바이러스 합성에 관한 발견으로 프랑수아 야콥, 앙드레 미셸 르보프, 자크 모노드에게 수여되었다.
개요
오퍼론은 주로 원핵생물에서 발생하지만 C. 엘레건과 초파리, 드로소필라 멜라노가스터와 같은 선충류를 포함한 일부 진핵생물에서도 발생한다.rRNA 유전자는 종종 코드네이트를 포함한 진핵생물 범위에서 발견된 오퍼론에서 존재한다.오퍼론은 공통 프로모터 아래에 배치되어 공통 오퍼레이터에 의해 조절되는 몇 가지 구조 유전자로 구성된다.그것은 일련의 인접한 구조 유전자와 구조 5[11]유전자의 전사에 영향을 미치는 인접한 조절 신호로 정의된다.억제기, 코어프레서 및 활성기를 포함한 특정 오퍼론의 규제자는 해당 오퍼론에 의해 반드시 코드화되지 않는다.조절자, 프로모터, 조작자 및 구조 DNA 배열의 위치와 조건은 일반적인 돌연변이의 영향을 결정할 수 있다.
오퍼론은 조절제, 각성제 및 모듈론과 관련이 있습니다; 반면에 오퍼론은 동일한 조작자에 의해 조절되는 유전자 세트를 포함하고, 조절제는 단일 조절 단백질에 의해 조절되는 유전자 세트를 포함하고, 각성제는 단일 세포 자극에 의해 조절되는 유전자 세트를 포함합니다.저자에 따르면, "operon"이라는 용어는 "작동하다"[12]라는 동사에서 유래했다.
전사 단위로서
오퍼론은 일반적으로 하나의 폴리시스트론 mRNA(하나 이상의 단백질을 코드하는 단일 mRNA 분자)로 전사되는 하나 이상의 구조 유전자를 포함한다.그러나 오퍼론의 정의에서는 mRNA가 폴리시스트론일 필요는 없지만 실제로는 폴리시스트론일 경우가 많습니다.[5]구조유전자의 상류에는 RNA중합효소가 결합하고 전사를 시작하는 부위를 제공하는 프로모터 배열이 있다.프로모터 근처에 연산자라고 불리는 DNA 부분이 있다.
오퍼론 대 원핵 유전자 클러스터링
오퍼론의 모든 구조 유전자는 그들 상류에 있는 단일 프로모터와 오퍼레이터에 의해 함께 켜지거나 꺼지지만, 때때로 유전자 발현에 대한 더 많은 제어가 필요하다.이러한 측면을 달성하기 위해, 몇몇 박테리아 유전자들은 서로 가까이 위치하지만, 그들 각각에 대한 특정 프로모터가 있습니다; 이것은 유전자 클러스터링이라고 불립니다.보통 이 유전자들은 신진대사 경로와 같은 동일한 경로에서 함께 작용하는 단백질을 암호화한다.유전자 클러스터링은 원핵세포가 올바른 [13]순서로 대사 효소를 생성하도록 돕는다.
일반구조
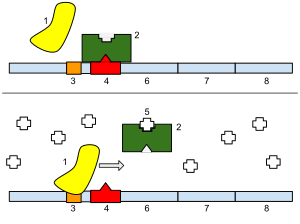
오퍼론은 다음의 3가지 기본 DNA 성분으로 구성됩니다.
- 프로모터 – 유전자의 전사를 가능하게 하는 뉴클레오티드 배열.프로모터는 RNA 중합효소에 의해 인식되며, RNA 중합효소는 전사를 시작합니다.RNA 합성에 있어서, 촉진제는 메신저 RNA 생성에 어떤 유전자를 사용해야 하는지 지시하고, 나아가 세포가 생성하는 단백질을 조절합니다.
- 연산자 – 억제제가 결합하는 DNA 세그먼트.이는 락 오퍼론에서 프로모터와 오퍼론의 [14]유전자 사이의 세그먼트로 고전적으로 정의된다.라크 오퍼론의 주 측정 시스템(O1)은 프로모터의 약간 하류에 위치하며, 두 개의 추가 측정 시스템(O2 및 O3)은 각각 -82와 +412에 위치합니다.억제제의 경우 억제단백질은 RNA중합효소가 유전자를 전사하는 것을 물리적으로 방해한다.
- 구조 유전자 – 오퍼론에 의해 공동 조절되는 유전자.
오퍼론에 항상 포함되는 것은 아니지만, 그 기능에서 중요한 것은 조절 유전자, 억제 단백질을 코드하는 끊임없이 발현되는 유전자입니다.조절 유전자는 [15]그것을 제어하기 위해 오퍼론 안에, 인접하거나 심지어 근처에 있을 필요가 없습니다.
유도체(소분자)는 조작자 부위(DNA)로부터 억제체(단백질)를 치환하여 억제되지 않는 오퍼론을 발생시킬 수 있다.
또는 코어프레서는 오퍼레이터 사이트에 바인드 할 수 있도록 리프레서에 바인드 할 수 있다.trp operon에서는 이러한 유형의 규제의 좋은 예를 볼 수 있습니다.
규정
오퍼론의 제어는 유기체가 환경 조건에 따라 다양한 유전자의 발현을 조절할 수 있도록 하는 유전자 조절의 한 종류이다.오퍼론 조절은 유도 또는 [14]억제에 의해 음수 또는 양수일 수 있습니다.
음성 제어는 전사를 방지하기 위해 억제제를 연산자에 결합하는 것을 포함합니다.
- 음성유도오페론에서는 조절억제단백질이 통상 오퍼론에 결합되어 있어 오퍼론상의 유전자의 전사를 방해한다.유도체 분자가 존재하면 억제체에 결합하고 그 구조를 변화시켜 연산자와 결합할 수 없다.이것에 의해, 오퍼론의 표현이 가능하게 됩니다.락 오퍼론은 유도체 분자가 알로락토스인 음성 제어 유도체 오퍼론이다.
- 음의 억제 가능한 오퍼론에서는, 통상, 오퍼론의 전사가 행해집니다.억제단백질은 조절유전자에 의해 생성되지만, 정상적인 형태에서는 조작자와 결합할 수 없다.그러나 코어프레서라고 불리는 특정 분자는 억제 단백질에 의해 결합되어 활성 부위에 구조 변화를 일으킨다.활성화된 억제 단백질은 조작자에게 결합되어 전사를 방지합니다.트립토판의 합성에 관여하는 trp 오퍼론은(그 자체가 코어프레서 역할을 함) 음으로 제어되는 억제 가능한 오퍼론입니다.
오퍼론도 확실하게 제어할 수 있습니다.양성 대조군에서는 활성제 단백질이 DNA에 결합함으로써 전사를 자극합니다(일반적으로 조작자 이외의 부위).
- 양성 유도 오퍼론에서 활성제 단백질은 일반적으로 관련 DNA에 결합할 수 없다.유도체가 활성제 단백질에 결합하면 DNA에 결합하고 전사를 활성화하기 위해 구조 변화를 겪는다.
- 양성 억제성 오퍼론에서 활성제 단백질은 일반적으로 관련 DNA 세그먼트에 결합됩니다.그러나 활성제에 의해 억제제가 결합되면 DNA와 결합하는 것이 방지된다.이것에 의해, 시스템의 액티브화와 전사가 정지됩니다.
랙 오퍼론
모델 박테리아인 대장균의 라크 오퍼론은 최초로 발견된 오퍼론이며 오퍼론 기능의 전형적인 예를 제공한다.그것은 프로모터, 터미네이터, 연산자 등 세 개의 인접한 구조 유전자로 구성되어 있다.라크 오퍼론은 포도당과 유당의 가용성을 포함한 여러 요인에 의해 조절됩니다.그것은 알로락토스에 의해 활성화 될 수 있다.유당은 억제 단백질에 결합하고 유전자 전사를 억제하는 것을 막습니다.이것은 억제할 수 없는(위에서: 음의 유도) 모델의 예입니다.그래서 그것은 유당이나 알로락토스의 존재에 의해 유도되는 음의 유도성 오퍼론이다.
trp 오퍼론
1953년 Jacques Monod와 동료들에 의해 발견된 대장균의 trp 오퍼론은 억제할 수 있는 오퍼론 중 처음으로 발견되었다.라크 오퍼론은 화학 물질(알로락토스)에 의해 활성화될 수 있지만 트립토판(Trp) 오퍼론은 화학 물질(트립토판)에 의해 억제된다.이 오퍼론은 트립토판 합성효소를 코드하는 trp E, trp D, trp C, trp B 및 trp A의 5가지 구조 유전자를 포함합니다.또한 RNA 중합효소에 결합하는 프로모터와 해당 오퍼레이터에 결합하는 억제 유전자(trp R)에 의해 합성되는 단백질에 결합되었을 때 전사를 차단하는 오퍼레이터를 포함한다.락오페론에서는 유당이 억제단백질에 결합해 유전자 전사를 억제하는 것을 막는 반면 트립토판은 억제단백질에 결합해 유전자 전사를 억제하는 것을 가능하게 한다.또한 라크 오퍼론과 달리 trp 오퍼론은 단계적 조절을 [16]가능하게 하는 리더펩타이드 및 감쇠기 배열을 포함한다.이것은 코어 압축 가능 모델의 예입니다.
오퍼레이터의 수와 조직 예측
오퍼론의 수와 구성은 대장균에서 가장 비판적으로 연구되어 왔다.그 결과, 유기체의 게놈 배열에 근거해 예측을 실시할 수 있다.
하나의 예측방법은 게놈 내 오퍼론 수의 일차적인 예측자로서 판독 프레임 사이의 유전자 간 거리를 사용한다.이 분리에 의해 프레임이 변경되어 읽기 효율이 향상됩니다.더 긴 스트레칭은 오퍼론이 시작되고 멈추는 곳에 존재하며, 종종 최대 40~[17]50개의 베이스가 있습니다.
오퍼론을 예측하는 다른 방법은 두 개 이상의 [18]게놈에서 유전자 순서와 배향이 보존된 유전자 클러스터를 찾는 것이다.
분자의 기능적 클래스를 고려한다면 오퍼론 예측은 훨씬 더 정확하다.박테리아는 단백질 복합체, 공통 경로 또는 공유 기질과 운반체에 대한 공동 개입에 의해 격리된 단위로 판독 프레임을 묶었다.따라서 정확한 예측에는 이러한 모든 데이터가 포함되며, 이는 정말 어려운 작업입니다.
파스칼 코사트의 실험실은 미생물인 리스테리아 모노사이토제네스의 모든 오퍼론을 실험적으로 확인한 최초의 실험실이었다.517 폴리시스트론 오퍼론은 다양한 조건에서 [19]L. 모노사이토겐에서 발생하는 전사의 전지구적 변화를 설명하는 2009년 연구에 나열되어 있습니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Sadava DE, Hillis DM, Heller HC, Berenbaum M (2009). Life: The Science of Biology (9th ed.). Macmillan. p. 349. ISBN 978-1-4292-1962-4.
- ^ Lodish H, Zipursky L, Matsudaira P, Baltimore D, Darnel J (2000). "Chapter 9: Molecular Definition of a Gene". Molecular Cell Biology. W. H. Freeman. ISBN 978-0-7167-3136-8.
- ^ Spieth J, Brooke G, Kuersten S, Lea K, Blumenthal T (May 1993). "Operons in C. elegans: polycistronic mRNA precursors are processed by trans-splicing of SL2 to downstream coding regions". Cell. 73 (3): 521–32. doi:10.1016/0092-8674(93)90139-H. PMID 8098272. S2CID 26918553.
- ^ Brogna S, Ashburner M (April 1997). "The Adh-related gene of Drosophila melanogaster is expressed as a functional dicistronic messenger RNA: multigenic transcription in higher organisms". The EMBO Journal. 16 (8): 2023–31. doi:10.1093/emboj/16.8.2023. PMC 1169805. PMID 9155028.
- ^ a b Blumenthal T (November 2004). "Operons in eukaryotes". Briefings in Functional Genomics & Proteomics. 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
- ^ "Definition of Operon". Medical Dictionary. MedicineNet.com. Retrieved 30 December 2012.
- ^ Liu J, Mushegian A (July 2004). "Displacements of prohead protease genes in the late operons of double-stranded-DNA bacteriophages". Journal of Bacteriology. 186 (13): 4369–75. doi:10.1128/JB.186.13.4369-4375.2004. PMC 421614. PMID 15205439.
- ^ "Bacteriophage Use Operons". Prokaryotic Gene Control. Dartmouth College. Archived from the original on 28 January 2013. Retrieved 30 December 2012.
- ^ a b Jacob F, Perrin D, Sanchez C, Monod J (February 1960). "[Operon: a group of genes with the expression coordinated by an operator]" [Operon: a group of genes with the expression coordinated by an operator] (PDF). Comptes Rendus Hebdomadaires des Séances de l'Académie des Sciences (Facsimile version reprinted in 2005) (in French). 250 (6): 1727–9. PMID 14406329. Archived from the original (PDF) on 2016-03-04. Retrieved 2015-08-27.
- ^ Shafee, Thomas; Lowe, Rohan (2017). "Eukaryotic and prokaryotic gene structure". WikiJournal of Medicine. 4 (1). doi:10.15347/wjm/2017.002. ISSN 2002-4436.
- ^ Miller JH, Suzuki DT, Griffiths AJ, Lewontin RC, Wessler SR, Gelbart WM (2005). Introduction to genetic analysis (8th ed.). San Francisco: W.H. Freeman. p. 740. ISBN 978-0-7167-4939-4.
- ^ Jacob F (May 2011). "The birth of the operon". Science. 332 (6031): 767. Bibcode:2011Sci...332..767J. doi:10.1126/science.1207943. PMID 21566161.
- ^ Lee JM, Sonnhammer EL (May 2003). "Genomic gene clustering analysis of pathways in eukaryotes". Genome Research. 13 (5): 875–82. doi:10.1101/gr.737703. PMC 430880. PMID 12695325.
- ^ a b Lewin B (1990). Genes IV (4th ed.). Oxford: Oxford University Press. pp. 243–58. ISBN 978-0-19-854267-4.
- ^ Mayer G. "Bacteriology – Chapter Nine Genetic Regulatory Mechanisms". Microbiology and Immunology Online. University of South Carolina School of Medicine. Retrieved 30 December 2012.
- ^ Cummings MS, Klug WS (2006). Concepts of genetics (8th ed.). Upper Saddle River, NJ: Pearson Education. pp. 394–402. ISBN 978-0-13-191833-7.
- ^ Salgado H, Moreno-Hagelsieb G, Smith TF, Collado-Vides J (June 2000). "Operons in Escherichia coli: genomic analyses and predictions". Proceedings of the National Academy of Sciences of the United States of America. 97 (12): 6652–7. Bibcode:2000PNAS...97.6652S. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
- ^ Ermolaeva MD, White O, Salzberg SL (March 2001). "Prediction of operons in microbial genomes". Nucleic Acids Research. 29 (5): 1216–21. doi:10.1093/nar/29.5.1216. PMC 29727. PMID 11222772.
- ^ Toledo-Arana A, Dussurget O, Nikitas G, Sesto N, Guet-Revillet H, Balestrino D, Loh E, Gripenland J, Tiensuu T, Vaitkevicius K, Barthelemy M, Vergassola M, Nahori MA, Soubigou G, Régnault B, Coppée JY, Lecuit M, Johansson J, Cossart P (June 2009). "The Listeria transcriptional landscape from saprophytism to virulence". Nature. 459 (7249): 950–6. Bibcode:2009Natur.459..950T. doi:10.1038/nature08080. PMID 19448609. S2CID 4341657.
외부 링크
- 결핵균 H37Rv 오퍼론 상관 브라우저
- OBD - Operon 데이터베이스(단, 사용하기에 다소 불편함)



