트립토판합성효소
Tryptophan synthase| 트립토판 합성효소 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
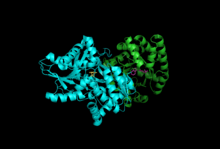 서브유닛:베타 서브유닛, PLP를 포함한 알파 서브유닛, IGP | |||||||||
| 식별자 | |||||||||
| EC 번호 | 4.2.1.20 | ||||||||
| CAS 번호 | 9014-52-2 | ||||||||
| 데이터베이스 | |||||||||
| 인텐츠 | IntEnz 뷰 | ||||||||
| 브렌다 | 브렌다 엔트리 | ||||||||
| ExPASy | NiceZyme 뷰 | ||||||||
| 케그 | KEGG 엔트리 | ||||||||
| 메타사이크 | 대사 경로 | ||||||||
| 프라이머리 | 프로필 | ||||||||
| PDB 구조 | RCSB PDB PDBe PDBum | ||||||||
| 진 온톨로지 | AmiGO / QuickGO | ||||||||
| |||||||||
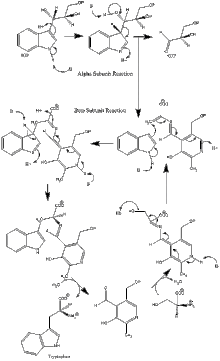
트립토판 합성효소 또는 트립토판 합성효소는 트립토판의 [1][2]생합성에서 마지막 두 단계를 촉매하는 효소(EC 4.2.1.20)이다.그것은 유박테리아,[3] 고세균,[4] 원생동물,[5] 곰팡이,[6][7] 플랜태에서 흔히 발견된다.그러나 Animalia에는 [8]없다.일반적으로 α2β2 [9][10]사량체로 발견된다.α 서브유닛은 인돌-3-글리세롤인산(IGP)에서 인돌과 글리세린알데히드-3-인산(G3P)의 가역적 형성을 촉매한다.β 서브유닛은 인돌과 세린의 비가역적 축합을 촉매하여 피리독살 인산염(PLP)의존성 반응에서 트립토판을 형성한다.각 α활성부위는 효소 내에 포함된 25앵스트롬 길이의 소수성 채널에 의해 β활성부위에 접속된다.이것은 기질 [11]채널링으로 알려진 과정에서 α 활성 부위에 형성된 인돌을 β 활성 부위로 직접 확산시키는 것을 촉진한다.트립토판 합성효소의 활성 부위는 알로스테릭하게 [12]결합되어 있다.
효소구조
서브유닛:트립토판 합성효소는 전형적으로 α-β-α 복합체로 존재한다.α와 β 서브유닛은 각각 27과 43kDa의 분자량을 가진다.α 서브유닛은 TIM 배럴 구성을 가집니다.β 서브유닛은 접이식 II 배치와 1가 양이온을 [13]위한 활성 부위에 인접한 결합 부위가 있다.복합체로의 결합은 양쪽 서브유닛의 구조적 변화로 이어지며 상호 활성화로 이어집니다.서브 유닛간 통신에는, 주로 2개의 메카니즘이 있습니다.먼저 β-서브유닛의 COMM 도메인과 α-루프2가 상호작용한다.또한 αGly181 잔기와 βSer178 [14]잔기 사이에는 상호작용이 있다.활성 부위는 알로스테릭하게 조절되며 열린 상태,[12] 비활성 상태 및 닫힌 상태, 활성 상태 간에 전환됩니다.
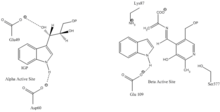
인돌-3-글리세롤 결합 부위:그림 1을 참조해 주세요.
인돌 및 세린 결합 부위:그림 1을 참조해 주세요.
소수성 채널:α 및 β 활성 부위는 인돌의 확산을 허용하는 효소 내에 포함된 25앵스트롬 길이의 소수성 채널에 의해 분리된다.채널이 존재하지 않으면 α 활성 부위에서 형성된 인돌은 빠르게 확산되어 소수성이며 쉽게 막을 통과할 수 있기 때문에 세포로 손실된다.따라서 채널은 효소 복합기능에 [15]필수적이다.
효소 메커니즘
α 서브유닛 반응:α 서브유닛은 IGP의 역알돌 분해로부터 인돌과 G3P의 형성을 촉매한다.αGlu49와 αAsp60은 [11]그림과 같이 촉매 작용에 직접 관여하는 것으로 생각된다.환율 제한 스텝은 IGP의 [16]이성화입니다.그림 2를 참조해 주세요.
β 서브유닛 반응:β 서브유닛은 인돌과 세린이 응축되어 PLP 의존성 반응에서 트립토판을 형성하는 β 치환 반응을 촉매한다.βLys87, βGlu109 및 βSer377은 [11]그림과 같이 촉매 작용에 직접 관여하는 것으로 생각된다.다시 말하지만, 정확한 메커니즘은 결정적으로 결정되지 않았다.그림 2를 참조해 주세요.
순반응:그림 3을 참조해 주세요.
생물학적 기능
트립토판 합성효소는 에우박테리아, 고세균, 원생동물, 곰팡이, 식물에서 흔히 발견된다.그것은 인간과 같은 동물에게는 존재하지 않는다.트립토판은 20개의 표준 아미노산 중 하나이며 인간을 위한 9개의 필수 아미노산 중 하나이다.따라서 트립토판은 인간의 식단에 꼭 필요한 요소이다.
기판범위
또한 트립토판 합성효소는 인돌 유사체, 예를 들어 불소화 또는 메틸화 인돌을 기질로 수용하여 대응하는 트립토판 [17]유사체를 생성하는 것으로 알려져 있다.
질병 관련성
인간에게는 트립토판 합성효소가 없기 때문에 이 효소는 잠재적인 약물 [18]표적으로 연구되어 왔다.그러나 박테리아는 아미노산을 생성하기 위한 대체 메커니즘을 가지고 있다고 생각되는데, 이는 이 접근 방식을 덜 효과적으로 만들 수 있다.어느 경우든, 비록 그 약이 박테리아를 약화시킬 뿐이지만, 그 박테리아는 이미 적대적인 숙주 환경에서 취약하기 때문에 여전히 유용할 수 있다.이와 같이 아미노산 대사에서 다른 PLP 효소와 함께 트립토판 합성효소의 억제는 의학적 [19]문제를 해결하는 데 도움을 줄 가능성이 있다.
아미노산 대사에서 트립토판 합성효소 및 기타 PLP 효소의 억제는 다음과 같은 이유로 제안되었다.
진화
진화 초기에 trpB2 유전자는 복제되었다고 생각된다.1개의 복사본이 trp 오퍼론에 trpB2i로 입력되어 trpA로 표현됩니다.TrpB2i는 TrpA와 과도 복합체를 형성하고 그 과정에서 단방향으로 TrpA가 활성화되었다.다른 카피는 trpB2o로 외부에 남아 기존 역할을 수행하거나 인돌의 인양 단백질 역할을 하는 등 새로운 역할을 수행했다.TrpB2i는 TrpB1로 진화하여 TrpA와 영구 복합체를 형성하여 양방향 활성화를 유발하였다.인돌 인양 단백질의 이점은 감소했고 TrpB 유전자는 손실되었다.마지막으로 TrpB1 및 TrpA 유전자가 융합되어 이관능성 [22]효소가 형성되었다.
역사적 의의
트립토판 합성효소는 광범위하게 연구된 두 가지 촉매 기능을 가진 것으로 확인된 첫 번째 효소였다.또한 기판 채널링을 최초로 사용한 것으로 확인되었습니다.이와 같이, 이 효소는 광범위하게 연구되어 큰 [11]관심을 받고 있는 주제이다.
「 」를 참조해 주세요.
레퍼런스
- ^ Dunn MF, Niks D, Ngo H, Barends TR, Schlichting I (June 2008). "Tryptophan synthase: the workings of a channeling nanomachine". Trends in Biochemical Sciences. 33 (6): 254–64. doi:10.1016/j.tibs.2008.04.008. PMID 18486479.
- ^ Miles EW (1991). "Structural basis for catalysis by tryptophan synthase". Advances in Enzymology and Related Areas of Molecular Biology. 64: 93–172. doi:10.1002/9780470123102.ch3. PMID 2053470.
- ^ Jablonski P, Jablonski L, Pintado O, Sriranganathan N, Howde C (September 1996). "Tryptophan synthase: Identification of Pasteurella multocida tryptophan synthase B-subunit by antisera against strain PI059". Microbiology. 142: 115–21. doi:10.1099/13500872-142-1-115. PMID 8581158.
- ^ Lazcano A, Diaz-Villgomez E, Mills T, Oro J (March 1995). "On the levels of enzymatic substrate specificity: Implications for the early evolution of metabolic pathways". Advances in Space Research. 15 (3): 345–56. doi:10.1016/S0273-1177(99)80106-9. PMID 11539248.
- ^ Anderson I, Watkins R, Samuelson J, Spencer D, Majoros W, Grey M, Loftus B (August 2005). "Gene Discovery in the Acanthamoeba castellanii Genome". Protist. 156 (2): 203–14. doi:10.1016/j.protis.2005.04.001. PMID 16171187.
- ^ Ireland C, Peekhaus N, Lu P, Sangari R, Zhang A, Masurekar P, An Z (April 2008). "The tryptophan synthetase gene TRP1 of Nodulisporium sp.: molecular characterization and its relation to nodulisporic acid A production". Appl Microbiol Biotechnol. 79 (3): 451–9. doi:10.1007/s00253-008-1440-3. PMID 18389234.
- ^ Sanjaya, Hsiao PY, Su RC, Ko SS, Tong CG, Yang RY, Chan MT (April 2008). "Overexpression of Arabidopsis thaliana tryptophan synthase beta 1 (AtTSB1) in Arabidopsis and tomato confers tolerance to cadmium stress". Plant Cell Environ. 31 (8): 1074–85. doi:10.1111/j.1365-3040.2008.01819.x. PMID 18419734.
{{cite journal}}: CS1 maint: 여러 이름: 작성자 목록(링크) - ^ Eckert SC, Kubler E, Hoffmann B, Braus GH (June 2000). "The tryptophan synthase-encoding trpB gene of Aspergillus nidulans is regulated by the cross-pathway control system". Mol Gen Genet. 263 (5): 867–76. doi:10.1007/s004380000250. PMID 10905354.
- ^ Ahmed SA, Miles EW, Davies DR (March 1985). "Crystallization and preliminary X-ray crystallographic data of the tryptophan synthase alpha 2 beta 2 complex from Salmonella typhimurium". The Journal of Biological Chemistry. 260 (6): 3716–3718. doi:10.1016/s0021-9258(19)83682-7. PMID 3882715.
- ^ Hyde CC, Ahmed SA, Padlan EA, Miles EW, Davies DR (November 1988). "Three-dimensional structure of the tryptophan synthase alpha 2 beta 2 multienzyme complex from Salmonella typhimurium". The Journal of Biological Chemistry. 263 (33): 17857–17871. doi:10.1016/s0021-9258(19)77913-7. PMID 3053720.
- ^ a b c d Raboni S, Bettati S, Mozzarelli A (April 2009). "Tryptophan synthase: a mine for enzymologists". Cell Mol Life Sci. 66 (14): 2391–403. doi:10.1007/s00018-009-0028-0. hdl:11381/2293687. PMID 19387555.
- ^ a b Fatmi MQ, Ai R, Chang CA (September 2009). "Synergistic regulation and ligand-induced conformational changes of tryptophan synthase". Biochemistry. 48 (41): 9921–31. doi:10.1021/bi901358j. PMID 19764814.
- ^ Grishin NV, Phillips MA, Goldsmith EJ (July 1995). "Modeling of the spatial structure of ornithine decarboxylases". Protein Sci. 4 (7): 1291–304. doi:10.1002/pro.5560040705. PMC 2143167. PMID 7670372.
- ^ Schneider TR, Gerhardt E, Lee M, Liang PH, Anderson KS, Schlichting I (April 1998). "Loop closure and intersubunit communication in tryptophan synthase". Biochemistry. 37 (16): 5394–406. doi:10.1021/bi9728957. PMID 9548921.
- ^ Huang X, Holden HM, Raushel FM (2001). "Channeling of Substrates and Intermediates in Enzyme-Catalyzes Reactions". Annu Rev Biochem. 70: 149–80. doi:10.1146/annurev.biochem.70.1.149. PMID 11395405.
- ^ Anderson KS, Miles EW, Johnson KA (May 1991). "Serine modulates substrate channeling in tryptophan synthase. A novel intersubunit triggering mechanism". J Biol Chem. 266 (13): 8020–33. PMID 1902468.
- ^ Wilcox M (June 1974). "The enzymatic synthesis of L-tryptophan analogues". Analytical Biochemistry. 59 (2): 436–440. doi:10.1016/0003-2697(74)90296-6. PMID 4600987.
- ^ a b c Chaudhary K, Roos DS (September 2005). "Protozoan genomics for drug discovery". Nat Biotechnol. 23 (9): 1089–91. doi:10.1038/nbt0905-1089. PMC 7096809. PMID 16151400.
- ^ Becker D, Selbach M, Rollenhagen C, Ballmaier M, Meyer TF, Mann M, Bumann D (March 2006). "Robust Salmonella metabolism limits possibilities for new antimicrobials". Nature. 440 (7082): 303–7. doi:10.1038/nature04616. PMID 16541065.
- ^ Caldwell HD, Wood H, Crane D, Baily R (June 2003). "Polymorphisms in Chlamydia trachomatis tryptophan synthase genes differentiate between genital and ocular isolates". J Clin Invest. 111 (11): 1757–69. doi:10.1172/JCI17993. PMC 156111. PMID 12782678.
- ^ Kulik V, Hartmann E, Weyand M, Frey M, Gierl A, Niks D, Dunn MF, Schlichting I (September 2005). "On the structural basis of the catalytic mechanism and the regulation of the α-subunit of tryptophan synthase from Salmonella typhimurium and BXI from maize, two evolutionarily related enzymes". J Mol Biol. 352 (3): 608–20. doi:10.1016/j.jmb.2005.07.014. PMID 16120446.
- ^ Leopoldseder S, Hettwer S, Sterner R (November 2006). "Evolution of Multi-Enzyme Complexes: The Case of Tryptophan Synthase". Biochemistry. 45 (47): 14111–9. doi:10.1021/bi061684b. PMID 17115706.