핵산 배열
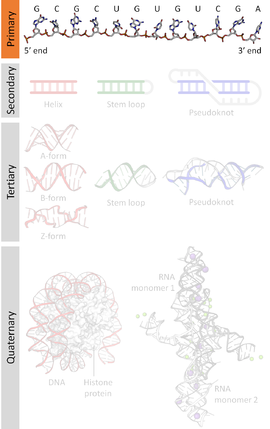
Nucleic acid sequence핵산 배열은 DNA(GACT) 또는 RNA(GACU) 분자 내에서 대립 유전자를 형성하는 뉴클레오티드의 순서를 나타내는 일련의 다른 다섯 글자로 나타나는 염기들의 연속이다.관례상 시퀀스는 보통 5' 끝에서 3' 끝까지 제시된다.DNA는 센스 스트랜드가 사용됩니다.핵산은 일반적으로 선형(분지되지 않은) 중합체이기 때문에, 염기서열을 지정하는 것은 전체 분자의 공유 구조를 정의하는 것과 같다.따라서 핵산 배열을 1차 구조라고도 한다.
시퀀스에는 정보를 나타내는 용량이 있습니다.생물학적 디옥시리보핵산은 유기체의 기능을 지시하는 정보를 나타낸다.
핵산은 또한 2차 구조와 3차 구조를 가지고 있다.프라이머리 구조를 프라이머리 시퀀스로 잘못 부르는 경우가 있습니다.반대로 2차 또는 3차 시퀀스의 병렬 개념은 없습니다.
뉴클레오티드
핵산은 뉴클레오티드라고 불리는 연결된 단위들의 사슬로 구성되어 있다.각 뉴클레오티드는 3개의 소단위: 인산기와 당(RNA의 경우 리보스, DNA의 디옥시리보스)으로 구성되어 있으며, 당에 결합되어 있는 핵염기 중 하나이다.핵염기는 유명한 이중 나선과 같은 높은 수준의 2차 및 3차 구조를 형성하기 위해 가닥의 염기쌍에 중요하다.
가능한 문자는 A, C, G, T이며, 포스포디에스테르 골격에 공유 결합되어 있는 DNA 가닥의 네 가지 뉴클레오티드 염기인 아데닌, 시토신, 구아닌, 티민을 나타낸다.전형적인 경우, 시퀀스 AAAGTCTGAC가 5'~3' 방향으로 왼쪽에서 오른쪽으로 읽히듯이, 갭 없이 서로 맞닿아 인쇄된다.전사에 관해서는 전사된 RNA와 순서가 같으면 코드 스트랜드 상에 배열이 있다.
한 시퀀스는 다른 시퀀스에 보완될 수 있으며, 이는 보완적(즉, A에서 T, C에서 G)의 각 위치에 기반을 두고 있다는 것을 의미한다.예를 들어, TTAC에 대한 보완 시퀀스는 GTAA입니다.이중 가닥 DNA의 한 가닥이 감지 가닥으로 간주되면, 반감지 가닥으로 간주되는 다른 가닥은 감지 가닥에 대한 상보적 서열을 가질 것입니다.
표기법
두 뉴클레오티드 배열 간의 % 차이 비교 및 결정.
- AATCC GCTAG
- AAACCCTTAG
- 두 개의 10-뉴클레오티드 배열이 주어졌을 때, 그것들을 일렬로 정렬하고 그들 사이의 차이를 비교하세요.다른 DNA 염기의 수를 뉴클레오티드의 총수로 나누어 유사도를 계산한다.이 경우 10뉴클레오티드 배열에는 3가지 차이가 있다.따라서 7/10을 나누면 70%의 유사성이 나오고 100%에서 빼면 30%의 차이가 납니다.
A, T, C 및 G는 특정 위치에서 특정 뉴클레오티드를 나타내지만, 그 위치에서 여러 종류의 뉴클레오티드가 발생할 수 있을 때 사용되는 모호성을 나타내는 문자도 있다.국제순수응용화학연합(IUPAC)의 규칙은 다음과 같다.[1]
| 기호[2] | 묘사 | 표시된 베이스 | 보완하다 | ||||
|---|---|---|---|---|---|---|---|
| A | 아데닌 | A | 1 | T | |||
| C | 시토신 | C | G | ||||
| G | 구아닌 | G | C | ||||
| T | 티민 | T | A | ||||
| U | 우라실 | U | A | ||||
| W | 약하다 | A | T | 2 | W | ||
| S | 강함 | C | G | S | |||
| M | a미노 | A | C | K | |||
| K | 케토 | G | T | M | |||
| R | 푸린 | A | G | Y | |||
| Y | 피리미딘 | C | T | R | |||
| B | A가 아니다(B는 A 뒤에 온다) | C | G | T | 3 | V | |
| D | C가 아님(D는 C 뒤에 있음) | A | G | T | H | ||
| H | G가 아님(H는 G 뒤에 있음) | A | C | T | D | ||
| V | T가 아님(V는 T와 U 뒤에 있음) | A | C | G | B | ||
| N | 모든 뉴클레오티드(틈이 아님) | A | C | G | T | 4 | N |
| Z | 제로 | 0 | Z | ||||
이러한 기호는 U(uracil)가 T(티민)[1]를 대체하는 경우를 제외하고 RNA에도 유효합니다.
아데닌(A), 시토신(C), 구아닌(G), 티민(T) 및 우라실(U) 외에 DNA 및 RNA는 핵산 사슬이 형성된 후 변경된 염기를 포함한다.DNA에서 가장 일반적인 변형 염기는 5-메틸시티딘(m5C)이다.RNA에는 의사우리딘(δ), 디히드로우리딘(D), 이노신(I), 리보시미딘(rT) 및 7-메틸구아노신(m7G)[3][4]을 포함한 많은 변성염기가 있다.히포산틴과 크산틴은 변이원 존재에 의해 생성된 많은 염기 중 두 가지이며, 둘 다 탈아미네이션(아민기를 카르보닐기로 대체)을 통해 생성된다.히포산틴은 아데닌에서 생성되며 크산틴은 [5]구아닌에서 생성된다.마찬가지로, 시토신의 탈아미노화는 우라실을 발생시킨다.
생물학적 의의
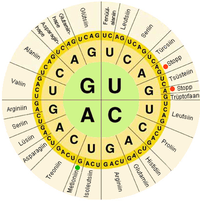
생물학적 시스템에서 핵산은 살아있는 세포에 의해 특정 단백질을 구성하기 위해 사용되는 정보를 포함한다.핵산 가닥 위의 핵염기 배열은 세포 기계에 의해 단백질 가닥을 구성하는 아미노산 배열로 변환됩니다.코돈이라고 불리는 세 개의 염기로 이루어진 각각의 그룹은 하나의 아미노산에 대응하고, 세 개의 염기의 각각의 가능한 조합이 특정한 아미노산에 대응하는 특정한 유전 코드가 있습니다.
분자생물학의 중심 교의는 핵산에 포함된 정보를 사용하여 단백질이 구성되는 메커니즘을 설명한다.DNA는 mRNA 분자로 전사되며, mRNA는 단백질 가닥을 구성하기 위한 템플릿으로 사용되는 리보솜으로 이동한다.핵산은 상보적인 염기서열을 가진 분자에 결합할 수 있기 때문에, 단백질을 코드하는 "감지" 염기서열과 그 자체로 기능하지는 않지만, 감지 가닥에 결합할 수 있는 "반감지" 염기서열 사이에는 차이가 있다.
시퀀스 결정
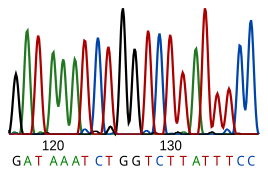
DNA 배열은 주어진 DNA 조각의 뉴클레오티드 배열을 결정하는 과정이다.생물의 DNA 배열은 그 생물이 생존하고 번식하는 데 필요한 정보를 암호화한다.그러므로, 염기서열을 결정하는 것은 응용 대상뿐만 아니라 유기체가 왜 어떻게 사는지에 대한 기초적인 연구에 유용하다.생물에게 DNA의 중요성 때문에, DNA 배열에 대한 지식은 실질적으로 어떤 생물학적 연구에서도 유용할 수 있다.예를 들어, 의학에서는 유전자 질환의 식별, 진단 및 잠재적으로 치료제 개발에 사용될 수 있다.마찬가지로 병원균에 대한 연구는 전염병 치료로 이어질 수 있다.바이오 테크놀로지는 많은 유용한 제품과 서비스의 가능성을 가진 급성장하고 있는 분야입니다.
RNA는 직접 배열되지 않습니다.대신 역전사효소에 의해 DNA로 복사되고 이 DNA는 배열된다.
현재의 배열 방법은 DNA 중합효소의 식별 능력에 의존하기 때문에 4개의 염기만 구별할 수 있다.이노신(RNA 편집 중 아데노신에서 생성)을 G로, 5-메틸시토신(DNA 메틸화에 의해 시토신에서 생성)을 C로 읽는다.현재의 기술로는 신호가 너무 약해서 측정하기 어렵기 때문에 소량의 DNA를 배열하는 것이 어렵다.이것은 중합효소 연쇄반응(PCR) 증폭에 의해 극복된다.
디지털 표현
생물로부터 핵산 배열을 얻은 후에는 디지털 형식으로 실리콘에 저장된다.디지털 유전자 배열은 배열 데이터베이스에 저장되고 분석되며(아래 배열 분석 참조), 디지털 방식으로 변경되며 인공 유전자 합성을 사용하여 새로운 실제 DNA를 생성하기 위한 템플릿으로 사용될 수 있습니다.
시퀀스 분석
디지털 유전자 배열은 그 기능을 결정하기 위해 생물정보학 도구를 사용하여 분석될 수 있다.
유전자 검사
유기체의 게놈에 있는 DNA는 유전병에 대한 취약성을 진단하기 위해 분석될 수 있고, 또한 아이의 아버지(유전자 아버지)나 사람의 조상을 결정하는 데 사용될 수 있다.보통, 모든 사람은 모든 유전자의 두 가지 변형을 가지고 있다. 하나는 어머니로부터 물려받은 것이고 다른 하나는 아버지로부터 물려받은 것이다.인간 게놈은 약 20,000-25,000개의 유전자를 가지고 있는 것으로 여겨진다.염색체를 개별 유전자의 수준으로 연구하는 것 외에, 넓은 의미의 유전자 검사에는 유전 질환의 존재 가능성, 즉 유전 질환의 발생 위험 증가와 관련된 돌연변이 형태의 유전자에 대한 생화학 검사가 포함됩니다.
유전자 검사는 염색체, 유전자 또는 [6]단백질의 변화를 식별한다.일반적으로 테스트는 유전적인 장애와 관련된 변화를 찾기 위해 사용됩니다.유전자 검사 결과는 의심되는 유전자 상태를 확인하거나 배제하거나 유전적인 장애가 발생하거나 유전될 가능성을 결정하는 데 도움을 줄 수 있다.현재 수백 개의 유전자 검사가 사용되고 있으며,[7][8] 더 많은 유전자 검사가 개발되고 있다.
시퀀스 얼라인먼트
생물정보학에서 배열 정렬은 DNA, RNA 또는 단백질의 배열로 [9]배열들 사이의 기능적, 구조적 또는 진화적 관계 때문일 수 있는 유사성 영역을 식별하는 방법입니다.얼라인먼트의 두 시퀀스가 공통의 조상을 공유하는 경우, 미스매치는 점 돌연변이와 갭으로 해석될 수 있으며, 서로 분기한 이후 한 계통 또는 두 계통에 도입된 삽입 또는 삭제 돌연변이(인델)로 해석될 수 있습니다.단백질 배열에서 배열의 특정 위치를 차지하는 아미노산 간의 유사도는 특정 영역 또는 배열 모티브가 계통 간에 얼마나 보존되어 있는지를 대략적으로 측정하는 것으로 해석할 수 있다.치환의 부재 또는 매우 보수적인 치환(즉, 곁사슬이 유사한 생화학적 특성을 가진 아미노산의 치환)만이 배열의 특정 영역에 존재하는 것은[10] 이 영역이 구조적 또는 기능적 중요성을 가지고 있음을 시사한다.DNA와 RNA 뉴클레오티드 염기는 아미노산보다 서로 더 유사하지만, 염기쌍의 보존은 유사한 기능적 또는 구조적 [11]역할을 나타낼 수 있다.
컴퓨터 계통유전학은 계통수 구성 및 해석에 배열배열을 광범위하게 이용하며, 이는 다른 종의 게놈에 나타나는 상동 유전자 간의 진화관계를 분류하는데 사용된다.쿼리 세트의 시퀀스가 다른 정도는 시퀀스 간의 진화적 거리와 질적으로 관련되어 있습니다.대략적으로 말하면, 높은 염기서열 정체성은 문제의 염기서열이 비교적 최근의 공통조상을 가지고 있다는 것을 암시하는 반면 낮은 염기서열 정체성은 그 차이가 더 오래되었다는 것을 암시한다.두 유전자가 처음 분리된 이후 경과된 시간(즉, 결합 시간)을 추정하기 위해 대략 일정한 비율의 진화적 변화를 사용할 수 있다는 "분자 시계" 가설을 반영하는 이 근사치는 돌연변이와 선택의 효과가 시퀀스 계통에 걸쳐 일정하다고 가정합니다.따라서 DNA 수복률이나 특정 영역의 기능적 보존률에서 생물이나 종 간의 가능한 차이를 설명하지 않는다.(뉴클레오티드 배열의 경우, 가장 기본적인 형태의 분자 시계 가설은 또한 주어진 코돈의 의미를 바꾸지 않는 침묵 돌연변이와 단백질에 다른 아미노산이 통합되는 결과를 초래하는 다른 돌연변이 사이의 수용률 차이를 감소시킨다.)통계적으로 더 정확한 방법은 계통수의 각 가지에 대한 진화 속도를 변화시켜 유전자의 결합 시간을 더 잘 추정할 수 있게 한다.
시퀀스 모티브
주요 구조는 기능적으로 중요한 모티브를 인코딩하는 경우가 많습니다.배열 모티브의 예로는 snoRNA의 C/D[12] 및 H/ACA 박스[13], U1, U2, U4, U5, U6, U12 및 U3, 샤인-달가르노 배열,[14] 코작 컨센서스[15] III 중합체 RNA가 있다.
시퀀스 엔트로피
생체정보학에서 배열 엔트로피(sequence complexity) 또는 정보 프로파일([17]information profile)이라고도 하는 배열 엔트로피는 처리 방향과는 독립적으로 DNA 배열의 국소 복잡도에 대한 정량적 측정을 제공하는 수치 배열이다.정보 프로파일을 조작하면, 예를 들면, 모티프나 재배열 [17][18]검출등의 정렬이 필요 없는 기술을 사용해 시퀀스를 분석할 수 있습니다.[19]
「 」를 참조해 주세요.
레퍼런스
- ^ a b Nomenclature for Uncomplete Specified Bases in Nuclear Acid Sequence, NC-IUB, 1984.
- ^ Nomenclature Committee of the International Union of Biochemistry (NC-IUB) (1984). "Nomenclature for Incompletely Specified Bases in Nucleic Acid Sequences". Retrieved 2008-02-04.
- ^ "BIOL2060: Translation". mun.ca.
- ^ "Research". uw.edu.pl.
- ^ Nguyen, T; Brunson, D; Crespi, C L; Penman, B W; Wishnok, J S; Tannenbaum, S R (April 1992). "DNA damage and mutation in human cells exposed to nitric oxide in vitro". Proc Natl Acad Sci USA. 89 (7): 3030–034. Bibcode:1992PNAS...89.3030N. doi:10.1073/pnas.89.7.3030. PMC 48797. PMID 1557408.
- ^ "What is genetic testing?". Genetics Home Reference. 16 March 2015. Archived from the original on 29 May 2006. Retrieved 19 May 2010.
- ^ "Genetic Testing". nih.gov.
- ^ "Definitions of Genetic Testing". Definitions of Genetic Testing (Jorge Sequeiros and Bárbara Guimarães). EuroGentest Network of Excellence Project. 2008-09-11. Archived from the original on February 4, 2009. Retrieved 2008-08-10.
- ^ Mount DM. (2004). Bioinformatics: Sequence and Genome Analysis (2nd ed.). Cold Spring Harbor Laboratory Press: Cold Spring Harbor, NY. ISBN 0-87969-608-7.
- ^ Ng, P. C.; Henikoff, S. (2001). "Predicting Deleterious Amino Acid Substitutions". Genome Research. 11 (5): 863–74. doi:10.1101/gr.176601. PMC 311071. PMID 11337480.
- ^ Witzany, G (2016). "Crucial steps to life: From chemical reactions to code using agents". Biosystems. 140: 49–57. doi:10.1016/j.biosystems.2015.12.007. PMID 26723230.
- ^ Samarsky, DA; Fournier MJ; Singer RH; Bertrand E (1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". The EMBO Journal. 17 (13): 3747–57. doi:10.1093/emboj/17.13.3747. PMC 1170710. PMID 9649444.
- ^ Ganot, Philippe; Caizergues-Ferrer, Michèle; Kiss, Tamás (1 April 1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes & Development. 11 (7): 941–56. doi:10.1101/gad.11.7.941. PMID 9106664.
- ^ Shine J, Dalgarno L (1975). "Determinant of cistron specificity in bacterial ribosomes". Nature. 254 (5495): 34–38. Bibcode:1975Natur.254...34S. doi:10.1038/254034a0. PMID 803646. S2CID 4162567.
- ^ Kozak M (October 1987). "An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs". Nucleic Acids Res. 15 (20): 8125–48. doi:10.1093/nar/15.20.8125. PMC 306349. PMID 3313277.
- ^ Bogenhagen DF, Brown DD (1981). "Nucleotide sequences in Xenopus 5S DNA required for transcription termination". Cell. 24 (1): 261–70. doi:10.1016/0092-8674(81)90522-5. PMID 6263489. S2CID 9982829.
- ^ a b Pinho, A; Garcia, S; Pratas, D; Ferreira, P (Nov 21, 2013). "DNA Sequences at a Glance". PLOS ONE. 8 (11): e79922. Bibcode:2013PLoSO...879922P. doi:10.1371/journal.pone.0079922. PMC 3836782. PMID 24278218.
- ^ Pratas, D; Silva, R; Pinho, A; Ferreira, P (May 18, 2015). "An alignment-free method to find and visualise rearrangements between pairs of DNA sequences". Scientific Reports. 5: 10203. Bibcode:2015NatSR...510203P. doi:10.1038/srep10203. PMC 4434998. PMID 25984837.
- ^ Troyanskaya, O; Arbell, O; Koren, Y; Landau, G; Bolshoy, A (2002). "Sequence complexity profiles of prokaryotic genomic sequences: A fast algorithm for calculating linguistic complexity". Bioinformatics. 18 (5): 679–88. doi:10.1093/bioinformatics/18.5.679. PMID 12050064.