유전성
Heritability유전성은 번식 및 유전학 분야에서 사용되는 통계로 개체 [1]간의 유전적 변이에 기인하는 개체군의 표현형 특성의 변화 정도를 추정한다.유전성의 개념은 다음과 같은 질문의 형태로 표현될 수 있다:[2] "환경이나 우연에 의해 설명되지 않는 모집단 내에서 주어진 특성에서 변화의 비율은?"
특성에서 측정된 변동의 다른 원인은 관찰 오류를 포함한 환경적 요인으로 특징지어집니다.유전성에 대한 인간연구에서는 같은 가구에서 자란 사람이 그렇지 않은 사람과 비슷하거나 비슷해지는 경향이 있는지 여부에 따라 종종 "공유 환경"과 "비공유 환경"의 요소로 분류된다.
유전성은 집단 내 관련 개체 간의 개별 표현형 변화를 비교하거나, 개별 표현형과 유전자형 [3][4]데이터 간의 연관성을 검사하거나, 심지어 게놈 전체 연관성 연구(GWAS)[5]의 요약 수준 데이터를 모델링함으로써 추정된다.유전성은 양적 유전학, 특히 선택적 교배와 행동 유전학(예: 쌍둥이 연구)에서 중요한 개념이다.그것은 그것의 기술적 정의가 일반적으로 이해되는 민속 정의와 다르다는 사실 때문에 많은 혼란의 원인이 되고 있다.따라서, 이것의 사용은 행동 특성이 [6]유전자에 의해 "상속"되거나 구체적으로 유전된다는 잘못된 인상을 전달한다.행동 유전학자들은 또한 유전자와 환경이 행동 특성에 [7]별개로, 부가적인 방식으로 기여한다는 가정에 기초하여 유전율 분석을 실시한다.
개요
유전성은 유전자 변이에 기인할 수 있는 표현형 변동의 비율을 측정합니다.이것은 개인의 표현형 중 이 부분이 유전학에 의해 발생한다고 말하는 것과 같지 않다.예를 들어, 성격 특성의 유전율이 0.6 정도이기 때문에, 성격의 60%는 부모님으로부터 물려받고 40%는 환경으로부터 물려받은다는 것은 잘못된 것입니다.또한, 유전성은 환경이 더 많은 변화를 일으키기 시작할 때처럼 어떠한 유전자 변화도 일어나지 않고 변할 수 있다.좋은 예로, 유전자와 환경 둘 다 지능에 영향을 미칠 수 있는 잠재력을 가지고 있다고 생각해라.유전적 변이가 증가하면 유전성이 증가하여 개인들이 다른 수준의 지능을 보이는 것과 같은 더 많은 표현형 변이를 보이게 할 수 있다.반면, 환경 변화가 감소하면 유전율도 증가하여 개인들이 더 유사한 수준의 지능을 보이는 것과 같은 표현형 변화를 덜 보이게 할 수 있다.유전자가 더 많은 변이에 기여하거나 비유전적인 요인이 덜 변이에 기여하기 때문에 유전성이 증가한다; 중요한 것은 상대적 기여이다.유전성은 특정 환경의 특정 모집단에 고유합니다.그 결과, 높은 유전율은 그 특성이 환경에 영향을 [8]받기 쉽다고는 할 수 없습니다.유전성은 환경 변화, 이주, 근친교배 또는 연구 [9]대상 개체에서 유전성 자체를 측정하는 방법에 따라 변경될 수도 있습니다.유전성은 [10][11]유전적으로 개체에서 유전적으로 결정되는 정도를 나타내는 척도로 해석되어서는 안 된다.
환경에 대한 표현형의 의존도 또한 관련된 유전자의 함수일 수 있다.유전자가 표현형을 관화시켜 모든 환경에서 발현을 거의 피할 수 없게 만들 수 있기 때문에 유전성의 문제는 복잡하다.같은 유전자형을 가진 사람들은 또한 표현형 가소성이라고 불리는 메커니즘을 통해 다른 표현형을 보일 수 있는데, 이것은 경우에 따라서는 유전성을 측정하기 어렵게 만든다.분자생물학의 최근 통찰력은 환경 변화와 관련된 개별 유전자의 전사 활동의 변화를 확인했습니다.그러나 환경에 [12]영향을 받지 않는 유전자가 많다.
유전율 추정치는 통계 분석을 사용하여 개인 간 차이의 원인을 식별하는 데 도움이 됩니다.유전성은 분산과 관련이 있기 때문에, 그것은 필연적으로 모집단 내 개인 간의 차이에 대한 설명입니다.유전성은 단변량 - 단일 특성 검사 - 또는 다변량 검사 - 여러 특성 간의 유전적 및 환경적 연관성을 한 번에 검사하는 것일 수 있습니다.이것은 예를 들어 머리카락 색깔과 눈 색깔과 같은 다른 표현형들 사이의 유전적 중복을 검사할 수 있게 해준다.환경과 유전학도 상호작용할 수 있으며, 유전율 분석은 이러한 상호작용을 테스트하고 검사할 수 있습니다(GxE 모델).
유전성 분석의 전제조건은 고려해야 할 모집단 변동이 있다는 것이다.이 마지막 포인트는 유전율이 모집단에서 불변하는 요인의 영향을 고려할 수 없다는 사실을 강조한다.특정 항생제에 접근할 수 있는 사람이 없거나 모든 사람이 커피를 마시는 것처럼 어디에나 존재하기 때문에 요인들이 부재하고 인구에 존재하지 않는 경우에는 요인들이 불변할 수 있다.실제로, 모든 인간의 행동 특성은 다양하며 거의 모든 특성은 어느 정도 [13]유전성을 보인다.
정의.
특정 표현형은 유전적 영향과 환경적 [14]영향의 합으로 모델링할 수 있다.
- 표현형(P) = 유전자형(G) + 환경(E).
마찬가지로 특성 내 표현형 분산 Var(P)는 다음과 같은 효과의 합계이다.
- Var(P) = Var(G) + Var(E) + 2 Cov(G,E)입니다.
계획된 실험에서는 Cov(G,E)를 0으로 제어하고 유지할 수 있다.이 경우 유전율 {\ H는 다음과 같이 정의됩니다[15].
H는2 광의 유전율이다.이것은 부가적, 지배적, 그리고 서신적 (다유전자적 상호작용)뿐만 아니라 개인이 포유류의 우유 생산과 같이 부모의 표현형에 직접적으로 영향을 받는 모성 및 부성 효과를 포함한 모집단의 표현형 분산에 대한 모든 유전적 기여를 반영한다.
유전자 분산의 특히 중요한 요소는 첨가 분산인 Var(A)이며, 이는 대립 유전자의 평균 효과(가산 효과)에 의한 분산이다.각 부모가 각 자손에게 궤적당 하나의 대립 유전자를 전달하기 때문에, 부모-부모의 유사성은 단일 대립 유전자의 평균 효과에 달려 있습니다.따라서 가법적 분산은 부모-외부 유사성에 책임이 있는 분산의 유전적 구성요소를 나타낸다.표현형 분산의 부가적 유전자 부분은 협감각 유전성으로 알려져 있으며 다음과 같이 정의된다.
대문자 H는2 넓은 의미를 나타내고 소문자2 h는 좁은 의미를 나타낸다.
연속적이지 않지만 추가 발가락이나 특정 질병과 같이 이분법적인 특징에 대해서는 다양한 대립 유전자의 기여가 역치를 지나 특성으로 나타나며 유전성을 추정할 수 있는 책임 역치 모델을 제공할 수 있다.
가법 분산은 선택에 중요합니다.가축의 개량 등 선택적인 압력이 가해지면 그 특성의 반응은 협감 유전성과 직결된다.특성의 평균은 다음 세대에 선택된 부모의 평균이 선택된 부모의 평균과 얼마나 다른지에 대한 함수로 증가할 것이다.선택에 대한 관찰된 반응은 좁은 감각의 유전율(실현된 유전율이라고 함)의 추정으로 이어집니다.이것은 인위적인 선택이나 교배의 기초가 되는 원칙이다.
예
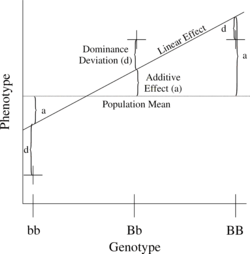
가장 단순한 유전자 모델은 하나의 정량적 표현형에 영향을 미치는 두 개의 대립 유전자(b와 B)를 가진 단일 궤적을 포함한다.
B 대립 유전자의 수는 0, 1, 또는 2가 될 수 있습니다.B와j B가 0 또는 1인 유전자형i(Bi, Bj)의 경우, 기대 표현형은 전체 평균, 선형 효과 및 우성 편차의 합으로 기록될 수 있다(우성 항을 B와j B 사이의i 상호작용으로 생각할 수 있다).
이 궤적에서 첨가 유전자 분산은 첨가 효과의 제곱에 대한 가중 평균이다.
서f ( ) +( ) . \ f ( )_ { } + ( )_ { } + ( )a_ { { displaystyle f ( b )
우세 편차의 분산에 대해서도 유사한 관계가 있습니다.
서f ( ) + ( ) + ( ) f}+f(BB)d_}
유전자형에 대한 표현형의 선형 회귀는 그림 1과 같다.
전제 조건
인간 특성의 총 유전성의 추정치는 "추가성의 가정"이라고 불리는 인식의 부재를 가정한다.일부 연구자들은 알려진 유전자적 위치에 의해 설명되지 않는 "유전성 누락"의 존재를 뒷받침하는 그러한 추정치를 인용했지만, 부가성의 가정은 이러한 추정치를 [16]무효로 할 수 있다.청소년 지능과 학업 [17]성취에 대한 행동 유전 연구에서 가감성 가정이 자주 위반된다는 경험적 증거도 있다.
유전율 추정
P만 직접 관찰하거나 측정할 수 있기 때문에 유전적 또는 환경적 유사성 수준에 따라 다양한 피험자에서 관찰된 유사성으로부터 유전성을 추정해야 한다.분산의 유전적 및 환경적 요소를 추정하는 데 필요한 통계 분석은 표본 특성에 따라 달라진다.간단히 말해서, 더 멀리 떨어져 있는(따라서 덜 유사한) 대상보다 쌍둥이, 형제자매, 부모 및 자손과 같은 매우 다양한 수준의 유전적 관계를 가진 개인으로부터 데이터를 사용하여 더 나은 추정치를 얻을 수 있다.유전성 추정치의 표준 오차는 표본 크기가 클수록 개선됩니다.
비인간 인구에서는 통제된 방법으로 정보를 수집하는 것이 종종 가능하다.예를 들어, 가축들 사이에서 황소가 많은 수의 소로부터 새끼를 낳고 환경을 통제하도록 하는 것은 쉽다.이러한 실험 제어는 일반적으로 자연적으로 발생하는 관계와 환경에 의존하여 인간 데이터를 수집할 때 가능하지 않다.
고전 양적 유전학에서는 유전율의 추정에 관한 두 가지 학파가 있었다.
한 학파는 시카고 대학의 Sewall Wright에 의해 개발되었고, C. C. Li (시카고 대학)와 J. L. L. Lush (아이오와 주립 대학)에 의해 더욱 대중화되었다.이 값은 상관 분석, 나아가 회귀 분석을 기반으로 합니다.경로 분석은 유전율을 추정하는 방법으로 Sewall Wright에 의해 개발되었습니다.
두 번째는 원래 R.A.에 의해 개발되었습니다. 피셔는 에든버러 대학, 아이오와 주립 대학, 노스캐롤라이나 주립 대학 및 기타 학교에서 사업을 확장했습니다.그것은 친척들의 클래스 내 상관관계를 이용하여 번식 연구의 분산 분석을 기반으로 한다.이러한 분석에는 분산 분석의 분산 성분(따라서 유전율)을 추정하는 다양한 방법이 사용됩니다.
오늘날, 유전성은 선형 혼합 모델을 사용한 일반 혈통과 유전자 표지자로부터 추정된 게놈 관련성으로부터 추정할 수 있다.
인간 유전성에 대한 연구는 종종 입양 연구 설계를 활용한다. 종종 일란성 쌍둥이는 어릴 때 헤어지고 다른 환경에서 자랐다.그러한 개인들은 동일한 유전자형을 가지고 있으며 유전자형과 환경의 영향을 분리하는데 사용될 수 있다.이 설계의 한계는 일반적인 태아 환경과 상대적으로 낮은 수의 쌍둥이가 떨어져 자란다는 것이다.두 번째 일반적인 설계는 일란성 쌍둥이와 이란성 쌍둥이의 유사성을 사용하여 유전성을 추정하는 쌍둥이 연구입니다.이 연구들은 일란성 쌍둥이가 유전적으로 완전히 동일하지 않다는 사실에 의해 제한될 수 있고, 잠재적으로 유전성을 과소평가하게 된다.
관찰 연구에서, 또는 유발 효과 때문에, G와 E는 공존할 수 있다: 유전자 환경 상관 관계.유전율을 추정하는 데 사용되는 방법에 따라 유전인자와 공유 또는 비공유 환경 간의 상관관계가 [18]유전성과 혼동될 수도 있고 그렇지 않을 수도 있다.
회귀/상관 추정 방법
첫 번째 추정 학교에서는 회귀와 상관 관계를 사용하여 유전율을 추정합니다.
가까운 친척의 비교
친척들의 비교에서, 우리는 일반적으로
여기서 r은 관련성 계수, b는 회귀 계수, t는 상관 계수라고 생각할 수 있습니다.
부모-오프프링 회귀 분석
유전성은 부모와 자녀 특성을 비교하여 추정할 수 있다(그림 2 참조).선의 기울기(0.57)는 자식 값이 부모의 평균 특성에 대해 회귀되었을 때의 특성 유전성에 근사합니다.부모 값이 하나만 사용되는 경우 유전율은 기울기의 두 배가 됩니다.(자녀 값은 항상 모집단의 평균값으로 회귀하는 경향이 있기 때문에, 즉 기울기가 항상 1보다 작기 때문에 이것이 "회귀"라는 용어의 근원이라는 점에 유의하십시오).이 회귀 효과는 영향을 [19]받는 한 구성원에 대해 선택된 쌍둥이를 분석하기 위한 DeFries-Fulker 방법의 기초가 된다.
형제간 비교
유전성에 대한 기본적인 접근은 완전한 Sib 설계를 사용하여 취할 수 있습니다: 생물학적 어머니와 [20]아버지를 모두 공유하는 형제자매 간의 유사성을 비교하는 것입니다.가법 유전자 작용만 있을 때, 이 형제 표현형 상관관계는 친숙한 지표이다. 즉, 가법 유전자 분산의 절반과 공통 환경의 완전한 효과의 합이다.따라서 전체 Sib 표현형 상관의 두 배인 첨가 유전률에 대한 상한을 설정한다.Half-Sib 설계는 한 부모를 다른 형제 그룹과 공유하는 형제 자매의 표현형 특성을 비교합니다.
쌍둥이 연구
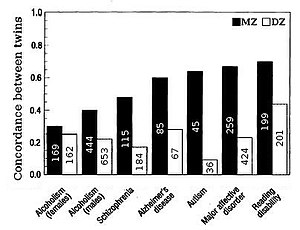
인간의 특징에 대한 유전성은 쌍둥이 사이의 유사성을 비교함으로써 가장 자주 추정됩니다."쌍둥이 연구의 장점은 총 편차를 유전자, 공유 환경 또는 공통 환경 및 고유한 환경 구성요소로 나눌 수 있어 유전성을 정확하게 추정할 수 있다는 것입니다."[21]이란성 또는 DZ 쌍둥이는 평균적으로 유전자의 절반을 공유합니다. 그래서 일란성 또는 일란성 쌍둥이는 평균적으로 DZ 쌍둥이보다 유전적으로 두 배 더 유사합니다.따라서 유전성의 대략적인 추정치는 MZ와 DZ 쌍둥이 간의 상관 관계에서 약 두 배 차이가 난다.매사냥꾼의 공식2 H=2(r(MZ)-r(DZ)).
공유환경의 영향2 c는 형제자매가 자란 환경의 공통성으로 인해 형제자매 간의 유사성에 기여합니다.공유 환경은 DZ 쌍둥이가 같은 유전자2 c=DZ-1/2h를2 공유하는 정도인 DZ 상관에서 반유전율을 뺀 값으로 근사한다.고유한 환경 분산 e는2 함께 양육된 일란성 쌍둥이가 다른 정도인2 e=1-r(MZ)를 반영한다.
분산 추정법 분석
두 번째 유전율 추정 방법 집합에는 분산 분석 및 분산 성분 추정 방법이 포함됩니다.
기본 모델
우리는 켐프손의 [14]기본적인 논의를 이용한다.유전자 모델의 가장 기본적인 것만 고려한다면, 우리는 유전자형i G를 가진 단일 궤적의 양적 기여를 볼 수 있습니다.
서 g ig_})는 유전자형i G의 이고 e e는 환경 영향이다.
랜덤 댐의 시어 그룹과 그 자손을 대상으로 한 실험을 고려해 보십시오.자손들은 유전자의 절반은 아버지로부터, 절반은 어머니로부터 물려받기 때문에, 자손 방정식은 다음과 같습니다.
클래스 내 상관 관계
위의 실험을 고려해 보십시오.비교할 수 있는 두 그룹의 자손들이 있습니다.첫 번째는 각각의 종(종(종(종(種) 그룹 내))에 대한 다양한 자손을 비교하는 것입니다.분산에는 유전적 분산(모두 동일한 유전자형을 얻은 것은 아니기 때문에)과 환경적 분산에 대한 항이 포함됩니다.이는 오류항으로 간주됩니다.
두 번째 자손 집단은 (시어 집단에서) 서로 반자매의 수단을 비교한 것이다.시어 그룹 내와 같은 오차항 외에, 우리는 다른 반자매들의 평균들 간의 차이로 인한 추가 항을 가지고 있다.클래스 내 상관관계는
- (z , ) r (μ + 2 + , + + ) 4 g \ style \ {} ( , z ' ) = \ { \ mu + { \ } {g + \ } )
환경 영향은 서로 독립적이기 때문입니다.
분산 분석
n n 및 r{\ r 자손을 대상으로 한 에서 V {\을(를) 유전자 변이로, e {\를 환경 변이로 하여 다음과 같은 분산 분석을 계산할 수 있습니다.
| 원천 | d.f. | 평균 제곱 | 기대 평균 제곱 |
|---|---|---|---|
| 시어 그룹 간 | |||
| 시어 그룹 내 |
g 은 이복 자매 간의 클래스 내 상관 관계입니다. + e ( - ) + ( - )W{ H} = + }} = {을 쉽게 할 수 있습니다. 기대 평균 제곱은 개체 간의 관계(예: 시어 내의 프로게니스는 모두 반시브)와 클래스 내 상관관계에 대한 이해로부터 계산됩니다.
분산 분석을 사용하여 유전율을 계산하면 직접 [22]효과보다 상호작용 효과에 대한 테스트에 대한 통계적 힘이 훨씬 낮기 때문에 종종 유전자-환경 상호작용의 존재를 고려하지 못한다.
가법 및 우세 항이 있는 모형
가법 항과 우세 항이 있는 모형에서 단일 궤적에 대한 방정식은 다음과 같습니다.
어디에
i_는 ith th 유전자의 첨가 효과, αth j(\displaystyle \alpha _{})는 j 대립 유전자의 우위 편차 e(\displaystyle d_{ij는 환경이다
실험은 표 1에 제시된 것과 유사한 설정으로 실행할 수 있다.서로 다른 관계 그룹을 사용하여 서로 다른 클래스 내 상관 관계를 평가할 수 있습니다.Using as the additive genetic variance and as the dominance deviation variance, intraclass correlations become linear functions of these parameters.일반적으로는
- 클래스 내 상관 a+ d , =
서r\r 및"\는 다음과 같이 표시됩니다.
{\ r=} [관계 쌍에서 무작위로 추출된 대립 유전자가 혈통별로 동일]
\ =} P[ 쌍에서 무작위로 추출된 유전자형은 혈통별로 동일]
몇 가지 일반적인 관계와 그 계수는 표 2에 제시되어 있다.
| 관계 | ||
|---|---|---|
| 일란성 쌍둥이 | ||
| 부모-오프링 | ||
| 이복형제 | ||
| 형제자매 | ||
| 퍼스트 커즌스 | ||
| 더블 퍼스트 커즌스 |
선형 혼합 모형
선형 혼합 모델을 사용하는 다양한 접근법이 문헌에 보고되었다.이러한 방법을 통해 표현형 분산은 유전성을 추정하기 위해 유전자, 환경 및 실험 설계 분산으로 구분된다.표현형 및 환경적 분산에서 유전적 분산의 추론이 특성에 영향을 미치는 전체 범위의 환경적 영향을 포착하는 어려움으로 인해 유전성을 과소평가할 수 있지만, 환경적 분산은 광범위한 환경에 걸쳐 개인을 연구함으로써 명시적으로 모델링될 수 있다.유전율을 계산하기 위한 다른 방법들은 유전인자에 의한 형질에 대한 영향을 추정하기 위해 게놈 전체의 연관성 연구의 데이터를 사용한다. 이것은 유전인자에 대한 추정적으로 연관된 유전자 위치(일반적으로 단핵다형증)의 속도와 영향에 의해 반영된다.그러나 이는 유전성을 과소평가하는 결과를 초래할 수 있다.이러한 차이를 "유전성 누락"이라고 하며, 유전성 [23]모델에서 유전적 및 환경적 분산을 정확하게 모델링해야 하는 과제를 반영한다.
크고 복잡한 혈통이나 앞서 언급한 다른 유형의 데이터를 이용할 수 있는 경우, 유전성 및 기타 정량적 유전자 매개변수는 제한된 최대우도(REML) 또는 베이지안 방법으로 추정할 수 있다.원시 데이터에는 일반적으로 각 개인에 대해 3개 이상의 데이터 포인트(시어 코드, 댐 코드 및 하나 이상의 특성 값)가 있습니다.특성 값이 다르면 특성 또는 측정 시점이 다를 수 있습니다.
현재 널리 사용되는 방법론은 시어와 댐의 정체성에 대한 높은 확실성에 의존한다. 시어 정체성을 확률적으로 처리하는 것은 일반적이지 않다.방법론은 야생 개체군에 거의 적용되지 않으며(여러 야생 유제류 및 조류 개체군에 사용되었지만), 시어는 번식 프로그램에 매우 높은 수준의 확실성으로 항상 알려져 있기 때문에 이것은 보통 문제가 되지 않는다.불확실한 친자 관계를 설명하는 알고리즘도 있다.
혈통은 Pedigree Viewer [1]와 같은 프로그램을 사용하여 볼 수 있으며 ASReml, VCE [2], WOMBAT [3], R 환경 [4] 내의 MCglmm 또는 BLUPF90 제품군 [5]과 같은 프로그램으로 분석할 수 있습니다.
혈통 모델은 역원인 관계, 태아 환경과 같은 모성 영향, 유전적 우위, 공유 환경, 모성 유전자 [24][9]효과의 교란과 같은 교란 문제를 해결하는데 도움이 된다.
게놈 유전율
게놈 전체의 유전자형 데이터와 많은 모집단 샘플의 표현형을 이용할 수 있는 경우, 유전자형을 바탕으로 개체 간의 관계를 추정할 수 있으며 유전자 표지에 의해 설명되는 차이를 선형 혼합 모델을 사용하여 추정할 수 있다.이것은 일반적인 유전자 [4]변종에 의해 포착된 분산을 바탕으로 게놈 유전율 추정치를 제공한다.대립 유전자 빈도와 연결불균형에 대해 서로 다른 조정을 하는 방법은 여러 가지가 있다.특히 HDL(High-Definition Lifility)이라는 방법은 GWAS 요약 [5]통계만을 사용하여 게놈 유전율을 추정할 수 있으므로 다양한 GWAS 메타 분석에서 사용할 수 있는 큰 표본 크기를 통합하는 것이 더 쉽다.
선택에 대한 응답
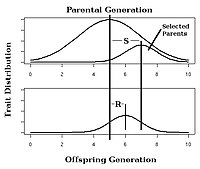
식물과 동물의 선택적 교배에서 좁은 의미의 유전성이알려진 특성 선택에 대한 예상 반응은 사육자의 [25]방정식을 사용하여 추정할 수 있다. (2){style ( }
이 방정식에서는 선택에 대한 응답(R)은 부모 세대와 다음 세대의 실현된 평균 차이로 정의되며, 선택 차분(S)은 부모 세대와 선택된 [14]: 1957 [26]부모 세대 간의 평균 차이로 정의됩니다.
예를 들어, 식물 증식자가 옥수수 이삭당 알맹이 수를 늘리기 위한 선택적 번식 프로젝트에 관여하고 있다고 가정해 보십시오.논거를 위해, 부모 세대의 옥수수 이삭의 평균이 100개의 알맹이를 가지고 있다고 가정해 보자.또한 선택된 부모가 귀당 평균 120개의 알맹이를 가진 옥수수를 생산한다고 가정해 보자.h가2 0.5이면 다음 세대는 이삭당 평균 0.5(120-100) = 10개의 추가 알맹이를 가진 옥수수를 생산하게 됩니다.따라서 옥수수 이삭당 총 알맹이 수는 평균 110개입니다.
인공 선택 실험에서 선택에 대한 반응을 관찰하면 그림 4와 같이 실현된 유전율을 계산할 수 있다.
위 방정식의 유전율은 유전자형과 환경 노이즈가 가우스 분포를 따르는 경우에만 V (A) / () \ / \ 과 같습니다.
논쟁
이 섹션은 특정 시점을 기준으로 균형이 맞지 않을 수 있습니다.(2016년 8월) |
Steven [27]Rose,[28] Jay Joseph 및 Richard Bentall과 같은 유전성 추정치는 주로 행동과학 및 사회과학에서의 유전성 추정치에 초점을 맞추고 있습니다.Bentall은 그러한 상속 가능성 점수 일반적으로 counterintuitively 숫자로 높은 점수를 도출하기 위해 계산된다 주장했다고 그 유전적 유전적 결정으로 아동 학대 후 정신병되는 등 연구 더 원인이 되어 중요한 알았다. 다른 요인들이 이 편향 혼란이 잘못 해석된다.[29][30]유전율 추정치는 유전자나 환경이 연구 대상 형질의 발달에 더 큰 역할을 하는지 여부에 대한 정보를 제공하지 않기 때문에 본질적으로 제한적이다.이러한 이유로, 데이비드 무어와 데이비드 셴크는 행동 유전학의 맥락에서 "유전성"이라는 용어를 "과학 역사상 가장 오해의 소지가 있는 것 중 하나"라고 묘사하고 매우 드문 [31]경우를 제외하고는 가치가 없다고 주장한다.복잡한 인간의 특성을 연구할 때, 유전자와 환경의 상대적 기여도를 결정하기 위해 유전율 분석을 사용하는 것은 불가능하다. 이러한 특성은 상호작용하는 [32]여러 원인에서 비롯되기 때문이다.특히, Feldman과 Lewontin은 유전성 자체가 환경 [33]변화의 함수라고 강조한다.그러나 일부 연구자들은 이 둘을 [34]분리하는 것이 가능하다고 주장한다.
유전율 추정치에 대한 논란은 주로 두 가지 연구에서 근거를 두고 있다.그러한 집단-유전학 연구의 결론을 확증하기 위한 분자-유전학 연구가 거의 성공하지 못한 것은 누락된 유전성 문제이다.[35]에릭 터크하이머는 새로운 분자 방법이 유전자와 [36]행동 사이의 관계를 설명하는 방법은 대부분 불분명하지만 쌍둥이 [35]연구의 전통적인 해석을 정당화했다고 주장했습니다.Turkheimer에 따르면 유전자와 환경 모두 유전되고, 유전적인 기여는 환경에 따라 다르며, 유전성에 대한 집중은 다른 중요한 [37]요소들로부터 주의를 분산시킨다.그러나 전반적으로 유전성은 널리 적용되는 [9]개념이다.
「 」를 참조해 주세요.
레퍼런스
- ^ Wray N, Visscher P (2008). "Estimating Trait Heritability". Nature Education. 1 (1): 29. Archived from the original on 2 August 2015. Retrieved 24 July 2015.
- ^ Gazzaniga MS, Heatherton TF, Halpern DF (February 2015). Psychological science (5th ed.). New York. ISBN 978-0-393-26313-8. OCLC 908409996.
- ^ Yang J, Lee SH, Goddard ME, Visscher PM (January 2011). "GCTA: a tool for genome-wide complex trait analysis". American Journal of Human Genetics. 88 (1): 76–82. doi:10.1016/j.ajhg.2010.11.011. PMC 3014363. PMID 21167468.
- ^ a b Yang J, Zeng J, Goddard ME, Wray NR, Visscher PM (August 2017). "Concepts, estimation and interpretation of SNP-based heritability" (PDF). Nature Genetics. 49 (9): 1304–1310. doi:10.1038/ng.3941. PMID 28854176. S2CID 8790524. Archived (PDF) from the original on 2020-10-05. Retrieved 2020-09-06.
- ^ a b Ning Z, Pawitan Y, Shen X (June 2020). "High-definition likelihood inference of genetic correlations across human complex traits" (PDF). Nature Genetics. 52 (8): 859–864. doi:10.1038/s41588-020-0653-y. hdl:10616/47311. PMID 32601477. S2CID 220260262. Archived (PDF) from the original on 2021-04-15. Retrieved 2021-02-08.
- ^ Stoltenberg SF (June 1997). "Coming to terms with heritability". Genetica. 99 (2–3): 89–96. doi:10.1007/BF02259512. hdl:2027.42/42804. PMID 9463077. S2CID 18212219. Archived from the original on 2020-12-02. Retrieved 2020-12-24.
- ^ Wahlsten D (1994). "The intelligence of heritability" (PDF). Canadian Psychology. 35 (3): 244–260. doi:10.1037/0708-5591.35.3.244. ISSN 1878-7304. Archived (PDF) from the original on 2018-10-24. Retrieved 2019-12-05.
- ^ Maccoby EE (February 2000). "Parenting and its effects on children: on reading and misreading behavior genetics". Annual Review of Psychology. 51 (1): 1–27. doi:10.1146/annurev.psych.51.1.1. PMID 10751963. S2CID 43435035.
- ^ a b c Visscher PM, Hill WG, Wray NR (April 2008). "Heritability in the genomics era--concepts and misconceptions" (PDF). Nature Reviews. Genetics. 9 (4): 255–66. doi:10.1038/nrg2322. PMID 18319743. S2CID 690431. Archived (PDF) from the original on 2016-03-24. Retrieved 2015-08-28.
- ^ Sauce B, Matzel LD (January 2018). "The paradox of intelligence: Heritability and malleability coexist in hidden gene-environment interplay". Psychological Bulletin. 144 (1): 26–47. doi:10.1037/bul0000131. PMC 5754247. PMID 29083200.
- ^ Block N (August 1995). "How heritability misleads about race". Cognition. 56 (2): 99–128. doi:10.1016/0010-0277(95)00678-r. PMID 7554794. S2CID 204981536.
- ^ Wills C (2007). "Principles of Population Genetics, 4th edition" (PDF). Journal of Heredity (Book Review). 98 (4): 382. doi:10.1093/jhered/esm035. Archived (PDF) from the original on 2020-10-05. Retrieved 2018-04-20.
- 리뷰 대상:
- ^ Turkheimer E (October 2000). "Three Laws of Behavior Genetics and What They Mean" (PDF). Current Directions in Psychological Science. 9 (5): 160–164. doi:10.1111/1467-8721.00084. ISSN 0963-7214. S2CID 2861437. Archived (PDF) from the original on 19 October 2013. Retrieved 29 October 2013.
- ^ a b c Kempthorne O (1957). An introduction to genetic statistics (1st ed.). Ames, Iowa: Iowa State Univ. Press. OCLC 422371269.
- ^ Stephen Downes and Lucas Matthews. "Heritability". Stanford Encyclopedia of Philosophy. Stanford University. Archived from the original on 2020-02-25. Retrieved 2020-02-20.
- ^ Zuk O, Hechter E, Sunyaev SR, Lander ES (January 2012). "The mystery of missing heritability: Genetic interactions create phantom heritability". Proceedings of the National Academy of Sciences of the United States of America. 109 (4): 1193–8. Bibcode:2012PNAS..109.1193Z. doi:10.1073/pnas.1119675109. PMC 3268279. PMID 22223662.
- ^ Daw J, Guo G, Harris KM (July 2015). "Nurture net of nature: Re-evaluating the role of shared environments in academic achievement and verbal intelligence". Social Science Research. 52: 422–39. doi:10.1016/j.ssresearch.2015.02.011. PMC 4888873. PMID 26004471.
- ^ Cattell RB (November 1960). "The multiple abstract variance analysis equations and solutions: for nature-nurture research on continuous variables". Psychological Review. 67 (6): 353–72. doi:10.1037/h0043487. PMID 13691636.
- ^ DeFries JC, Fulker DW (September 1985). "Multiple regression analysis of twin data". Behavior Genetics. 15 (5): 467–73. doi:10.1007/BF01066239. PMID 4074272. S2CID 1172312.
- ^ Falconer DS, Mackay TF (December 1995). Introduction to Quantitative Genetics (4th ed.). Longman. ISBN 978-0582243026.
- ^ Gielen M, Lindsey PJ, Derom C, Smeets HJ, Souren NY, Paulussen AD, Derom R, Nijhuis JG (January 2008). "Modeling genetic and environmental factors to increase heritability and ease the identification of candidate genes for birth weight: a twin study". Behavior Genetics. 38 (1): 44–54. doi:10.1007/s10519-007-9170-3. PMC 2226023. PMID 18157630.
- ^ Wahlsten, Douglas (March 1990). "Insensitivity of the analysis of variance to heredity-environment interaction" (PDF). Behavioral and Brain Sciences. 13 (1): 109–120. doi:10.1017/S0140525X00077797. ISSN 1469-1825. S2CID 143217984. Archived (PDF) from the original on 2020-10-05. Retrieved 2020-09-06.
- ^ Heckerman D, Gurdasani D, Kadie C, Pomilla C, Carstensen T, Martin H, Ekoru K, Nsubuga RN, Ssenyomo G, Kamali A, Kaleebu P, Widmer C, Sandhu MS (July 2016). "Linear mixed model for heritability estimation that explicitly addresses environmental variation". Proceedings of the National Academy of Sciences of the United States of America. 113 (27): 7377–82. Bibcode:2016PNAS..113.7377H. doi:10.1073/pnas.1510497113. PMC 4941438. PMID 27382152.
- ^ Hill WG, Goddard ME, Visscher PM (February 2008). MacKay TF, Goddard ME (eds.). "Data and theory point to mainly additive genetic variance for complex traits". PLOS Genetics. 4 (2): e1000008. doi:10.1371/journal.pgen.1000008. PMC 2265475. PMID 18454194.

- ^ Plomin R, DeFries JC, McClearn GE, McGuffin P (2017). Behavioral Genetics: A Primer (2nd ed.). New York: W.H. Freeman. ISBN 978-0-7167-2056-0.
- ^ Falconer DS, Mackay TF (1998). Introduction to quantitative genetics (4th ed.). Essex: Longman. ISBN 978-0-582-24302-6.
- ^ Rose SP (June 2006). "Commentary: heritability estimates--long past their sell-by date". International Journal of Epidemiology. 35 (3): 525–7. doi:10.1093/ije/dyl064. PMID 16645027.
- ^ Joseph J (2004). "Chapter 5". The Gene Illusion. New York: Algora. p. 141. ISBN 978-1-898059-47-9. Archived from the original on 2017-07-19. Retrieved 2016-04-02.
- ^ Bentall RP (2009). Doctoring the Mind: Is Our Current Treatment of Mental Illness Really Any Good?. New York: New York University Press. pp. 123–127. ISBN 978-0-8147-8723-6. Archived from the original on 2020-10-05. Retrieved 2016-04-02.
- ^ McGrath M (5 July 2009). "Doctoring the Mind: Review". The Telegraph. Archived from the original on 28 September 2011. Retrieved 4 April 2018.
- ^ Moore DS, Shenk D (January 2017). "The heritability fallacy". Wiley Interdisciplinary Reviews: Cognitive Science. 8 (1–2): e1400. doi:10.1002/wcs.1400. PMID 27906501.
- ^ Feldman MW, Ramachandran S (April 2018). "Missing compared to what? Revisiting heritability, genes and culture". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 373 (1743): 20170064. doi:10.1098/rstb.2017.0064. PMC 5812976. PMID 29440529.
...all complex human traits result from a combination of causes. If these causes interact, it is impossible to assign quantitative values to the fraction of a trait due to each, just as we cannot say how much of the area of a rectangle is due, separately, to each of its two dimensions. Thus, in the analyses of complex human phenotypes...we cannot actually find ‘the relative importance of genes and environment in the determination of phenotype’.
- ^ Marcus W. Feldman; Richard C. Lewontin (1975). "The Heritability Hang–Up". Science. 190 (4220): 1163–1168. Bibcode:1975Sci...190.1163F. doi:10.1126/science.1198102. PMID 1198102. S2CID 6797128. Archived from the original on 20 May 2021. Retrieved 20 May 2021.
- ^ 트레두, 가반 '사각형의 본질과 육성' (2019).
- ^ a b Turkheimer E (2011). "Still missing". Research in Human Development. 8 (3–4): 227–241. doi:10.1080/15427609.2011.625321. S2CID 14737438.
- ^ Turkheimer E (2015). "Genetic Prediction". The Hastings Center Report. 45 (5 Suppl): S32–8. doi:10.1002/hast.496. PMID 26413946.
- ^ Joseph J (2014). The Trouble with Twin Studies: A Reassessment of Twin Research in the Social and Behavioral Sciences (PDF). New York: Routledge. p. 81. ISBN 978-1-317-60590-4. Archived (PDF) from the original on 2016-04-04. Retrieved 2016-04-02.
추가 정보
- Lynch M, Walsh B (1998). Genetics and analysis of quantitative traits. Sunderland, Mass.: Sinauer Assoc. ISBN 978-0-87893-481-2.
- Johnson W, Penke L, Spinath FM (2011). "Understanding Heritability: What it is and What it is Not". European Journal of Personality. 25 (4): 287–294. doi:10.1002/per.835. ISSN 0890-2070. S2CID 41842465.

 다음과 같이 정의됩니다
다음과 같이 정의됩니다








 환경 영향이다.
환경 영향이다. 


 (를) 유전자 변이로,
(를) 유전자 변이로,  환경 변이로
환경 변이로 













 다음과 같이 표시됩니다.
다음과 같이 표시됩니다.









 같습니다.
같습니다. 

