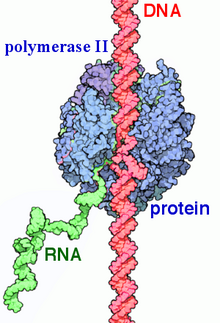
RNA중합효소II
RNA polymerase IIRNA 중합효소 II(RNAP II 및 Pol II)는 DNA를 메신저 RNA(mRNA)와 대부분의 작은 핵 RNA(snRNA) 및 마이크로RNA의 [1][2]전구체로 전사하는 다단백질 복합체이다.그것은 진핵세포의 [3]핵에서 발견되는 세 가지 RNAP 효소 중 하나이다.12개의 서브유닛으로 이루어진 550kDa 복합체인 RNA 중합효소 중 가장 많이 연구된 유형이다.업스트림 유전자 촉진제에 결합하고 전사를 시작하기 위해서는 광범위한 전사 인자가 필요하다.
검출
초기 연구들은 최소 두 개의 RNA를 제안했다. 하나는 핵에서 rRNA를 합성한 것이고, 다른 하나는 핵의 일부인 핵플라스마의 [5]다른 RNA를 합성한 것이다.1969년, 과학 실험가 로버트 로더와 윌리엄 러터는 핵플라스마에서 RNA의 전사를 담당하는 추가적인 RNA를 확실히 발견했다.이 연구결과는 DEAE 코팅 Sephadex 비즈를 통해 이온 교환 크로마토그래피를 사용하여 얻어졌습니다.이 기술은 황산암모늄의 농도를 증가시켜 해당 용출량인 ι, ι, , 순으로 효소를 분리하였다.효소는 용출, RNAPI, RNAPII, RNAPII의 [3]순서로 명명되었다.이 발견은 RNAP II와 RNAP [6]III 사이의 분화를 가능하게 하는 추가적인 효소가 핵플라스마에 존재한다는 것을 증명했다.
RNA 중합효소 II(RNAP2)는 초기 신장 중에 조절된 전사 일시정지 과정을 거친다.다양한 연구에서 전사 연장의 교란은 암, 신경 퇴화, HIV 잠복기 [7]등과 관련이 있는 것으로 나타났다.
서브유닛

진핵핵핵 RNA 중합효소 II는 전사 [9]분석을 사용하여 최초로 정제되었다.정제 효소는 전형적으로 10~12개의 소단위(인간과 효모에서 12개)를 가지며 특정 프로모터를 [10]인식할 수 없다.많은 서브유닛-서브유닛 상호작용이 [11]알려져 있다.
- DNA 지향 RNA 중합효소 II 서브유닛 RPB1 – 사람에서 POLR2A 유전자에 의해 코드되고 효모에서 RPO21에 의해 코드되는 효소. RPB1은 RNA 중합효소 II의 가장 큰 서브유닛이다.중합효소 [12]활성에 필수적인 최대 52개의 헵타펩타이드 반복(YSPTSPS)으로 구성된 카르복시 말단 도메인(CTD)을 포함한다.CTD는 토론토 대학의 C.J. 잉글스와 존스 홉킨스 대학의 JL 코든의 연구실에서 처음 발견되었다.RPB1 서브유닛은 다른 몇 가지 중합효소 서브유닛과 조합하여 중합효소의 DNA 결합 도메인을 형성하며, 이는 DNA 템플릿이 [13]RNA로 전사되는 홈이다.RPB8과 [11]강하게 상호 작용합니다.
- RPB2(POLR2B) – 최소 2개의 다른 중합효소 서브유닛과 결합하여 DNA 템플릿과 새로 합성된 RNA 사이의 효소 활성 [14]부위에서 접촉을 유지하는 중합효소 내 구조를 형성하는 두 번째로 큰 서브유닛이다.
- RPB3(POLR2C)– 세 번째로 큰 서브유닛코어 서브어셈블리를 형성하는 또 다른 중합효소 서브유닛인 POLR2J와 함께 헤테로다이머로 존재한다.RPB3는 RPB1-5, 7, 10-12와 [11]강하게 상호 작용합니다.
- RNA 중합효소 II 서브유닛 B4(RPB4) – POLR2D[15] 유전자에 의해 인코딩된 서브유닛은 네 번째로 크며 스트레스 보호 역할을 할 수 있다.
- RPB5 – 사람 내에서는 POLR2E 유전자에 의해 부호화된다.각 RNA 중합효소 [16]II에는 2개의 분자가 존재하며,[11] RPB5는 RPB1, RPB3, RPB6와 강하게 상호작용한다.
- RPB6(POLR2F) – DNA [17]템플릿에서 전사 중합효소를 안정화시키는 최소 2개의 서브유닛과 구조를 형성합니다.
- RPB7 – POLR2G에 의해 인코딩되며 중합효소 [18]기능을 조절하는 역할을 할 수 있습니다.RPB7은 RPB1 및 RPB5와 강력하게 [11]상호 작용합니다.
- RPB8(POLR2H): 서브유닛 RPB1-3, 5, 및7과 [11]상호작용합니다.
- RPB9 – DNA 템플릿이 RNA로 전사되는 홈은 RPB9(POLR2I)와 RPB1로 구성됩니다.
- RPB10 – 유전자 POLR2L의 산물.RPB1-3 및 5와 대화하며 RPB3와 [11]강력하게 대화합니다.
- RPB11 – RPB11 서브유닛 자체는 인간의 경우 POLR2J(RPB11-a), POLR2J2(RPB11-b) 및 POLR2J3[19](RPB11-c)의 3개의 서브유닛으로 구성됩니다.
- RPB12 – RPB3는 RPB12(POLR2K)[11]와도 대화합니다.
어셈블리
RPB3는 RNA 중합효소 II [20]어셈블리에 관여한다.서브유닛 [20]합성 직후에 RPB2와 RPB3의 서브콤플렉스가 나타난다.이 복합체는 그 후 RPB1과 [20]대화합니다.RPB3, RPB5, 및 RPB7은 서로 대화하여 호모디머를 형성하고 RPB3 및 RPB5는 함께 RPB9를 [11]제외한 다른 모든 RPB 서브유닛에 접속할 수 있습니다.RPB1만이 RPB5에 [11]강하게 바인드 됩니다.또, RPB1 서브 유닛은, RPB7, RPB10, 및 보다 약하지만,[11] RPB8에 가장 효율적으로 접속합니다.RPB1이 컴플렉스에 들어가면 RPB5나 RPB7 등의 다른 서브유닛이 들어갈 수 있습니다.여기서 RPB5는 RPB6 및 RPB8에 바인드되어 RPB3은 RPB10, RPB11, RPB12.[11] RPB4 및 RPB9가 조립됩니다.RPB4는 RPB7과 [11]콤플렉스를 형성합니다.
동력학
효소는 초당 수 백만 개의 반응을 촉매할 수 있다.효소율은 용액 상태와 기질 농도에 따라 달라집니다.다른 효소들과 마찬가지로 POLR2는 포화 곡선과 최대 속도(Vmax)를 가지고 있다.K(반쪽max V에 필요한 기질 농도)와m k(초당 활성 부위 1개가 처리하는 기질 분자 수)를cat 가진다.특이성 상수는 k/K로catm 지정됩니다.특이성 상수에 대한 이론적 최대값은 약 108~109(Ms−1−1)의 확산 한계이며, 효소와 기질이 충돌할 때마다 촉매작용이 발생한다.효모에서 가장 큰 서브유닛의 트리거-루프 도메인에서의 돌연변이는 효소의 [21]역학을 변화시킬 수 있다.
RNA 중합효소 II의 친척인 박테리아 RNA 중합효소는 [22]DNA를 따라 앞뒤로 이동함으로써 비활성 상태와 활성 상태를 전환합니다.[NTP]eq = 10 μM GTP, 10 μM UTP, 5 μM ATP, 2.5 μM CTP의 농도는 RNA 중합효소 [22]II의 상대인 세균 RNA의 평균 신장률, 회전수(NTP)−1를 생성한다.
RNA 중합효소 II는 전사 [23][24]신장 중에 광범위한 동시 전사 일시정지 과정을 거친다.이러한 일시정지는 특히 뉴클레오솜에서 두드러지며, 부분적으로 전사적으로 무능력한 역추적 [23]상태로 들어가는 중합효소를 통해 발생합니다.이러한 일시정지 시간은 초에서 분 또는 그 이상이며, TFIIS와 같은 신장 [25]인자에 의해 긴 일시정지 종료가 촉진될 수 있습니다.이어서 전사속도는 전사된 뉴클레오솜의 히스톤이 염색질에서 제거되는지 또는 전사중합효소 [26]뒤에 재삽입되는지 여부에 영향을 준다.
알파아마니틴
RNA중합효소 II는 α-아마니틴[27] 및 기타 아마톡신에 의해 억제된다.α-아마니틴은 많은 [5]버섯에서 발견되는 매우 독성이 강한 물질이다.버섯 독은 각각의 RNA 중합효소인 I, II, III에 다른 영향을 미칩니다. RNAPI는 물질에 전혀 반응하지 않고 정상적으로 기능하지만 RNAPIII는 중간 정도의 민감성을 가집니다.그러나 RNAP II는 독소에 의해 완전히 억제된다.알파-아마니틴은 RPB-1 서브유닛의 [28]핵심 "브릿지 알파-나선" 영역과 효소의 "퍼널", "클리프트" 영역에서의 강한 상호작용에 의해 RNAP II를 억제한다.
홀로엔자임
RNA 중합효소 II 홀로엔자임은 살아있는 [10]세포의 단백질 코드화 유전자의 촉진제에게 모집되는 진핵 RNA 중합효소 II의 한 형태이다.그것은 일반적인 전사 인자의 하위 집합인 RNA 중합효소 II와 SRB 단백질로 알려진 조절 단백질로 구성됩니다.
홀로엔자임 조립체의 일부는 전사가 시작되기 전에 유전자 프로모터에서 이루어지기 때문에 사전 개시 복합체라고 불린다.매개체 복합체는 RNA 중합효소 II와 전사 인자 사이의 가교 역할을 합니다.
염색질 구조에 의한 제어
이것은 크로마틴 구조와 히스톤 번역 후 변형이 RNA 중합효소 II에 의한 유전자 전사를 조절하고 기록하는 효모세포의 예시 메커니즘의 개요이다.
이 경로는 다음과 같은 전사 지점에서 조절의 예를 제시합니다.
- 사전 개시(Bre1, 히스톤 수정에 의한 촉진)
- 개시(TFIIH에 의한 프로모션, Pol II 수정 및 COMPASS에 의한 프로모션, 히스톤 수정)
- 신장(세트2, 히스톤 수정에 의한 촉진)
이는 프로세스의 다양한 단계를 규제 단계라고 합니다.규제에 사용되는지는 증명되지 않았지만, 매우 가능성이 높습니다.
RNA Pol II 신장촉진제는 3가지 등급으로 요약할 수 있다.
- 약물/배열 의존성 정지 영향인자(다양한 간섭단백질)
- 크로마틴 구조 지향 인자(히스톤 전사 후 수식제, 예를 들어 히스톤 메틸전달효소)
- RNA Pol II 촉매 개선 인자(다양한 간섭 단백질 및 Pol II 보조 인자, RNA 중합효소 II 참조).
전사 메커니즘
- 염색질 구조 지향 요인:
(HMT(Histone MethylTransferase):
COMPlex – (Set1과 관련된 단백질의 COMPlex)– 히스톤 H3의 메틸레이트 리신 4: 전사 억제/소멸을 담당합니다.RNAP II [29]내 세포 성장과 전사 조절의 정상적인 부분. - Set2 – 히스톤 H3의 메틸레이트 리신 36: Set2는 CTD와의 [30]직접 접촉을 통해 조절 전사 연장에 관여한다.
(재미없는 예: Dot1*" – 히스톤 H3의 리신 79를 메틸화함) - Bre1 – 히스톤 H2B의 유비쿼터스(유비퀴틴을 첨가) 리신 123.사전 시작 및 RNA Pol II 결합 허용과 관련이 있습니다.
RNA중합효소CTD
RPB1의 C 터미널이 추가되어 C 터미널 도메인(CTD)이 형성됩니다.RNA 중합효소 II의 카르복시 말단 도메인은 일반적으로 Tyr-Ser-Pro-Thr-Ser-Pro-Ser 배열의 [31]최대 52회 반복으로 구성됩니다.도메인은 RNAPII 효소의 핵심에서 출구 채널까지 확장되며, 이러한 위치는 "RNA 처리 반응, RNA 처리 [32]기계의 구성 요소와의 직접적 또는 간접적 상호작용을 통한" 인덕션으로 인해 효과적이다.CTD 도메인은 RNA 중합효소 I 또는 RNA 중합효소 [3]III에 존재하지 않습니다.RNA 중합효소 CTD는 C.J.의 연구실에서 처음 발견되었다.토론토 대학과 존스 홉킨스 대학의 J 코든 연구실에서 효모와 마우스로부터 각각 RNA 중합효소의 RPB1 서브유닛을 코드하는 DNA의 염기서열 분석 과정 중 잉글스.다른 단백질들은 종종 중합효소 활성을 활성화하기 위해 RNA 중합효소의 C 말단 도메인을 결합한다.이것은 전사의 시작, RNA 전사체의 캡핑 및 RNA 스플라이싱을 [12]위한 스플라이세오솜에 대한 부착에 관여하는 단백질 도메인이다.
CTD 도메인의 인산화
RNA 중합효소 II는 [5][3]비인산화 및 인산화, IIO의 두 가지 형태로 존재한다.두 형태 사이의 전환은 전사를 위한 다른 기능을 용이하게 합니다.CTD의 인산화는 6가지 일반적인 전사 인자 중 하나인 TFIIH에 의해 촉매된다. TFIIH는 두 가지 목적을 제공한다. 하나는 전사 시작 부위에서 DNA를 풀어내는 것이고 다른 하나는 인산화이다.중합효소 IIA는 프리이니티시네이션 복합체에 결합하는데, 이는 IIA가 중합효소 IIO 형태보다 일반 전사인자 TFIID의 서브유닛인 TBP(TATATA-box binding protein)에 높은 친화력으로 결합하기 때문이다.중합효소 IIO 형태는 RNA [5]사슬의 신장을 촉진합니다.신장 개시 방법은 TFIIH를 통해 위치 5(Ser5)에서 세린의 인산화로 이루어진다.새롭게 인산화 된 Ser5는 새롭게 합성된 RNA의 5' 말단과 "폴리(A) [32]부위에 대한 3' 처리 인자를 차단하기 위한 효소를 모집한다.두 번째 세린이 인산화되면 Ser2가 활성화된다.신장을 종료하기 위해서는 탈인화가 일어나야 한다.일단 도메인이 완전히 탈인화되면, RNAP II 효소는 "재순환"되고 다른 시작 [32]부위와 동일한 과정을 촉매한다.
전사결합재조합수리
산화적 DNA 손상은 RNA 중합효소 II의 전사를 차단하고 가닥을 끊을 수 있습니다.DNA [33]손상으로부터 보호할 수 있는 RNA 템플릿 전사 관련 재조합 과정이 설명되었습니다.세포주기의 G1/G0단계에서 세포는 능동적으로 전사된 영역 내의 이중사슬단절에서 상동재조합인자의 집합을 나타낸다.전사는 RNA 템플릿화된 상동 재조합에 의한 DNA 이중 가닥 파괴의 복구와 결합되는 것으로 보인다.이 복구 과정은 RNA 중합효소 II에 의해 활발하게 전사되는 유전자의 이중 가닥 절단을 효율적이고 정확하게 다시 결합합니다.
「 」를 참조해 주세요.
레퍼런스
- ^ Kornberg RD (December 1999). "Eukaryotic transcriptional control". Trends in Cell Biology. 9 (12): M46–9. doi:10.1016/S0962-8924(99)01679-7. PMID 10611681.
- ^ Sims RJ, Mandal SS, Reinberg D (June 2004). "Recent highlights of RNA-polymerase-II-mediated transcription". Current Opinion in Cell Biology. 16 (3): 263–71. doi:10.1016/j.ceb.2004.04.004. PMID 15145350.
- ^ a b c d Young, Richard A. (2003-11-28). "RNA Polymerase II". Annual Review of Biochemistry. 60 (1): 689–715. doi:10.1146/annurev.bi.60.070191.003353. PMID 1883205.
- ^ Meyer PA, Ye P, Zhang M, Suh MH, Fu J (June 2006). "Phasing RNA polymerase II using intrinsically bound Zn atoms: an updated structural model". Structure. 14 (6): 973–82. doi:10.1016/j.str.2006.04.003. PMID 16765890.
- ^ a b c d Weaver, Robert Franklin (2012-01-01). Molecular biology. McGraw-Hill. ISBN 9780073525327. OCLC 789601172.
- ^ Roeder RG, Rutter WJ (Oct 1969). "Multiple forms of DNA-dependent RNA polymerase in eukaryotic organisms". Nature. 224 (5216): 234–7. Bibcode:1969Natur.224..234R. doi:10.1038/224234a0. PMID 5344598. S2CID 4283528.
- ^ Cermakova, Katerina; Demeulemeester, Jonas; Lux, Vanda; Nedomova, Monika; Goldman, Seth R.; Smith, Eric A.; Srb, Pavel; Hexnerova, Rozalie; Fabry, Milan; Madlikova, Marcela; Horejsi, Magdalena (2021-11-26). "A ubiquitous disordered protein interaction module orchestrates transcription elongation". Science. 374 (6571): 1113–1121. Bibcode:2021Sci...374.1113C. doi:10.1126/science.abe2913. PMC 8943916. PMID 34822292. S2CID 244660781.
- ^ Armache, Karim-Jean; Mitterweger, Simone; Meinhart, Anton; Cramer, Patrick (2019). "Structures of complete RNA polymerase II and its subcomplex, Rpb4/7" (PDF). Journal of Biological Chemistry. 280 (8): 7131–1734. doi:10.2210/pdb1wcm/pdb. PMID 15591044.
- ^ Sawadogo M, Sentenac A (1990). "RNA polymerase B (II) and general transcription factors". Annual Review of Biochemistry. 59: 711–54. doi:10.1146/annurev.bi.59.070190.003431. PMID 2197989.
- ^ a b Myer VE, Young RA (October 1998). "RNA polymerase II holoenzymes and subcomplexes". The Journal of Biological Chemistry. 273 (43): 27757–60. doi:10.1074/jbc.273.43.27757. PMID 9774381.
- ^ a b c d e f g h i j k l m Acker J, de Graaff M, Cheynel I, Khazak V, Kedinger C, Vigneron M (July 1997). "Interactions between the human RNA polymerase II subunits". The Journal of Biological Chemistry. 272 (27): 16815–21. doi:10.1074/jbc.272.27.16815. PMID 9201987.
- ^ a b Brickey WJ, Greenleaf AL (June 1995). "Functional studies of the carboxy-terminal repeat domain of Drosophila RNA polymerase II in vivo". Genetics. 140 (2): 599–613. doi:10.1093/genetics/140.2.599. PMC 1206638. PMID 7498740.
- ^ "Entrez Gene: POLR2A polymerase (RNA) II (DNA directed) polypeptide A, 220kDa".
- ^ "Entrez Gene: POLR2B polymerase (RNA) II (DNA directed) polypeptide B, 140kDa".
- ^ Khazak V, Estojak J, Cho H, Majors J, Sonoda G, Testa JR, Golemis EA (April 1998). "Analysis of the interaction of the novel RNA polymerase II (pol II) subunit hsRPB4 with its partner hsRPB7 and with pol II". Molecular and Cellular Biology. 18 (4): 1935–45. doi:10.1128/mcb.18.4.1935. PMC 121423. PMID 9528765.
- ^ "Entrez Gene: POLR2E polymerase (RNA) II (DNA directed) polypeptide E, 25kDa".
- ^ "Entrez Gene: POLR2F polymerase (RNA) II (DNA directed) polypeptide F".
- ^ "Entrez Gene: POLR2G polymerase (RNA) II (DNA directed) polypeptide G".
- ^ "POLR2J3 polymerase (RNA) II (DNA directed) polypeptide J3".
- ^ a b c Kolodziej PA, Young RA (September 1991). "Mutations in the three largest subunits of yeast RNA polymerase II that affect enzyme assembly". Molecular and Cellular Biology. 11 (9): 4669–78. doi:10.1128/mcb.11.9.4669. PMC 361357. PMID 1715023.
- ^ Kaplan CD, Jin H, Zhang IL, Belyanin A (April 12, 2012). "Dissection of Pol II trigger loop function and Pol II activity-dependent control of start site selection in vivo". PLOS Genetics. 8 (4): e1002627. doi:10.1371/journal.pgen.1002627. PMC 3325174. PMID 22511879.
- ^ a b Abbondanzieri EA, Greenleaf WJ, Shaevitz JW, Landick R, Block SM (November 2005). "Direct observation of base-pair stepping by RNA polymerase". Nature. 438 (7067): 460–5. Bibcode:2005Natur.438..460A. doi:10.1038/nature04268. PMC 1356566. PMID 16284617.
- ^ a b Hodges, Courtney; Bintu, Lacramioara; Lubkowska, Lucyna; Kashlev, Mikhail; Bustamante, Carlos (2009-07-31). "Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II". Science. 325 (5940): 626–628. Bibcode:2009Sci...325..626H. doi:10.1126/science.1172926. ISSN 1095-9203. PMC 2775800. PMID 19644123.
- ^ Churchman, L. Stirling; Weissman, Jonathan S. (2011-01-20). "Nascent transcript sequencing visualizes transcription at nucleotide resolution". Nature. 469 (7330): 368–373. Bibcode:2011Natur.469..368C. doi:10.1038/nature09652. ISSN 1476-4687. PMC 3880149. PMID 21248844.
- ^ Galburt, Eric A.; Grill, Stephan W.; Wiedmann, Anna; Lubkowska, Lucyna; Choy, Jason; Nogales, Eva; Kashlev, Mikhail; Bustamante, Carlos (2007-04-12). "Backtracking determines the force sensitivity of RNAP II in a factor-dependent manner". Nature. 446 (7137): 820–823. Bibcode:2007Natur.446..820G. doi:10.1038/nature05701. ISSN 1476-4687. PMID 17361130. S2CID 4310108.
- ^ Bintu, Lacramioara; Kopaczynska, Marta; Hodges, Courtney; Lubkowska, Lucyna; Kashlev, Mikhail; Bustamante, Carlos (2011-11-13). "The elongation rate of RNA polymerase determines the fate of transcribed nucleosomes". Nature Structural & Molecular Biology. 18 (12): 1394–1399. doi:10.1038/nsmb.2164. ISSN 1545-9985. PMC 3279329. PMID 22081017.
- ^ Kaplan CD, Larsson KM, Kornberg RD (June 2008). "The RNA polymerase II trigger loop functions in substrate selection and is directly targeted by alpha-amanitin". Molecular Cell. 30 (5): 547–56. doi:10.1016/j.molcel.2008.04.023. PMC 2475549. PMID 18538653.
- ^ Gong, Xue Q.; Nedialkov, Yuri A.; Burton, Zachary F. (2004-06-25). "α-Amanitin Blocks Translocation by Human RNA Polymerase II". Journal of Biological Chemistry. 279 (26): 27422–27427. doi:10.1074/jbc.M402163200. ISSN 0021-9258. PMID 15096519.
- ^ Briggs, Scott D.; Bryk, Mary; Strahl, Brian D.; Cheung, Wang L.; Davie, Judith K.; Dent, Sharon Y. R.; Winston, Fred; Allis, C. David (2001-12-15). "Histone H3 lysine 4 methylation is mediated by Set1 and required for cell growth and rDNA silencing in Saccharomyces cerevisiae". Genes & Development. 15 (24): 3286–3295. doi:10.1101/gad.940201. ISSN 0890-9369. PMC 312847. PMID 11751634.
- ^ Li, Bing; Howe, LeAnn; Anderson, Scott; Yates, John R.; Workman, Jerry L. (2003-03-14). "The Set2 Histone Methyltransferase Functions through the Phosphorylated Carboxyl-terminal Domain of RNA Polymerase II". Journal of Biological Chemistry. 278 (11): 8897–8903. doi:10.1074/jbc.M212134200. ISSN 0021-9258. PMID 12511561.
- ^ Meinhart A, Cramer P (July 2004). "Recognition of RNA polymerase II carboxy-terminal domain by 3'-RNA-processing factors". Nature. 430 (6996): 223–6. Bibcode:2004Natur.430..223M. doi:10.1038/nature02679. hdl:11858/00-001M-0000-0015-8512-8. PMID 15241417. S2CID 4418258.
- ^ a b c Egloff, Sylvain; Murphy, Shona (2008). "Cracking the RNA polymerase II CTD code". Trends in Genetics. 24 (6): 280–288. doi:10.1016/j.tig.2008.03.008. PMID 18457900.
- ^ Wei L, Levine AS, Lan L (2016). "Transcription-coupled homologous recombination after oxidative damage". DNA Repair (Amst.). 44: 76–80. doi:10.1016/j.dnarep.2016.05.009. PMID 27233112.
외부 링크
(웨이백 머신 복사)